基于DNA染料EMA的RT-PCR技术定量检测海产品中病原性副溶血弧菌活细胞
祝儒刚,吕淑霞*,刘月萍,张 良
(1.沈阳农业大学食品学院,辽宁 沈阳 110866;2.辽宁大学轻型产业学院,辽宁 沈阳 110036;3.沈阳农业大学生物科学技术学院,辽宁 沈阳 110866)
基于DNA染料EMA的RT-PCR技术定量检测海产品中病原性副溶血弧菌活细胞
祝儒刚1,2,吕淑霞1,3,*,刘月萍3,张 良3
(1.沈阳农业大学食品学院,辽宁 沈阳 110866;2.辽宁大学轻型产业学院,辽宁 沈阳 110036;3.沈阳农业大学生物科学技术学院,辽宁 沈阳 110866)
将一种DNA染料叠氮溴化乙锭(ethidium bromide monoazide,EMA)与实时定量聚合酶链式反应(RT-PCR)技术相结合,建立一种能选择性定量检测牡蛎中trh阳性副溶血弧菌活细胞的新方法。结果表明:使EMA成功插入死细胞DNA并且光解溶液中游离EMA的最佳曝光时间为20min;不抑制副溶血弧菌活细胞DNA扩增的最大EMA质量浓度为2.0μg/mL;完全抑制热致死细胞DNA扩增的最小EMA质量浓度为1.0μg/mL;人工污染牡蛎样品,不经过富集,在2.0×103~2.0×107CFU范围内细胞数的常用对数值与Ct值之间呈严格的负相关性,并且纯培养与人工污染牡蛎样品的RT-PCR检测限均为2×103CFU,即人工污染牡蛎样品的RT-PCR检测灵敏度为每克牡蛎样品400个活细胞;冻融实验表明,在温度低于55℃的水浴中对冷冻海产品进行解冻时,冻融过程对副溶血弧菌活细胞几乎没有影响。该方法是一种快速、灵敏且能有效鉴别并定量检测病原活细胞的新方法。
叠氮溴化乙锭(EMA);实时定量聚合酶链式反应(RT-PCR);海产品;副溶血弧菌;活细胞
副溶血弧菌(Vibrio parahaemolyticus)是一种革兰氏阴性嗜盐菌,它作为人类食源性致病菌能引起肠胃炎和败血症。副溶血弧菌广泛分布于自然界,尤其以海产品携带率最高,因此副溶血弧菌是沿海一带引起食物中毒和急性腹泻的重要病原菌,近年来,我国尤其是南部沿海地区均有因食用副溶血弧菌引起食物中毒报道。目前,副溶血弧菌引起食物中毒的发生规模以及人群暴露规模呈明显上升趋势,已成为我国首要的食源性致病菌[1-2]。
目前的研究认为,副溶血弧菌产生的毒素有溶血素和尿素酶。其致病因子主要有耐热直接溶血毒素(thermostabile direct hemolysin,TDH)、耐热直接相关溶血毒素(TDH-related hemolysin,TRH)、不耐热溶血毒素(thermolabile hemolysin,TLH),分别由相关的tdh、trh和tlh基因编码。TDH和TRH与副溶血弧菌的致病能力关系密切。临床分离的副溶血弧菌中超过90%为tdh阳性菌株,而环境中食物感染的潜在致病菌均为trh阳性副溶血弧菌,而且由该菌引起的食物感染和中毒事件呈上升趋势[3-5]。快速检测和诊断是及早预防控制副溶血弧菌食物中毒的发生的关键。
随着人们食品安全意识的提高,聚合酶链式反应(ploymerase chain reaction,PCR)技术以其特异性强、灵敏度高等优势在食品安全控制和临床诊断上得到越来越多的运用。实时定量PCR的出现,更是将PCR检测技术提高到定量的水平,并且检测灵敏度得到进一步提高[6-7]。然而,用PCR方法检测病原性副溶血弧菌污染时,它不能区分所检测到的副溶血弧菌是活细胞还是死细胞。因此,利用PCR选择性扩增副溶血弧菌活细胞DNA面临巨大挑战。近年来,人们尝试各种方法通过PCR技术仅仅检测样品中的活菌,例如mRNA的检测,以及利用能够与核酸共价结合的染料,这种染料能够选择性地渗透到死细胞内部与细胞内的DNA共价结合。在所有这些方法中,一种DNA插入型染料叠氮溴化乙锭(ethidium bromide monoazide,EMA),展现出了很大的应用潜力。许多研究[8-11]表明,EMA与PCR技术相结合,能够成功地选择性抑制样品中死细胞的DNA扩增,以便于更加精确地检测样品中活菌细胞的存在。
目前,EMA与RT-PCR检测技术相结合来检测并定量海产品中病原性副溶血弧菌活细胞仍未见报道。本研究将EMA选择渗透性和RT-PCR特异性和灵敏性相结合,从而建立一种快速、灵敏并能够有效检测并定量海产品中病原性副溶血弧菌活细胞的新方法。
1 材料与方法
1.1 菌种
实验用标准菌株,副溶血弧菌ATCC17802,本实验室保存。
1.2 试剂及培养基
叠氮溴化乙锭 美国Sigma公司;LightCycler 480 SYBR GreenⅠ(cat. no. 04 707 516 001)、荧光染料试剂盒美国Roche Diagnostics公司;胰胨肉汤(蛋白胨2%,NaCl 4%,0.01%的结晶紫0.5%,pH9.0);T1N3(1%蛋白胨,3% NaCl,2%琼脂)琼脂。
1.3 仪器与设备
1.4 副溶血弧菌细胞热处理
取37℃、150r/min条件下过夜培养的副溶血弧菌菌悬液,10000r/min离心5min。菌体沉淀用0.85%生理盐水洗两次,悬浮于0.85%的生理盐水中。取定量菌悬液梯度稀释,测定每个梯度的OD600nm并进行平板计数。3次独立实验,每次独立实验3次重复。
调整菌悬液浓度为4×108CFU/mL,取0.5mL于离心管中,95℃水浴加热10min,将经过热处理的0.5mL菌悬液冷却至室温后全部涂T1N3平板,37℃培养48h检测是否仍有活菌存在。
1.5 完全抑制死细胞DNA扩增的最小EMA质量浓度确定[12-14]
用ddH2O配制0.1mg/mL的EMA溶液,-20℃避光保存。将EMA(0.1mg/mL)分别加入装有0.5mL热致死副溶血弧菌菌悬液(4×108CFU/mL)的12支离心管中,使EMA终质量浓度分别达到0.2、0.4、0.6、0.8、1.0、1.2、1.4、1.6、1.8、2.0、2.2μg/mL。不加EMA 的一管作为阳性对照,0.5mL生理盐水(不含细菌细胞)做为阴性对照。充分混匀后,将离心管黑暗中室温放置约5min,让EMA充分渗透到死细胞内部并插入到细胞内DNA双螺旋上。然后将离心管开盖放置冰上,距离灯管16cm,曝光20min。
1.6 不抑制活细胞DNA扩增的最大EMA质量浓度确定[15-16]
取EMA溶液(1mg/mL)分别加入装有0.5mL活细胞菌悬液(4×108CFU/mL)的10支离心管中,使EMA终质量浓度分别达到1、2、4、6、8、10、12、14、16μg/mL。充分混匀后,将离心管黑暗中室温放置约5min。不加EMA的一管作为阳性对照,0.5mL生理盐水(不含细菌细胞)做为阴性对照。然后将离心管开盖放置冰上,距离灯管16cm,曝光20min。
1.7 EMA最佳曝光时间优化
分别取0.5mL活细胞菌悬液(4×108CFU/mL)于9个离心管,加入4μL EMA使每管EMA(0.1mg/mL)终质量浓度达到1.0μg/mL。充分混匀后,将离心管黑暗中室温放置5min。然后将离心管开盖放置冰上,距离灯管16cm,分别曝光0、5、10、15、20、15、30、35、40min。不加EMA的0.5mL活细胞菌悬液(4×108CFU/mL)做为阳性对照,0.5mL生理盐水(不含细菌细胞)做为阴性对照。
1.8 RT-PCR鉴别死活细胞混合菌悬液中活细胞
完全一样的9只离心管中分别加入0.25mL固定数量(2×107CFU)的热致死细胞菌悬液与0.25mL变化数量(2×107、2 ×106、2 ×105、2 × 104、2 ×103、2 ×102CFU和20CFU)的活细胞菌悬液(生理盐水洗涤2次),混合均匀。另一组同样9只完全相同的离心管,仅仅含有0.5mL变化数量(2×107、2×106、2×105、2×104、2×103、2×102CFU和20CFU)的活细胞菌悬液。第3组实验,9只相同的离心管中分别加入0.25mL固定数量(2×107CFU)的活细胞菌悬液与0.25mL变化数量(2×107、2×106、2×105、2×104、2×103、2×102CFU和20CFU)的热致死细胞菌悬液。
每只离心管中加入0.1mg/mL EMA,使其终浓度达到1.0μg/mL。将菌悬液在黑暗中室温放置5min,然后将离心管开盖放置冰上,距离灯管16cm曝光20min。
1.9 人工污染实验
1.9.1 标准曲线制作[17-18]
取过夜培养的副溶血弧菌(ATCC17802)菌悬液,菌体用灭菌生理盐水洗涤两次,调整菌悬液浓度为4×108CFU/mL。稀释10倍,得到菌浓度范围4×107~4CFU/mL。
无菌操作,每个浓度取1mL分别接种49mL灭菌胰胨肉汤液体培养基,混匀。另一组实验,牡蛎样品来自当地海产品零售市场,在无菌均质袋内,无菌操作剪碎5g样品于44mL灭菌胰胨肉汤液体培养基,利用高效自动均质器均质,同样方法每个浓度取1mL分别接种、混匀。
吸取每个样品10mL,800r/min离心10min,除去牡蛎细胞碎片,上清液转移到新的离心管10000r/min离心10min收集菌体。将菌体重悬于0.5mL无菌水中,添加EMA,使其终质量浓度达到1.0μg/mL。将菌悬液在黑暗中室温放置5min,然后将离心管开盖放置冰上,距离灯管16cm曝光20min。曝光后的混合菌悬液在10000r/min离心5min,收集菌体。倒掉上清液后将菌体重悬在0.5mL的无菌水中,10000r/min再次离心5min,收集菌体,倒掉上清液,再次重悬在50μL无菌水中。
为确保牡蛎样品不含trh阳性副溶血弧菌,取1g样品加入50mL胰胨肉汤培养基中,37℃、150r/min过夜培养后,取菌液做RT-PCR鉴定。
1.9.2 RT-PCR鉴别经冻融后菌悬液中的活细胞
过夜培养的副溶血弧菌菌悬液,菌体经生理盐水洗涤后调整其浓度为4×108CFU/mL。无菌操作分别取5g牡蛎组织于6只完全相同的无菌均质袋内,吸取1mL上述菌液人工污染,-20℃保存7d。冷冻样品分别在不同温度水浴中解冻,4℃解冻1h,25℃解冻30min,37、55、75、95℃分别解冻5min。解冻后的牡蛎组织添加无菌水至50mL,高效自动均质器均质。取10mL均质匀浆,如上述方法离心后将收集的菌体重悬在1mL无菌水中,取0.5mL经梯度稀释后涂T1N3平板,每个梯度涂3个平板,37℃培养3d后计数。剩余0.5mL菌悬液经EMA(终质量浓度1.0μg/mL)处理后进行RT-PCR扩增。1.10 DNA模板制备[19-20]
用TZ裂解液(2.0% Triton X-100,2.5mg/mL叠氮化钠,0.1mol/L Tris-HCl缓冲液,pH8.0)提取模板DNA。
经EMA处理曝光后的菌悬液与等体积的2×TZ裂解液混匀,沸水浴10min后冷却到室温,10000r/min离心10min去掉菌体碎片沉淀,取上清液直接用作RT-PCR模板。
1.11 引物及RT-PCR扩增
实验采用实时荧光定量PCR仪。病原性副溶血弧菌致病基因trh检测引物上游(P1)为:5′-TTGCTTT CAGTTTGCTATTGGCT-3′;下游引物(P2)为:5′-GCT ACT TTC TAG CAT TTT CTC TGC-3′,扩增片段长度为300bp。20μL的扩增体系包括:10μL荧光染料混合液,5μL模板DNA,上下游引物各1μL(1μmol/L),3μL PCR级无菌水。RT-PCR反应体系:94℃预变性10min,40个循环,每个循环94℃ 20s、55℃ 20s、72℃ 30s。每个RT-PCR反应重复3次,得到平均Ct值和标准背离值。
1.12 数据分析
所得数据以均数±标准差(x±s)表示,应用SPSS 11.0统计软件对实验数据进行t检验,P<0.05表示差异具有显著性。
2 结果与分析
2.1 副溶血弧菌热处理
将菌悬液梯度稀释,测定各梯度的OD600nm值,并取适量菌液涂平板,每个梯度设3个重复,37℃过夜培养,计算每个梯度的菌浓度。得到活菌的菌浓度随OD600nm变化曲线,结果如图1所示。可知,当OD600nm为0.7时,菌浓度为4×108CFU/mL。
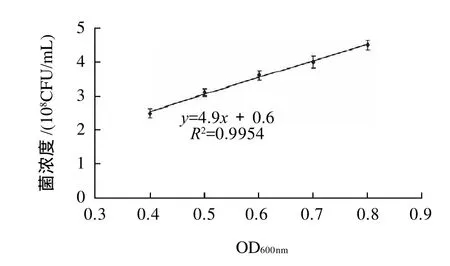
图1 副溶血弧菌浓度与OD600nm的标准曲线Fig.1 Concentration dependence of OD600nm values in Vibrio.parahaemoliticus suspension
95℃水浴处理副溶血弧菌菌悬液(OD600nm为0.7,菌浓度4×108CFU/mL)10min,冷却至室温后将0.5mL菌液完全涂T1N3平板,37℃培养48h,无菌落生长。因此,浓度为4×108CFU/mL的副溶血弧菌菌悬液在95℃水浴处理10min,可将所有副溶血弧菌细胞全部被杀死,平板计数为零。
2.2 最佳EMA曝光时间

图2 激活和完全光解游离EMA的最佳曝光时间优化Fig.2 Optimization of light exposure time to activate DNA-bound EMA and to achieve complete photolysis of free EMA
在RT-PCR中,当每个反应管内的荧光信号到达设定的荧光阈值时所经历的循环数,定义为Ct值,荧光阈值的缺省设置是3~15个循环的荧光信号的标准偏差的10倍。研究表明,每个模板的Ct值与该模板的起始拷贝数的对数存在线性关系,起始拷贝数越少,扩增曲线向右移动,其Ct值越大;相反,起始拷贝数越多,扩增曲线向左移动,其Ct值越小。如图2所示,阴性对照(不加模版)的Ct值是30.42±0.60。当活菌细胞浓度为4×108CFU/mL时,随着曝光时间的延长,各样品Ct值逐渐减小,在曝光时间为15min时,其Ct值为22.94±0.66。经曝光以后,未被光解的游离EMA会与细胞裂解释放出来的DNA共价结合,从而抑制RT-PCR扩增,导致Ct值增大。统计分析表明,当曝光时间大于等于15min时,各样品的Ct值与阳性对照(22.44±0.56)没有显著差异(P<0.05)。因此,为了使游离EMA完全曝光光解,选择20min为最佳曝光时间。
2.3 不抑制活细胞DNA扩增的最大EMA质量浓度
EMA的不同添加量对活菌RT-PCR扩增的抑制作用如图3所示。随着EMA质量浓度的不断增大,各样品Ct值也逐渐增加,表明EMA质量浓度足够大时对活菌的RT-PCR扩增也有一定抑制作用。由图3分析可知,当EMA质量浓度小于等于2μg/mL时,样品Ct值与阳性对照Ct值(为18.67±0.45,不添加EMA)不具有统计显著差异性,此时EMA对活细胞RT-PCR扩增几乎没有抑制作用(P>0.05)。然而,当EMA质量浓度大于等于4μg/mL时,EMA对活细胞RT-PCR扩增具有显著抑制作用,此时样品Ct值与阳性对照具有统计显著性差异(P<0.05)。
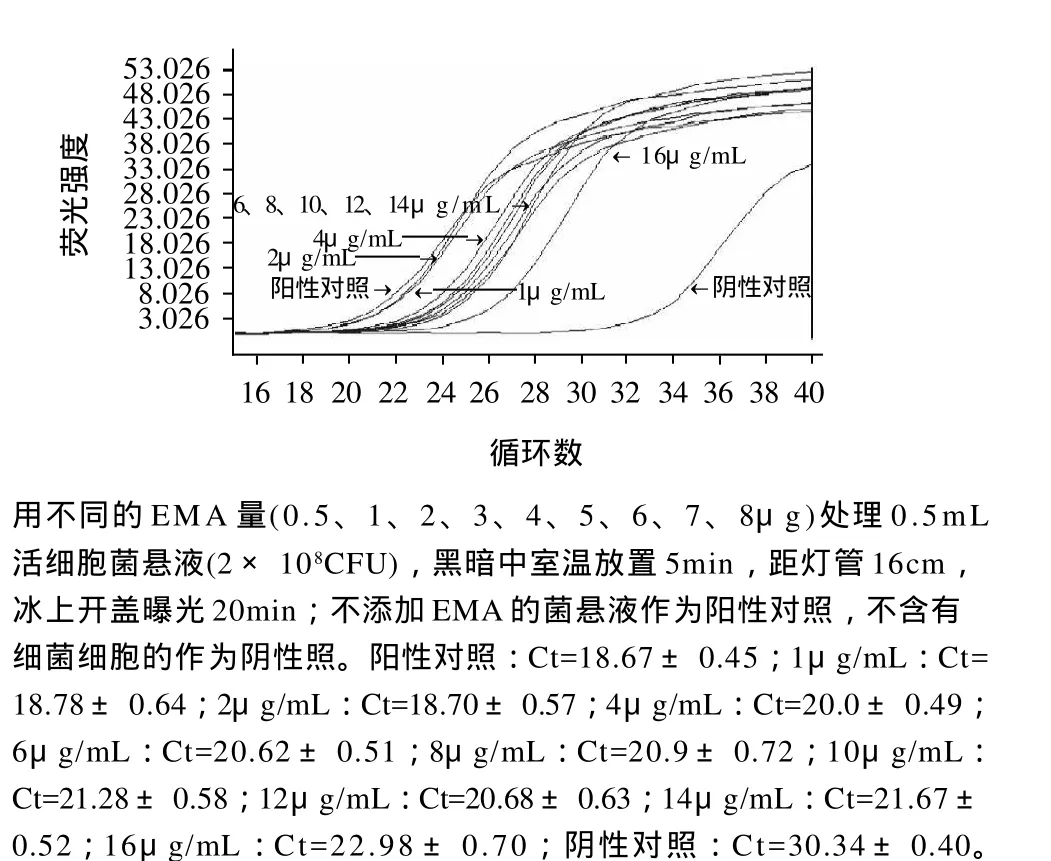
图3 不抑制活细胞DNA扩增的最大EMA质量浓度优化Fig.3 Optimization of maximum EMA amount without inhibitory effect on DNA amplification from viable cells
2.4 完全抑制死细胞DNA扩增的最小EMA浓度
如图4所示,当EMA质量浓度为0.8μg/mL或更高时,所得到的Ct值均大于等于28.30±0.41,此时的Ct值与阴性对照(28.76±0.63)不具有统计显著差异性,其产物溶解曲线Tm值为75℃(数据未提供),可能为引物二聚体(P>0.05)。表明,此时死细胞DNA的RT-PCR被完全抑制。相反,当EMA质量浓度分别为0.2、0.4、0.6μg/mL时产物的溶解曲线Tm值均为81℃,此时的Ct值与阴性对照具有统计极显著差异(P<0.01)。此时,0.8μg/mL的EMA质量浓度远远小于可以抑制活细胞RTPCR扩增的EMA质量浓度2μg/mL,因此,为了能够使死细胞DNA的RT-PCR彻底被EMA抑制,同时又不会抑制活细胞DNA的RT-PCR扩增,实验中选择EMA质量浓度1.0μg/mL为完全抑制死细胞DNA RT-PCR扩增时EMA的最适质量浓度。
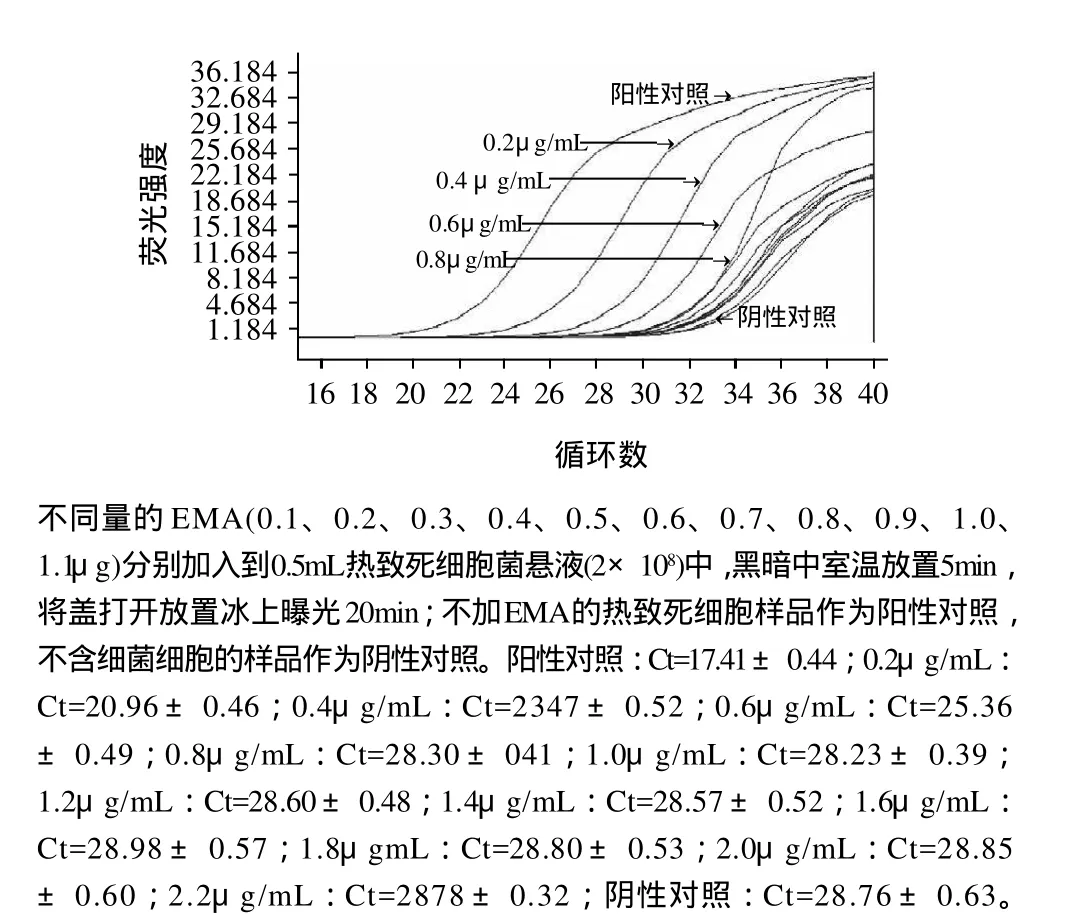
图4 完全抑制死菌细胞DNA扩增的最小EMA添加量的优化Fig.4 Optimization of minimum EMA amount to inhibit DNA amplification completely from heat killed cells
2.5 RT-PCR鉴别死活细胞混合液中的活细胞
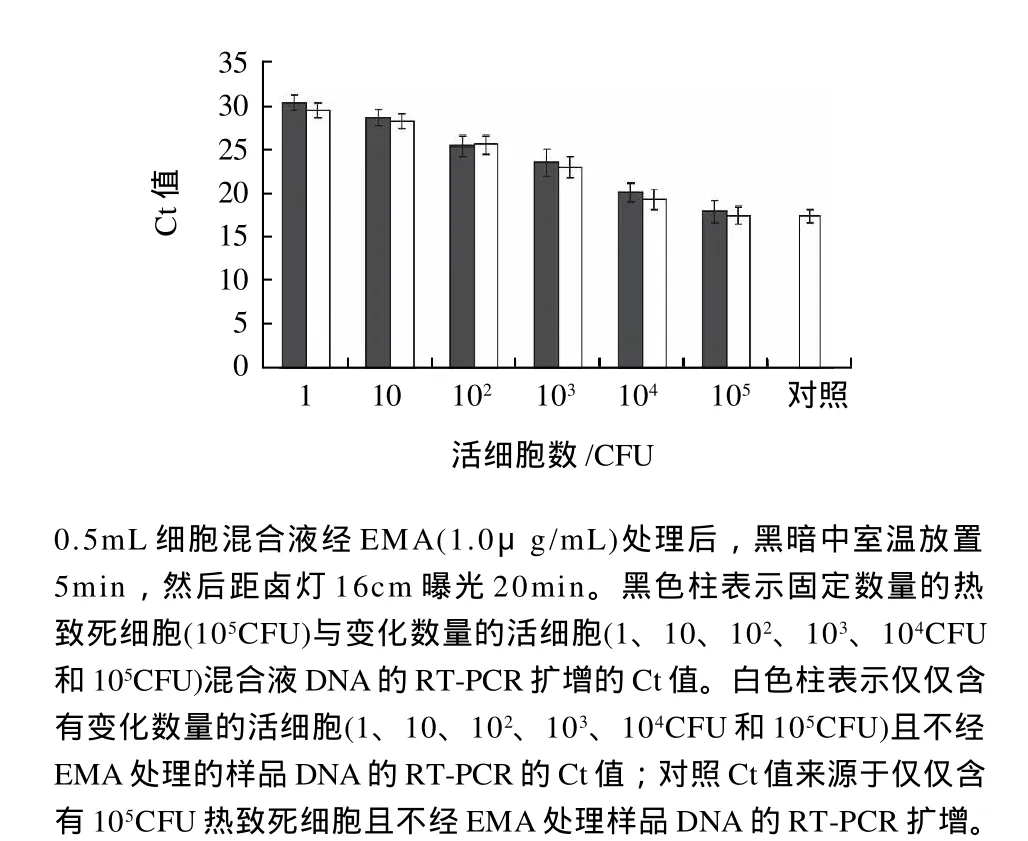
图5 固定量的热致死细胞与变化量的活细胞混合液中DNA的不同RT-PCR扩增Fig.5 Differential DNA amplification by RT-PCR in the mixtures of a constant number of heat-killed cells and a varying number of viable cells
由图5可知,当EMA质量浓度为1.0μg/mL时,在固定量的热致死细胞(105CFU)与变化数量的活细胞(105、104、103、102、10CFU和1CFU)混合液中,热致死细胞的DNA的RT-PCT扩增被完全抑制。而活细胞DNA的RT-PCR没有受到影响,随着细胞数量的增大,Ct值逐渐减小。对照样品含105个热致死细胞,不经EMA处理,其Ct值(17.25±0.72)与不经EMA处理且含相同活细胞数样品的Ct值(17.34±1.01)几乎相同。
而在固定量的活细胞(105CFU)与变化数量的热致死细胞(105、104、103、102、10CFU 和 1CFU)混合液 DNA的RT-PCR扩增中,各样品的Ct值没有统计显著差异性(P>0.05)。这是由于EMA完全抑制了热致死细胞DNA的RT-PCR扩增,而对活细胞DNA的RT-PCR扩增没有影响。其Ct值与相同活细胞浓度下不经EMA处理的对照样品几乎相同(图6)。
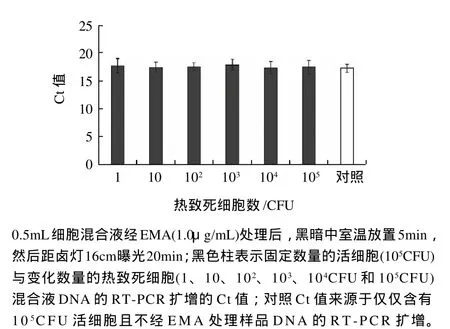
图6 固定量的活细胞与变化量的热致死细胞混合液中DNA的不同RT-PCR扩增Fig.6 Differential DNA amplification by RT-PCR in the mixtures of a constant number of viable cells and a varying number of heat-killed cells
2.6 标准曲线
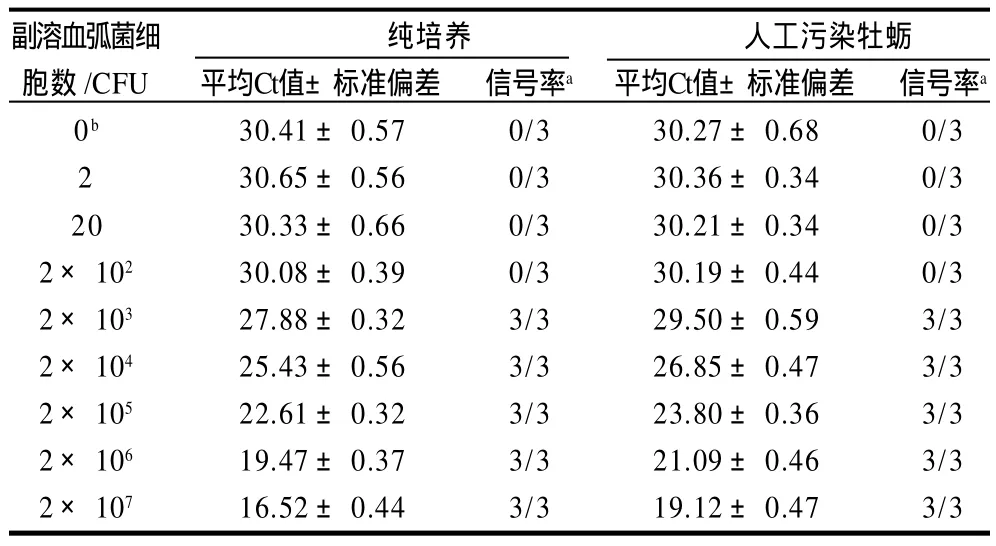
表1 纯培养和人工污染牡蛎样品的Ct值与菌落数关系Table 1 The relationship between CFU and Ct values in pure culture and artificially contaminated oyster samples
添加不同的活细胞数做人工污染,纯培养和人工污染牡蛎样品各Ct值以及RT-PCR阳性信号率如表1所示。由表可知,随着人工污染细胞数的不断增大(2~2×107CFU),纯培养(30.65±0.56~16.52±0.44)和牡蛎样品(30.36±0.34~19.12±0.47)的Ct值逐渐减小。并且,相同的细胞数进行人工污染,牡蛎样品的Ct值比纯培养要高1~3个循环。根据RT-PCR的阳性信号率,该方法对于纯培养和人工污染牡蛎样品的RT-PCR检测限均为2×103个活细胞,人工污染牡蛎样品检测限可以达到400个细胞/克牡蛎样品。由图7可知,在2×103~2×107个细胞范围内,纯培养和人工污染牡蛎样品的Ct值与细胞数的对数均具有很好的线性关系。
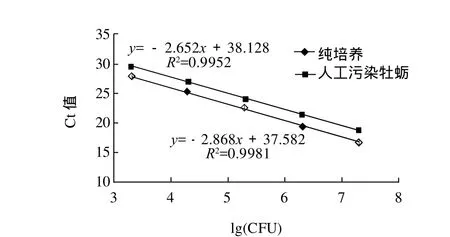
图7 纯培养和人工污染牡蛎样品中副溶血弧菌细胞数的对数与RT-PCR反应Ct值线性关系的标准曲线Fig.7 Standard curves showing the linear relationship between Ct values and Log CFU (3.3~7.3) of V. parahaemolyticus in pure culture and artificially contaminated oyster samples
2.7 RT-PCR研究人工污染冻-融牡蛎样品中病原性副溶血弧菌细胞存活情况
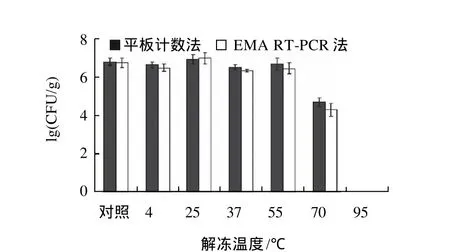
图8 RT-PCR检测经冻融后每克牡蛎组织中的病原性副溶血弧菌活细胞数Fig.8 The numbers of viable cells per gram of oyster tissue as a result of freezing and thawing determined by RT-PCR
1mL副溶血弧菌菌悬液(4×108CFU/mL)人工污染5g牡蛎组织,-20℃冷冻贮存7d后,在4℃水浴1h、25℃水浴30min、37℃和55℃分别水浴5min解冻时,通过平板计数和EMA RT-PCR测得的活细胞数的对数值与对照(不经过冷冻,约6.8)没有显著性变化(P>0.05)(图8)。然而,在70℃水浴5min解冻时,平板计数和EMA RTPCR常用对数值分别下降至4.3和4.7。这可能是由于热对一部分细菌的破坏作用,在不完全损坏细胞膜的情况下导致酶失活或者核糖体的降解。95℃解冻5min时,细菌细胞几乎被完全杀死,平板计数和EMA RT-PCR都没有检测到活细胞的存在。
3 结 论
3.1 副溶血弧菌浓度为4×108CFU/mL时,激活和光解菌悬液中EMA的最佳曝光时间为20min,不抑制副溶血弧菌活细胞RT-PCR扩增的最大EMA质量浓度是2μg/mL,完全抑制副溶血弧菌死细胞RT-PCR扩增的最小EMA浓度为1.0μg/mL。
3.2 人工污染牡蛎样品,不经过富集培养,接种细胞数的对数值与对应人工污染样品RT-PCR的Ct值之间具有严格的线性相关性,纯培养与人工污染牡蛎样品标准曲线的相关系数分别为0.9981和0.9952。纯培养与人工污染牡蛎样品的RT-PCR检测限均为2×103CFU,即人工污染牡蛎样品的RT-PCR检测灵敏度为每克牡蛎样品400个活细胞。
3.3 在温度低于55℃的水浴中对冷冻海产品进行解冻时,冻融过程对副溶血弧菌活细胞基本没有影响。
[1] TYAGI A, SARAVANAN V, KARUNASAGAR I, et al. Detection of Vibrio parahaemolyticus in tropical shellfish by SYBR green real-time PCR and evaluation of three enrichment media[J]. International Journal of Food Microbiology, 2009, 129(5): 124-130.
[2] NORDSTROM J L, VICKERY M C L, BLACKSTONE G M, et al.Development of a multiplex real-time pcr assay with an internal amplification control for the detection of total and pathogenic Vibrio parahaemolyticus bacteria in Oysters[J]. Applied and Environental Microbiology, 2007, 73(1): 5840-5847.
[3] 金周浩, 宋达锋, 顾青. 副溶血弧菌致病因子与耐热直接溶血毒素的研究进展[J]. 水产科学, 2008, 27(6): 320-324.
[4] 金周浩, 宋达锋, 顾青. 副溶血弧菌毒力基因的检测研究[J]. 中国食品学报, 2008, 8(3): 143-146.
[5] 张蔚, 孟冬梅, 潘劲草, 等. 杭州地区临床和环境分离副溶血弧菌菌株携带毒力基因的特征[J]. 中华预防医学杂志, 2004, 6(5): 77-81.
[6] 李志峰, 聂军, 陈义忠, 等. 一种快速检测副溶血弧菌的PCR方法[J]. 解放军预防医学杂志, 2004, 22(6): 443-444.
[7] 杨文鸽, 孙翠玲, 潘云娣, 等. 水产品中致病微生物的快速检测方法[J]. 中国食品学报, 2006, 6(1): 402-406.
[8] GU W M, LEVIN R E. Quantification of viable Plesiomonas shigelloides in a mixture of viable and dead cells using ethidium bromide monoazide and conventional PCR[J]. Food Biotechnology, 2007, 21(2): 145-149.
[9] LEE J M, LEVIN R E. Use of ethidium bromide monoazide for quantification of viable and dead mixed bacterial flora from fish fillets by polymerase chain reaction[J]. Journal of Microbiological Methods, 2006,67(3): 456-462.
[10] NOGVA H, DROMTORP S, NISSEN H, et al. Ethidium monoazide for DNA-based differentiation of viable and bacteria by 5′-nuclease PCR[J].Biotechniques, 2003, 34(7): 804-813.
[11] RUDI K, MOEN B, DROMTORP S, et al. Use of ethidium monoazide and PCR in combination for quantification of viable and dead cells in complex samples[J]. Appl Environ Microbiol, 2005, 71(3): 1018-1024.
[12] 冯建军, 金志娟, 刘西莉, 等. 一种DNA染料结合聚合酶链反应检测鉴别植物病原细菌死活细胞[J]. 高等学校化学学报, 2008, 29(5):944-948.
[13] LEE J L, LEVIN R E. Discrimination of viable and dead Vibrio vulnificus after refrigerated and frozen storage using EMA, sodium deoxycholate and real-time PCR[J]. Journal of Microbiological Methods, 2009, 79(7):184-188.
[14] LEE J L, LEVIN R E. Quantification of total viable bacteria on fish fillets by using ethidium bromide monoazide real-time polymerase chain reaction[J]. International Journal of Food Microbiology, 2007, 118(2):312-317.
[15] LEE J L, LEVIN R E. Use of ethidium bromide monoazide for quantification of viable and dead mixed bacterial flora from fish fillets by polymerase chain reaction[J]. Journal of Microbiological Methods, 2006,67(3): 456-462.
[16] WANG S, LEVIN R E. Discrimination of viable Vibrio vulnificus cells from dead cells in real-time PCR[J]. Journal of Microbiological Methods,2006, 64(9): 1-8.
[17] BLACKSTONE G M, NORDSTROM J L, VICKERY M C L, et al.Detection of pathogenic Vibrio parahaemolyticus in oyster enrichments by real time PCR[J]. Journal of Microbiological Methods, 2003, 53(10):149-155.
[18] NAM H M, SRINIVASAN B, GILLESPIE B E, et al. Application of SYBR green real-time PCR assay for specific detection of Salmonella spp. in dair farm environmental samples[J]. International Journal of Food Microbiology, 2005, 102(4): 161-171.
[19] 刘晓侠, 林建平, 岑沛霖. 微生物基因组DNA提取方法的比较与改进[J]. 嘉兴学院学报, 2007, 19(3): 48-50.
[20] ABOLMAATY A, VU C, OLIVER J, et al. Development of a new lysis solution for releasing genomic DNA from bacterial cells for DNA amplification by polymerase chain reaction[J]. Microbios, 2000, 101(6): 181-189.
Quantification of Pathogenic Viable Cells of Vibrio parahaemolyticus in Seafood by Ethidium Bromide Monoazide Staining and Real-time Polymerase Chain Reaction
ZHU Ru-gang1,2,LU Shu-xia1,3,*,LIU Yue-ping3,ZHANG Liang3
(1. College of Food Science, Shenyang Agricultural University, Shenyang 110866, China;2. College of Light Industry, Liaoning University, Shenyang 110036, China;3. College of Biological Science and Techology, Shenyang Agricultural University, Shenyang 110866, China)
A new method for selectively quantitative detection of trh-positive viable cells of Vibrio parahaemolyticus in oysters was developed using ethidium bromide monoazide (EMA) staining in combination with real-time PCR (RT-PCR, real-time polymerase chain reaction). The results showed the optimum light exposure time to achieve DNA crosslinking by EMA in dead cells and to photolyze the free EMA in solution was 20 min. The use of 2.0μg/mL EMA or less did not inhibit the RT-PCR amplification of DNA derived from viable cells of Vibrio parahaemolyticus. The minimum amount of EMA to completely inhibit the RT-PCR amplification of DNA derived from heat-killed cells was 1.0μg/mL. In artificial contaminated oyster samples without enrichment process, there was a strict negative correlation between the log cell number and the Ct values in the range of 2.0 × 103to 2.0 × 107CFU. The detection limit of the real-time PCR assay was 2 × 103CFU in both pure cultures and artificial contaminated oyster samples, which indicated the sensitivity of RT-PCR was 400 CFU/g in artificial contaminated oyster sample. The freezing/thawing experiments showed thawing in water bath at temperature below 55 ℃had little effect on viable cells of Vibrio parahaemolyticus. Therefore, this method avoided the defection in traditional PCR,which could not distinguish viable bacteria cells from dead ones. It may offer a fast, sensitive method to identify andquantify pathogenic viable cells effectively.
EMA;RT-PCR (real-time polymerase chain reaction);seafood;Vibrio parahaemolyticus;viable cells
2010-05-26
教育部留学回国人员基金项目(2006331);辽宁省科技厅项目(20062109);沈阳市科技局项目(1063312-1-00;090009)
祝儒刚(1980—),男,讲师,博士研究生,研究方向为食品微生物与分子生物学。E-mail:zrg_luck@163.com
*通信作者:吕淑霞(1963—),女,教授,博士,研究方向为微生物生物化学与分子生物学。E-mail:lushuxia@hotmail.com
TS201.6
A
1002-6630(2011)08-0219-07

