用于食源性疾病病原菌诊断的基因芯片研究及评价
任莉莉,从彦丽*
用于食源性疾病病原菌诊断的基因芯片研究及评价
任莉莉,从彦丽*
(深圳职业技术学院应用化学与生物技术学院,广东 深圳 518055)
目的:建立应用基因芯片结合酪胺信号放大法检测食源性疾病病原菌的方法。方法:针对细菌16S和23S rDNA基因设计各种病原菌诊断的特异性探针和通用引物。被检测的病原菌经过生物素标记的引物进行PCR扩增,然后与制备的基因芯片探针区杂交,再用酪胺-Cy3进行显色。最后通过荧光扫描仪扫描杂交图像。结果:建立的基因芯片检测方法用于诊断沙门菌、志贺菌、副溶血性弧菌、空肠弯曲菌、肠出血性大肠杆菌O157:H7、单增李斯特菌、霍乱弧菌、腊样芽孢杆菌等8种细菌性传染病病原菌。纯培养物的检测灵敏性达到5×102CFU/mL,对20例模拟双盲样本进行检测,结果与预期的完全相符。结论:本方法特异性强、灵敏度高,可以用于传染病防控和临床诊断等多个领域。
病原菌;基因芯片;酪胺信号放大
细菌性食源性疾病在全世界公共卫生事件中占有较大的比例,也是我国传染病预防控制的重点。其中病因主要有沙门菌、志贺菌、肠出血性大肠杆菌O157:H7、副溶血性弧菌、空肠弯曲菌等病原菌所导致[1]。早期快速确诊病原菌类型对于传染病的防控、疾病治疗均具有重要意义。病原菌形态和培养技术是作为细菌鉴定的“金标准”,但是由于培养周期长,培养条件各不相同,同时还要结合生化试验及免疫学试验才能对细菌类型进行判定,因此无法满足传染病防控的需要[2]。为研究一种能够高通量、高灵敏性检测多种细菌性传染病病原菌的方法,本实验选取细菌16S和23S rDNA基因建立对8种病原菌检测的基因芯片方法,同时采用酪胺信号放大方法提高基因芯片检测的灵敏度。
1 材料与方法
1.1 标准菌株及基因组DNA提取
肠出血性大肠杆菌O157:H7(ATCC43889)、志贺菌(ATCC25931)、副溶血性弧菌(ATCC20511)、沙门菌(ATCC43975)、腊样芽孢杆菌(ATCC33019)、空肠弯曲菌(ATCC33560)、结肠弯曲菌(ATCC43478)、O1群霍乱弧菌(569B)、O139群霍乱弧菌(MO45)、单增李斯特菌(ATCC54001)、英诺克李斯特氏菌(ATCC33090)、西尔李斯特氏菌(ATCC35967)、格氏李斯特氏菌(ATCC25401)、威尔李斯特氏菌(ATCC35897)、粪肠球菌(ATCC14506) 标准菌株均购自上海汉尼生物技术有限公司,按照每种细菌的标准方法进行接种、分离培养。菌株基因组DNA提取采用上海超世生物科技有限公司试剂盒,严格按照试剂盒说明书操作。提取的基因组DNA放置于-20℃保存,待用。
1.2 试剂与仪器
细菌常规培养试剂;Taq酶、dNTPs等PCR相关试剂 法国梅里埃公司;基因芯片片基、显色液、杂交液 宝生物工程(大连)有限公司;酪胺-Cy3试剂 博奥生物科技有限公司。
9700 PCR仪 基因科技有限公司;4000B荧光扫描仪 美国ABI公司;凝胶成像系统 美国分子仪器公司;离心机 美国Bio-Rad公司;7500点样仪 美国Cartesian公司。
1.3 引物和探针设计

表1 基因芯片引物与探针序列Table 1 Oligonucleotide primers and probes used in the present study
根据参考文献[3-5]设计细菌16S和23S rDNA的通用引物,通过GenBank下载细菌相关16S rDNA 和 23S rDNA基因序列,采用clustalW和Genedoc软件对序列进行比对,设计引物与文献报道略有不同。下游引物5′端标记生物素基团,16S引物扩增长度为559bp,23S引物扩增长度为632bp。寡核苷酸探针设计在每种目的病原菌基因组两对引物之间的可变区,在5′端标记氨基基团,同时连接一定长度的连接臂(10个T碱基)用于增加探针杂交的动力[6];序列见表1,引物和探针在上海生工生物工程有限公司合成。
1.4 靶基因扩增
采用双重PCR进行扩增,体系如下:10×buffer 2.0μL,25mmol/L Mg2+1.2μL,200μmol/L dNTPs 1.6 μL,上游引物(23S-F和16S-F)分别为0.5μmol/L,下游引物(16S-R和16S-R)分别为0.3μmol/L,Taq聚合酶0.3U;模板基因组DNA 2.0μL(20~50ng/μL),用去离子水补至20μL。在PCR仪上进行扩增,PCR反应条件为:95℃ 5min;进入循环95℃ 30s、55℃ 30s、72℃30s,共35个循环;72℃ 1min。扩增后的产物可以用1.5%的琼脂糖凝胶电泳进行检测。
1.5 基因芯片制备
参照文献[3]报道,采用醛基化处理的玻璃片和点样仪,基因芯片制备流程参照文献[7]进行。制备的基因芯片矩阵图如图1所示。

图1 基因芯片点样矩阵图Fig.1 Layout of oligonucleotide probes
1.6 基因芯片杂交与显色
参照文献[8-10]进行操作。吸取PCR产物10μL与10μL杂交液混匀后于100℃加热变性10min,迅速滴加于基因芯片上探针区,将基因芯片放入铺有吸水纸的盒中,保持一定湿度,避光,在55℃温箱中放置1.5h。取出杂交盒,甩掉芯片上剩余的杂交液,用洗涤液清洗基因芯片,重复3次。待晾干后,在杂交区加入用PBS溶液按照1:1500稀释的链酶亲和素标记HRP,37℃避光放置30min;取出后用含0.05% 吐温-20的PBS-T清洗1min,重复3次,置室温晾干。用1×PBS加质量分数1% BSA配制的稀释液将TSA-Cy3稀释成1:1000,加入杂交区,37℃避光放置30min。取出后再用洗液洗涤,置室温晾干。将基因芯片放入荧光扫描仪中扫描,图像与基因芯片矩阵图对比,对杂交结果进行判定。
1.7 模拟样本制备
采用临床健康人群粪便离心上清液混合不同的病原菌,在双盲状态下制备20例临床模拟样本。
2 结果与分析
2.1 靶病原菌特异性杂交
对8种细菌性传染病病原菌沙门菌、志贺菌、副溶血性弧菌、空肠弯曲菌、肠出血性大肠杆菌O157:H7、单增李斯特菌、霍乱弧菌、腊样芽孢杆菌以及属内细菌进行基因芯片杂交。结果显示筛选的探针具有很高的特异度。如图2所示,靶病原菌对应探针和阳性对照探针均出现特异性信号,同时阴性探针没有信号出现。其他非靶病原菌杂交只有阳性对照探针出现信号,说明本实验选取的探针可以特异性的检测8种靶病原菌。过培养和菌落计数,取培养到5×107CFU/mL,依次梯度10倍稀释,制备成5×107、5×106、5×105、5×104、5×103、5×102、5×101CFU/mL等一系列灵敏性评价标准品。PCR扩增和基因芯片杂交后,检测结果见图3。从结果看出,5×102CFU/mL出现微弱的荧光信号,而5×101CFU/mL则没有荧光信号产生,说明本项目所建立的检测体系的灵敏性可达到5× 102CFU/mL,每次实验重复3次。
2.3 模拟样本检测
为验证基因芯片检测的有效性,在双盲状态下制备20例临床模拟样本,对样本进行基因组DNA提取,PCR扩增和基因芯片杂交。结果显示与制备的病原菌类型完全一致,一种包括2例混合样本,1例混合样本含有沙门菌和肠出血性大肠杆菌O157:H7,1例含有副溶血性弧菌和霍乱弧菌,部分见图4。

图2 靶病原菌杂交结果Fig.2 Typical hybridization profiles from pure bacterial culture
2.2 基因芯片灵敏性评价

图3 基因芯片检测副溶血性弧菌灵敏性评价结果Fig.3 Evaluation of sensitivity for detecting Vibrio parahaemolyticus using DNA chip
选取副溶血性弧菌对基因芯片灵敏性进行考核,经
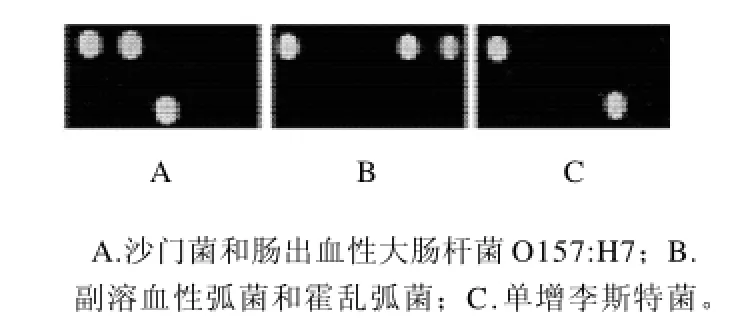
图4 部分模拟样本基因芯片检测结果Fig.4 Partial DNA chip results for detecting intestinal pathogens from model samples
3 讨 论
基因芯片是在20世纪90年代中期发展出来的一种集生物学、分子生物学、微电子学、材料学等多个学科的高科技技术。基因芯片基质一般是经过处理后的玻璃片、尼龙膜、塑料等,处理的方法一般有醛基化和氨基化[2]。目前,基因芯片检测致病菌的方法均采用了通用引物扩增方法和特异性基因多重扩增方法,扩增的靶基因分为特异性毒力基因和通用基因。采用的检测方法有荧光标记检测方法和可见光标记显色方法等等,标记的方法分为末端标记法、掺入法等[11]。上述诸多方法各具特点,本实验选取病原菌核糖体16S和23S亚基基因,参照文献设计引物和探针,采用酪胺-Cy3进行显色,由于HRP(辣根过氧化物酶)能够催化酪胺沉淀,因此相对于其他末端比较荧光素的方法,会在一定程度上提高检测的灵敏性。
对于基因芯片检测方法,筛选特异性高、结合能力强的探针是比较关键的。据文献报道,核糖体基因具有高度保守的特性,可以作为细菌分类的依据。因此,选取病原菌核糖体基因作为检测多种病原菌的靶基因,可以依靠通用性引物在少量PCR次数的基础上实现对多种病原菌检测。从本实验结果可以看出,选取的特异性探针可以检测和诊断对应的8种细菌性传染病病原菌,对纯培养细菌检测的灵敏度可达到5×102CFU/mL,相当于在1个PCR扩增体系中少于1个CFU的细菌量。同时在对20例模拟双盲样本进行考核时,检测的结果完全符合,其中包括2例模拟多种病原菌感染的样本,说明该方法完全可以应用到临床样本的检测。在后期的工作中,应该收集大量临床样本进行基因芯片检测实际样本的考核,同时研究更灵敏的显色技术提高检测的灵敏性,进一步优化并完善该基因芯片检测的效果。
本实验建立的基因芯片结合酪胺信号放大法检测细菌性传染病病原菌的方法,可以对沙门菌、志贺菌、副溶血性弧菌、空肠弯曲菌、肠出血性大肠杆菌O157: H7、单增李斯特菌、霍乱弧菌、腊样芽孢杆菌等8种细菌性传染病病原菌进行鉴定。纯培养细菌检测的灵敏性达到5×102CFU/mL。这种方法的建立为今后进一步扩大检测范围,增加靶病原菌数量提供参考。
[1]TAYLOR D N, ECHEVERRIA P. Diarrheal disease: current concepts and future challenges. Molecular biological app roaches to the epidemiology of diarrheal diseases in developing countries[J]. Trans R Soc Trop Med Hyg, 1993, 87(Suppl 3): 3-5.
[2]刘毅, 韩金祥. 生物芯片技术在临床病原菌检测中的新进展[J]. 中国实验诊断学, 2006, 10(3): 325-328.
[3]JIN Dazhi, WEN Siyuan, CHEN Suhong, et al. Detection and identification of intestinal pathogens in clinical specimens using DNA microarrays [J]. Molecular and Cellular Probes, 2006, 20(6): 337-347.
[4]高爽, 谢明杰, 金大智, 等. 运用基因芯片技术建立检测水产品食物中常见病原微生物方法的研究[J]. 生物技术通讯, 2007(18): 72-76.
[5]ANTHONY R M, BROWN T J, FRENCH G L. Rapid diagnosis of bacteremia by universal amplification of 23S ribosomal DNA followed by hybridization to an oligonucleotide array[J]. Journal of Clinical Microbiology, 2000, 38(2): 781-788.
[6]顾鸣, 韩伟, 关嵘, 等. 常见食源性致病菌基因芯片鉴定技术[J]. 中国卫生检验杂志, 2005, 15(11): 1309-1312.
[7]毛正果, 郑浩轩, 王新颖, 等. 基因芯片检测常见肠道致病菌感染的研究与评价[J]. 胃肠病学和肝病学杂志, 2008, 17(10): 809-812.
[8]SPEEL E J M, HOPMAN A H N, KOMMINOTH P. Amplification methods to increase the sensitivity of in situ hybridization: play CARD (S)[J]. The Journal of Histochemistry and Cytochemistry, 1999, 47(3): 281-288.
[9]SPEEL E J M, RAMACEKERS F C S, HOPMAN A H N. Sensitivity multicolor fluorescence in situ hybridization using catalyzed reporter deposition (CARD) amplification[J]. The Journal of Histochemistry and Cytochemistry, 1997, 45(10): 1439-1446.
[10]KERSTENS H M, PODDIGHE P J, HANSELAAR A G. A novel in situ hybridization signal amplification method based on the deposition of biotinylated tyramine[J]. J Histochem Cytochem, 1995, 43(4): 347-352.
[11]SCHENA M. Microarray Biochip Technology[M]. Natick, USA: Eaton Publishing, 2000.
Preliminary Study and Evaluation of DNA Chip for Diagnosing Foodborne Pathogenic Bacteria
REN Li-li,CONG Yan-li*
(School of Applied Chemistry and Biological Technology, Shenzhen Polytechnic, Shenzhen 518055, China)
Objective: To establish a method for detecting pathogenic bacteria quickly and accurately combinedly using DNA chip and tyramide signal amplification method. Methods: According to the sequences of 16S and 23S rDNA gene, common primer pairs and specific probes were designed. The primer pairs labeled with biotin group were used for PCR amplification, and then PCR products were hybridized with probes on the DNA chip. After the above process, the coloration was done using tyramide-Cy3. Finally, hybridization images were scanned by a fluorescence scanner. Results: Eight pathogenic bacteria were detected using this assay described here. The targets were Salmonella spp, Shigella spp, Vibrio parahaemolyticus, Campylobacter jejuni, Escherichia coli O157:H7, Listeria monocytogenes, Vibrio cholera, Bacillus cereus. The sensitivity of this assay was approximately 5 ×102CFU/mL for Vibrio parahaemolyticus. When 20 double-blind samples were detected, the results were in accordance with those of conventional methods. Conclusion: The specific and sensitive assay reported in the article can be applied for disease controlling and clinical diagnosis.
pathogenic bacteria;DNA chip;tyramide signal amplification
TS207.4;Q343.1
A
1002-6630(2011)04-0208-04
2010-03-19
深圳市科技计划项目(07KJba165)
任莉莉(1977—),女,讲师,硕士,主要从事营养和食品安全、分子生物学研究。E-mail:475946790@qq.com
*通信作者:从彦丽(1966—),女,副教授,硕士,主要从事生物活性物质、食品营养和安全研究。E-mail:congyl@szpt.net

