蒺藜苜蓿EST-SSRs分布特征及标记的开发
屠德鹏,魏臻武,武自念,雷艳芳,张 栋,邱伟伟
(1.扬州大学动物科学与技术学院 扬州大学草业科学研究所,江苏 扬州 225009; 2.上海鼎牛饲料有限公司,上海 200436)
随着分子生物学、基因组学和功能基因组学的快速发展,分子标记技术的应用也越来越广泛[1-3]。苜蓿(Medicagosativa)分子标记辅助育种是加速苜蓿育种进程的重要手段之一。魏臻武[4]利用SSR、ISSR、RAPD标记技术构建了苜蓿的指纹图谱,指纹图谱可以鉴定苜蓿品种、品系和选择杂交亲本,为育种工作提供保障。在众多的分子标记中,SSR(simple sequence repeat)标记以含量丰富、遍布整个基因组、共显性、多态性高等优点被广泛应用[5-7]。传统SSR引物开发一般是通过构建基因组文库、Southern杂交、基因克隆和测序等程序进行的,该方法工作量大、成功率低且成本较高[5]。
蒺藜苜蓿(M.truncatula)作为豆科模式植物,具有遗传转化效率高、生长期短、基因组小、自花授粉、固氮等优点[8-10]。蒺藜苜蓿与苜蓿亲缘关系很近,与苜蓿基因组具有很高的同源性。因而从蒺藜苜蓿获得的信息可以用于苜蓿中,这对于促进豆类作物和苜蓿等牧草的育种有重要意义。随着对蒺藜苜蓿基因组学的深入研究,SSR标记已不能满足蒺藜苜蓿高密度遗传图谱构建、基因克隆等研究的需要。因此,需要开发大量新的标记。
表达序列标签(expressed sequence tags,EST)是指从不同组织来源的cDNA文库中随机挑选克隆进行5′或3′端测序后得到的长约300~500 bp的基因表达序列片段[11-12]。EST-SSRs是基于EST的新型分子标记,EST-SSRs与基因组SSR标记相比不但具有可以直接获得基因表达信息,为功能基因提供可靠标记的优点,而且具有通用性高、开发简单且成本较低等优点[13]。在小麦(Triticumaestivum)[14]、大豆(Glycinemax)[15]、苜蓿[4,16]、高羊茅(Festucaarundinacea)[13]、鸭茅(Dactylisglomerata)[17]、鹰嘴豆(Cicerarietinum)[18]、高粱(Sorghumbicolor)[19-20]等植物的EST序列中检测到2%~10%的EST-SSRs。从而可见,EST中含有大量的SSR,为SSR标记的开发提供了一个巨大的DNA序列来源。目前,NCBI上公布的蒺藜苜蓿EST已达285 285条,大量EST的存在为蒺藜苜蓿EST-SSRs标记的开发奠定了基础。
该试验利用NCBI上公布的蒺藜苜蓿EST序列,查找EST-SSRs,分析SSR在蒺藜苜蓿EST中的分布特征。设计EST-SSRs引物,利用蒺藜苜蓿重组自交系 (RIL)群体及其亲本进行电泳检测,开发新的EST-SSRs标记。这些新的EST-SSRs标记,为蒺藜苜蓿高密度遗传图谱的构建、遗传多样性、基因克隆、数量性状基因位点(QTL)定位及引物通用性等方面的研究提供材料来源。
1 材料与方法
1.1材料 试验材料为来自法国的蒺藜苜蓿Jemalong中的A20和A17构建的RIL-8群体及亲本,该群体目前有143个家系。群体和亲本2009年10月种植在扬州大学草业科学实验基地,试验地土壤肥力中等偏上,有机质含量为1.2%,全氮含量0.12%,碱解氮100.4 mg/kg,速效磷88.7 mg/kg,速效钾36.3 mg/kg。条播,行距0.7 m,小区面积为1 m×2 m。全年不施肥。适时排灌水,人工除草。
1.2方法
1.2.1DNA的提取 采用魏臻武[4]稍加改进的CTAB法提取基因组DNA。用0.8%琼脂糖凝胶和紫外分光光度计对其质量和浓度进行检测。提取的DNA溶解于100 μL TE后放入4℃冰箱备用。
1.2.2蒺藜苜蓿EST序列中SSR的检索 从NCBI(http://www.ncbi.nlm.nih.gov/)网上下载285 285条蒺藜苜蓿EST序列。利用SSRIT(simple sequence repeat identification tool)软件检索重复单元为二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸的SSR,标准为重复序列长度≥20 bp,即二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸重复次数分别大于或等于10、7、5、4、4。
1.2.3EST-SSRs引物设计 利用Primer Premier 5.0软件进行引物设计,引物设定的条件如下:引物长度范围为18~22 bp;引物退火温度在50~60℃,上下引物退火温度相差不超过2℃;GC含量在40%~60%,最佳为50%,且上下引物序列GC含量的差异不能太大,3′端最后5个碱基最好不要富含GC,特别是连续的3个G或C;扩增产物在100~600 bp;尽量避免形成稳定的引物二聚体(dimer and cross dimer)和发夹结构(hairpin)。设计的引物由上海英骏生物技术有限公司合成。
1.2.4SSR扩增反应体系 DNA模板(20~90 ng/μL)3 μL;引物(10 pmol/μL)3 μL;反应体系混合物配比:0.24 μL dNTP (10 mmol/L) +1.5μL 10×Buffer + 0.9 μL Mg2+(20 mmol/L) + 0.16 μL TaqDNA聚合酶(1U) +1.2 μL ddH2O[11]。反应体系为10 μL(每孔添加量)。
扩增反应程序:94℃预变性3 min,95℃变性1 min,52℃退火1.5 min(不同引物退火温度不同),72℃延伸1 min,共循环35次,最后72℃保温8 min,4℃保存。
1.2.5凝胶电泳及银染 PCR扩增产物中加入1 μL Loading buffer(溴酚蓝缓冲液),扩增产物用8%非变性聚丙烯酰胺凝胶(PAGE)电泳,150 V恒电压,然后银染检测,拍照保存。
2 结果与分析
2.1蒺藜苜蓿EST-SSRs的检索 从285 285条EST序列中共检测SSR序列6 688条,分布于6 512个EST序列中。EST-SSRs占所有EST总数的2.28%,其中二、三、四、五和六核苷酸重复基元分别占总SSR的34.4%、29.6%、10.2%、11.8%和14.0%,二核苷酸和三核苷酸出现频率最高,四核苷酸重复基元出现的频率最低,为10.2%(表1)。
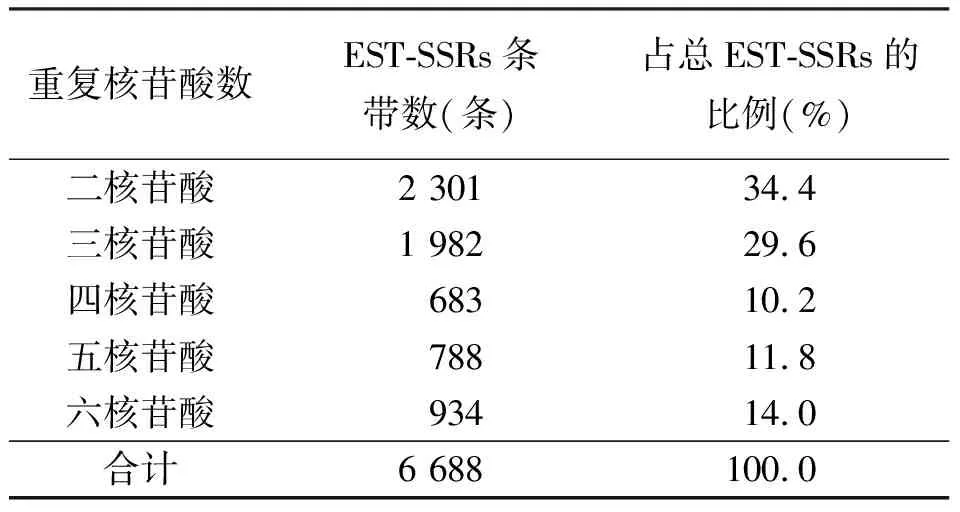
表1 EST-SSRs序列中不同重复核苷酸数的数量及百分比
2.2蒺藜苜蓿EST-SSRs的分布特征 在检测到的2 301条含有二核苷酸重复基元的EST-SSRs中,含GA/TC和AG/CT重复基元最多,分别占SSRs总数的17.49%(1 170条)和11.21%(55条)(图1);其中CG/CG重复基元没有出现。三核苷酸重复基元中以GAA/TTC出现的频率最高,为7.10%;其次是AAG/CTT、AGA/TCT、ATG/CAT和AAT/ATT,占总SSRs比例分别为4.28%、3.75%、1.67%和1.50%,其他重复基元出现的频率较低。四核苷酸、五核苷酸和六核苷酸出现频率较高的为AATT/AATT(1.58%)、CTCA/TGAG(1.48%)、AAATC/GATTT(0.57%)和TCTAAA/TTTAGA(1.06%)。其余各重复基元频率都较低。
对于重复次数,二核苷酸基元的重复次数类型多、跨度大。其中GA/TC基元跨度最大,达72种,重复次数为10~87次;其次为AG/CT基元,跨度为47种,重复次数为10~79次;AT/AT和TA/TA基元跨度也较大,重复次数分别为10~34和10~37。三核苷酸基元重复次数类型和跨度比二核苷酸小,其中GAA/TTC(11种)、AGA/TCT(8种)、 AAG/CTT(7种)和ACT/AGT(6种),其他重复基元的重复次数多以7、8、9次出现。四核苷酸中ATAG/CTAT(4种)和CTCA/TGAG(4种)跨度较大,其他类型重复次数多以4、5次出现。五核苷酸和六核苷酸重复基元的重复次数多以4、5、6次中的一种或两种出现(表2)。
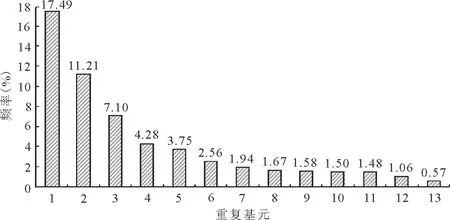
图1 蒺藜苜蓿EST-SSRs主要重复基元分布图

表2 蒺藜苜蓿EST-SSRs各基元类型的重复次数分布
2.3蒺藜苜蓿EST-SSRs电泳筛选结果 利用Primer Premier 5.0软件对6 688条EST-SSRs进行引物设计,去除重复等不合适的引物共设计出3 090对,随机选取100对引物合成,以RIL-8群体亲本A17和A20的DNA为模板进行扩增、筛选,筛选出多态性的引物在RIL-8群体中扩增、电泳(图2)。在A17和A20上有清晰条带为85对,占所设计引物比例的85%。其中有多态性的引物29对(表3),占34.1%(图3)。多态性引物均能在RIL-8群体中扩增出稳定的产物。
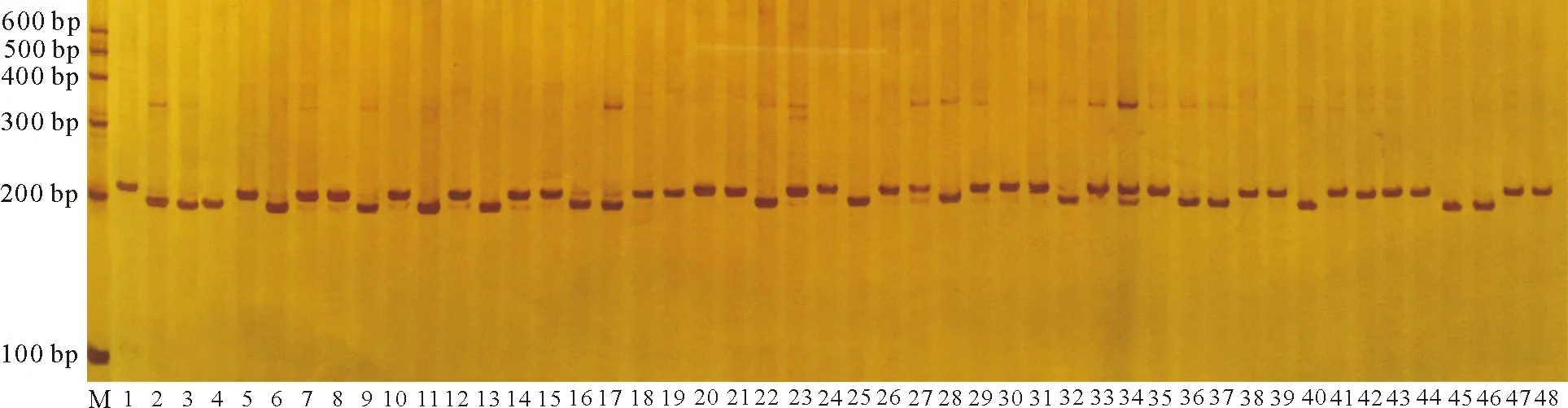
图2 MES028在部分蒺藜苜蓿RIL-8群体上的电泳图
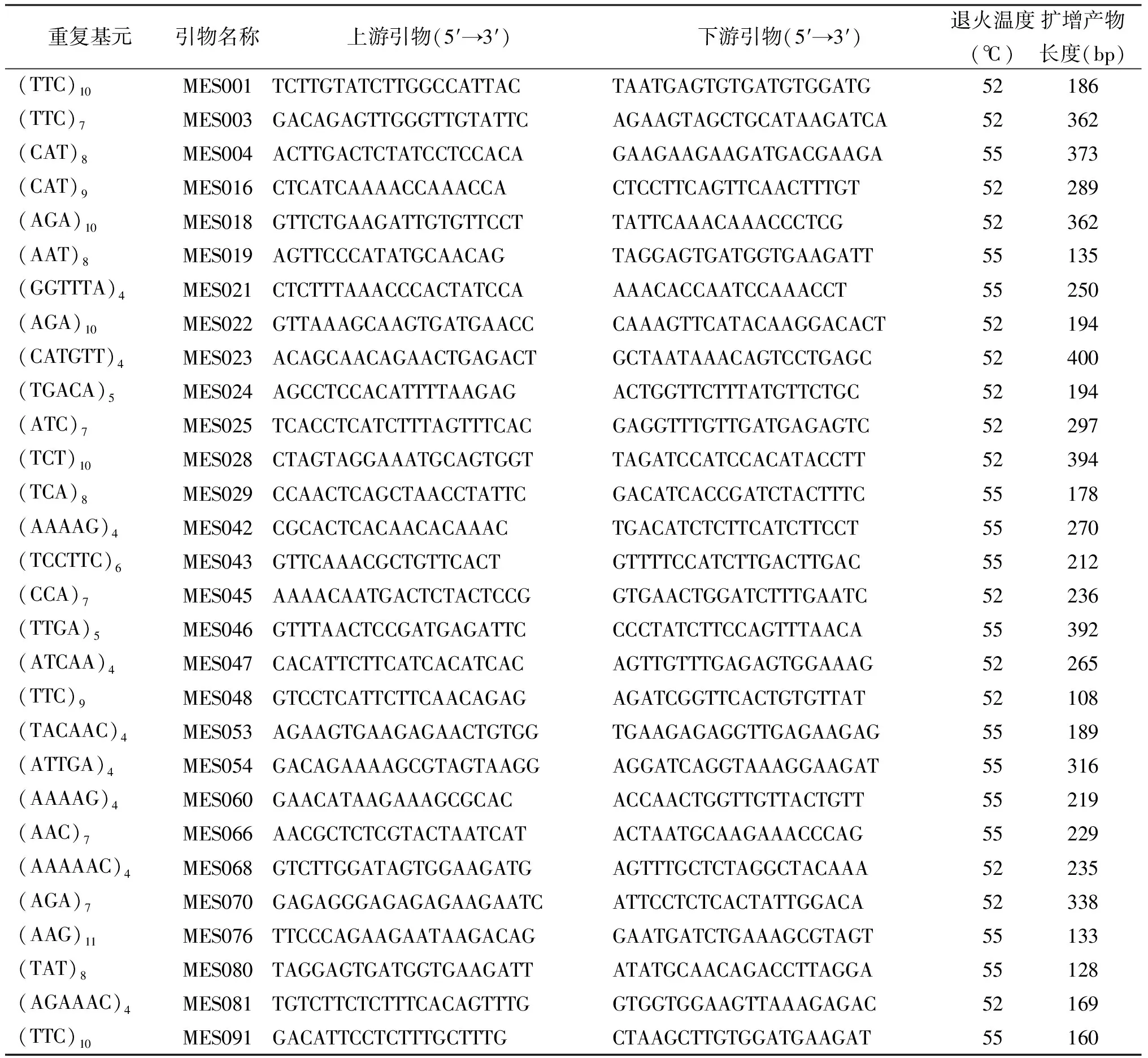
表3 29对蒺藜苜蓿EST-SSRs多态性引物

图3 MES001~MES024在蒺藜苜蓿A17和A20上的电泳图
3 讨论
3.1EST-SSRs的分布特征 在小麦[14]、大豆[15]、莴苣(Lactucasativa)[21]等大多数植物的EST-SSRs中发现,三核苷酸重复出现的频率比二核苷酸重复高。而在本研究中EST-SSRs的分布二核苷酸基元(34.4%)略大于三核苷酸基元(29.6%)。在鹅掌楸(Liriodendronchinense)[22]、芝麻(Sesamumindicum)[23]、砂梨(Pyruspyrifolia)[24]等中也得到了同样的结论。这可能是与本研究中检索SSR的标准为重复序列≥20 bp有关。Thiel等[25]的研究结果中显示18 bp长度时,三核苷酸重复基元出现的频率高于二核苷酸重复基元。大多数作物中二核苷酸重复基元以GA/TC出现的频率最高,而CG/CG重复基元出现的频率最低,如水稻(Oryzasativa)、玉米(Zeamays)、大豆、高粱等[26-27]。本试验中GA/TC出现的频率是最高的,而CG/CG没有出现,该结论与大多数植物的研究结果一致。不同植物三核苷酸重复基元主要的重复类型变化较大,小麦以AAC/GTT出现的频率最高[14],大麦(Hordeumvulgare)、玉米、水稻和高粱中则出现的频率最高是GGC/GCC[28];鹅掌楸中是以AAG/CTT重复基元出现频率较高,占三核苷酸重复基元的40.2%[22](表4);本研究检测到的1 982条三核苷酸重复基元中GAA/TTC出现的频率最高,为475条,占总检测SSR的7.1%,占三核苷酸重复基元的24%,本研究结论与在大豆上的检测结果是一致的[15]。
3.2EST-SSRs标记的开发 随着分子标记和测序技术的快速发展,EST-SSRs的应用也越来越普遍,为EST-SSRs标记的开发提供了广阔的前景。截至到2010年6月11日,从NCBI中的dbEST数据库中蒺藜苜蓿的EST序列已达到285 285条,与2002年12月31日的147 000条EST相比,增加了将近一倍。目前,在多种作物中EST-SSRs都展开了研究,陈海梅等[14]在小麦的EST中检测到1.34%的序列中含SSR,并将43个EST-SSRs位点绘制到小麦的遗传图谱上;常玮等[15]在大豆的458 220条EST中获得无冗余的SSR序列8 190条,设计的200对引物中148对有清晰且单一条带扩增产物,开发了148个新的SSR标记。在牧草中EST-SSRs的研究也比较普遍,Eujayl等[6]研究蒺藜苜蓿EST-SSRs在苜蓿属中的通用性,结果表明74%的EST-SSRs引物在苜蓿属中有扩增产物;Sim等[29]研究了谷物的EST-SSRs在黑麦草(Loliumspp.)中的通用性,来自谷物的165个EST-SSRs中,57%的EST-SSRs在黑麦草中有扩增产物,且多态率达67%; Xie等[17]利用50对谷物(玉米、小麦和高粱)的EST-SSRs和15对鸭茅SSR引物对74个鸭茅材料进行护增,其多态率为84.63%;Simko[21]从莴苣的19 523条EST中检测到4.5%的EST含有SSR序列(重复序列≥20 bp)。本研究在285 285条EST中检测到6 512条EST含有SSR序列,SSR序列为6 688条。随机设计的100对引物中,在蒺藜苜蓿RIL-8群体亲本A17和A20中85对引物有扩增产物,开发了85个新的SSR标记。具有多态性的引物为29对。

表4 部分植物出现频率最高的核苷酸重复基元
4 结论
1)从NCBI数据库285 285条EST序列中检测到6 688条SSR序列。占 EST总数的2.28%,二核苷酸出现频率最高,其次为三核苷酸,二、三核苷酸占所有核苷酸的64%。四核苷酸分布最少,为10.2%。
2)蒺藜苜蓿基因组中GA/TC和AG/TC重复基元占SSRs总数的17.49%和11.21%,在各种重复基元中最多。蒺藜苜蓿基因组中CG/CG重复基元没有出现。
3)利用EST-SSRs序列随机设计100对引物,在蒺藜苜蓿RIL-8群体亲本A17和A20中扩增、电泳,85对引物具有清晰的扩增条带,开发了85个新的SSR标记。具有多态性的引物为29对。这些EST-SSRs标记可以为蒺藜苜蓿高密度遗传图谱的构建、遗传多样性、基因克隆、QTL定位及引物通用性等方面的研究提供材料来源。
[1] 魏臻武,盖钧镒.豆科模式植物蒺藜苜蓿基因组研究进展[J].中国草地学报,2006,28(6):83-90.
[2] Town C D.Annotating the genome ofMedicagotruncatula[J].Current Opinion in Plant Biology,2006,9:122-127.
[3] Young N D,Udvardi M.TranslatingMedicagotruncatulagenomics to crop legumes[J].Current Opinion in Plant Biology,2009,12:193-201.
[4] 魏臻武.利用SSR、ISSR和RAPD技术构建苜蓿基因组DNA指纹图谱[J].草业学报,2004,13(3):62-67.
[5] 陈怀琼,隋春,魏建和.植物SSR引物开发策略简述[J].分子植物育种,2009,7(4):845-851.
[6] Eujayl I,Sledge M K,Wang L,etal.MedicagotruncatulaEST-SSRs reveal cross-species genetic markers forMedicagospp.[J].Theoretical and Applied Genetics,2004,108:414-422.
[7] 谢文刚,张新全,马啸,等.中国西南区鸭茅种质遗传变异的SSR分析[J].草业学报,2009,4(18):138-146.
[8] 魏臻武,盖钧镒.豆科模式植物——蒺藜苜蓿[J].草业学报,2008,17(1):114-120.
[9] May G D,Dixon R A.Medicagotruncatula[J].Current Biology,2000,14(5):181-182.
[10] Cook D.Medicagotruncatula——a model in the making[J].Current Opinion in Plant Biology,1999,2:301-304.
[11] 胡松年.基因表达序列标签[M].杭州:浙江大学出版社,2005.
[12] 王晓娜,卢欣石.表达序列标签的应用现状及分析方法研究[J].草业科学,2010,27(5):76-84.
[13] Tehrani M S,Mardi M.Genetic diversity and structure among Iranian tall fescue populations based on genomic-SSR and EST-SSR marker analysis[J].Plant Systematics and Evolution,2009,282:57-70.
[14] 陈海梅,李林志,卫宪云,等.小麦EST-SSR标记的开发、染色体定位和遗传作图[J].科学通报,2005,50(20):2208-2216.
[15] 常玮,赵雪,李侠,等.大豆EST-SSR标记开发及与Genomic-SSR的比较研究[J].中国油料作物学报,2009,31(2):149-156.
[16] Sledge M,Ray I,Mian M A.EST-SSRs for genetic mapping in alfalfa[J].Molecular Breeding of Forage and Turf,2004,12:239-243.
[17] Xie W G,Zhang X Q,Cai H W,etal.Genetic diversity analysis and transferability of cereal EST-SSR markers to orchardgrass (DactylisglomerataL.)[J].Biochemical Systematics and Ecology,2010,38:740-749.
[18] Choudhary S,Sethy N K,etal.Development of chickpea EST-SSR markers and analysis of allelic variation across related species[J].Theoretical and Applied Genetics,2009,118:591-608.
[19] Shiringani A L,Frisch M,Friedt W.Genetic mapping of QTLs for sugar-related traits in a RIL population ofSorghumbicolorL.Moench[J].Theoretical and Applied Genetics,2010,121:323-336.
[20] 李杰勤,王丽华,詹秋文,等.高粱EST-SSR标记的建立及其在苏丹草中的应用初探[J].草业科学,2010,27(3):112-117.
[21] Simko I.Development of EST-SSR markers for the study of population structure in Lettuce (LactucasativaL.)[J].Journal of Heredity,2009,100(2):256-262.
[22] Xu M,Sun Y G,Li H G.EST-SSRs development and paternity analysis forLiriodendronspp.[J].New Forests,2010,40(3):361-382.
[23] 魏利斌,张海洋,郑永战,等.芝麻EST-SSR标记的开发和初步研究[J].作物学报,2008,34(12):2077-2084.
[24] 崔海荣,刘金义,佟兆国,等.砂梨EST-SSR引物开发及其应用[J].西北植物学报,2010,30(8):1551-1556.
[25] Thiel T,Michalek W,Varshney R K,etal.Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (HordeumvulgareL.)[J].Theoretical and Applied Genetics,2003,106:411-422.
[26] Li L Z,Wang J J,Guo Y,etal.Development of SSR markers from ESTs of gramineous species and their chromosome location on wheat[J].Progress in Natural Science,2008,18:1485-1490.
[27] Liu Y L,Li Y H,Zhou G A,etal.Development of soybean EST-SSR markers and their use to assess genetic diversity in the subgenus soja[J].Agicultural Sciences in China,2010,9(10):1423-1429.
[28] Kantety R V,Rota M L,Matthews D E,etal.Data mining for simple sequence repeats in expressed sequence tags from barley,maize,rice,sorghum and wheat[J].Plant Molecular Biology,2002,48:501-510.
[29] Sim S C,Yu J K,Jo Y K,etal.Transferability of cereal EST-SSR markers to ryegrass[J].Genome,2009,52:431-437.

