香蒲拟发网菌12S rDNA的PCR扩增及序列分析1)
张旭 潘景芝 刘福杰 王琦 李玉
(食药用菌教育部工程研究中心(吉林农业大学),长春,130118)(长春市传染病医院)(食药用菌教育部工程研究中心(吉林农业大学))
黏菌(Myxomycetes)在生物界中是介于原生动物与真菌之间的一个特殊类群,其名称、类群范围及分类地位,在学者们中的见解始终不一致。虽然现代生物分类学的发展日益趋于多界系统,但黏菌仍然是一个存在争议的类群[1]。黏菌的分布是世界性的,其生境最常见于林中阴凉湿润的地方。黏菌在原生质团时期生活在潮湿的木头缝隙和树皮里面,爬行摄食,形成子实体时移到较干燥的基物表面,如腐朽木段、枯枝落叶、树皮草茎。本研究在其它黏菌分子生物学研究的基础上[2-7],将现代分子生物学方法引入香蒲拟发网菌的系统分类,采用CTAB法对基物培养[8-12]获得的研究标本进行DNA提取,用White等[13]设计的引物对其rDNA片段进行扩增和序列测定分析。探讨了香蒲拟发网菌的分子系统学关系,为其系统演化研究提供理论依据。
1 材料与方法
香蒲拟发网菌的基物培养:将所采基物放于铺满滤纸的搪瓷盘中,用无菌水充分湿润浸透,加塑料布遮盖,给予一定散射光,在室温(15~25℃)下培养,观察黏菌原生质团和子实体的发育过程,至子实体成熟时,收集,阴干后放于标本盒中保存,镜检鉴定。
总DNA的提取:①CTAB DNA提取液的配制。CTAB 20 g/L,Tris-HCl(pH值8.0)50 mmol/L,Na2EDTA 10 mmol/L,NaCl 0.7 mmol/L。②TE缓冲液的配制。10 mmol/L Tris,1 mmol/L EDTA(pH值8.0)。③破壁。取少量孢子(0.1 mg)置于1.5 mL离心管中,加入100 μL CTAB提取液,用玻璃研磨杵研磨。④消化。向离心管中再加入300 μL CTAB提取液,轻轻摇动。将管置于65℃水浴中消化1.0~1.5 h,在此过程中轻轻摇动2~3次。⑤抽提:加入等体积预冷的氯仿—异戊醇(V(氯仿)∶V(异戊醇)=24∶1),剧烈振荡使之形成悬乳状。12 000 r/min离心10 min,将上清液移至一干净的离心管中,重复2次。⑥沉淀。加入3 mol/L NaAc(100~130 μL)使溶液终浓度为0.1 mol/L。加入2倍体积预冷的无水乙醇,上下转动混匀,放入-20℃冰箱过夜,沉淀DNA。⑦洗涤。12 000 r/min离心15 min,弃去上清液,用预冷的75%乙醇洗涤DNA沉淀,然后弃去冷乙醇。共洗涤2次。管中乙醇倒净后,将管口倾斜倒置在滤纸上,使乙醇完全挥发。⑧溶解。加入30 μL TE缓冲液溶解DNA沉淀,-20℃保存备用。
PCR扩增:PCR扩增引物由北京三博远志生物技术有限责任公司合成,12S上游引物(5'-AAG GAG CCG GTA TCA AGT A-3'),12S下游引物(5'-TAG AGG GAT GTG AAG TGC C-3')[13]。扩增反应体系为10倍PCR Buffer 5.0 μL,25 mmol/L MgCl25.0 μL,dNTP 5.0 μL,引物各2.0 μL,Taq酶0.6 μL,模板4~6 μL,最后以ddH2O水补足至总体积50 μL。PCR反应条件为94℃变性50 s;48~55℃退火50 s,72℃延伸1 min,共循环35次,最后再72℃延伸10 min。在上述PCR的反应条件下,将DNA模板原液用TE缓冲液按1∶5,1∶10,1∶15梯度稀释,改变引物浓度及各项反应条件,从而获取最佳的DNA扩增条件。
PCR扩增产物的检测:取PCR反应产物2.5 μL,在经溴化乙锭染色的1%的琼脂糖凝胶上电泳25 min(200 V)后取出,在FR-200紫外与可见电泳分析装置上观察,并照相。
PCR产物的序列测定:将PCR原液送至北京三博远志生物技术有限责任公司进行序列测定。
序列比对和系统发育树的构建:采用Clustal X 1.83对NCBI上已注册的黏菌12S rDNA基因序列片段进行比对,经BioEdit version7.0.9.0人工调整后,运用分析软件MEGA4中基于缺失位点百分数的临位相连法构建系统发育树,同时进行2000次Bootstrap自举法检验。
2 结果与分析
2.1 香蒲拟发网菌子实体及其孢丝、孢子形态
孢囊群生,有柄,窄圆柱形,顶端稍窄或钝圆,直立,少数弯曲,全高3~5 mm,宽0.5~2.0 mm。囊被凋落。柄黑褐色,长为全高的1/3~1/2,囊轴黑色,向上渐细。孢丝稠密,弯曲,分枝并联结。基质层膜质,褐色。孢子成堆时浅紫褐色,球形,有小疣,直径7~9 μm。根据《中国真菌志——黏菌卷二》[14]鉴定为香蒲拟发网菌(图1)。
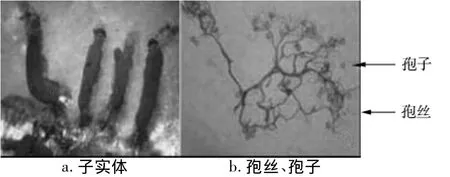
图1 香蒲拟发网菌及其孢丝、孢子
2.2 DNA提取
通过CTAB法得到高质量的黏菌DNA溶液,可以用于下一步试验。DNA电泳结果见图2。
2.3 PCR扩增
PCR反应适宜条件:所用DNA模板浓度以1∶5稀释扩增效果最好。最佳的反应体系为10倍PCR Buffer 5.0 μL,25 mmol/L MgCl25.0 μL,dNTP 5.0 μL,引物各2.0 μL,Taq酶0.6 μL,模板4 μL,最后以ddH2O补足至50 μL。
最佳反应温度与时间:94℃变性50 s,在退火温度为48℃、时间50 s时出现清晰的扩增条带(图3),然后72℃延伸1 min,共35个循环,最后72℃再延伸10 min。
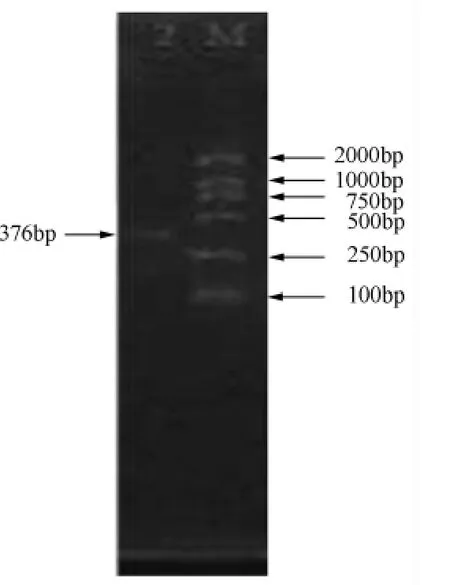
图3 香蒲拟发网菌PCR扩增结果
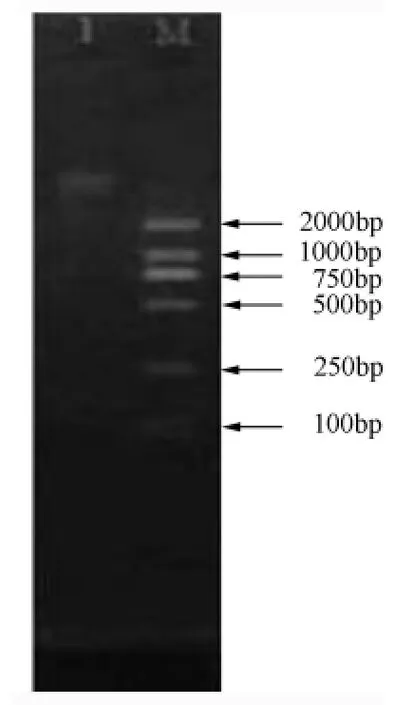
图2 香蒲拟发网菌DNA电泳结果
2.4 测序结果
香蒲拟发网菌12S rDNA片断序列见图4。该序列全长376 bp,GenBank登录号为HM102318。

图4 香蒲拟发网菌12S rDNA片断序列
2.5 序列比对和系统发育树的构建
香蒲拟发网菌与NCBI上查到的9个黏菌的12S rDNA序列片断比对结果见图5。
粉瘤属的大粉瘤菌和筒菌属的筒菌聚在一起,但筒菌的分化早于大粉瘤菌。粉瘤属的大粉瘤菌,绒泡菌属的圈绒泡菌、钙皮菌属的黑柄钙皮菌和半网菌属的棒形半网菌聚集在一起,其Bootstrap支持率为100%,显示出十分密切的亲缘关系。团网菌属的灰团网菌与双皮菌属的辐射双皮菌聚在一个分支上。拟发网菌属的香蒲拟发网菌与煤绒菌属的煤绒菌及半网菌属蛇形半网菌聚在一个分支上,其支持率分别为66%和46%。
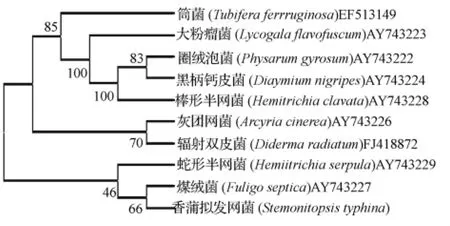
图5 基于12S rDNA序列分析得到的NCBI已注册黏菌的系统发育树
3 结论与讨论
基物培养是黏菌研究的一种必备手段,通过它可以丰富所考察地区的黏菌分布,弥补野外采集工作的不足。本试验所采用的湿室培养方法简便,无需对器皿和试剂进行消毒灭菌,使得以后的培养工作易于进行。从培养条件上看,变温条件下湿室培养产生黏菌的频率较高,温度恒定或过于极端则不利于黏菌子实体的产生,可能是由于变温的条件更接近于采集地自然的温度,有利于黏菌子实体的形成。培养中对光照无苛求,只要保证水份湿度,让其在自然的变温和散射光条件下就可生长。
在提取黏菌DNA过程中,最重要因素即为细胞壁破壁过程,适当增加研磨时间可以提高提取DNA的质量。相对于玻片压碎法[3]及匀浆器研磨法[15],应用玻璃研磨杵在离心管中直接研磨黏菌孢子[16],既可以避免匀浆器研磨后转入离心管过程中标本的损失,又可以弥补玻片压碎法中玻片仅能对黏菌孢子破壁的不足,从而可以更好地利用有限的标本资源,提取高质量黏菌DNA。
线粒体DNA是生物体内的核外遗传信息载体,系共价闭环的环状分子,分子量小,基因组中一般没有间隔序列,结构简单,为严格的母系遗传,几乎不发生倒位、易位等畸变与重组,使之容易被检测。因而线粒体DNA越来越广泛地被应用于各类群的系统进化研究。而12S rDNA基因是线粒体DNA上2个rRNA基因之一,在结构上存在4个结构域,第三结构域是相当保守的。该结构域包括32至48号茎,高度保守的侧翼序列使它成为12S rDNA基因中最常被扩增的区域。同时,12S rDNA基因进化速度较16S、18S、28S rDNA快。因此12S rDNA基因序列适用于研究生物类群的种属间系统发生关系[17]。
目前,GenBank中关于黏菌12S序列的报道仅有10个种,且关于香蒲拟发网菌分子生物学方面的研究尚未见报道。本研究首次成功地对香蒲拟发网菌的12S rDNA片段进行了PCR扩增。该序列的测得为进一步研究黏菌的系统演化提供了依据。由于部分黏菌的地域性及其培养条件的不成熟在一定程度上限制了黏菌标本的来源,进而限制了其分子生物学方面的发展,虽然基物培养在一定程度上解决了这个问题,但其培养条件及基物的选择还有待进一步研究。
与传统的形态学分类不同,在对已知的10个黏菌12S rDNA的序列比对中可以看出团网菌属、半网菌属、粉瘤属、筒菌属、绒泡菌属、钙皮菌属和双皮菌属黏菌的关系较近,在演化的过程中交替出现。拟发网菌属与煤绒菌属、半网菌属聚在一个分支上,但是其Bootstrap支持率不高,只能作为参考。黏菌系统发育的研究需要大量的黏菌基因序列对其分类进行支持,为以后的研究提出了思路。选用不同的分子标记也会造成截然不同的结论,这种现象在分子系统学研究中并不少见,可能是因不同的DNA标记在进化的各阶段所起作用不同,或是统计分析方法中存在的某种缺陷所致,仍有待于作进一步研究。在研究某一特定类群的详细系统亲缘关系时,单凭某一种或某几种方法是不够的,必须综合多方面资料(形态、生理、生化、古生物等)进行全面的考查与分析。DNA序列比对结果做为研究中的证据之一,还须结合形态学等多种手段,相互印证,才可得出符合客观规律的结论,从而为物种的系统发育、生态学等研究提供重要的信息。
[1]Kirk P M,Cannon P F,Minter D W,et al.Ainsworth&Bisby's dictionary of the fungi[M].10th ed.U K:CABI Bioscience,CAB International,2008.
[2]刘淑艳,李玉.首次用PCR法扩得长尖团毛菌小亚基rDNA片段[J].吉林农业大学学报,2004,26(3):291-292.
[3]刘淑艳,李玉.首次用一对引物扩得大粉瘤菌小亚基rDNA长片段[J].菌物系统,2003,22(1):74-76.
[4]刘淑艳,李玉.黑发菌核糖体DNA小亚基片段的序列测定[J].菌物研究,2003,1(1):5-8.
[5]白秀娟,李玉,刘淑艳.粘菌基因组DNA提取方法的研究[J].吉林农业大学学报,2003,25(2):158-160.
[6]Kamono A,Fukui M.Rapid PCR-based method for detection and differentiation of Didymiaceae and Physaraceae(myxomycetes)in environmental samples[J].Journal of Microbiological Methods,2006,67(3):496-506.
[7]Kamono A,Matsumoto J,Kojima H,et al.Characterization of myxomycete communities in soil by reverse transcription polymerase chain reaction(RT-PCR)-based method[J].Soil Biology&Biochemistry,2009,41(6):1324-1330.
[8]王琦.中国团毛菌科四个属的分类研究及双毛菌属DITRICHIA的建立[D].沈阳:沈阳农业大学,1994.
[9]王琦,李玉.中国团毛菌目黏菌分支分类研究[J].吉林农业大学学报,2005,27(6):617-620.
[10]周宗璜,张志澄,刘宗麟.从基物培养获得的几种粘菌[J].吉林农业大学学报,1981,3(2):1-9.
[11]赵曰锋.粘菌基物培养的研究[J].吉林农业大学学报,1983,5(2):67-74.
[12]陈双林,李玉.粘菌湿室培养的初步研究[J].吉林农业大学学报,1995,17(3):33-37.
[13]White T J,Bruns T,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]//Innis M A,Gelfand D H,Sninsky J J,et al.PCR Protocols:A guide to methods and applications.San Diego,California:Academic Press,1990:315-322.
[14]李玉,李惠中,王琦,等.中国真菌志:2卷[M].北京:科学出版社,2007:127-128.
[15]刘朴,白秀娟,王琦.筒菌12S rDNA的PCR扩增及序列测定[J].吉林农业大学学报,2007,29(6):633-635.
[16]潘景芝,张旭,刘福杰,等.蓝灰团网菌ITS rDNA的PCR扩增及序列测定[J].东北林业大学学报,2010,38(10):103-105.
[17]郑冬,刘学东,马建章.12S rRNA基因及其二级结构在系统学研究中的应用[J].东北林业大学学报,2003,31(3):59-61.

