两种蛤仔线粒体16S rRNA基因和COⅠ基因的序列比较
刘相全,包振民,方建光,王如才
(1.山东省海洋水产研究所,山东 烟台 264006; 2.中国海洋大学,山东 青岛 266003; 3.中国水产科学研究院 黄海水产研究所,山东 青岛 266071)
线粒体DNA(mtDNA) 由于具有分子质量小、结构简单、表现为母系遗传、进化速度快等特点,成为动物群体遗传学和系统进化研究中的重要对象。对于选定的基因或基因片段,可通过互补于高度保守区域的通用引物进行 PCR 扩增,然后进行分子克隆测序或 PCR产物直接测序,对序列数据进行分析。在海洋贝类方面,根据mtDNA区段或基因的序列来构建分子系统树,并进而进行分类和进化分析的研究已有许多报道。
Canapa等[1-3]根据16S rRNA基因序列分别对8种、14种帘蛤科贝类进行聚类分析表明,缀锦蛤亚科的 6个种类尽管可以聚在一起,但在属的水平上的分类则不能很好的对应; 对 8种扇贝科贝类的系统发生进行了研究,发现有两种扇贝应为同一种类。Barucca等[4]分析了 23种扇贝科贝类的 16S和 12S基因片段,两个片段反应的扇贝间的亲缘关系基本一致。Matsumoto等[5-6]根据 COⅠ的氨基酸序列对17种扇贝科贝类的分类进行了研究,结果支持Waller的形态学分类系统; 对翼形亚纲的分类进行了研究,并与18S rDNA的分类结果进行了比较。Yu等[7]根据16S和COⅠ两个片段的序列分析了中国沿海 4种牡蛎的分类情况。杨建敏等[8]研究了 3 种鲍的 16S rRNA 基因。Therriault等[9]根据 16S和COⅠ两个片段的序列对黑海饰贝科的分类进行了研究,分清了同物异名的两个种; 16S和 COⅠ两个片段对种内生态型之间的差异则不能很好地区分。
蛤仔属(Ruditapes)作为帘蛤科的一个属,是Fischer-Piette等对缀锦蛤亚科订正后提出的。在我国蛤仔属只有两个种,即菲律宾蛤仔(R.philippinarum)和杂色蛤仔(R.variegata)。为了进一步比较这两种在外形上酷似的帘蛤科贝类,本文通过对线粒体 DNA的16S和COⅠ两个片段进行扩增、测序,分析了两个序列的基本特征,并引用Genbank中其他2种贝类的序列,进行了系统发生的比较。
1 材料与方法
1.1 样品收集和DNA提取
菲律宾蛤仔(R.philippinarum,简称 RP)取自青岛,杂色蛤(R.variegata,简称RV)取自湛江硇洲岛。常规酚氯仿法提取基因组DNA。
1.2 PCR扩增、克隆和测序
PCR扩增用引物:16S采用Palumbi等[10]设计的通用引物,16sar-L (5′-CGCCTGTTTATCAAAAACAT-3′)和 16sbr-H (5′-CCGGTCTGAACTCAGATCACGT-3′); COⅠ采用 Folmer 等[11]设计的引物COIL1490/COIH2198:GGTCAACAAATCATAAAGATATTGG/TAAACTTCAGGGTGACCAAAAAATCA。PCR扩增采用 25μL体系,包括 30~50 ng基因组DNA,0.2 mmol/L dNTP,1 μmol/L 引物,缓冲液 (10 mmol/L Tris-HCl,1.5 mmol/LMgCl2,50 mmol/L KCl),以及 0.025 UTaq酶/μL。94℃变性2 min,然后35个循环(94 ℃ 40 s,56℃1min,72℃1min)最后72℃延伸5 min。扩增片段在2%的琼脂糖胶上检测。PCR产物连接到PMD18-T,然后转化到E.coliDH5α细胞,37℃培养,涂平板,挑克隆,载体引物检测,双向测序。序列提交到Genbank。
1.3 序列分析
测序结果去掉两端引物序列后,用CLUSTALX 1软件进行比对,用MEGA 2软件(Kumar等,2001)的距离法对序列进行分析。根据Kimura(1980)双参数法计算距离,产生的矩阵根据 NJ法(Saitou and Nei,1987)分析系统发生的关系。系统发生关系的置信度用自展(bootstrap)重复分析验证(重复次数 NJ为 1 000,MP为 100)。引用的序列包括硬壳蛤(Mercenaria mercenaria,简称 MM。16S:AJ548773,COⅠ:U47648)和白斑乌贼(Sepia latimanus,简称Sla。16S:AB192322,COⅠ:AB192338)以及菲律宾蛤仔16S的序列AF484296。菲律宾蛤仔COⅠ和杂色蛤仔 COⅠ、16S的序列号为 JN248565~JN248567。
2 结果
每种蛤仔用10只个体进行PCR扩增,所有扩增结果均为单一产物。两种蛤仔的序列长度和 4种碱基的含量见表1、表2。菲律宾蛤仔和杂色蛤仔16S序列长度分别为511 bp和476 bp,COⅠ序列的长度都是 658 bp,结合对软体动物相关片段的分析,这两个片段具有明显的长度保守性。两个片段的GC含量菲律宾蛤仔都低于杂色蛤仔,而 GC含量都低于AT含量,符合线粒体DNA的碱基组成特点。两个片段的GC含量比较接近,但COⅠ基因片段碱基T的含量要明显高于16S rRNA基因片段核苷酸中T碱基的含量。

表1 两种蛤仔16S序列的长度以及碱基组成Tab.1 Size and GC content of the 16S rRNA gene

表2 两种蛤仔COⅠ序列的长度以及碱基组成Tab.2 Size and GC content of the COⅠgene
两种蛤仔 COⅠ基因片段 658个核苷酸编码的20种氨基酸组成的219个氨基酸序列中,都是亮氨酸(Leu)含量最高,分别为 16.44%(RP)和14.16%(RV)。排在其后的氨基酸含量两种蛤仔略有不同,菲律宾蛤仔依次是甘氨酸(Gly),甲硫氨酸(Met),而杂色蛤仔为甲硫氨酸(Met),甘氨酸(Gly)。从总体看,两种蛤仔的氨基酸组成还是具有较高的相似性(表 3)。
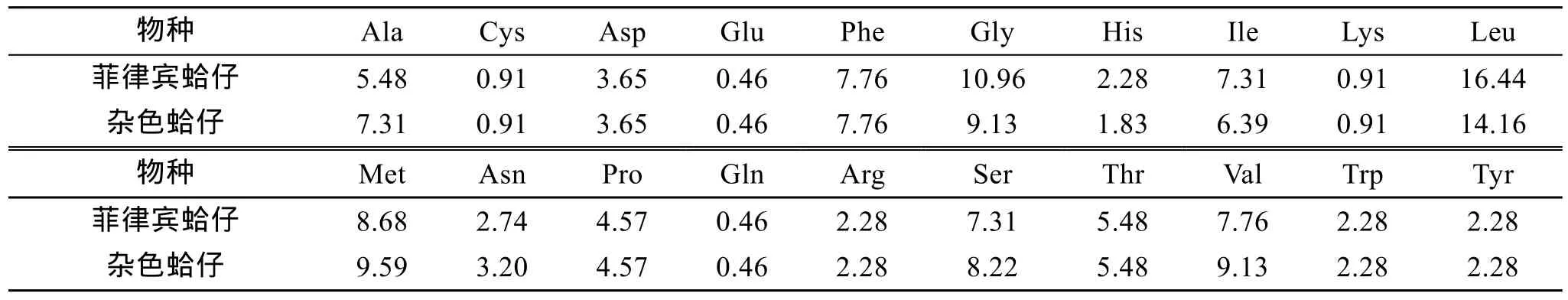
表3 两种蛤仔COⅠ基因的氨基酸组成Tab.3 The amino acid composition(%) of the COⅠgene
两种蛤仔16S序列比对结果见图1。516个位点中共有 117个变异位点(其中,插入缺失 44,多态位点73个),COⅠ序列658个位点中共有160个变异位点,全是多态位点,没有插入缺失(图2)。COⅠ基因编码的219个氨基酸序列,两种蛤仔有29个氨基酸不同(图 3)。

图1 两种蛤仔16S序列的比对图Fig.1 Alignment of 16S rDNA sequences of two clams
以 16S序列计算两种蛤仔与其他几种贝类的碱基替换/颠换率以及距离,以 COⅠ基因编码的氨基酸序列计算两两之间的遗传距离,结果见表4。两两之间的遗传距离最小值 16S的结果在菲律宾蛤仔与杂色蛤仔之间为0.17,而COⅠ氨基酸序列的结果在杂色蛤仔与硬壳蛤之间为 0.14,不过与两种蛤仔之间的遗传距离0.15非常接近。
系统关系分析分别用16S序列和COⅠ编码的蛋白序列进行。根据16S序列构建的系统树见图4,NJ树和MP树完全一致,两种蛤仔都聚为一支,硬壳蛤与白斑乌贼单独聚为一支,这与传统的分类是一致的。而根据COⅠ基因编码的蛋白序列构建的系统树(图5),与16S序列的结果不太一致,NJ树虽将两种蛤仔聚在一起,但这一支的置信度比较低,也就是说,两种蛤仔与硬壳蛤的差异不是很大; 而MP树则将两种蛤仔与硬壳蛤聚为一支,说明这三种蛤之间的差异非常小。

图2 两种蛤仔COⅠ序列的比对图Fig.2 Alignment of COⅠgene sequences of two clams

图3 两种蛤仔COⅠ蛋白序列的比对图Fig.3 Alignment of the amino acid sequences of COⅠgene of two clams
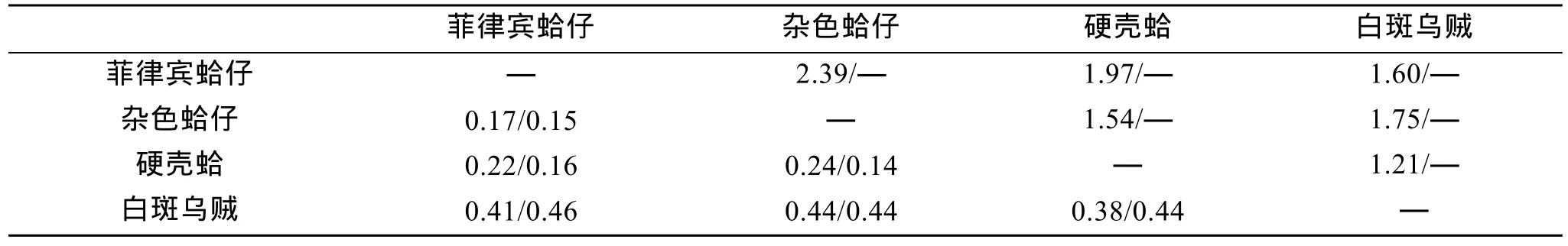
表4 4种贝类两两之间的遗传距离(分别对16S和COⅠ编码的氨基酸序列)Tab.4 The pair-wise genetic distance matrix of COⅠand 16S rRNA gene

图4 根据16S序列构建的系统树Fig.4 Phylogenetic relationships based on 16S rRNA sequences
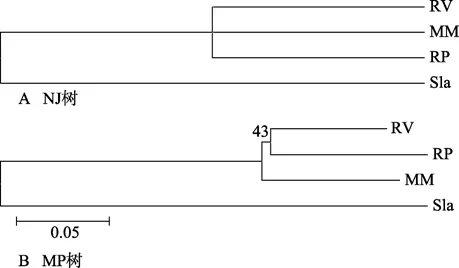
图5 根据COⅠ基因编码的蛋白序列构建的系统树Fig.5 Phylogenetic relationships based on the amino acid sequences of COⅠgenes
3 讨论
帘蛤科贝类在全世界广为分布,包括 500多个种类,许多种类都具有重要经济价值。帘蛤科贝类生活习性比较一致,形态也比较相似,这使得根据形态对系统发生关系的分析非常困难。因此,许多的生化手段被用来研究帘蛤科的分类问题,而分子生物学的研究还刚刚开始。本文得到了两种蛤仔线粒体DNA 16S rRNA和COⅠ两个片段的序列,分析了两种蛤仔这两个片段的序列特征,并从进化的角度分析了它们的变异,为利用分子生物学的方法研究帘蛤科的分类提供了一定的依据。
蛤仔属在帘蛤科中的分类地位一直存在着争议,其属下种的归属问题也曾经比较混乱。因为蛤仔属Ruditapes和缀锦蛤属Tapes、石蛤属Venerupis的亲缘关系很近,形态结构非常相似。1951年,Keen把Ruditapes作为Tapes的亚属。1971年,法国自然史博物馆Fischer-Piette和Metivier对缀锦蛤亚科各个属铰合部主齿板的形态进行了仔细比较研究后,把Ruditapes提升为属,根据他们的分类系统,菲律宾蛤仔与杂色蛤仔都应当是在Ruditapes的属下[12]。
本文对 16S序列的分析结果表明,两种蛤仔的亲缘关系比较接近,而与同为帘蛤科的硬壳蛤有明显的差异。但对COⅠ序列的分析结果则表明,两种蛤仔之间的关系以及它们与硬壳蛤之间的关系差异不明显。不同的DNA片段进行聚类分析会产生不同的结果[13],这说明蛤仔属与其他相近属种的亲缘关系非常接近。潘鹤婷等[14]的聚类结果也表明两种蛤仔不能聚为一支。Canapa等[3]对帘蛤科贝类根据 16S rRNA 基因序列的研究表明,缀锦蛤亚科的6个种类尽管可以聚在一起,但在属的水平上的分类则不能很好的对应,其中的菲律宾蛤仔与错纹蛤仔(Ruditapes decussatus)就不能在属的水平上聚类。一般认为,COⅠ序列比 16S序列具有更大的变异性,因此,在区分近缘种的分类关系上,COⅠ序列比16S序列更灵敏。Boudry等[15]用9种内切酶对太平洋牡蛎(Crassostrea gigas)和葡萄牙牡蛎(Crassostrea angulata)的253个个体的16S序列PCR产物进行酶切,未发现有任何酶切位点变异; 但用4种内切酶对同样个体的COⅠ序列PCR产物进行酶切,却检测到了明显的多态性。
对帘蛤科缀锦蛤亚科各个属及属内包含的种进行细致明确的分类,需要对更多相近种分析更多的DNA序列,包括核DNA和线粒体DNA的序列。在系统演化研究中,采用多组分子生物学数据并结合形态特征、解剖结构等分析,将有助于正确阐述物种间真实的进化关系以及某个物种在系统演化中的确切地位。
[1]Canapa A,Barucca M,Marinelli A,et al.Molecular data from the 16S rRNA gene for the phylogeny of Pectinidae (Mollusca:Bivalvia) [J].J Mol Evol,2000,50:93-97.
[2]Canapa A,Marota I,Rollo F,et al.Phylogenetic analysis of Veneridae (Bivalvia):Comparison of molecular and palaeontological data[J].J Mol Evol,1996,43:517-522.
[3]Canapa A,Schiaparelli S,Marota,et al.Molecular data from the 16S rRNA gene for the phylogeny of Veneridae (Mollusca:Bivalvia) [J].Marine Biology,2003,142:1125-1130.
[4]Barucca M,Olmo E,Schiaparelli S,et al.Molecular phylogeny of the family Pectinidae (Mollusca:Bivalvia)based on mitochondrial 16S and 12S rRNA genes[J].Molecular Phylogenetics and Evolution,2004,31:89-95.
[5]Matsumoto M.Phylogenetic analysis of the subclass Pteriomorphia (Bivalvia) from mtDNA COI sequences[J].Molecular Phylogenetics and Evolution,2003,27:429-440.
[6]Matsumoto M,Hayami I.Phylogenetic analysis of the family Pectinidae (Bivalvia) based on mitochondrial cytochrome C oxidase subunit I[J].J Moll Stud,2000,66:477-488.
[7]Yu Z N,Kong X Y,Zhang L S,et al.Taxonomic status of four Crassostrea oysters from China as inferred from mitochondrial DNA sequences[J].Journal of Shellfish Research,2003,22(1):31-38.
[8]杨建敏,郑小东,王如才,等.3种鲍16S rRNA 基因片段序列的初步研究[J].青岛海洋大学学报,2003,33(1):36-40.
[9]Therriault T W,Docker M F,Orlova M I,et al.Molecular resolution of the family Dreissenidae (Mollusca:Bivalvia) with emphasis on Ponto-Caspian species,including first report ofMytilopsis leucophaeatain the Black Sea basin[J].Molecular Phylogenetics and Evolution,2004,30(3):479-489.
[10]Palumbi S R,Martin A,Romano S,et al.The simple fool’s guide to PCR[J].Honolulu:University of Hawaii Press,1991.
[11]Folmer O,Black M,Hoeh W,et al.DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J].Mol Mar Biol Biotechnol,1994,3:294-299.
[12]庄启谦.中国动物志:软体动物门 双壳纲 帘蛤科[M].北京:科学出版社,2001:41-52.
[13]刘相全,包振民,胡景杰,等.几种帘形目贝类 rDNA ITS序列的比较[J].高技术通讯,2007,17(4):435-440.
[14]潘鹤婷,袁媛,吴琪,等.缀锦蛤亚科(Tapetinae)贝类线粒体 DNA序列的系统学分析[J].海洋与湖沼,2008,39(3):284-290.
[15]Boudry P S,Heurtebise S,Collet B,et al.Differentiation between population of the Portuguese oyster,Crassostrea angulata(Lamark)and the Pacific oyster,Crassostrea gigas(Thunberg),revealed by mtDNA RFLP analysis[J].J Exp Mar Biol Eco,1998,226:279-291.

