SYBR Green I荧光检测痕量宿主DNA
蔺 芳,付瑞敏,皮川真,时 凯,叶 刚,江景玉,陈慧珍
(1.新乡学院生命科学与技术系,河南 新乡 453000;2.河南教育学院人口与生命科学系,郑州 450046;3.邦和药业股份有限公司,郑州 450001)
近年来,灵敏、准确的DNA痕量定量方法在生物学研究及应用领域中发挥着重要的作用。痕量定量方法的灵敏度及准确度均需达到较高的标准。紫外分光光度法是以往使用较多的DNA定量方法,此方法操作简便,成本低,但存在灵敏度低、测样量大等问题。目前,检测外源DNA含量常用的方法是Southern blotting杂交法[1],此法准确性高、特异性强,但存在费时费力的缺点。而荧光定量法是一种灵敏可靠的DNA定量方法,它将荧光染料与DNA结合,利用荧光分光光度计采集结合后增强的荧光信号,通过DNA标准样品计算得出样品DNA的初始浓度。本研究基于SYBR Green I荧光发光原理,来建立一种痕量DNA快速检测方法。
1 材料与方法
1.1 样品
狂犬疫苗(宁波荣安生物药业有限公司)。
1.2 主要试剂和仪器
Protein Precipitation solution和DNA Hydration solution(为Puregene Cell and Tissue Kit中的两种成分)(德国Qiagen公司);DNA检测试剂盒(瑞士Roche公司);SYBR Green I、Pico Green和 λDNA(100 μg·mL-1)(美 国 Invitrogen 公 司);糖 原(Glycogen)(碧云天生物技术研究所);蛋白酶K、RNA酶、三羟甲基氨基甲烷、异丙醇和十二烷基硫酸钠(美国Sigma公司);EDTA(天津市化学试剂三厂);无水乙醇(天津市科密欧化学试剂有限公司)。多功能酶标仪Varioskan Flash(美国Thermo公司);LS-45/55荧光/磷光/发光分光光度计(美国Perkin Elmer公司);3-18K高速台式离心机(德国赛多利斯公司);紫外交联仪(新芝科技股份有限公司);电热恒温水浴箱(上海申贤恒温设备厂);96孔荧光微量滴定板(美国Costar公司)。
1.3 DNA的抽提
500 μL 样品加入 10 μL SDS,10 μL Tris-HCl和 15 μL蛋白酶 K,55℃孵育 3 h;加入 5 μL RNAase 37 ℃ 孵 育 1 h;加 入 167 μL Protein Precipitation solution剧烈震荡20 s,冰浴10 min,16 000 r·min-1离心 5 min;取 620 μL 上清液加入500 μL 异丙醇和 0.5 μL Glycogen于-80℃放置 2 h,16 000 r·min-1离心 30 min;弃上清,加 500 μL 70%乙醇进行洗涤沉淀,16 000 r·min-1离心10 min;弃去上清,待乙醇挥发尽,用100 μL DNA Hydration solution溶解沉淀,65℃孵育1 h,室温轻微震荡过夜,充分溶解DNA[2]。
1.4 SYBR Green I工作浓度的选择
为了选择合适SYBR Green I工作浓度,我们将SYBR Green I分别稀释为 1∶1 000,1∶10 000 和 1∶100 000来绘制DNA的标准曲线(DNA浓度分别为1 600、 800、 400、 200、 100、 50、 25、 12.5、6.25、3.125、1.56、0.78、0.39、0.2和0 ng·mL-1)。
1.5 不同缓冲液体系对SYBR Green I荧光定量的影响
采用不同的缓冲液(即TE缓冲液,PBS缓冲液和PBS+0.5 mol·L-1(NH4)2SO4缓冲液)对标准λDNA稀释成标准系列(400、200、100、50、25、12.5、6.25、3.125、1.56、0.78、0.39、0.2 和 0 ng·mL-1),SYBR Green I采用对应的缓冲液稀释10 000倍。2 mL DNA溶液+2 mL SYBR Green I稀释液混合后避光10 min,倒入比色皿中用LS-45/55荧光/磷光/发光分光光度计在激发光为485 nm,发射光为518 nm下测定。每个梯度测两个值。
1.6 SYBR Green I荧光定量
1.6.1 样品稀释
用TE将标准λDNA稀释成标准系列(400、200、 100、 50、 25、 12.5、 6.25、 3.125、 1.56、0.78、0.39、0.2 和 0 ng·mL-1)。荧光染料 SYBR Green I荧光染料用TE缓冲液稀释10 000倍。
1.6.2 荧光数据采集
96孔荧光微量滴定板内分别加100 μL标准和样品稀释液,每孔再加100 μL用TE稀释10 000倍的SYBR Green I混合,避光静止15 min。用多功能酶标仪Varioskan Flash在激发光为485 nm,发射光为518 nm下测值。每个标准和样品做2孔[3-4]。
1.6.3 数据处理
计算每个标准和样品的OD值,即OD=(OD测定1+OD测定2)/2,以标准DNA各浓度的吸光值为横坐标,以相应的浓度为纵坐标作图绘制标准曲线,然后将样品的吸光值在标准曲线上找到相应的y值,则原样品 DNA的浓度(ng·mL-1)=y×稀释倍数/5。
1.7 Southern杂交
按窦志勇等采用的杂交方法处理硝酸纤维素膜[5],并用点样器点样,待检及已知浓度的DNA对照样品均先用沸水煮10 min变性,迅速冷却,20 μL点样。室温晾干膜,紫外线照射膜5 min,然后进行预杂交→杂交→洗膜→封闭→加抗体→洗膜→显色→终止反应,记录结果。由于采用的500 μL体系抽提样品 DNA,用 100 μL DNA Hydration solution去溶解DNA,在点膜前根据样品DNA进行适当的稀释,点膜用20 μL,故原样品DNA的浓度(ng·mL-1)=膜上直读样品 DNA 浓度×10×稀释倍数。
2 结果与分析
2.1 不同缓冲液体系对SYBR Green I荧光定量的影响
采用不同缓冲体系对DNA和SYBR Green I进行稀释并测值,绘制标准曲线(见图1),记录读数(见表1)。
从图1可以看出,三种缓冲体系的线性关系比较好,相关系数的平方均达到0.99以上。其中,含 0.5 mol·L-1(NH4)2SO4缓冲体系的线性最好,相关系数的平方达到0.9997。但从表1可以看出含0.5 mol·L-1(NH4)2SO4缓冲体系只能检测DNA浓度高于1.56 ng·mL-1的,它的读数相对于其他缓冲体系值远远偏小;TE缓冲体系能检测到50 pg·mL-1,对于检测痕量DNA很有帮助。
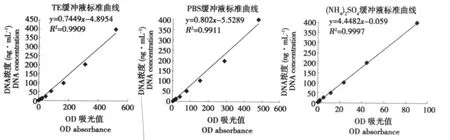
图1 不同缓冲体系下DNA标准曲线Fig.1 Standard curve of DNA in different buffer system

表1 不同缓冲体系下DNA标准系列吸光读数Table 1 Absorbance of DNA standard series in different buffer system
2.2 不同稀释度下SYBR Green I DNA标准曲线
将 SYBR Green I分别稀释为 1∶1 000,1∶10 000和1∶100 000来绘制DNA的标准曲线。从图2中可以看出荧光染料在1∶1 000稀释度时DNA浓度在3.125~1 600 ng·mL-1之间,线性关系较好,相关系数的平方达到0.9991;在1∶10 000稀释度时DNA浓度在1.56~1 600 ng·mL-1之间,线性关系较好,相关系数的平方达到0.9999;而1∶100 000稀释度时DNA浓度在0.78~400 ng·mL-1之间,线性关系较好,相关系数的平方达到0.9991。由此可知,荧光染料在1∶10 000稀释度下其线性范围比1∶1 000稀释度广,这样可以节约成本,效果更好。荧光染料在1∶10 000稀释度时虽然灵敏度高,但线性范围窄,所以我们选择荧光染料1∶10 000的稀释度。

图2 不同稀释度下SYBR Green I DNA标准曲线Fig.2 Standard curve of DNA in different SYBR Green I dilution
2.3 荧光法和Southern blotting杂交法的比较
为了进一步验证建立的痕量DNA测定方法,将4个样品经改进的方法抽提后同时经荧光法和Southern blotting杂交法检测DNA的浓度。在Southern blotting杂交法时我们根据样品DNA的浓度进行稀释后点膜(样品3、4在前点膜稀释10倍)。Southern blotting杂交的结果见图3。
表2为荧光法和Southern blotting法通过换算后各个样品抽提前样品DNA的浓度。由于Southern blotting法是半定量的,不能具体读出DNA含量。从表2可以看出Southern blotting结果和荧光检测结果基本可以相互印证。
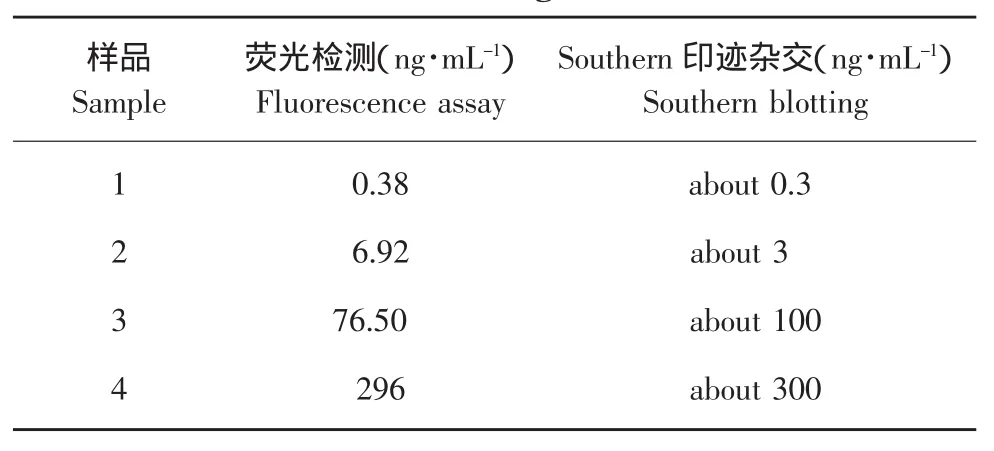
表2 荧光法和Southern blotting法的对比Table 2 Comparison of fluorescence assay and Southern blotting
3 讨论
目前,用于双链DNA定量的荧光染料主要有溴化乙啶(EB)、Hoechst33258、Picogreen及SYBR Green I。EB是一种常用的凝胶染色荧光染料,由于其自身有较高的本底荧光,与RNA及单链DNA也有较强的结合能力,因此不适用于特异性双链DNA准确定量研究[6]。Hoechst 33258是一选择性与dsDNA结合的荧光染料,可检测到0.01 ng·μL-1的dsDNA样品[7],并受蛋白影响较小[8];但该染料对A-T富含区有较强的选择性[9],并且受DNA长度的限制(<200 bp)[7]。Picogreen是一种嵌入型荧光染料,可以检测到pg级的dsDNA[10],由于受ssDNA及蛋白影响较小,可以用于dsDNA的定量[11],但为了增加方法的灵敏度,定量时需要加入较高浓度的染料[12],增加了实验成本。SYBR Green I与Picogreen具有相似的化学性质[13],但比Picogreen具有更好的热稳定性,因此可作为dsDNA定量检测。
4 结论
Southern blotting杂交法只是半定量的方法,而荧光法可以根据DNA的标准曲线准确读出DNA的浓度。荧光法和Southern blotting杂交法相比,更快速,精度更高。荧光法检测只需30 min,而Southern blotting杂交法需要2 d。同时,利用SYBR Green I进行dsDNA定量时,所需染料浓度较低,受pH、定量体积及时间影响较小,因此是一种灵敏、快速、经济的dsDNA定量方法。
[1]侯静,马凤鸣,陈胜勇,等.甜菜基因组DNA的提取及Southern杂交分析[J].东北农业大学学报,2008,39(12):14-18.
[2]邵强,皮川真,于翔,等.痕量DNA荧光检测方法建立[J].中国公共卫生,2010,26(3):340-341.
[3]Leggate J,Allain R,Isaac L,et al.Microplate fluorescence assay for the quantification of double stranded DNA using SYBR Green I dye[J].Biotechnol Lett,2006,28:1587-1594.
[4]刘歆,徐根明,郭江峰,等.基于SYBR Green I的双链DNA定量方法[J].中国生物工程杂志,2008,28(1):55-60.
[5]窦志勇,鲁宏,钱浩.应用地高辛标记探针检测以Vero传代细胞制备人用狂犬疫苗中残余细胞DNA含量[J].中国公共卫生学报,1997,16(4):251.
[6]Waring M J.Complex formation between ethidium bromide and nucleic acids[J].Journal of Molecular Biology,1965,13(1):269-282.
[7]Teare J M,Islam R,Flanagan R,et al.Measurement of nucleic acid concentrations using the DyNA Quant and the GeneQuant[J].Bio Techniques,1997,22(6):1170-1174.
[8]Moe D,Garbarsch C,Kirkeby S.The protein effect on determination of DNA with Hoechst 33258[J].Journal of Biochemical and Biophysical Method,1994,28(4):263-276.
[9]Loontiens F G,Regenfuss P,Zechel A.Binding characteristics of Hoechst 33258 with calf thymus DNA,poly[d(A-T)],and d(CCGGAATTCCGG):Multiple stoichiometries and determination of tight binding with a wide spectrum of site affinities[J].Biochemistry,1990,29(38):9029-9039.
[10]Singer V L,Jones L J,Yue S T,et al.Characterization of PicoGreen reagent and development of a fluorescence-based solution assay for double-strandedDNAquantitation[J].AnalyticalBiochemistry,1997,249(2):228-238.
[11]陈旭,齐凤坤,康立功,等.实时荧光定量PCR技术研究进展及其应用[J].东北农业大学学报,2010,41(8):148-155.
[12]Vitzthum F,Geiger G,Bisswanger H.A quantitative fluorescencebased microplate assay for the determination of double-stranded DNAusingSYBRGreenIandastandardultraviolettransilluminator gel imaging system[J].Analytical Biochemistry,1999,276(1):59-64.
[13]Zipper H,Brunner H,Bernhagen J.Investigations on DNA intercalation and surface binding by SYBR Green I,its structure determination and methodological implications[J].Nucleic Acids Research,2004,32(12):e103.

