不同国家菜豆核DNA ITS序列分析
欧立军 ,张人文 ,谭智文 ,蒋向辉 ,佘朝文
(1.民族药用植物资源研究与利用湖南省重点实验室,湖南怀化,41800;
2.湘西药用植物与民族植物学湖南省高校重点实验室;3.怀化学院生命科学系)
菜豆(Phaseolus vulgarisL.)又名四季豆、梅豆、芸豆,因其色泽嫩绿、肉荚肥厚、味道鲜美、极富营养价值而深受广大消费者的喜爱,是一种鲜嫩可口,色、香、味俱全,营养丰富的优质蔬菜。19世纪末期之前,人们认为普通菜豆起源于亚洲[1]。根据形态学与生物化学的证据显示,秘鲁南部、玻利维亚和阿根廷地区是野生菜豆的一个起源中心,即安第斯中心;墨西哥、危地马拉、巴拿马、洪都拉斯、尼瓜拉加、哥斯达黎加和哥伦比亚等地区是另一个起源中心,即中美洲中心[2]。野生菜豆在这2个中心分别被驯化成栽培植物,然后才逐渐被引入世界各地,包括南美、北美、非洲、欧洲和亚洲的一些地区[3]。普通菜豆于16世纪初传到欧洲,16世纪和17世纪在欧洲迅速传播,并且在西班牙和葡萄牙北部地区形成了大粒白芸豆的次级多样性中心[4~5]。随后,普通菜豆由这一地区传播到欧洲其他地区,再由欧洲传入中东、西亚以及世界上其他国家和地区[6]。普通菜豆传入非洲的具体时间不太清楚,但是2个起源中心的材料在非洲都存在并且主要以安第斯中心的材料为主[4]。郑卓杰等[7]认为,中国栽培的菜豆是15世纪直接从美洲引入的,但尚缺乏直接有力的证据。
高等植物中的rRNA基因是高度重复的串联序列,18S、5.8S和26S rDNA连结在一起作为一个转录单位,拷贝数可达500~40 000,ITS序列包括被5.8S隔开的ITS1和ITS2。编码18S、5.8S和26S的序列为高度保守区,ITS序列为进化速度较快的中度保守序列,且各重复单元间具有同步进化的特点。因此,ITS作为基因标记很快成为在序列水平上探讨系统发育和进化研究的有效手段[8~11]。
本文基于ITS序列对来自不同国家的菜豆进行聚类分析,从分子水平探讨菜豆种质的亲缘关系,以期为菜豆核心种质筛选和优异种质培养提供理论依据,同时为确定菜豆的起源及次级起源中心提供参考。
1 材料与方法
1.1 试验材料
16份菜豆种质来自亚洲、欧洲、美洲和非洲,每份材料取3个样本(表1),由美国农业部种子库和民族药用植物资源研究与利用湖南省重点实验室提供。
1.2 试验方法
采取CTAB法提取DNA。ITS引物采用参考White 等[12],5'-TCCTCCGCTTATTGATATGC-3',5'-GGAAGGTAAAAGTCAAGG-3',反应体系包括10×PCR buffer 5 μL,10 mmol/L dNTP 1 μL,50 mmol/L 引物各 1 μL,DNA 40 ng,Taq DNApolymerase 0.4 μL,补充双蒸水至50 μL。反应程序为:95℃预变性4 min;94℃变性 45 s,56℃退火 45 s,72℃延伸 45 s,35 个循环;72℃后延伸10 min。PCR产物纯化用购自上海生工的DNA纯化试剂盒。纯化产物鉴定后送往上海生工双向测序。所得序列采用 DNAMAN和MEGA.4软件进行分析并提交Genebank得到登录号。
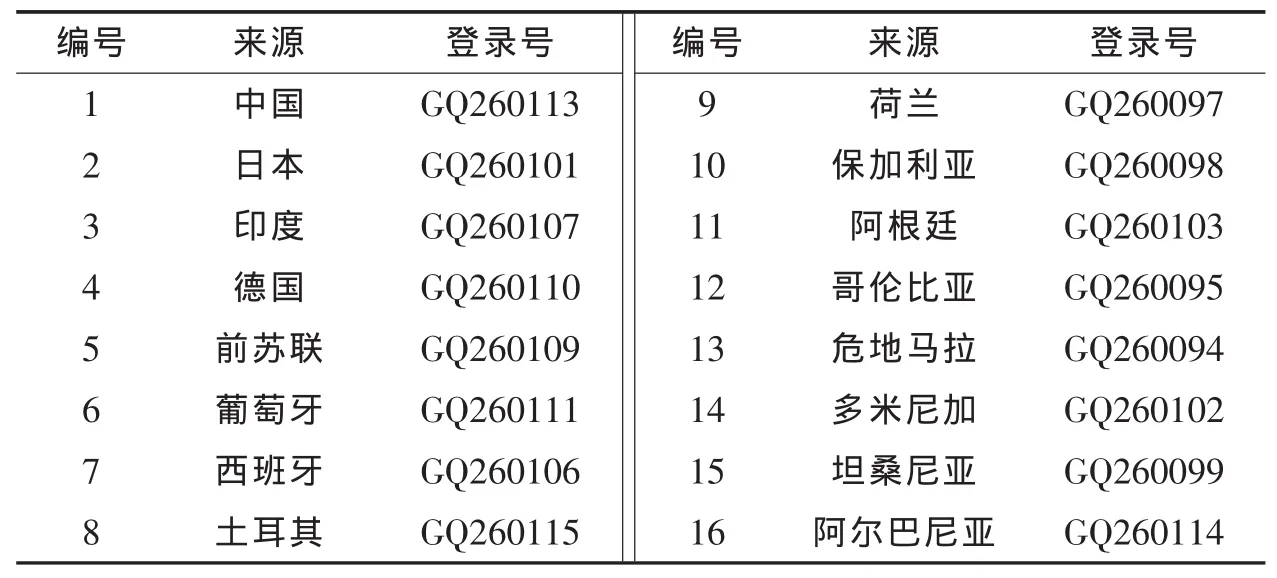
表1 材料及来源
2 结果与分析
2.1 ITS序列分析
PCR扩增结果显示,ITS片段大小在700 bp左右(图1a),纯化后条带清晰,达到测序要求(图1b)。测序分析发现,ITS序列长度很保守,为645 bp或635 bp,片段大小相差10 bp,变异为1.5%;GC含量变化较大,在52.8%~57.1%波动;ITS1片段为 250 bp或259 bp,GC含量较高,变化范围不大,在58.7%~59.9%波动;ITS2片段长度为222 bp或223 bp,相差仅1个bp,GC含量在57.9%~60.3%波动(表2)。
2.2 遗传相似度分析
16份种质的ITS1相似度为88.13%,ITS2相似度为 85.14%,ITS1的相似度高于ITS2。除种质14和16外,其他14份种质的相似度很高,其中ITS1的相似度达到99.35%,ITS2则为93.56%,以上结果说明ITS2片段的遗传多态性高于ITS1片段。
2.3 同源树分析
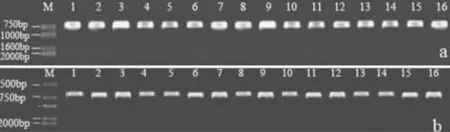
图1 菜豆ITS PCR扩增(a)和纯化(b)
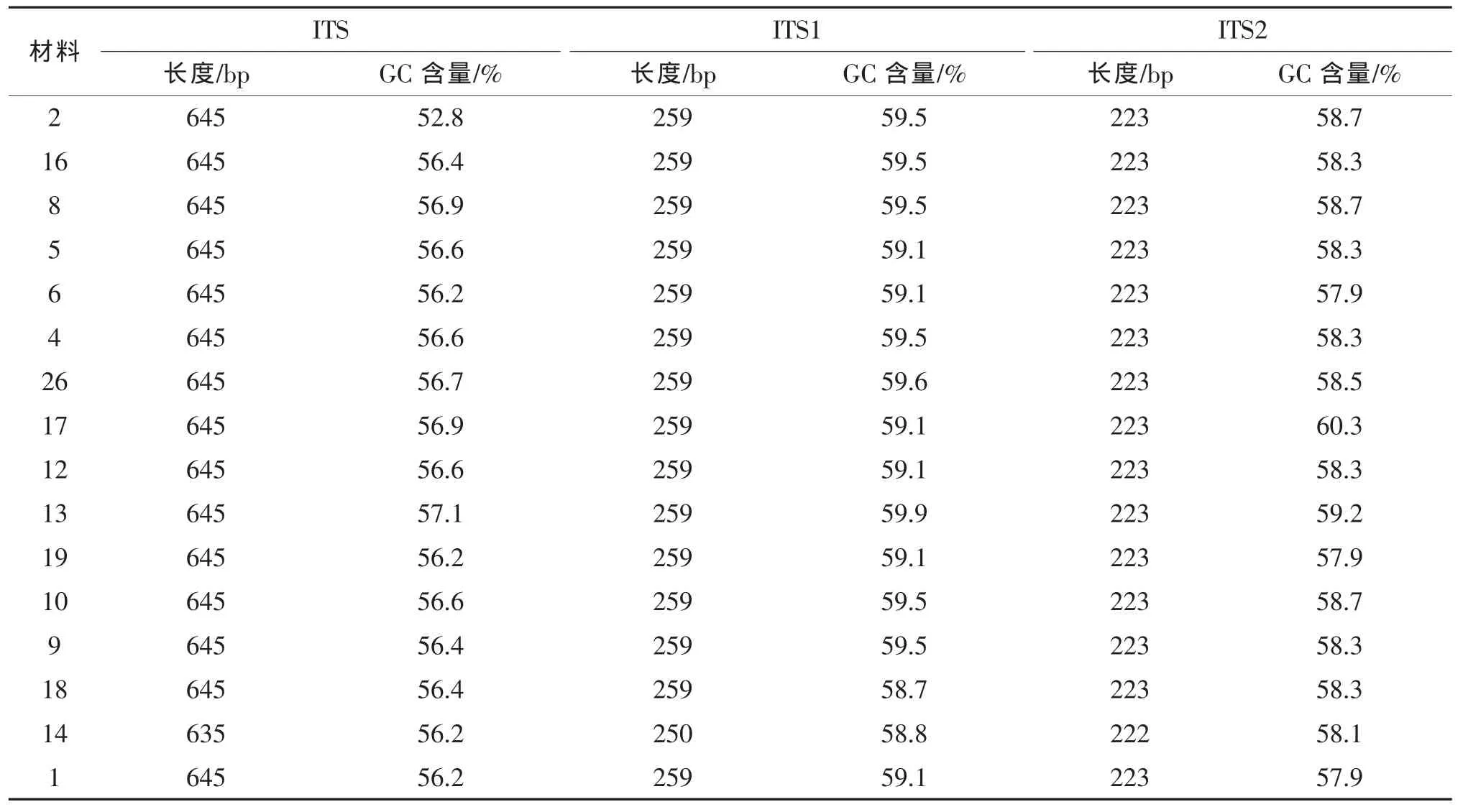
表2 菜豆ITS序列长度和GC含量
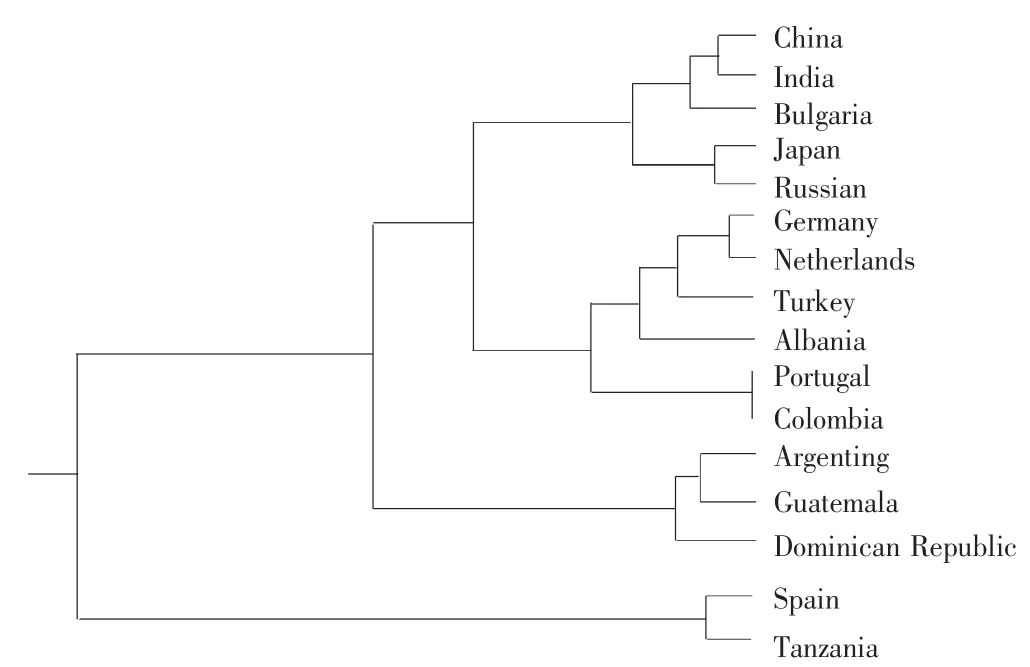
图2 基于ITS序列构建的同源树
基于ITS序列构建同源树,不同菜豆种质被分为2大支,来自西班牙的菜豆与来自坦桑尼亚的菜豆为1大支,其他种质为另1大支。第2大支中,来自中国的菜豆优先与来自印度的菜豆聚类,然后与来自保加利亚的菜豆聚类;来自日本的菜豆与来自俄罗斯的菜豆优先聚类,其他来自欧洲的菜豆优先聚类后与来自亚洲的菜豆聚类,然后与来自美洲的菜豆聚类(图2)。
3 小结与讨论
来自葡萄牙和哥伦比亚菜豆种质ITS序列完全相同,这与Santalla等[5]提出的普通菜豆于16世纪初由美洲传到欧洲,然后16世纪和17世纪在欧洲迅速传播,并且与在西班牙和葡萄牙北部地区形成了大粒白芸豆的次级多样性中心的说法一致。来自坦桑尼亚和西班牙的菜豆的ITS序列特点相近,序列长度均为635 bp(其他种质都为645 bp),在进化树上聚为一类,表明坦桑尼亚的菜豆可能直接来自于欧洲的西班牙。来自非洲阿尔巴尼亚的菜豆则与来自欧洲的菜豆优先聚类,说明非洲的菜豆可能直接来自欧洲,这与Schoonhoven等[13]提出非洲是菜豆第2遗传中心的说法不相一致,因此非洲是否为菜豆的次级起源中心有待深入研究。
来自中国的菜豆优先与来自欧洲的菜豆聚类后,再与来自美洲的菜豆聚类,表明中国的菜豆与欧洲的菜豆之间的亲缘关系更为密切,与郑卓杰[7]通过形态学比较提出中国的菜豆是15世纪直接从美洲引进的说法不一致。考证历史发现,1497年,葡萄牙航海家达·伽马经过加那利群岛,绕好望角,经莫桑比克等地,于1498年到达印度西南部卡利卡特,1869年苏伊士运河通航前,欧洲对印度洋沿岸各国和中国的贸易,主要通过这条航路,因此中国的菜豆可能是通过这条航路传播过来的。通过基因组得到的结果比形态上得到的结果更可靠,虽然本研究仅仅分析了1个DNA片断,但这个片段是常用来进行物种内遗传进化的分子标记,因此本研究认为中国的菜豆是否直接从美洲引进值得商榷,并且更有可能是来自于欧洲。
[1]Linnaeus C.Species plantarum[M].London:England,Ray Society,1753.
[2]Gepts P,Bliss F A.Dissemination pathways of common bean(Phaseolus vulgrisFabaceae)deduced from phaseolin electrophoretic variability.II.Europe and Africa[J].Economic Botany,1988,42(1):86-104.
[3]Broughton W J,Hernández G,Blair M,et al.Vanderleyden Beans(Phaseolusspp.)-model food legumes[J].Plant and Soil,2003,252:55-128.
[4]Singh S P.Patterns of variation in cultivated common bean(Phaseolus vulgarisFabaceae)[J].Economic Botany,1989,43:39-57.
[5]Santalla M,Rodino A,De Ron A.Allozyme evidence supporting southwestern Europe as a secondary center of genetic diversity for common bean[J].Theoretical and Applied Genetics,2002,104:934-944.
[6]Smartt J,Simmonds N W.Evolution of Crop Plants[M].UK:Longman Scientific and Technical,1995:287-296.
[7]郑卓杰.中国食用豆类学[M].北京:中国农业出版社,1997.
[8]Mao S,Kenji K,Eiji M,et al.Identification of medicinal A-tractylodes based on ITS sequences of nrDNA [J].Biol Pharm Bull,2006,29(2):315-320.
[9]Christopher H J,Kenneth J S,Harvey E B J.Evolutionary relationships,interisland biogeography,and molecular evolution in the Hawaiian violets(Viola:Violaceae)[J].American Journal of Botany,2009,96:2 087-2 099.
[10]Vijaykumar A,Saini A,Jawali N.Phylogenetic analysis of subgenus vigna species using nuclear ribosomal RNA ITS:evidence of hybridization among vigna unguiculata subspecies[J].Journal of Heredity,2010,101:177-188.
[11]Yan J,Deng J,Zhou C J,et al.Phenotypic and molecular characterization ofMadurella pseudomycetomatissp.nov.,a novel opportunistic fungus possibly causing black-grain mycetoma[J].Journal of Clinical Microbiology,2010,48:251-257.
[12]White T J,Bruns T,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]//In:Innes M,Gelfand D,Sninsky J,et al.eds.PCR protocols:a guide to methods and applications,1990,315-322.
[13]Schoonhoven A V,Voyest O.Common beans:Research for Crop Improvement[M].CABI,1991.

