西菲律宾海比科尔陆架深海沉积物古菌多样性研究
格根塔娜, 萨仁高娃, 于心科, 李铁刚, 周伟光
(1.内蒙古农业大学 动物科学与医学学院, 内蒙古 呼和浩特 010018; 2.中国科学院 海洋研究所, 海洋地质与环境重点实验室, 山东 青岛 266071)
西菲律宾海比科尔陆架深海沉积物古菌多样性研究
格根塔娜1, 萨仁高娃2, 于心科2, 李铁刚2, 周伟光1
(1.内蒙古农业大学 动物科学与医学学院, 内蒙古 呼和浩特 010018; 2.中国科学院 海洋研究所, 海洋地质与环境重点实验室, 山东 青岛 266071)
通过构建16S rRNA基因文库, 对西太平洋西菲律宾海东板比科尔陆架5个不同层位沉积物样品中的古菌的多样性进行了研究, 并获得了465个有效克隆63个OTUs (Operational Taxonomic Units)。通过16S rRNA序列与GenBank已知序列的同源性比较及构建系统进化树的结果显示, 古菌序列分别来自泉古菌(Crenarchaeota)和广古菌(Euryarchaeota), 以Marine Benthic Group B(MBGB)、Marine Group I(MGI)、Miscellaneous Crenarchaeotic Group(MCG)和Marine Benthic Group D(MBGD)为主。少量序列为Marine Hydrothermal Vent Group(MHVG)。研究结果表明, 该垂直分布的5个不同层位的沉积物样品仅有 5个古菌类群, 古菌群落多样性并不高, 且 5个层位中的优势古菌类群稍有差异。研究结果揭示了比科尔陆架深海古菌垂向分布特征, 为今后大范围自然海区古菌生态学研究提供科学参考。
西太平洋; 沉积物; 古菌; 多样性
海洋约占地球表面积的70%左右, 而深海沉积物覆盖了地球表层的50%以上, 由于巨大的广度和深度, 海底沉积物和上部洋壳是地球上最大的生态系统之一。海洋生态环境独特, 具有高盐、高压、低温、营养贫乏和光照强度变化大等特点[1], 因此在这种复杂的独特的海洋环境下生存的大量微生物为适应环境, 在物种类型、代谢类型、功能基因组成和生态功能上具有多样性。
深海微生物主要包括古菌和细菌这两类原核生物,占了全球生物圈原核生物总数的 70%[2], 同时占有1/10~1/3的地球总生物量[3]。其中, 古菌(Archaea)又分为泉古菌(Crenarchaeota)和广古菌(Euryarchaeota)两个界[4], 前者包括极端嗜热菌、产甲烷菌、嗜盐菌; 后者有极端嗜热型古菌、硫酸盐还原型古菌等。古菌在地球上分布广泛, 不仅仅只存在于一些高温、高盐等极端环境, 还在包括湖泊、开放大洋、热泉以及沉积物, 温和的海洋水域中, 极地海洋水域中[5-8], 发现古菌都广泛地存在。但 Britschgi[9]在 1991年,Delong[5-6]在1992年、1994年的研究均表明, 古菌的绝大多数种难以纯化培养, 即使能培养的也往往是非典型的种属。传统的微生物学方法由于需要纯化培养, 就无法对古菌进行定性分析。目前所报道分离成功的古菌几乎都是在极端环境条件下生存的古菌[10-13]。近几年来, 随着分子生物学的发展, 分子生物学方法在分类上的应用日益广泛, 特别是 PCR扩增和16S rRNA基因序列同源性比较的应用。利用分子生物学方法研究深海微生物, 发现在深海环境中微生物的种类数目要比传统的分离培养方法分离到的种类要广泛得多。通过 16S rRNA 基因核酸序列分析和系统进化分析研究微生物的多样性和分布情况能够比较全面地反映环境中微生物群落的结构,已广泛应用于土壤、海洋、湖泊等多种生态系统中,已取得良好成效[14-15]。因此通过比较古菌16S rRNA序列的最大相似性, 揭示它们之间及与真核生物之间的进化关系, 为古菌的形态、生理生化特性、生态作用的研究奠定基础。
本文利用 16S rRNA技术对西太平洋西菲律宾海比科尔陆架深海沉积物中古菌多样性作了系统发育分析, 研究了深海沉积物中古菌的种群结构及其相互间的进化关系, 此项工作的完成为开展深海生物圈在物质循环中作用的研究提供了前提。
1 材料与方法
1.1 沉积物样品的采集
比科尔陆架(Bicol shelf)样品来自西太平洋西菲律宾海东板 MD06-3052 岩心(14°48.60′N, 123°29.40′E),水深 703 m。该岩心是由法国极地研究所 Marion Dufresne号科学考察船于2006年6月12日借助中法合作航次 Marco Polo 2, 利用国际上独一无二的calypso重力活塞取心器采集。工作人员在船上立即对沉积物进行分样并冷冻于-20℃, 回到实验室后保存于-80℃。
1.2 主要试剂
土壤 DNA快速提取试剂盒(FastDNA Spin Kit for Soil, QBIOgene, USA)、华舜胶回收试剂盒、pMD-19T Vector (TaKaRa)、Escherichia colibacillusTOP10感受态细胞、凝胶成像仪(BIO-RAD)、限制性内切酶HhaⅠ和MspⅠ(TaKaRa)、核酸提取仪Fast PrepTM FP120(Thermo Electron Corp, USA)、DYY-6C型电泳仪、TP-600型PCR仪 (TaKaRa)。
1.3 沉积物样品中总DNA的提取
从西太平洋菲律宾海东板比科尔陆架MD06-3052站位的沉积物柱状样选取 5个不同层位样品。样品H, V, Z, J, R分别对应于0.05, 1, 3, 5, 6 m深度的沉积物样品。沉积物样品基因组DNA的提取均采用土壤DNA快速提取试剂盒及Fast PrepTM FP120核酸提取仪进行提取。各沉积物样品均取0.4 g泥样按照试剂盒的操作步骤提取 DNA。提取的基因组 DNA用1%琼脂糖凝胶电泳进行检测。将得到的 DNA溶液分装到多个离心管中, 短期使用的于4 ℃保存, 长期储存的于-80 ℃放置。
1.4 PCR 扩增
用提取的DNA进行PCR扩增, 古菌16S rRNA基因的扩增引物为 Arch 21F (TTCCGGTTGATCCYGCCGGA)和Arch 958R (YCCGCGTTGAM TCCAATT)。PCR 反应体系(终体积 12.5 µL)为 10×Taq buffer with KCl 1.25µL, 1.5µmol/L MgCl21µL, 100µmol/L dNTP 1µL, 每个引物浓度为 1 µmol/L 1µL,BSA 1µL, Taq酶0.2 µL以及梯度浓度的模板。PCR条件为94℃ 变性1 min, 55℃复性1 min, 72℃ 延伸1 min, 共35个循环, 最后72℃延伸10 min。扩增产物通过1%琼脂糖凝胶电泳进行检测。选取最适条带的模板浓度进行平行样扩增。
1.5 基因文库的构建
采用上海华舜生物技术公司的胶回收试剂盒回收纯化沉积物样品中扩增出来的所有古菌16S rRNA,根据TaKaRa公司生产的pMD19-T载体手册上的方案将回收到的PCR产物连接到pMD19-T载体上, 经转化后涂在LB板上培养并初筛出阳性克隆。用水煮法快速提取细胞中的基因组DNA, 通过菌落PCR对初筛的克隆进行插入片段大小的验证, 所用引物为pMD19-T 载体引物 RV-M (5′-GAGCGGATAACAATTTCACACAGG-3′)和 M13-D (5′-AGGGTTTTCCCAGTCACGACG-3′)。PCR 条件为 94 ℃变性 45 s,58 ℃复性1 min, 72℃延伸1 min 20 s, 共35个循环,最后72 ℃延伸10 min。
1.6 RFLP分析
将上述片断大小正确的PCR产物分别用Msp I和Hha I (MBI, USA)两种限制性内切酶进行酶切。酶切反应于37℃过夜。酶切产物用3%的琼脂糖凝胶(Akeaibao, Spain)在0.5×TBE缓冲液中进行电泳。分析产物酶切后的图谱, 计算每种酶切RFLP带型的出现频率, 将不同RFLP带型的克隆子进行测序。
1.7 古菌16S rRNA序列分析
将测序成功的所有基因序列分别与 NCBI Gen-Bank数据库中nucleotide blast程序(http://www.ncbi.nlm.nih.gov/blast/Blast.cgi)进行比对, 搜索与数据库中最相似的核苷酸序列。根据搜索结果并参照有关文献选取参照序列。将古菌16S rRNA序列中, 序列同源性不小于 97%时, 定义为一种 OTUs(Operation Taxonomic Units), 并从NCBI GenBank 数据库中比对的最相似序列用 Clustal X(version 1.8)软件对齐,截齐后的序列通过邻接法和最大简约法, 运用DNASTAR和MEGA4程序构建系统进化树, 并递交GenBank, 获取序列登录号。本研究所得到的GenBank 序列号为:GQ994250~GQ994363。
2 结果
2.1 古菌16S rRNA多样性指数及稀释曲线分析
本文从MD06-3052站位的5个文库得到具有正确插入片段的克隆共465个(去掉杂合子后), 每个层位的样品91~ 96个克隆不等(表1)。对具有不同谱型的114个克隆进行测序。测序成功的基因序列与NCBI GenBank数据库中nucleotide blast程序进行比对, 搜索与数据库中最相似的核苷酸序列。根据这些古菌的16S rRNA序列之间的相似性比较, 通过Dotur软件在氨基酸 97%相似度水平上归类后共得到 63个OTUs。
从表1可看出, 所构建的5个古菌16S rRNA克隆文库包含了78.01%~95.60%的古菌多样性。其中深层(样品 J)的覆盖率最高, 达 95.60%; 上层(样品 V)的覆盖率最低, 为78.01%。由所获得的不同16S rRNA基因型数目可知, 在整个柱状样中, MD3052V层深海沉积物中微生物的多样性相对较高, 深层中相对较低。小尺度上呈现出随深度增加而多样性减少的趋势。
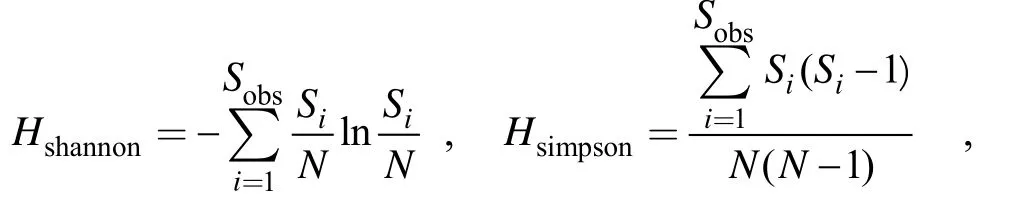
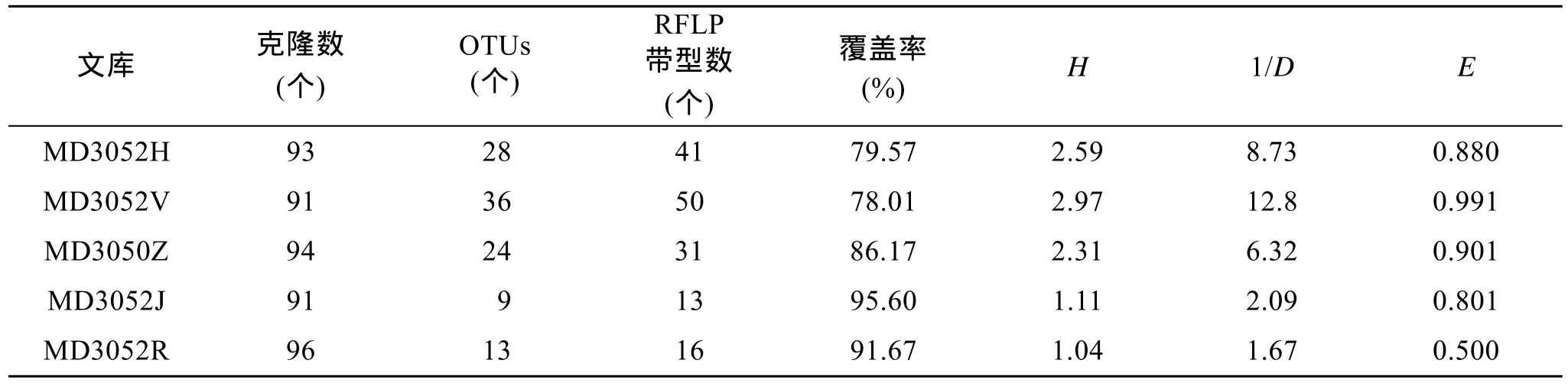
表1 深海沉积物古菌16S rRNA克隆文库分析Tab. 1 Analyses of 16S rRNA clone libraries of the archaea in deep sea sediments
另外从古菌 16S rRNA基因克隆文库的稀释曲线(图1)可以看出, MD3052V(1 m)的斜率较高,MD3052J的斜率较低, 趋于平缓, 这一结果进一步证明了该环境中深层的优势菌突出, 接近饱和这一结果。
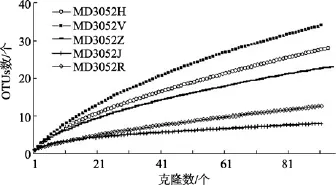
图1 古菌16S rRNA基因文库稀释曲线Fig. 1 Rarefaction curves of 16S rRNA clone libraries of the archaea
2.2 古菌16S rRNA系统进化分析
据表2显示, 古菌 16S rRNA 基因序列主要来自泉古菌, 占92.9%。泉古菌又分为4个类群, 主要为Marine Benthic Group B (MBGB) (58.7%), Miscellaneous Crenarchaeothic Group (MCG) (20%), Marine Group I (MGI) (13.5%)和仅少量的Marine Hyolrothermal Vent Group (MHVG) (0.65%); 且MBGB随着沉积物的深入而增多的趋势。广古菌则以Marine Benthic Group D (MBGD) (7.1%)为主。
根据已测定的古菌 16S rRNA序列之间的同源性比较构建系统发育树(图2), 反映古菌彼此间的系统进化关系。系统进化分析表明该沉积物古菌群落的多样性较低。在 5个不同层位样品所构建的文库中共有 12个 OTUs(代表273个克隆)属于 MBGB。无论是从数量还是多样性上看, MBGB均广泛分布于 MD3052站位深层沉积物的各个层位。MCG在V(1 m)层中占主要优势, MGI则在表层中占优势。
MBGB也称为DSAG (Deep-Sea Archaeal Group),最初发现于深海沉积物和热液口[17-18]。事实上该类群广泛分布于多种深海环境, 如大西洋深海沉积物、海底泥火山、黑海碳酸盐壳、贫有机物的深海海盆以及大陆边缘有机物丰富含甲烷或甲烷水合物的沉积物, 是秘鲁边缘 1230站位甲烷-硫酸盐转换带中古菌的优势类群[3,18-22]。从本文构建的系统进化树中可以看出, 同属于MBGB的古菌同源性序列主要来源于 4种环境, 即太平洋深海、鄂霍次克海、加利福尼亚州圣巴巴拉盆地大陆边缘沉积物和冷泉环境。
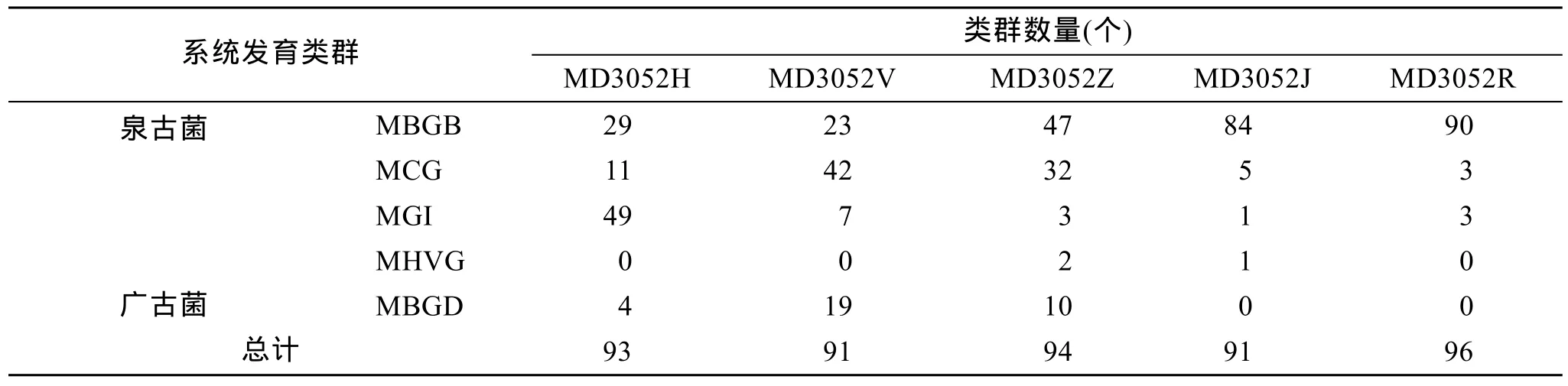
表2 沉积物古菌16S rRNA基因文库克隆系统发育归属关系Tab. 2 Analyses of the archaeal 16S rRNA gene clone libraries from sediments of the West Pacific Ocean
MCG广泛存在于多种陆地和海洋环境, 在海水和沉积物中均有大量分布[17,20,23-25]。本文中其同源性序列来源于太平洋深海、加利福尼亚州圣巴巴拉盆地大陆边缘。
MGI发现于海水中[5], 是海水中古菌的主要类群[4,6-7]。本文中其同源性序列主要来源于韩国的海绵共生的古菌, Nankai海槽、地中海、鄂霍次克海、冷泉环境中的古菌。
MBGD最初发现于大西洋陆坡和新英格兰离岸深海平原[18]。本文中其同源性序列来源环境同MBGB相同, 主要来自太平洋深海、鄂霍次克海、加利福尼亚州圣巴巴拉盆地大陆边缘沉积物和冷泉环境。
2.3 聚类分析
本文利用 PRIMER 6.0软件创建 Principal Component Analysis(PCA)聚类图(图3)。从图3可以看出MD3052R和 MD3052J聚在一起, 而它们属深层位沉积物, 古菌多样性相对来说不高, 优势菌群较为突出。
3 讨论
本文以PCR技术为基础的构建基因文库的方法对西太平洋菲律宾东板比科尔陆架5个不同层位的沉积物古菌多样性进行了研究。对沉积物的总DNA直接进行分析, 避免了人工培养所造成的偏差, 能够较好地评估样品中的微生物多样性。基因文库分析方法研究生物多样性能够获得大量目的基因的序列信息, 可以详细地揭示样本中的微生物多样性直至种的水平。得到的序列信息亦可以作为建立特异性寡核苷酸探针的基础, 为原位杂交等方法提供依据。尽管他有其缺点(如:样品总DNA的提取效率偏差; PCR过程中产生的偏差; 挑取克隆的随机性),但到目前, 这种方法广泛用于深海微生物多样性研究, 特别是通过16S rRNA基因文库反映微生物群落结构的研究, 这种方法也广泛用于古菌功能基因多样性研究。以16S rRNA基因为研究对象的现代分子生物学方法, 把对微生物多样性的研究推进到一个新的时代。
古菌16S rRNA基因文库分析表明, 西太平洋菲律宾东板比科尔陆架泥质区在6 m深度沉积物范围内, 古菌多样性不高, 仅有5种古菌类群。序列来自两大类, 泉古菌和广古菌, 以泉古菌为主。有报道指出海洋中古菌主要为泉古菌门, 仅有小部分广古菌门种类[4]。本文做出的结果符合上述观点。该区泉古菌又以MBGB为主。各个层位中的优势类群又稍有不同, MCG在V(1 m)层位中较为优势, MGI则在表层 H(0.05 m)中占优势, 随着沉积物的深入, MBGB占优势地位, 接近饱和, 其他古菌类群则逐渐减少。导致这一结果的原因可能是沉积物中的古菌群落结构受海域和菌群的适应性所影响。此区沉积物古菌的同源序列大部分来自太平洋深海、鄂霍次克海、加利福尼亚州圣巴巴拉盆地过渡区大陆边缘、地中海、Nankai海槽、韩国海绵共生的古菌, 冷泉环境。另外, 古菌群落垂向分布结构总体没有显著差异,只在小尺度上有些不同。
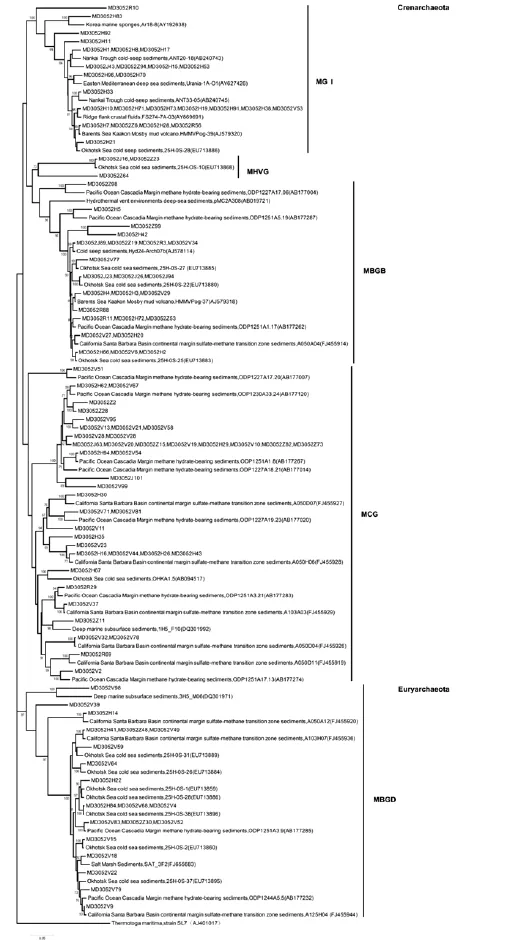
图2 比科尔陆架深海沉积物中古菌16S rRNA基因序列系统发育树Fig.2 Phylogenetic tree of the archaeal 16S rRNA gene sequences recovered from the Bicol shelf mud wedge

图3 沉积物古菌16S rRNA基因PCA分析Fig. 3 The principal component analysis of 16S rRNA genes obtained from the archaea in deep sea sediments
根据 PCA聚类分析结果显示 MD06-3052站位J(5 m)、R(6 m)层位沉积物的古菌16S rRNA基因序列最为相似聚在一起(图3)。MBGB在这两层中占的比例也较大, 将近饱和状态, 是这两层位中的优势菌群。另外根据多样性指数及稀释曲线, 也可诠释上述结果。
本文的研究结果揭示了比科尔陆架深海古菌垂向分布特征, 为今后大范围自然海区古菌生态学研究提供科学参考。且西太平洋海域深海沉积物古菌多样性的研究对了解深海环境特点、开发和利用深海微生物资源提供了重要的理论依据和基础。
[1]陈秀兰, 张玉忠, 高培基. 深海微生物研究进展[J].海洋科学, 2004, 28(1):61-66.
[2]Whitman W B, Coleman D C, Wiebe W J. Prokaryotes:The unseen majority [J]. Proc Nat Acad Sci USA, 1998,95:6578-6583.
[3]Parkes R J, Webster G, Cragg B A, et al. Deep sub-seafloor prokaryotes stimulated at interfaces over geological time[J]. Nature, 2005, 436:390-394.
[4]Karner M B, DeLong E F, Karl D M. Archaeal dominance in the mesopelagic zone of the Pacific Ocean[J].Nature, 2001, 409:507-510.
[5]Delong E F. Archaea in coastal marine environments[J]. Proc Natl Acad Sci USA, 1992, 89:5685-5689.
[6]Delong E F, Wu K Y, Prezdin B B, et al. High abundance of Archaea in Antarctic marine picoplankton [J].Nature, 1994, 371:695-697.
[7]Fuhrman J A, Ouverney C C. Marine microbial diversity studied via 16S RNA sequences:cloning results from coastal waters and counting of native archaea with fluorescent single cell probes[J]. Aquatic Ecology,1998, 32:3-15.
[8]Pearson A, Huang Z, Ingalls A E, et al. Non-marine crenarchaeol in Nevada hot springs[J]. Appl Environ Microbiol, 2004, 70:5229-5237.
[9]Britschgi T B, Giovannoni S J. Phylogenetic analysis of a natural marine bacterioplankton population by rRNA gene cloning and sequencing [J]. Appl Environ Microbiol, 1991, 57:1707-1713.
[10]Itoh T, Suzuki K, Sanchez P C, et al. Caldisphaera lagunensis gen. Nov. sp. Nov., a novel thermoacidophilic crenarchaeote isolated from a hot spring at Mt Maquiling, Philippines [J]. Int J Syst Evol Microbiol, 2003,53:1149-1154.
[11]Volkl P, Huber R, Drobner E, et al. Pyrobaculum aerophilum sp. Nov., a novelnitrate-reducing hyperthermophilic archaeum [J]. Appl Environ Microbiol,1993, 59:2918-2926.
[12]Nakagawa S, Takai K, Horikoshi K, et al. Aeropyrum camini sp. Nov., a strictly aerobic, hyperthermophilic archaeon from a deep-sea hydrothermal vent chimney[J]. Int J Syst Evol Microbiol, 2004, 54:329-335.
[13]Prokofeva M I, Miroshnichenko M L, Kostrikina N A,et al. Acidilobus aceticus gen. Nov, sp. Nov., a novel anaerobic thermoacidophilic archaeon from continental hot vents in Kamchatka [J]. Int J Syst Evol Microbiol,2000, 50:2001-2008.
[14]Kemp P F, Aller J Y. Bacterial diversity in aquatic and other environments:what 16S rDNA libraries can tell us[J]. FEMS Microbiology Ecology, 2004, 47(2):161-177.
[15]Kormas K A, Tivey M K, Von Damm K, et al. Bacterial and archaeal phylotypes associated with distinct mineralogical layers of a white smoker spire from a deep-sea hydrothermal vent site (9 degrees N, East Pacific Rise) [J]. Environmental Microbiology, 2006, 8(5):909-920.
[16]Krebs C J. Ecologieal Methodology [M]. 2thed. Menlo Park, CA:Benjamin Cummings, 1998.
[17]Takai K, Horikoshi K. Diversity of archaea in deep-sea hydrothermal vent environments[J]. Genetics, 1999,152:1285-1297.
[18]Vetriani C, Jannasch H W, MacGregor B J, et al.Population structure and phylogenetic characterization of marine benthic archaea in deep-sea sediments[J].Appl Environ Microbiol, 1999, 65(10):4375-4384.
[19]Sørensen K B, Lauer A, Teske A. Archaeal phylotypes in a metal-rich, low-activity deep subsurface sediment[J]. Geobiology, 2004,2:151-161.
[20]Knittel K, Losekann T, Boetius A, et al. Diversity and distribution of methanotrophic archaea at cold seeps[J].Appl Environ Microbiol, 2005, 71:467–479.
[21]Inagaki F, Nunoura T, Nakagawa S, et al. Biogeographical distribution and diversity of microbes in methane hydrate-bearing deep marine sediments on the Pacific ocean margin[J]. Proc Natl Acad Sci USA, 2006,103:2815-2820.
[22]Biddle J F, Lipp J S, Lever M A, et al. Heterotrophic Archaea dominate sedimentary subsurface ecosystems off Peru[J]. Proc Natl Acad Sci USA, 2006, 103:3846-3851.
[23]Coolen M J L, Cypionka H, Sass A M, et al. Ongoing modification of Mediterranean sapropels mediated by prokaryotes[J]. Science, 2002, 296:2407-2410.
[24]Takai K, Moser D P, DeFlaun M, et al. Archaeal diversity in waters from deep South African Gold Mine[J].Appl Environ Microbiol, 2001, 67:5750-5760.
[25]Inagaki F, Suzuki M, Takai K, et al. Microbial communities associated with geological horizons in coastal subseafloor sediments from the Sea of Okhotsk[J].Appl Environ Microbiol, 2003, 69:7224-7235.
The diversity of archaea in deep-sea sediments from the West Philippine Sea Bicol shelf
Gegentana1, Sarengaowa2, LI Tie-gang2, ZHOU Wei-guang2, YU Xin-ke1
(1. College of Animal Science and Veterinary Medicine of Inner Mongolia Agricultural University, Hohhot 010018,China; 2. Key Laboratory of Marine Geology and Environment, Institute of Oceanology, the Chinese Academy of Sciences, Qingdao 266071, China)
Feb., 1, 2010
the West Pacific Ocean; deep-sea sediments; archaea; diversity
The archaeal diversity and phylogenesis of the sediments samples from the Bicol shelf of the West Philippine Sea for the West Pacific Ocean were analyzed by the 16S rRNA gene clone library method. Totally 465 cloning sequences were obtained and divided into 63 OTUs. Phylogenetic analysis showed that the archaeal sequences were from Crenarchaeota and Euryarchaeota. The majority of the archaeal phylotypes were Marine Benthic Group B (MBGB), Marine Group I (MGI), Miscellaneous Crenarchaeotic Group (MCG), and Marine Benthic Group D. A few sequences fell into Marine Hydrothermal Vent Group (MHVG). The results indicated that the sediment samples of five different layers contained only five groups of archaea. Achaeal community diversity was low, and dominant archaea had some differences in each layers.
Q16 文献标识码:A 文章编号:1000-3096(2011)11-0004-07
2010-02-01;
2011-07-21
中国科学院知识创新工程领域前沿项目(MGE2008KG06);中国科学院知识创新工程重要方向项目(KZCX2-YW-JC201,KZCX2-YW-211-03)
格根塔娜(1982-), 女, 蒙古族, 内蒙古呼伦贝尔人, 硕士研究生, 海洋地质微生物研究, 电话:0532-82898835, E-mail:x.tn2008@163.com; 于心科, 通信作者, 电话:0532-82896726, E-mail:geomicrobiology@126.com
刘珊珊)

