应用 ISSR分子标记探讨酵母菌种间亲缘关系*
徐晓波, 蒋冬花, 李 杰, 后家衡
(浙江师范大学化学与生命科学学院,浙江金华 321004)
酵母菌是一类十分重要的微生物,对其分类与鉴定是人类开发利用酵母菌的基础.传统的酵母菌种鉴定主要依据其形态特征,尤其是生理特征,但是这些特征易受培养条件等影响而出现不确定的结果.近年来,DNA分子标记技术的引入给微生物分类鉴定产生了巨大的推动作用,使其分类鉴定工作由一般的表型特征鉴定深化到分子水平和遗传型特征的鉴定[1].
简单重复序列区间 (ISSR)标记技术是由 Zietkiewicz等[2]提出的,该技术检测的是 2个简单重复序列(SSR)之间的一段短DNA序列上的多态性[3].ISSR技术的基本原理就是利用真核生物基因组中广泛存在的 SSR序列,设计出各种能与 SSR序列结合的聚合酶链式反应 (PCR)引物,对 2个相距较近、方向相反的 SSR序列之间的DNA区段进行扩增.一般在引物的 5′或 3′端接上 2~4个嘌呤或嘧啶碱基,以对具有相同重复形式的许多 SSR座位进行筛选,使得最终扩增出的 ISSR片段不会太多[4-6].ISSR技术所用的 PCR引物长度在 20个核苷酸左右,因此可以采用与常规 PCR相同的反应条件,该技术克服了限制性内切酶片段长度多态性 (RFLP)标记、SSR标记和随机扩增多态性 DNA标记 (RAPD)的技术限制,具有操作简单、可重复性高、模板 DNA用量少等优点[7-10].目前,ISSR分子标记在植物上应用较为广泛,但在微生物中的应用还不多,将 ISSR分子标记用在酵母菌种的分子鉴定中尚未见有报道.
本研究应用 ISSR标记对实验室保藏的 19株待鉴定的酵母菌进行了分子标记鉴定,检测这种分子标记技术用于酵母菌菌种鉴别的可行性,为酵母菌的遗传分析和分类研究提供DNA水平的证据.
1 材料及方法
1.1 材料与试剂
从梨、苹果、葡萄、猕猴桃等水果表皮的不同部位,以及果园土、泡菜、酒曲等中分离筛选得到 19个酵母菌菌株,分别进行编号为 Y-1-Y-19.
用于 ISSR-PCR反应的 dNTPs,Taq DNA聚合酶及 DL 2000TMDNA Marker均购自宝生物工程 (大连)有限公司.从 100条 ISSR引物中选取了 27条引物进行试验,引物 (其序列见表 1)由上海生工生物技术有限公司合成.
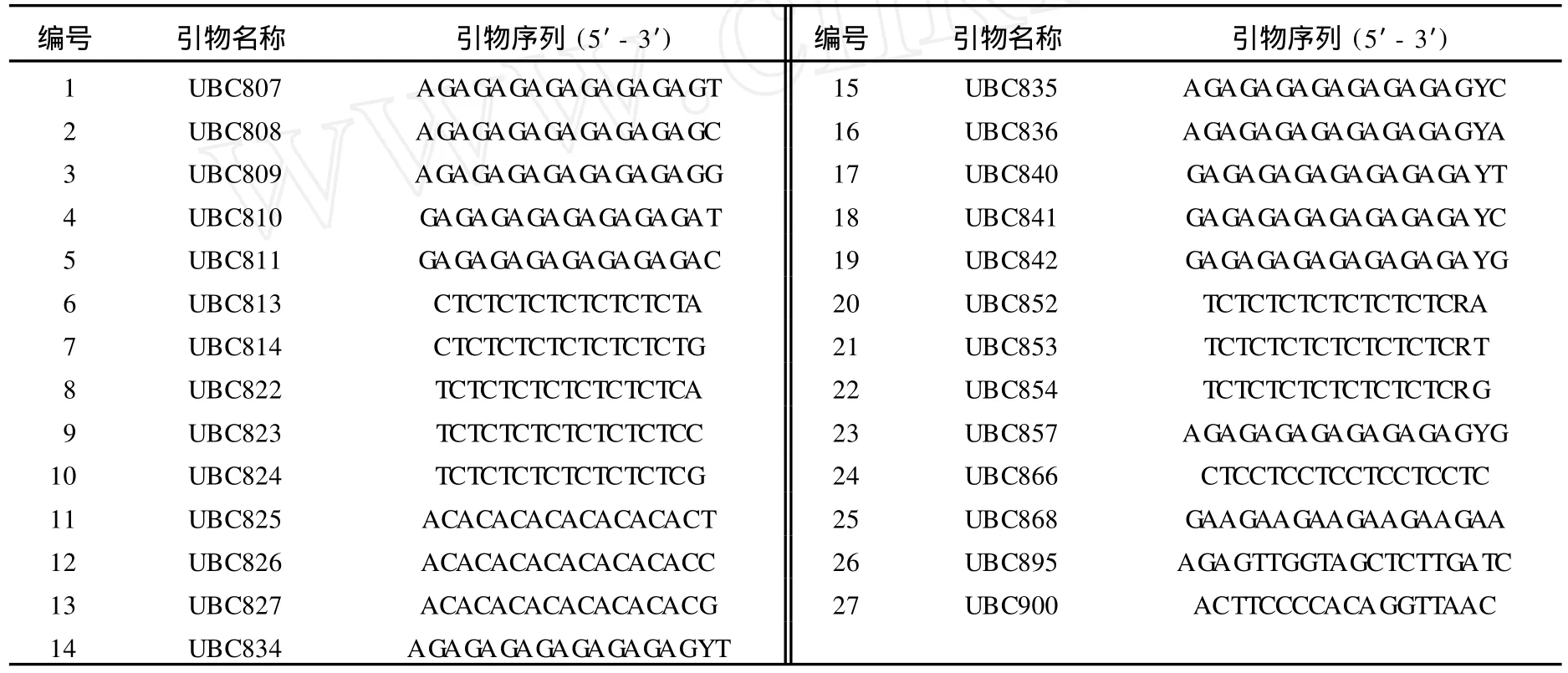
表 1 ISSR标记实验的引物序列
1.2 基因组 DNA的提取
使用 TI ANamp Yeast DNA Kit提取酵母菌基因组 DNA,试剂盒购于北京科百奥生物科技有限责任公司.用 0.8%的琼脂糖凝胶电泳检测 DNA的质量,通过紫外分光光度计测定 DNA的浓度和纯度,并将 DNA溶液稀释至 50 ng/μL.
1.3 引物的筛选
[11-12].用 27条 ISSR引物对酿酒酵母 (Saccharom yces cerevisiae)进行扩增实验,从中筛选出带形清晰、多态性丰富的引物.扩增反应体系为 25μL,含 50 ng模板 DNA,50 ng引物,1.0μL dNTPs溶液 (2.5 mmol/L),1.5μL MgCl2溶液 (25 mmol/L),1.0 U Taq DNA聚合酶,1×PCR缓冲液.反应程序为:94℃预变性 5 min;94℃变性 30 s,52℃退火 45 s,72℃延伸 90 s,扩增 45个循环;72℃延伸7 min.
1.4 PCR正交设计实验
参考文献 [13-15].采用 L9(34)正交法,对影响 ISSR-PCR扩增的主要因素:Mg2+,dNTPs,引物和Taq DNA聚合酶设计 4因素 3水平的正交试验,方案见表 2和表 3.反应体系总体积为 25μL,包括 1×PCR缓冲液,50 ng模板 DNA,其他各成分按照表 2加样,最后用重蒸馏水 (ddH2O)补足.每个处理做 2次重复.
反应结束后,PCR产物在 1.5%的琼脂糖凝胶中 120 V电泳 1 h,在含有 0.5μg/mL EB(溴化乙锭)的染色液中染色后,置凝胶成像系统中进行拍照分析.

表 2 ISSR-PCR体系各因素和水平
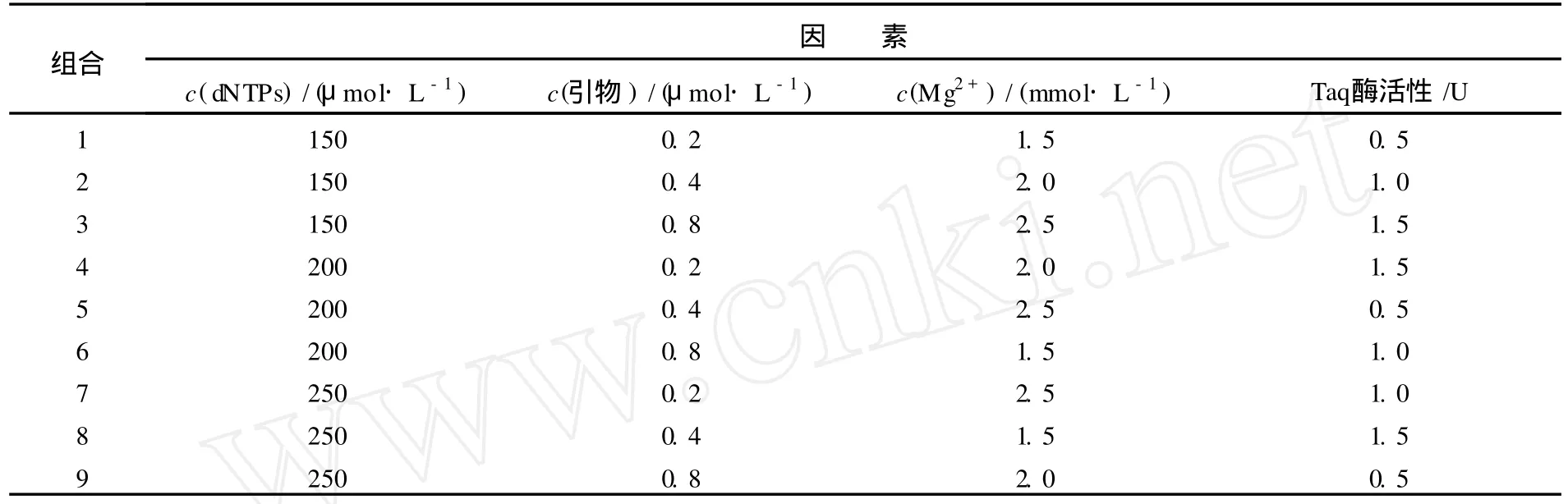
表 3 ISSR-PCR正交试验设计 (L9(34))
1.5 退火温度和模板DNA浓度的筛选
根据正交试验找出最佳组合后,使用梯度 PCR,在 48~55℃进行退火温度的筛选,选出最佳的退火温度.利用 ISSR-PCR的最佳组合进行 DNA浓度对 ISSR扩增结果的影响试验,25μL反应体系中模板设 10,20,30,40,50,60,70,80和 90 ng共 9个处理.2个实验所用的引物均为 UBC809.
1.6 DNA多样性聚类分析
参照文献[16].以最适条件进行 PCR扩增,PCR扩增产物用 1.5%琼脂糖凝胶电泳分离获得扩增图谱,根据同一位点带的有无编成 1,0矩阵输入计算机,用 NTSYSpc软件中的 SI MQUA I程序计算菌株间的相似系数,最后用UPG MA法进行聚类分析,构建相似聚类树状图.
2 结果与分析
2.1 引物筛选结果
ISSR技术中很重要的步骤是对引物的筛选,合适的引物是扩增高产量、高分辨率分子标记的前提.用 27条 ISSR引物对酵母菌株进行 PCR扩增,电泳结果见图 1,其中 UBC807(1),UBC808(2),UBC809(3),UBC810(4),UBC811(5),UBC835(15),UBC836(16),UBC841(18),UBC842(19)和UBC857(23)共 10条引物扩增产物条带清晰、重复性和稳定性好,且多态性条带相对较多,故选用这 10条引物进行19个未知酵母菌株的 ISSR扩增实验.

图 1 ISSR引物筛选结果
2.2 PCR正交实验结果分析
依据琼脂糖凝胶电泳条带的强弱和杂带的多少做直观分析.PCR的扩增结果是 dNTP,引物,Mg2+和 Taq酶等因素综合作用的结果.从图 2可以看出:在 9个处理中,由于受 4个因素的浓度及组合不同的影响,扩增结果也存在明显的差异.通过比较发现,组合 2扩增的谱带不但多态性好,且谱带清晰,是最理想的组合.本实验确定 2号组合为最佳组合,即 25μL PCR反应体系中含:150μmol/L dNTPs,2.0 mmol/L Mg2+,0.4μmol/L ISSR引物和 1U Taq酶.
2.3 退火温度对 ISSR扩增结果的影响
利用 PCR正交实验得到的最佳组合 2,引物选择 UBC809,使用梯度 PCR法,在 48~55℃进行退火温度的筛选,结果见图 3,通过比较得出最佳的退火温度为 52~54℃.
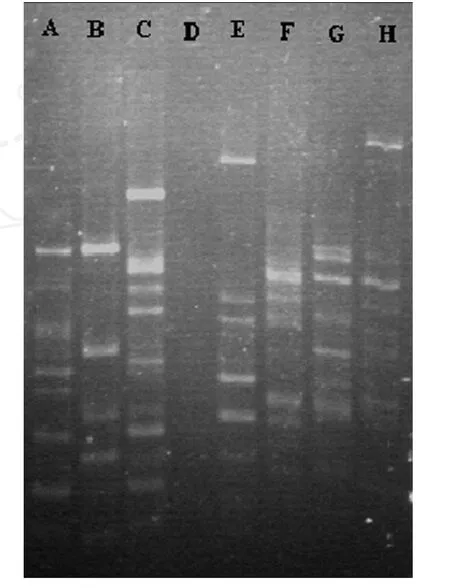
图 3 退火温度对 ISSR扩增结果的影响

图 2 正交设计 ISSR-PCR反应体系扩增结果
2.4 模板 DNA用量对 ISSR扩增结果的影响
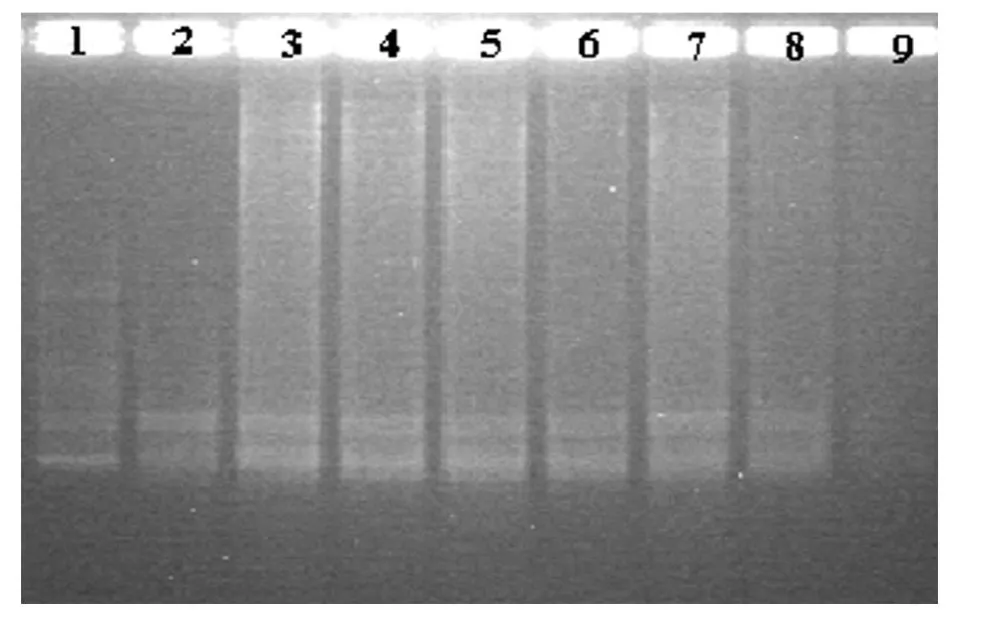
图 4 模板 DNA用量对 ISSR扩增结果的影响
利用 PCR正交实验得到的最佳组合2,在 25μL反应体系中,设 DNA含量分别为 10,20,30,40,50,60,70,80和 90 ng共 9个处理,引物选择 UBC809,扩增结果如图 4.结果表明:DNA模板的适宜用量有一个较大的范围,电泳条带差异不明显,DNA用量在 10~80 ng均可扩增出条带.本实验最终选择在 25μL反应体系中DNA的用量为 50 ng.
2.5 ISSR扩增结果
在最适的 ISSR扩增条件下,对供试的 19株酵母菌 DNA进行了 10条引物的ISSR扩增实验,经重复试验 (3次)扩增结果稳定.部分引物对供试菌株的扩增结果见图 5.
10条引物共扩增出 108条带,其中 90.7%呈多态性 (见表 4).Y-5酵母的 ISSR扩增图谱与其他菌株的图谱差异较大,扩增的条带较多.在后面的聚类分析中,以上述 10个引物扩增的产物图谱为依据,对供试的 19个酵母菌株进行聚类分析.
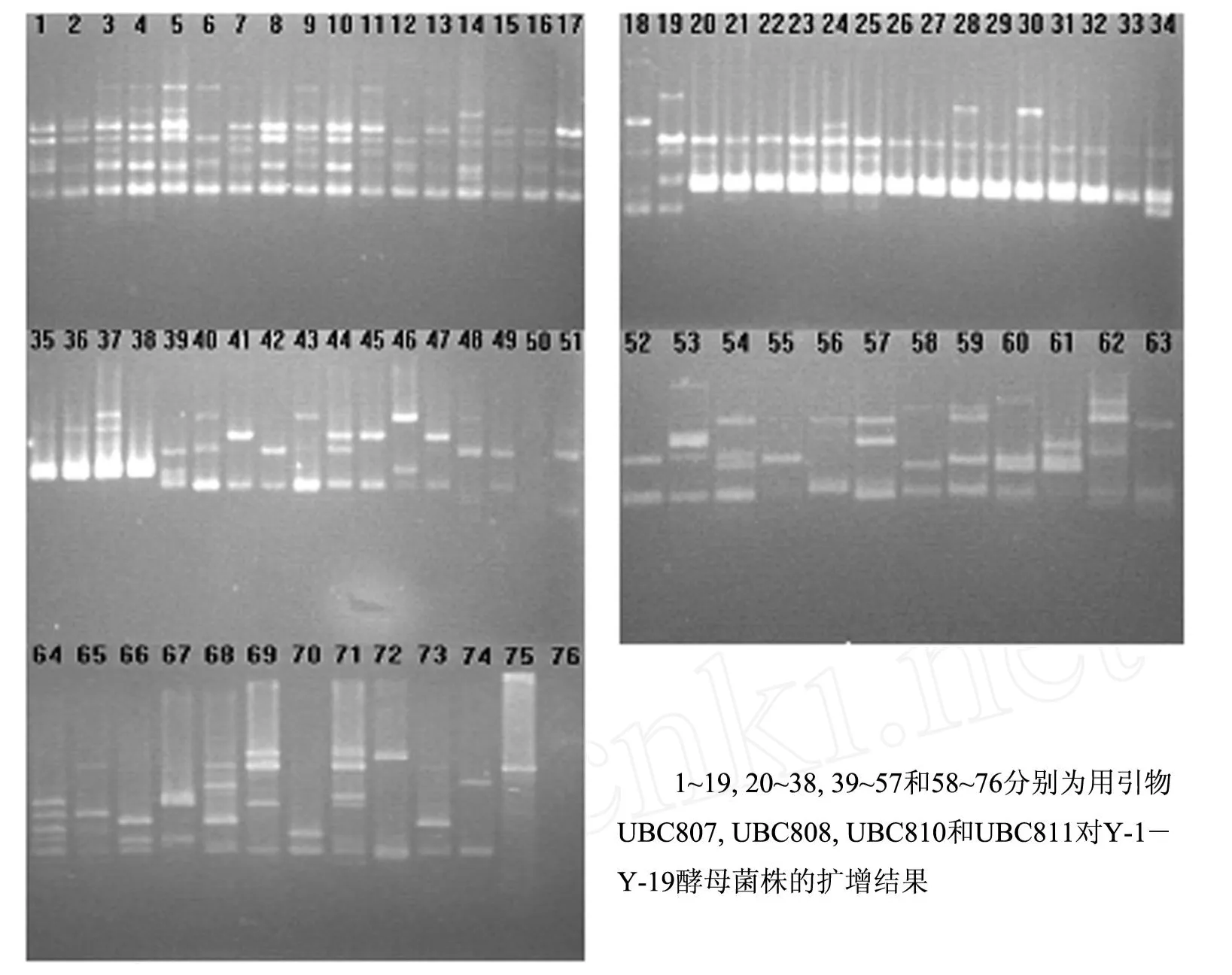
图 5 部分引物对 19株酵母菌的扩增结果
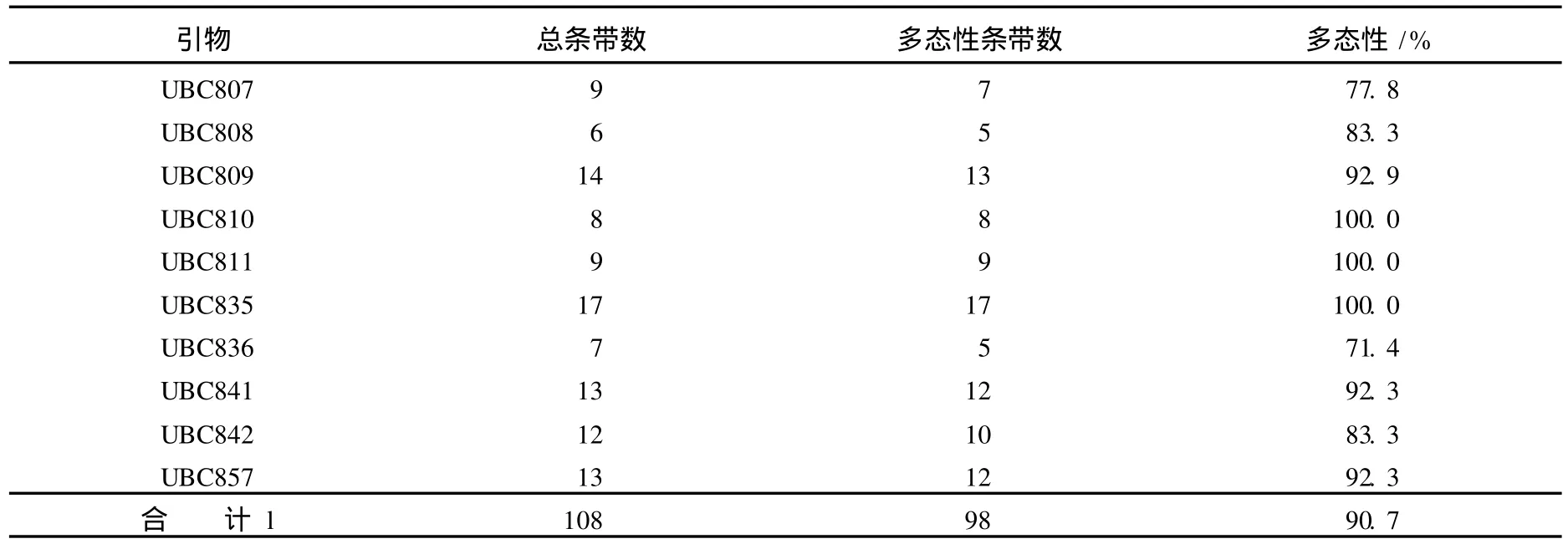
表 4 19株酵母菌 ISSR扩增的总条带数和多态性条带数
2.6 ISSR扩增产物的聚类分析
采用 NTSYSpc软件中的 SI MQUA I程序进行数据分析,计算两两菌株间的遗传相似系数.从不同水果表皮和自然环境中分离得到的 19株酵母菌的遗传相似系数为 0.48~1.00,最小的是酵母Y-18与其他酵母的相似系数 (0.48),相似系数最高的是酵母 Y-16和 Y-17,Y-9和 Y-13,两者的相似系数都达到了1.00.
如图 6所示,在相似水平为 0.61处,19个酵母菌株可以划分为 3个群,第Ⅰ群:Y-1,Y-2,Y-5,Y-6,Y-19,Y-8,Y-16,Y-17,Y-9,Y-13,Y-3,Y-4,Y-7;第Ⅱ群:Y-10,Y-11,Y-12,Y-14,Y-15;Y-18酵母单独代表一个群.
3 讨 论
ISSR分子标记多态性水平高,易于分析,不受环境影响,在品种鉴定中显示出巨大的应用潜力[17].ISSR引物为重复序列,由于重复序列在基因组中是变异最快的成分,不受或很少受到自然选择的影响,故其变异保留下来,所以重复序列区域的多态性远高于基因区域的多态性,这是 ISSR标记多态性较高的原因所在[18].
本实验利用正交试验设计方法,初步建立并优化了酵母菌 ISSR分子标记的 PCR反应体系,这一反应体系受诸多因素的影响,包括 Taq酶,Mg2+,dNTP,引物和模板 DNA浓度,以及退火温度都会影响PCR产物的生成和质量.ISSR标记技术已经开发出 100多条通用引物,但不是每条引物都适合于所有物种,因此,运用该标记时对引物的筛选也是非常必要的.本研究结果显示:不同的 ISSR引物对不同的酵母菌种间的区分度有所不同.在利用该标记技术进行分类鉴定及亲缘关系研究时,应尽量选用多种引物进行扩增反应,在综合大量谱带信息的基础上再进行聚类分析.
本研究利用 ISSR分子标记获得的多态性条带,对本实验室分离纯化得到的 19株酵母菌可在一定程度上进行区分,比较适合于酵母菌的种间鉴别.因此,ISSR分子标记技术应用于酵母菌菌种鉴别是可行的,可以作为酵母菌分类鉴定的辅助手段.要将这 19株酵母菌准确鉴定到种的水平,还需结合其他辅助手段,如 26 S rDNA D1/D2区序列分析和 5.8 S r DNA-ITS区域的 PCR-RFLP分析等,每种技术手段都有各自的优缺点,多种技术手段的结合才能大大提高酵母菌种鉴定的准确性.

图 6 19株酵母菌 ISSR聚类分析树形图
参考文献:
[1]Kurtzman C P,Fell J W.The yeasts:A taxonomic study[M].New York:Elsevier,1998.
[2]Zietkiewicz E,RafalskiA,Labuda D.Genome fingerprinting by simple sequence repeat(SSR)-anchored polymerase chain reaction amplification[J].Genomics,1994,20:176-183.
[3]朴红梅,李万良,穆楠,等.ISSR标记的研究与应用[J].吉林农业科学,2007,32(5):28-30.
[4]谢佳燕,张知彬.ISSR标记技术及其在遗传多样性研究中的应用[J].兽类学报,2004,24(1):71-77.
[5]陈龙,王家良,杨贤松.ISSR分子标记及其在植物分子生物学中的应用[J].种子,2007,26(10):49-52.
[6]姚明哲,黄海涛,余继忠,等.ISSR在茶树品种分子鉴别和亲缘关系研究中的适用性分析[J].茶树科学,2005,25(2):153-157.
[7]关萍,马丹炜,王贵侠,等.利用 ISSR标记对天麻的贵州种群遗传多样性分析[J].北京林业大学学报,2007,29(6):35-40.
[8]宋培玲,杨家荣,毛岚.正交设计优化棉花黄萎病菌 ISSR-PCR反应体系[J].西北农业学报,2009,18(1):194-197.
[9]Ge X J,SunM.Reproductive biology and genetic diversityof a cryptoviviparousmangroveAegiceras corniculatum(Myrsinaceae)using allozyme and intersimple sequence repeat(ISSR)analysis[J].Molecular Ecology,1999,8(12):2061-2069.
[10]王玉民,姜昱,康岭生,等.利用 ISSR技术探讨大豆属植物的亲缘关系[J].吉林农业科学,2007,32(6):30-32;38.
[11]王俊平,郭卫东,倪开诚,等.矮化佛手的形态特征及遗传背景分析[J].浙江农业科学,2007(4):407-410.
[12]余艳,陈海山,葛学军.简单重复序列区间 (ISSR)引物反应条件优化与筛选[J].热带亚热带植物学报,2003,11(1):15-19.
[13]刘海河,侯喜林,张彦萍.西瓜 ISSR-PCR体系的正交优化研究[J].果树学报,2004,21(6):615-617.
[14]王佳,梁国华,缪旻珉,等.正交设计优化黄瓜 ISSR体系[J].分子植物育种,2006,4(3):439-442.
[15]陈大霞,钱敏,李隆云,等.正交试验优化黄连 ISSR反应体系的研究[J].重庆中药研究,2006(2):4-7.
[16]冷言峰,马云芳,何桥,等.几个槐树植物的 ISSR鉴定[J].西南师范大学学报:自然科学版,2007,32(6):83-86.
[17]侯渝嘉,何桥,李中林,等.应用 ISSR分子标记对茶树种质资源进行分子鉴定[J].西南农业学报,2007,20(6):1272-1276.
[18]张立荣,徐大庆.SSR和 ISSR分子标记及其在植物遗传育种中的应用[J].河北农业大学学报,2002,25(1):90-94.

