基于Web Service的生物学实体映射数据库
张 智 张正国
(中国医学科学院基础医学研究所、北京协和医学院基础学院,北京 100005)
引言
随着生物医学的不断快速发展,产生了海量的在线(online)生物医学数据资源,其涉及的生物学实体有基因、蛋白质、小分子物质、化合物和药物等。生物学实体映射就是要实现对上述实体的不同标识符和名称之间的相互转换。现在,生物医学研究者越来越希望将他们的实验结果与存在于海量在线信息中的潜在相关数据联系在一起。获取生物学实体映射信息可以部分地满足研究者们的这种需求。同时,从事生物医学文本挖掘和信息检索的研究者在命名实体识别(named entity recognition,NER)和查询关键词扩展方面也需要生物学实体映射的支持。在实际工作中,Cote等建立了基于Web Service的蛋白质标识符交叉引用(protein identifier cross-reference,PICR)服务,成功地实现了不同蛋白质数据库标识符的映射[1]。本研究致力于实现更多生物学实体的映射,不仅仅局限于蛋白质。
网络程序接口一般分为基于HTML和基于XML两类。传统的基于HTML的网络程序接口存在一个严重的不足,即对于单个的网络应用程序的访问很方便,但是对于集成多个相关网络应用程序,进而创建一个新的工作流程就非常困难[2]。此时,需要对从一个服务器返回的数据进行分析处理和重新格式化后才能提交到下一个,并且,连续不断地进行类似的笨重工作。为了解决上述问题,基于XML的网络程序接口解决方案应运而生,而Web Service就是其中最成功的一种。Web Service是一个独立于平台的、松耦合的、可编程的网络应用程序,使用开放的XML标准描述、发布、发现和配置这些应用程序,适用于开发分布式的互操作的应用程序[3]。近些年来,随着信息技术的快速发展以及研究者需求的不断增加,生物医学数据网络访问接口逐渐从基于HTML的方式发展到基于Web Service的方式。Cote等建立了基于Web Service的生物医学本体查找服务(ontology lookup service,OLS)[4],研究者可以将OLS用于生物医学各领域内数据的整合与分析,以及作为规范词表(controlled vocabulary)用于生物医学文本挖掘领域。Angelo等构建了提供表型和基因型数据集成及挖掘服务的Web Service框架[5],而 Galvez等提出的“下一代生物信息学”就是使用多核处理器架构为序列比对(sequence alignment)开发 Web Service[6]。
为了给生物医学研究者提供日常的生物学实体映射服务,同时给生物医学文本挖掘和信息检索提供技术支持,本研究构建了一个生物学实体映射数据库,该数据库存储了基因、蛋白质、小分子物质、化合物和药物等实体的映射信息。同时,为了方便用户使用生物学实体映射数据库,特别是为了实现数据共享和软件复用,本研究构建了一个基于Web Service的生物学实体映射网络应用系统,该系统为一般用户提供通过浏览器方式访问生物学实体映射数据库途径,为从事相关软件开发的用户提供通过Web Service方式访问生物学实体映射数据库。
1 材料和方法
1.1 设备和数据
本研究采用服务器的硬件配置:中央处理器为英特尔至强(Intel Xeon)E5530;内存类型为DDR2 ECC,容量为4 GB;硬盘类型为SATA,容量为5 TB。服务器的系统软件全部采用免费软件或开源软件。操作系统采用运行稳定、高效的Linux系统Debian 5;数据库服务器采用关系型数据库MySQL 5.1.41;网络服务器采用 Tomcat 6.0.20;Java编程环境为Java EE 6。
本研究采用的数据来源于6个生物学实体映射数据集,如表1所示。这些数据集提供的原始数据文件类型是以制表符(TAB)作为分隔符的文本文件。

表1 生物学实体映射数据集Tab.1 Biological entity mapping data sets
1.2 方法
1.2.1 构建生物学实体映射数据库
本研究通过解析和清洗原始数据,并将其用转换为MySQL数据库的方法构建生物学实体映射数据库,具体流程如图1所示。首先,通过文件解析模块获取6个原始数据集中的数据内容。解析原始数据文件的同时,还需要进行数据清洗的工作。数据清洗模块将对解析得到的文件内容进行有效甄别。第一,保留生物学实体相关内容,去除其他与本研究无关的信息;第二,改正或删除格式不正确的生物学实体标识符(ID)或名称;第三,去除原始数据中可能存在的无效字符,如多余的空格、制表符及null字符等。然后,通过MySQL提供的驱动程序,使用Java数据库互连(Java database connectivity,JDBC)应用程序编程接口,把清洗好的生物学实体映射数据存储到MySQL数据库中,并建立必要的索引以优化数据库的检索。最后,使用消除冗余模块,对已构建好的数据库进行全盘扫描,消除冗余数据。
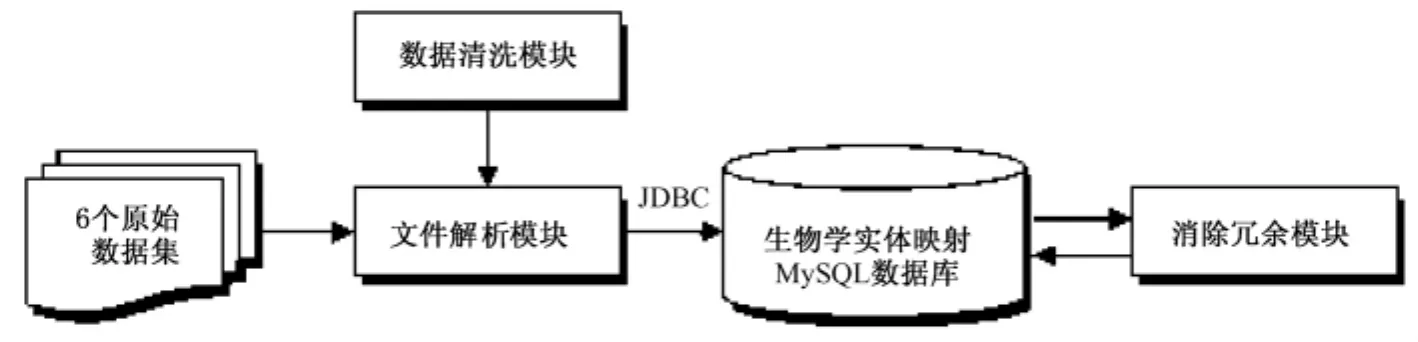
图1 构建生物学实体映射数据库的流程Fig.1 The procedure of constructing biological entity mapping database
1.2.2 构建生物学实体映射Web Service
本研究采用Apache CXF作为Web Service的实现平台,构建的Web Service框架如图2所示。Web Service的接口类模块定义了暴露给用户的服务,Web Service的实现类模块完成了接口类中定义的服务。由于本系统提供的Web Service返回结果是生物实体模型对象列表,该列表属于网络服务描述语言(web services description language,WSDL)规范中的复杂类型(complex type),CXF不能直接支持,因此需要建立复杂类型的适配器,告知CXF如何将生物实体模型对象列表转化为CXF支持的对象,从而返回正确的结果数据。
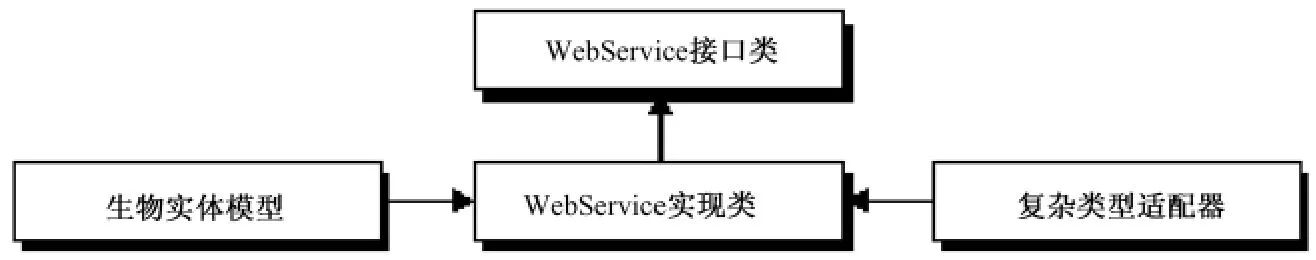
图2 生物学实体映射Web Service框架Fig.2 The framework of biological entity mapping web service
1.2.3 构建生物学实体映射网络应用系统
使用Java语言平台构建了一个网络应用系统,其架构如图3所示。系统分为4个部分:展现层(presentation layer)、Web Service接口层(web service interface layer)、服务层(service layer)和持久层(persistence layer)。展现层实现了用户使用浏览器,通过HTTP协议与服务器进行数据交互,以JSP作为展现层的技术实现。Web Service接口层实现了用户使用自编的程序调用系统提供的Web Service,通过SOAP协议与服务器进行数据交互。其中,服务契约模块为用户提供Web Service的描述信息,服务适配器模块负责根据用户选择的服务调用服务层的Web Service组件。服务层实现了整个网络应用系统的业务逻辑,该层包括生物实体模型、生物实体映射和Web Service共3个组件。生物实体模型是整个系统的业务实体,是基本的数据存储结构和单位;生物实体映射组件负责根据用户提交的查询参数检索数据库,并根据生物实体模型构造返回结果;Web Service组件负责实现和维护系统提供的Web Service。服务层是网络应用系统的核心,它根据由展现层或Web Service接口层提交的用户查询请求,通过持久层获取用户所需数据,并将这些数据返回给展现层或Web Service接口层,进而返回给用户。持久层实现了对数据库的物理访问。数据访问组件负责对数据库的检索,而数据缓存组件负责缓存查询结果以提高系统的性能。
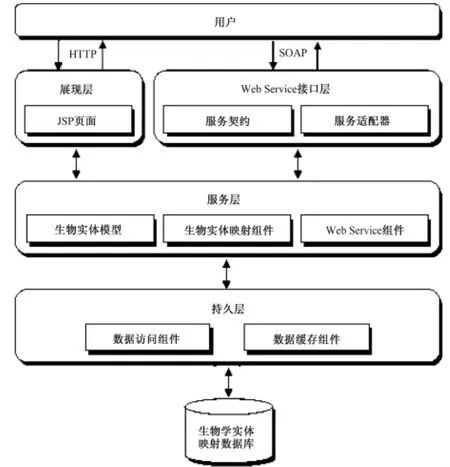
图3 网络应用系统架构Fig.3 Architecture of web application system
2 结果
2.1 生物学实体映射数据库
利用所编写的Java程序,完成了将6个原始生物学实体映射数据集转换存储到MySQL数据库的工作,形成了一个由MySQL统一管理、全新的生物学实体映射数据库,其数据存储量约为12 GB,相应索引的存储量约为15 GB,所存储的生物学实体映射记录超过2亿条。
2.2 生物学实体映射网络应用程序
构建了一个生物学实体映射网络应用程序,该系统查询界面如图4(a)所示。可以在区域①处以单列列表的形式输入生物学实体标识符或名称;可以在区域②处对映射的数据库进行限定,默认为映射到可能的最大范围;点击区域③处的提交按钮进行查询。本系统以列表的形式返回查询结果,如图4(b)所示,表头从左至右分别为输入的实体参数、映射类型及映射结果。区域①处显示了用户查询参数;区域②处是数据结果操作按钮,从左至右分别为复制结果数据到剪贴板、保存结果数据为CSV格式文件、保存结果数据为Excel格式文件以及打印结果数据共4项功能;在区域③处输入字符,可以按输入字符过滤表格内容;在区域④处是一系列数据表格的分页显示操作按钮。
2.3 生物学实体映射Web Service
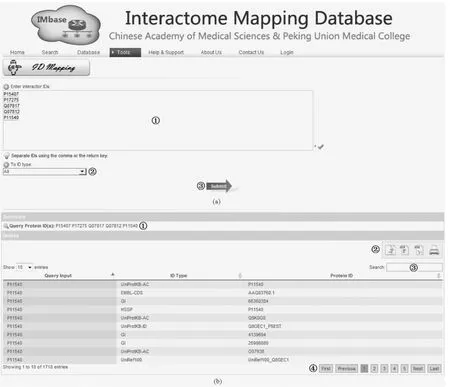
图4 生物学实体映射数据库Web应用程序。(a)查询提交界面;(b)查询结果界面Fig.4 Web application of biological entity mapping database.(a)query interface;(b)result interface
构建了一个生物学实体映射Web Service系统。用户可以通过自编程序,灵活地调用系统提供的Web Service,获取所需的映射数据信息。通过Web Service方式与通过上述网络应用程序的方式所获取的映射数据信息是完全等价的。以Java语言为例,用户编写的客户端程序的核心代码如下所示:

用户只需要以上代码就可以完成生物学实体的映射,进而可以实现将系统提供的Web Service集成到用户自己的系统中。
3 讨论和结论
本研究构建了一个生物学实体映射数据库,该数据库可以为研究者提供日常所需的生物学实体映射服务。例如,Entrez Gene、RefSeq和 UniProt等数据库的基因和蛋白质名称的互相映射,以及发现各类型实体之间的关联等多方面的应用。该数据库也可以用于生物医学文本挖掘,主要体现在两个方面:一方面,可提供命名实体识别所需要的词典(thesaurus);另一方面可用于扩展实体名称,以提高系统的召回率(recall),从而大幅提高文本挖掘系统的性能。同时,该数据库可以作为查询关键词的扩展支持,用于生物医学检索和搜索系统,提高其召回率,进而完善其性能。
本研究构建的网络应用系统采用网络应用程序和Web Service两种形式,为用户提供生物学实体映射服务。对于一般用户,可以使用浏览器,通过访问网络应用程序的方式使用该系统,直观地获取所需信息。对于从事相关软件开发的研究者,可以将系统提供的Web Service作为中间件服务,通过在其自编程序中调用Web Service的方式来使用该系统,使得该系统提供的Web Service嵌入到研究者自己的系统之中,实现了软件的复用和共享。特别地,一般的网络应用程序是无法满足第二类用户的需求的。
采用Web Service的方式构建网络应用系统有以下几点优势。
1)采用开放式的架构。Web Service的部署方式采用工业标准,独立于特定的供应商协议,依托现有的网络技术、数据交换技术、安全技术及其他已经存在的基础设施。因此,Web Service可以有效降低成本投入,并且可以进一步促进不同技术资源之间的相互整合利用。
2)语言和平台的无关性。Web Service和客户端可以分别由不同的计算机编程语言编写,分别运行在不同的操作系统平台上。例如,C/C++、C#、Java、Perl、Python、Ruby 和 VB 等计算机编程语言都可以实现Web Service的客户端,Windows、Linux和Unix平台都可以运行Web Service程序。
3)采用松耦合的模块化设计。这意味着一个新的服务可以方便地通过集成或层次化现有的服务而产生。
在实际应用中,选择适合研究者所开发系统的Web Service平台至关重要。现在,Web Service的主流平台有Axis 2,CXF和XFire等。Axis 2是一个重量级Web Service框架,其优势在于不但能制作和发布Web Service,而且可以生成Java和C/C++等多种语言下的Web Service客户端和服务端代码。但是,这也不可避免的导致了Axis 2使用和部署的复杂性。XFire是一个高性能的Web Service框架,其优点是简便易用,可以实现与现有Web开发框架的无缝整合。然而,XFire对Java之外的语言没有提供相关的代码工具,而且XFire已经停止维护,全面升级为CXF。CXF是一个新型Web Service框架,源于XFire项目,具备XFire的特点,同时实现了ESB(Enterprise Service Bus,企业服务总线),为 SOA(Service-Oriented Architecture,面向服务的体系结构)的实施提供了一种选择方案。综上所述,如果应用程序需要多语言的支持,则应该选择Axis 2;如果应用程序侧重于Java,而且希望遵循 Spring等Web框架进行开发,XFire就是一种更好的选择;如果在发布Web Service的同时还计划实施SOA,同时考虑应用程序的升级和可扩展性,则应该使用CXF。
本系统在追求完善系统架构的同时,还注重优化系统的性能。首先,在构建数据库时,消除了数据库中的冗余信息,保证了数据库内容简洁而高效;第二,对数据库中查询频繁的字段建立了全文索引(full-text index),大幅提高了查询速度;第三,在网络应用系统的持久层建立了数据缓存机制,减少了对数据库检索的磁盘I/O操作,缩短了系统的响应时间。
本研究涉及的所有软件均为免费软件或开源软件。本研究基于以下几点因素使用免费软件或开源软件:第一,安全性好;第二,可靠性和稳定性高;第三,杜绝盗版,遵守知识产权条约和世贸组织规定;第四,降低研究和开发成本。免费软件或开源软件满足了本研究涉及的异构数据库系统全部的设计需求,并在实际应用中取得出了令人满意的效果。
[1]Cote RG,Jones P,Martens L,et al.The Protein Identifier Cross-Reference(PICR)service:reconciling protein identifiers across multiple source databases[J].BMC Bioinformatics,2007,8(1):401.
[2]Neerincx PB,Leunissen JA.Evolution of web services in bioinformatics[J].Brief Bioinform,2005,6(2):178-188.
[3]Papazoglou MP.Web Service: Principles and Technology[M].Upper Saddle River,NJ: Pearson Education,2008.
[4]Cote RG,Jones P,Martens L,et al.The Ontology Lookup Service:more data and better tools for controlled vocabulary queries[J].Nucleic Acids Reseach,2008,36(Web Server issue):W372-376.
[5]Nuzzo A,Riva A,Bellazzi R.Phenotypic and genotypic data integration and exploration through a web-service architecture[J].BMC Bioinformatics,2009,Suppl 12:S5.
[6]Gálvez S,Díaz D,Hernández P,et al.Next-generation bioinformatics:using many-core processor architecture to develop a web service for sequence alignment[J].Bioinformatics,2010,26(5):683-686.
[7]Liu H,Hu ZZ,Zhang J,et al.BioThesaurus: a web-based thesaurus of protein and gene names[J].Bioinformatics,2006,22(1):103-105.
[8]Hettne KM,Stierum RH,Schuemie MJ,et al.A dictionary to identify small molecules and drugs in free text[J].Bioinformatics,2009,25(22):2983-2991.
[9]UniProt Consortium.The Universal Protein Resource(UniProt)in 2010[J].Nucleic Acids Research,2010,38(Database issue):D142-D148.
[10]Kuhn M,Szklarczyk D,Franceschini A,et al.STITCH 2: an interaction network database for small molecules and proteins[J].Nucleic Acids Research,2010,38(Database issue):D552-D556.
[11]Jensen LJ,Kuhn M,Stark M,et al.STRING 8-a global view on proteins and their functional interactions in 630 organisms[J].Nucleic Acids Research,2009,37(Database issue):D412-D416.

