根据mtDNA D-loop序列分析东海银鲳群体遗传多样性
彭士明, 施兆鸿, 陈 超, 侯俊利
(1. 中国水产科学研究院 东海水产研究所, 上海 200090; 2. 中国水产科学研究院 黄海水产研究所, 山东青岛 266071)
根据mtDNA D-loop序列分析东海银鲳群体遗传多样性
彭士明1, 施兆鸿1, 陈 超2, 侯俊利1
(1. 中国水产科学研究院 东海水产研究所, 上海 200090; 2. 中国水产科学研究院 黄海水产研究所, 山东青岛 266071)
根据线粒体D-loop序列对舟山群岛附近海域的银鲳(Pampus argenteus)群体(n=24)的遗传多样性进行了研究。通过PCR技术对线粒体D-loop序列进行扩增, 获得大小约为500 bp的扩增产物。PCR产物经纯化并进行序列测定后, 得到了357 bp的核苷酸片段。在24个个体中, 共检测到14个变异位点, 其中8个转换位点, 5个颠换位点, 1个转换与颠换同时存在的位点。运用MEGA软件计算出不同个体间的遗传距离, 并根据其遗传距离构建了UPGMA和NJ系统树。DNASP软件计算出的单倍型多样性(h)、核苷酸多样性(π)及平均核苷酸差异数(k)分别为 0.89、0.007与 2.57。此外, 岐点分布及中性检验显示, 东海银鲳群体在历史上可能经历过种群扩张。研究结果表明, 线粒体D-loop基因可用于银鲳群体内及群体间遗传多样性的分析。
银鲳(Pampus argenteus); 东海; 线粒体DNA; D-loop序列
银鲳(Pampus argenteus)是一种重要的海产经济鱼类, 具有巨大的市场需求, 广泛分布于东海、东南亚海、波斯湾、阿拉伯海和印度洋[1,2]。近年来, 由于过度捕捞其全球捕捞产量已显著降低[3]。中国鲳属鱼类资源的状况也并不容乐观, 从最新的调查和日常监测结果来看, 鲳鱼资源受到了严重的破坏, 过度捕捞不仅严重影响了捕捞种类的群体数量, 而且改变了鲳鱼的群体组成结构[4]。以往有关东海银鲳的研究主要集中在其渔业资源学[5,6]、繁殖生物学[7~10]及其人工育苗[11]等方面, 其遗传多样性方面的研究国内尚未见有相关报道。
线粒体基因组(mtDNA)具有结构简单、母系遗传、进化速率快、几乎不发生重组等特点, 业已成为研究种内、种间近缘群体间遗传分化关系的有效工具之一[12]。本研究对东海区银鲳线粒体 DNA控制区(D-loop)序列进行 PCR扩增和测序以分析东海区银鲳群体的遗传多样性, 研究结果可为制定合理的资源增殖与保护措施提供科学依据。
1 材料与方法
1.1 材料
24尾银鲳样品采自舟山群岛附近海域, 平均叉长为15.5 cm, 平均体质量为114.3 g, 取背部肌肉组织, 置于 95%乙醇中保存, 存放于-20℃中保存待用。
1.2 DNA的提取、扩增及测序
分别从24尾银鲳样品中取0.1 g肌肉用于提取总DNA。总DNA的提取采用常规的酚-氯仿方法[13],DNA经沉淀、洗涤并干燥后, 溶于50 μL TE缓冲液中, 于-20℃中保存备用。
扩增所用引物为, L15926:5′-TCAAAGCTTACACCAGTCTTGTAAACC-3′, H16498:5′-CCTGAAGTAGGAACCAGATG-3′[14]。每个 PCR反应总体积为50 μL, 其中 29.6 μL 超纯水、5 μL 10 × PCR 缓冲液、2 μL 10 mm dNTPs、5 μL 25 mm MgCl2、4 μL 10 mm引物(各 2 μL)、0.4 μL Taq DNA 聚合酶、4 μL 模板DNA。在GeneAmp PCR仪(9700)上进行PCR反应,反应程序为:94℃预变性2 min, 然后进行35个循环(94℃ 45 s, 52℃ 1 min, 72℃ 1 min), 72℃延伸 7 min,10℃保温。PCR产物用1%琼脂糖凝胶电泳分离, EB染色, 凝胶成像系统观察并拍照。
PCR产物经纯化后, 在全自动基因测序分析仪CEQ8000 (Beckman Coulter, US)上进行测序, 测序所用引物为扩增引物。
1.3 数据分析
利用Clustal X[15]程序对DNA序列进行多重对位排列并辅以手工校正。用 MEGA 4.0软件[16]分析不同序列间的碱基组成、变异位点、简约信息位点等, 并计算个体间的遗传距离, 构建 UPGMA与 NJ系统树。用DNASP4.0[17]计算单倍型多态性(h)和核苷酸多样性(π)等遗传多样性指数。使用 Arlequin 3.0[18]进行歧点分布分析(Mismatch distribution)[19]、Tajima检验(Dtest)[20]和 Fu 检验(Fsneutrality test)[21], 用以进行种群历史分析。
2 结果
2.1 D-loop片段的序列特征及其多态性
经电泳检测, PCR扩增产物为大小约为 500 bp的1条整齐而清晰的条带。本研究共测定了银鲳24个个体的mtDNA D-loop序列片段, 在除去引物及部分端部序列后, 经 BLAST对比, 获得银鲳线粒体D-loop序列5′端的长度为357 bp的片段。
A、T、C、G 4种碱基在24个个体中的平均比例分别为 40.1%、30.6%、16.7%、12.6%, AT比例(70.7%)高于 GC(29.3%), 符合脊椎动物 mtDNA D-loop区域碱基组成的特点。D-loop序列变异位点在银鲳群体中的分布如表1所示。357 bp长度的核苷酸序列中共检测到 14个变异位点, 其中 8个转换位点, 5个颠换位点, 1个转换与颠换同时存在的位点。

表1 变异位点在24条银鲳D-loop序列中的分布Tab. 1 Distribution of variable sites in 24 individuals of silver pomfret
2.2 群体的遗传多样性及其个体间的遗传关系
通过DNASP 4.0软件计算出这24个个体的遗传多样性参数为:多态位点数(S)为14, 单倍型个数(H)为 11, 单倍型多样性(h)为 0.89, 核苷酸多样性(π)为0.007, 平均核苷酸差异数(k)为2.57。由此可以看出,该群体银鲳具有较高的单倍型多样性, 但核苷酸多样性较低。
应用MEGA 4.0软件, 根据线粒体D-loop序列算出 24个个体间的 Kimura 2-parameter遗传距离,个体间的遗传距离在0.000~0.017之间, 其中遗传距离达到0.017的共有8组, 而遗传距离>0.01的至少有80组。以24个个体的D-loop序列构建的UPGMA系统树和NJ系统树分别如图1、图2所示。由图1和图2可以看出, 2种方法获得的系统树的拓扑结构基本一致, 24个个体均形成2大分支。
2.3 种群历史分析
歧点分布分析表明(图3), 歧点分布呈单峰状,图中曲线表示在突然扩张假设理论下歧点分布的模拟值, 同时无限突变位点模型的中性检验值Tajima’sD(-1.106,P= 0.138) 和Fs(-26.601,P= 0.000) 都是负值。结果提示东海银鲳群体在历史上可能经历过种群扩张事件。
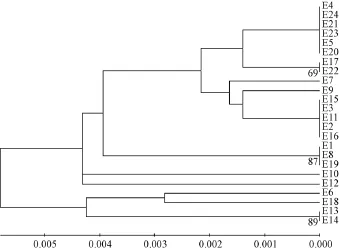
图1 由D-loop序列得到的UPGMA系统树Fig. 1 UPGMA phylogenetic tree based on D-loop sequences

图2 由D-loop序列得到的NJ系统树Fig. 2 NJ phylogenetic tree based on D-loop sequences
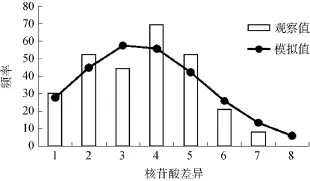
图3 东海银鲳线粒体DNA D-loop单倍型的歧点分布Fig. 3 Mismatch distribution based on control-region sequences of silver pomfret in the East China Sea
3 讨论
通常序列分析(测序)是最为有效的揭示群体遗传结构的方法之一[22]。线粒体DNA (mtDNA)具有分子小而稳定、母系遗传、进化速率快等特点, 成为群体遗传学的理想分子标记[12]。控制区(D-loop区)是mtDNA上的一段非编码区, 由于受选择压力小, 在进化过程中积累了较多变异, 因而被认为是线粒体基因组上进化最快的部分之一。本研究测序分析了东海野生银鲳群体线粒体D-loop区5′端的一部分片段, 以此研究分析东海银鲳群体的遗传多样性。
本研究结果显示, 东海银鲳群体具有较高的单倍型多样性(0.89)。已有的资料表明, 种群内能够维持较高单倍型多样性的原因可能在于较大的种群数量、环境的不均一性或者具有适应种群快速增长的生活特性[23]。对于海水鱼类讲, 较大的种群数量是维持较高遗传多样性的基础[24]。银鲳分布较广, 间接说明其具有较大的种群数量, 此可能是其维持较高遗传多样性的基础。本研究结果还显示, 东海银鲳群体虽具有较高的单倍型多样性, 但其核苷酸多样性较低(0.007)。根据不同单倍型多样性与核苷酸多样性间的组合, 海水鱼类大致可以分为4个类型:第一种类型是低的单倍型多样性与低的核苷酸多样性; 第二种类型是高的单倍型多样性与低的核苷酸多样性;第三种类型是低的单倍型多样性与高的核苷酸多样性; 第四种类型是高的单倍型多样性与高的核苷酸多样性[25]。本试验的结果为高的单倍型多样性与低的核苷酸多样性属于第二种类型。已有的研究表明,鲸鲨(Rhincodon typus)(h= 0.90,π= 0.005)[26], 尖吻鲭鲨(Isurus oxyrinchus)(h= 0.76,π= 0.0035), 西北太平洋毛鳞鱼(Mallotus villosus)、红拟石首鱼(Sciaenops ocellata)与西大西洋小沙丁鱼(Sardinella aurita) (h= 0.79~0.98, π= 0.0029~0.0068)均属于此种类型[25]。本研究中24个个体的D-loop序列所构建的UPGMA系统树和 NJ系统树表明, 两种方法所得到的系统树的拓扑结构基本一致, 24个个体均形成两大分支, 由于线粒体 DNA属于母系遗传, 由此可推断该群体的24个个体可能来源于两个不同的母系祖先。
通常有两种方法检验种群在历史上是否发生过种群扩张。一是采用Fu[21]的Fs中性检验显著偏离中性突变, 负的Fs值和差异显著的P值被认为种群在历史上有扩张的迹象。二是根据歧点分布曲线是否呈现多峰或单峰型。若核苷酸岐点分布曲线呈现单峰分布, 且中性检验值显著偏离中性, 则群体在过去可能经受了种群扩张[20]。本研究结果显示, 歧点分布呈单峰状, 且中性检验值 Tajima’sD为-1.106(P= 0.138),Fs为–26.601 (P= 0.000), 表明东海银鲳群体在历史上可能经历过种群扩张事件。
一个物种的遗传多样性高低与其适应能力、生存能力和进化潜力密切相关。丰富的遗传多样性意味着比较高的适应生存潜力, 蕴藏着比较大的进化潜能以及比较丰富的育种和遗传改良的潜力, 而贫乏的遗传多样性则会给物种生存、进化及种质资源的保护和利用带来许多不利影响[27]。本研究结果显示, 东海银鲳群体具有较高的单倍型多样性(0.89),遗传多样性较为丰富。近年来, 由于过度捕捞, 导致鱼体小型化、渔获物低龄化, 严重损害幼鱼资源, 造成产量不稳定[4]。从长远角度看, 过度捕捞势必破坏其遗传多样性和种质资源的稳定性。由于没有之前的相关数据, 本研究也无法判定东海银鲳遗传多样性水平是否由于近年来的过度捕捞而有所降低, 同样也无法确定东海银鲳遗传多样性水平受其影响的程度。然而, 值得注意的是, 不能因为目前相对丰富的遗传多样性水平而忽视对银鲳资源必要的保护与管理。目前应针对中国范围内不同群体银鲳资源的遗传多样性展开较为详细的研究工作, 以期为今后的定期遗传多样性检测奠定基础, 并根据其遗传多样性的变化趋势采取相应的保护措施, 科学的保护野生银鲳的种质资源, 使其资源能够达到可持续利用, 此也有助于推动渔业产业的长远发展。
本研究仅仅是针对舟山群岛附近海域银鲳资源遗传多样性的分析, 尚不能全面反映中国不同群体银鲳的遗传多样性水平与群体分化状态。今后应运用D-loop基因分析中国不同地区银鲳群体的遗传背景, 同时应选择 mtDNA其他不同的基因如Cytb、COⅠ等, 进一步比较研究银鲳群体内及群体间的遗传多样性, 以全面揭示银鲳资源的遗传多样性水平及其群体分化状况, 以期能够合理的开发利用我国的鲳鱼资源。
[1]Kagwade P V. Pomfret resources along north-west coast of India [A]. Bombay Research Centre CMFRI. Living Resources of the Indian Seas [C]. Bombay, India, 1988.219-224.
[2]Almatar S M, Al-Abdul-Elah K, Abu-Rezq T. Larval developmental stages of laboratory-reared silver pomfret,Pampus argenteus[J]. Ichthyological Research,2000, 47:137-141.
[3]Wen T Y, Jian L, Gen H Y. Multiplex genotyping of novel microsatellites from silver pomfret (Pampus argenteus) and cross-amplification in other pomfret species[J]. Molecular Ecology Notes, 2006, 6:1 073-1 075.
[4]郑元甲, 陈雪忠, 程家骅, 等. 东海大陆架生物资源与环境 [M]. 上海:上海科学技术出版社, 2003.379-400.
[5]柳卫海, 詹秉义. 东海区鲳鱼资源利用现状分析[J].湛江海洋大学学报, 1999, 19(1):30-34.
[6]何正侃, 孙振中, 洪波. 长江口南岸水域银鲳及幼鱼资源动态监测[J]. 水产科技情报, 2006, 33(2):81-83.
[7]龚启祥, 倪海儿, 李伦平, 等. 东海银鲳卵巢周年变化的组织学观察[J]. 水产学报, 1989, 13(4):316-325.
[8]倪海儿, 龚启祥. 东海银鲳个体生殖力的研究[J]. 浙江水产学院学报, 1995, 14(2):118-122.
[9]施兆鸿, 王建钢, 高露姣, 等. 繁殖生物学及人工繁育技术的研究进展[J]. 海洋渔业, 2005, 27(3):246-250.
[10]施兆鸿, 高露姣, 谢营梁, 等. 渔场银鲳和灰鲳繁殖特性的比较[J]. 水产学报, 2006, 30(5):647-652.
[11]施兆鸿, 马凌波, 高露姣, 等. 人工育苗条件下银鲳仔稚幼鱼摄食与生长特性[J]. 海洋水产研究, 2007,28(4):38-46.
[12]Aquadro C F, Greenberg B D. Human mitochondrial DNA variation and evolution:analysis of nucleotide sequences from seven individuals [J]. Genetics, 1983,103:287-312.
[13]Sambrook J, Fritsch E P, Maniatis T. Molecular Cloning:A Laboratory Manual [M]. NY:Cold Spring Harbor Laboratories Press, 1989.
[14]Kocher T D, Thomas W K, Meyer A,et al.Dynamics of mitochondrial DNA evolution in animals:amplification and sequencing with conserved primers [J]. Proc Natl Acad Sci, 1989, 86:6 196-6 200.
[15]Thompson J D, Gibson T J, Plewniak F,et al. The clustal–windows interface:Flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25:4 876-4 882.
[16]Tamura K, Dudley J, Nei M,et al.MEGA4:Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0 [J]. Molecular Biology and Evolution,2007, 24:1 596-1 599.
[17]Rozas J, Sanchez-Delbarrio C J, Messeguer X,et al.DnaSP, DNA polymorphism analyses by coalescent and other methods [J]. Bioinfomatics, 2003, 19:2 496-2 497.
[18]Excoffier L, Laval G, Schneider S. Arlequin ver. 3.0:An integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics Online,2005, 1:47-50.
[19]Rogers A R, Harpending H. Population growth makes waves in the distribution of pairwise genetic difference[J]. Molecular Biology and Evolution, 1992, 9:552- 569.
[20]Tajima F. Statistical methods for testing the neutral mutation hypothesis by DNA polymorphism [J]. Genetics, 1989, 123:585-595.
[21]Fu Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection [J]. Genetics, 1997, 147:915-925.
[22]Buonnacorsi V P, McDowell J R, Graves J E. Reconciling patterns of interocean molecular variance from four classes of molecular markers in blue marlin (Makaira nigricans) [J]. Mol Ecol, 2001, 10:1 179-1 196.
[23]Nei M. Molecular Evolutionary Genetics [M]. New York:Columbia University Press, 1987.
[24]Avise J. Phylogeography [M]. MA:Harvard University Press, Cambridge.1998.
[25]Grant W S, Bowen B W. Population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation [J].J Heredity, 1998, 89:415-426.
[26]Ramirez-Macias D, Vazquez-Juarez R, Galvan-Magana F,et al. Variations of the mitochondrial control region sequence in whale sharks (Rhincodon typus) from the Gulf of California, Mexico [J]. Fisheries Research,2007, 84:87-95.
[27]季维智, 宿兵. 遗传多样性研究的原理与方法[M].杭州:浙江科学技术出版社, 1999.
Genetic diversity analysis of silver pomfret (Pampus argenteus)in the East China Sea based on mtDNA D-loop sequence
PENG Shi-ming1, SHI Zhao-hong1, CHEN Chao2, HOU Jun-li1
(1.East China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Shanghai 200090, China;2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China)
Mar., 20, 2009
Pampus argenteus; East China Sea; mitochondrial DNA; D-loop sequence
Genetic diversity of silver pomfret (Pampus argenteus) in the East China Sea was investigated based on mtDNA D-loop sequences. The PCR technique was used to amplify the mtDNA D-loop in 24 individuals of silver pomfret collected from the East China Sea, near the coast waters of Zhoushan Islands. The PCR products were purified and sequenced. The results showed that 357bp nucleotide sequences of partial D-loop gene were obtained (the primer and some of the marginal sequences were excluded). Among the 24 individuals, 14 variation sites were observed, of which there were 8 transition sites, 5 transversion sites and 1 transition-transversion sites. The paiwise genetic distances were computed by MEGA software. The UPGMA and NJ phylogenetic trees were obtained through the cluster analysis over the pairwise genetic distance. The haplotype diversity (h), nucleotide diversity (π)and average number of pairwise nucleotide differences (k) were calculated by DNASP software, which were 0.89,0.007 and 2.57, respectively. Mismatch distribution analysis and Neutrality tests indicated a possible population expansion in silver pomfret population of the East China Sea. In conclusion, mtDNA D-loop may be one of the genetic markers available for scanning genetic diversity of intra-population and inter-populations of silver pomfret.
S968.3 文献标识码:A 文章编号:1000-3096(2010)02-0028-05
2009-03-20;
2009-07-03
中央级公益性科研院所基本科研业务费资助项目(东2008M14, 东2007Z02)
彭士明(1980-), 男, 山东东营人, 博士, 助理研究员, 研究方向:水生动物营养学与种质资源学, E-mail:shiming.peng@163.com;施兆鸿, 通信作者, E-mail:shizhh@sh163.net
康亦兼)

