基于微卫星技术的岩羊家域研究


关键词:岩羊;微卫星;家域;最小凸多边形法;贺兰山
家域(home range)是动物进行取食、交配和育幼等日常活动的区域,家域为野生动物提供各种必需的生存条件[1]。对野生动物的家域开展研究,有利于了解动物的资源需求、栖息地特征、社会组织结构等生态学和生物学信息[2−3],对于动物的生存和繁衍、保育和再引种具有重要意义[4−7]。野生动物家域的相关研究一直是国内外学者的研究重点[8−10]。自20世纪70年代起,我国开始重视野生动物家域的研究,主要集中在大熊猫(Ailuropoda melanoleuca)、川金丝猴(Rhinopithecus roxellana)、喜马拉雅小熊猫(Ailurus fulgens)、喜马拉雅扭角羚(Budorcas taxi⁃color)和麋鹿(Elaphurus davidianus)等珍稀濒危物种[11−12]。
微卫星(microsatellite)是以广泛分布于真核生物基因组的1 ~ 6个核苷酸为单位的短串联重复序列。微卫星具有核心序列多态性高、侧翼序列相对保守的特点,由于微卫星标记有丰富的多态性、较高的稳定性和易于检测分析等特点,在国内外广泛地应用于物种的遗传多样性检测、遗传图谱构建、群体遗传结构分析、个体识别、性别鉴定、亲缘鉴定以及种质资源保护等方面的研究[13−19],但结合微卫星技术用于物种家域的研究相对较少[2,20−22]。
岩羊(Pseudois nayaur)是国家二级重点保护野生动物,历史上岩羊曾遍布整个宁夏,而现在主要集中分布在宁夏贺兰山区域[23−24]。关于岩羊国内外已经开展了多方面研究,主要集中在种群结构、栖息地选择利用、食性、行为学以及遗传多样性等方面[18−19,25],而对家域研究较少[26],主要是因为贺兰山岩羊生存环境较为险峻,宏观手段研究其家域较为不易。为提升研究效率,验证微卫星技术应用于动物家域研究的可行性与准确性,本研究采用微卫星技术对贺兰山岩羊的家域大小进行研究,并与已有的研究成果比对,以期为岩羊的保护和管理提供更有效的手段。
1 研究区域
宁夏贺兰山国家级自然保护区(以下简称“保护区”)位于宁夏西北部(38°19′—39°22′ N,105°49′—106°41′ E),南北长170 km,东西宽20 ~ 40 km,保护区总面积为193 535. 68 hm2。保护区地处蒙古高原、黄土高原与青藏高原的交界地带,地跨温带草原与荒漠两大植被区域的交接处,是腾格里、毛乌素、乌兰布和三大沙漠的分界线,成为我国风沙干旱森林生态系统的典型代表地带。保护区独特的地理位置,复杂的地形组合,垂直分布明显的气候、土壤等自然因素,使保护区内保存着比较丰富的珍稀、濒危动植物物种,具有很强的特有性、典型性和珍稀性,具有重要的生态区位和特殊的保护价值。保护区目前记录到野生维管植物有84科329属647种,苔藓植物30科81属204种,大型真菌259种。贺兰山在动物地理区划上属于古北界中亚亚界蒙新区西部荒漠亚区和东部草原亚区的过渡地带,共有脊椎动物5纲24目56科139属218种,其中国家一级重点保护野生动物12 种,国家二级重点保护野生动物41种[27]。
2 研究方法
2. 1 粪便样品采集
采样区域位于保护区南端,主要为紫花沟、甘沟、驴路沟以及几条沟相交区域(图1)。岩羊粪便样品采样时间为2012年8月和9月,此时正是岩羊产仔季后期,雌雄个体还没有发情聚集,所以较适合研究各亚群岩羊个体的扩散情况。所选区域内各条沟段间为山体,样线沿着各沟段布设,采集沟底、山坡所遇岩羊粪便,并在采样处进行GPS定位,记录周围植被类型、距水源距离和人为干扰等生境因素。为了避免污染样品影响试验结果,在采样过程中戴口罩、一次性手套,并用已灭菌的牙签采集粪便样品。在粪便集堆处,随机选三四粒分装到不同采集管中,采集当天样品用无水乙醇浸泡,置于冰箱中-20 ℃保存,样品转移至实验室后,-80 ℃保存。为了不漏采和重复采样,采样时两人并行,每条样线只单向记录1次。为更好地获得遗传信息,采集当天的岩羊新鲜粪便,共计222份。
2. 2 岩羊粪便DNA 微卫星筛选
2. 2. 1 引物选择与合成
由于微卫星位点在近缘物种中具有保守性,所以本研究通过本实验室已有引物,查阅文献和公共数据库NCBI 得到36 个岩羊近缘物种的微卫星位点信息和引物序列。引物由生工生物工程(上海)股份有限公司合成,并用灭菌双蒸水配制成浓度为100 μmol/L的储存液备用。
2. 2. 2 筛选方法
微卫星位点的初步筛选采用PCR扩增的方法,先用36对引物分别对岩羊组织DNA进行扩增,扩增成功的引物再对粪便DNA进行扩增,最后扩增成功的引物再进行进一步筛选,筛选出多态性较好的引物,多肽信息含量(polymorphism information content,PIC) gt; 0. 50为高度多态性,0. 50 ≥ PIC ≥ 0. 25为中度多态性,PIC lt; 0. 25为低度多态性。
PCR 扩增采用15. 0 μL的反应体系:DNA 模板1. 5 μL、Premix Ex Taq 聚合酶(1. 25 U)7. 5 μL、BSA(20 mg/mL;TaKaRa,Otsu,Japan) 0. 5 μL、上下游引物(10 μmol/L)各1. 0 μL、双蒸水定容至15. 0 μL。PCR扩增程序:94 ℃预变性3 min;94 ℃变性30 s,退火30 s(温度因引物而异),72 ℃延伸45 s,35 个循环;最后72 ℃延伸20 min。在使用PCR扩增仪对各引物进行扩增时,设置梯度退火温度,以寻找各对引物的最适退火温度。PCR产物经2. 0%琼脂糖凝胶电泳检测,EB染色后,在凝胶成像系统中观察扩增效果,并拍照记录。为了确保结果准确可靠,对每个样本均重复扩增2次以上,统计2次扩增一致的条带。
采用毛细管电泳法确定位点的多态性。将初步筛选出的引物重新合成,在上游引物5′端分别标记三色荧光标记FAM、HEX和TAMARA。对222份粪便DNA样品重新扩增。取5 μL扩增产物进行2. 0%琼脂糖凝胶电泳(30 min),检验PCR反应是否成功。扩增后将PCR产物按比例混合成一组,每组10 ~ 15 μL,低温送至上海美吉生物医药科技有限公司测定等位基因的大小。等位基因结果用GeneMapper v. 4. 0软件(美国应用生物系统公司)分析。
在测得样品的基因型数据后,用MicrosatelliteTool kit程序来寻找数据中相匹配的基因型,并且判断不同的岩羊个体。
2. 3 牙釉蛋白基因PCR 扩增
2. 3. 1 样品
在采集的岩羊粪便样品中,选择至少扩增出一个微卫星位点的样品,采用牙釉蛋白基因PCR法进行性别鉴定。牙釉蛋白基因PCR扩增的阳性对照采用实验室已有的17份岩羊组织DNA样品(提取自野外自然死亡的岩羊),这17份样品分别于2011年采自宁夏贺兰山、西藏、青海、甘肃和新疆。另外,还有3份2013年采自上海动物园的岩羊粪便(已知性别)用来检验牙釉蛋白基因在岩羊粪便样品中的有效性。
2. 3. 2 PCR扩增
使用引物SE47(5′-CAGCCAAACCTCCCTCTGC-3′)和SE48(5′-CCCGCTTGGTCTTGTCTGTTGC-3′)扩增岩羊粪便DNA 和组织DNA,PCR 扩增采用15. 0 μL 的反应体系:DNA 模板1. 5 μL、Premix ExTaq 聚合酶(1. 25 U) 7. 5 μL、BSA (20 mg/mL; Ta‐KaRa,Otsu,Japan)0. 5 μL、上下游引物(10 μmol/L)各1. 0 μL、双蒸水定容至15. 0 μL。PCR扩增程序:94 ℃预变性5 min;95 ℃变性30 s,62 ℃退火30 s,72 ℃延伸45 s,35 个循环;最后72 ℃延伸7 min。PCR产物经2. 0%琼脂糖凝胶电泳检测(33 min),在凝胶成像系统中观察扩增效果,拍照记录。
牙釉蛋白法扩增雄性个体在进行电泳检测的时候会出现2条条带,而雌性只出现1条条带[28]。为了提高准确性,把222份岩羊粪便样品重复扩增6次,如果其中有2次出现2条条带,则判定该个体为雄性,否则为雌性。
2. 4 家域分析
根据个体识别和性别鉴定的结果,可以知道个体的重捕次数,选择重捕数大于4次的个体,根据同一个体不同位置的粪便,利用ArcGIS扩增模块homerange analysis 中的100% 最小凸多边形法(minimumconvex polygons,MCP)计算它们的家域面积。采用独立样本t 检验对雌雄家域的大小进行差异分析。
3 结果
3. 1 岩羊重捕个体筛选
通过9对微卫星引物对222份粪便DNA样品进行扩增后,共识别出岩羊个体183只,其中有93只雌性个体和90只雄性个体,雌雄比为93∶90=1. 03∶1。详细实验过程及结果见文献[18−19]。
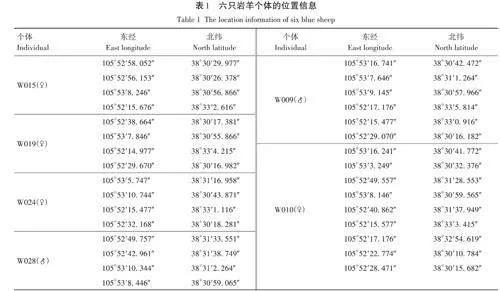
选出重捕数大于4次的6只个体来分析岩羊的家域范围,其中雌性个体4只(W010、W015、W019、W024),雄性个体2只(W009、W028),6只岩羊个体的位置信息见表1。
3. 2 岩羊个体家域
6只岩羊个体的重捕数为4 ~ 9次,平均重捕次数为5. 17次。采用MPC法计算岩羊个体家域面积(表2)。2只雄性(W009、W028)岩羊个体的家域面积分别为3. 23、0. 10 km2。4 只雌性(W010、W015、W019、W024)岩羊个体的家域面积分别为3. 57、1. 17、2. 72、2. 93 km2。6只岩羊个体的家域范围基本上全部重叠在一起(图2)。
对雌雄家域的大小进行差异分析(独立样本t 检验),结果显示,雌雄个体家域大小差异不显著(t =-0. 761,P gt; 0. 05)(表3)。
对重捕次数和面积关系进行分析,并绘制曲线图。由图3 可以看出,重捕次数多,面积也相应增加。
4 讨论
4. 1 微卫星技术用于家域分析
对动物家域进行估算的基础是收集其活动位点,而收集活动位点的方法越科学,家域面积估算的准确度则越高[29]。目前,收集动物活动位点的常用方法有直接观察法、无线电遥测、红外相机监测、项圈跟踪以及分子粪便学等[29−34]。其中无线电遥测法因贺兰山地势险峻应用比较困难,GPS项圈也因岩羊个体的生活环境不易被捕捉佩戴,红外相机监测法通过照片进行个体识别目前也存在一定困难,而且家域范围受相机放置位点影响较大。
基于粪便微卫星DNA分析技术进行野生动物家域研究是解决以上问题的有效手段[29]。国外科学家在20 世纪90 年代已经开始了一些研究,Kohnet al.[35]描述了家域的使用以及父权鉴定和亲缘关系鉴定都能用粪便基因型的分布和关系模式来推断;Taberlet et al.[36]和Smith et al.[37]分别用遗传学方法对棕熊(Ursus arctos)和胡安捷狐(Vulpes macro⁃tis mutica)的家域进行了研究,证明了方法的可靠性。近年来,国内研究人员也用粪便微卫星DNA分析技术对物种的家域进行了研究[2,20−22,38]。研究发现,微卫星技术可以应用于物种的家域分析,但是也同样存在着问题,如当样品量不足、重捕次数少时,有较多单独活动的个体家域面积会存在过小的现象[2];以及在粪便DNA质量比较差的情况下,错误的微卫星基因型会对个体鉴定结果造成严重的偏差[22]。本研究在采用微卫星技术对岩羊进行家域分析时,也同样发现了以上两个问题。此外,采样区域的地形地貌对采样的覆盖程度也影响较大,会间接影响分析出的家域面积的准确性。
4. 2 岩羊家域大小与影响因素
本研究选定6个重捕次数超过4次的个体作为研究岩羊个体家域的对象(4只雌性,2只雄性),雄性家域平均为1. 67 km2,雌性家域平均为2. 60 km2。其中除雄性个体W028家域面积偏小外,其余均与崔多英[26]研究所得的岩羊家域大小相符。本研究岩羊家域大小与其他有蹄类家域大小有所差别,东北马鹿(Cervus elaphus xanthopygus)雄性家域平均为2. 54 km2,雌性家域平均为1. 69 km2[39];梅花鹿(Cer⁃vus nippon)雄性家域为1. 93 km2,雌性为1. 16 km2[40];白尾鹿(Odocoileus virginianus)的家域为0. 50 km2[41];南汇东滩獐(Hydropotes inermis)的家域面积均值为6. 71 km2[42]。对比可知,岩羊家域与东北马鹿家域大小相差不大,相较梅花鹿和白尾鹿的家域偏大,较獐家域偏小。有蹄类动物的体型大小和栖息环境对其家域的大小有很大的影响,尤其是其中的食物资源影响更大,如草原鹿(Ozotoceros bezoarticus)的雌性家域为5. 9 km2,雄性家域为9. 9 km2,这与草原鹿所食食物营养含量低,需要采食更多食物才能满足需求有关[43]。贺兰山岩羊分布区域的植物种类较少,可利用低,岩羊需要寻找更多的食物资源来满足自身的需要,这就使得岩羊相比其他一些有蹄类家域要大。
除食物资源的分布与丰富度影响物种的活动与家域外,环境中其他资源不是广泛分布时,这种情况对家域的影响会更突出。Pages et al.[44]报道了食土习性对中缅灰叶猴(Trachypithecus melamera)活动范围的影响,由于食土地点不在正常的家域范围之内,所以致使猴群的活动范围由不采食土壤的73. 7 hm2增加到93. 4 hm2。本研究地点处于贺兰山较为干旱的区域,水资源较为稀缺,研究区域内有两处水源,分别位于图2中家域范围的最上顶点以及左下顶点,这也证明了水资源的分布会影响岩羊的家域范围。
4. 3 岩羊雌雄个体家域大小差异
本研究中雌雄岩羊家域面积差异不显著,从个体家域大小角度比较,雌性W010家域最大,这与其他有蹄类研究不同,在黑麂(Muntiacus crinifrons)、梅花鹿和马鹿等有蹄类的研究中,雄性的家域均大于雌性,原因是雄性个体需通过扩大家域面积来满足自身的需求[2,38,45−46],但因本研究数据量有限,且岩羊是偏雌扩散的物种[47],如果采样采集到扩散中的粪便样品,也会导致家域偏大,所以雌性家域是否偏大有待进一步验证。本研究6只岩羊个体家域有重叠,有研究表明岩羊是有家域但是没有领域性的动物[26],所以雌雄个体间存在家域重叠属于正常现象。
基于微卫星技术的动物家域研究,避免了对野生动物及其生存环境的干扰和破坏,这对于珍稀濒危动物的保护和管理非常重要。对于传统家域方法较难开展的研究,粪便分子生物学及微卫星技术是一种有效的研究手段。本研究在研究过程中还存在很多问题有待优化和解决,如用作家域分析的个体数较少,分析出的结果可能存在一定偏差;采样时间未覆盖全年,可能会导致分析结果未能完全反映实际家域大小;提取DNA对粪便样品要求较高,在采集样品时会有筛选舍弃,导致采样损失;无法保证所有个体的所有粪便均被采到,尽管已经对研究区域进行了仔细的搜寻,但难免会有遗漏,所以也可能导致结果有偏差,在结果分析中个体家域随着重捕次数增加而有所增大也说明了这点。这些是今后应用分子生物学手段研究物种家域需要去解决的问题。

