资源植物黑老虎的比较叶绿体基因组学研究

摘要:【目的】发掘我国重要资源植物黑老虎(Kadsura coccinea)的叶绿体基因组高变异区域及SSR变异位点,为其遗传多样性及种质资源评估奠定基础。【方法】获取5个黑老虎个体的叶绿体基因组,通过基因注释,采用生物信息学方法开展核苷酸多态性、简单重复序列(simple sequarce repeat,SSR)分析,并以2个五味子属植物为外类群分析南五味子属的系统发育关系。【结果】黑老虎完整叶绿体基因组为典型的四分体环状结构,长度为145 413~145 903 bp。其中,大单拷贝区的长度范围为94 457~94 757 bp,小单拷贝区的长度为18 032~18 047 bp,重复区为16 431~16 552 bp。黑老虎叶绿体基因组编码125个基因,蛋白编码基因、tRNA基因、rRNA基因分别为82、35、8个;总G(鸟嘌呤)和C(胞嘧啶)的碱基数占总碱基数的比例(GC占比)为39.7%。采用滑动窗口法分析发现黑老虎叶绿体基因组上petN-psbM和trnS-GCU-trnG-UCC具有很高的核苷酸多态性(Pi gt; 0.03)。该物种叶绿体基因组上共有212个SSR位点,其中24个SSR位点在5个黑老虎个体间存在多态变异,具有应用前景。系统发育分析表明,此次研究中黑老虎个体聚成一支,与同属其他物种关系较远。【结论】本研究首次系统比较了多个黑老虎个体的叶绿体基因组,发掘出基因组内的高变异区域及变异SSR位点,为其遗传多样性及种质资源评估奠定了基础。
关键词:黑老虎;质体基因组;遗传多样性;SSR位点;系统发育
中图分类号:S718;Q754"""" 文献标志码:A开放科学(资源服务)标识码(OSID):
文章编号:1000-2006(2024)06-0071-08
Comparative chloroplast genomics of the important resource plant Kadsura coccinea
ZHAI Xuechang1, PENG Li1, YAN Haifei2*, ZHU Kefan3, ZHANG Shuyan2,4, ZHANG Caiyun5, LU Xiankai2
(1. Jiangxi Environmental Engineering Vocational College, Ganzhou 341000, China; 2. Guangdong Provincial Key Laboratory of Applied Botany, South China Botanical Garden, Chinese Academy of Sciences, Guangzhou 510650, China;3. Xinfeng County Seed Farm of Jiangxi Province, Ganzhou 341624, China; 4. College of Life Sciences, South China Agricultural University, Guangzhou 510642, China; 5. Guangdong Food and Drug Vocational College, Guangzhou 510520, China)
Abstract: 【Objective】This study aims to investigate the chloroplast genomes and SSR loci of Kadsura coccinea, an important plant resource in China, to establish a basis for assessing its genetic diversity and germplasm resources. 【Method】The study"" obtained and analyzed the chloroplast genomes of five individuals of K. coccinea through genetical annotation, nucleotide polymorphism analysis, and SSR analysis using bioinformatics methods. Additionally, the phylogenetic"" analysis of Kadsura were reconstructed using two Schisandra spp. as outgroups. 【Result】The chloroplast genome of K. coccinea showed a typical quadripartite structure, with genome lengths ranging from 145 413 to 145 903 base pairs (bp). The large single-copy region (LSC) spaned from 94 457 to 94 757 bp, while the small single-copy region (SSC) encompassed 18 032 to 18 047 bp." It encodes a total of 125 genes, including 82 protein-coding genes, 35 tRNA genes, and 8 rRNA genes. The genome had a total GC content of 39.7% and demonstrated substantial nucleotide polymorphisms (Pi gt; 0.03) in the intergenic regions of petN-psbM and trnS-GCU-trnG-UCC. A total of 212 single sequence repeat (SSR) loci were identified across the chloroplast genomes of this species. Mononucleotide repeats were the most prevalent, followed by trinucleotide repeats, and pentanucleotide repeats were the least frequent. Among these loci, 24 polymorphic SSR loci were found among five individuals of K. coccinea, indicating their potential utility in future. Phylogenetic analysis robustly clusters K. coccinea individuals into a distinct group, revealing no close relationship with other congeneric species. 【Conclusion】This study presents the first systematic comparison of chloroplast genomes among multiple K. coccinea individuals. Our findings identified highly variable regions and SSR loci that can be valuable for evaluating the genetic diversity and germplasm resource of this species.
Keywords:Kadsura coccinea; plastid genome; genetic diversity; SSR loci; phylogeny
黑老虎(Kadsura coccinea)隶属于五味子科(Schisandraceae)南五味子属(Kadsura)[1],主要分布于我国长江以南,以及印度尼西亚、缅甸、越南、老挝和泰国等国家;生长于海拔400~1 900 m的山地疏林中[1-2],具有药用、食用、观赏等多种价值,是重要的资源植物[3-5],其果实富含多种氨基酸及微量元素,极具开发价值[6]。尽管黑老虎具有重要的应用价值和开发前景,但其种质资源方面的研究仍较薄弱[7-8]。近年来,我国有关黑老虎的遗传多样性研究取得了一些进展。例如,邹建文等[9]通过高通量测序开发了黑老虎的28对简单重复序列(simple sequence repeat,SSR)引物,并使用其中的17个SSR位点分析了湖南省内的11个黑老虎群体,发现了该物种群体内遗传变异小但群体间分化较大的特点,并建议对其加强就地和迁地保护[10]。Dong等[11]利用RAD-Seq对5个南五味子属的107个个体进行了分析,发现黑老虎与其他近缘种间具有较明显的遗传分化,且与其他种间无基因流。总之,对黑老虎的种质及遗传资源研究还不充分,不利于后续的资源保护及应用开发。
叶绿体基因组作为植物细胞内三大基因组之一,具有高拷贝数、保守的基因组结构和基因数量、基本上不发生基因重组、具适中的核苷酸替换率等特点,是研究植物遗传多样性及系统发育的重要材料[12-13]。高通量测序技术及生物信息工具的快速发展和迭代更新,促进植物叶绿体基因组的发表量快速增加[14]。虽然黑老虎的叶绿体基因组已有报道[15-18],但该物种在我国的分布区较广,对其叶绿体基因组的变异范围尚缺乏深入认识。此外,叶绿体基因组在绝大多数情况下都是母系遗传,通过黑老虎叶绿体基因组的变异研究有望还原出黑老虎的种子流(种子迁移)过程,为今后该植物的种源鉴定提供依据。因此,本研究新获取江西地区两个黑老虎个体的叶绿体基因组,并与已发表的叶绿体基因组进行比较,以期找出我国黑老虎叶绿体基因组的高变异区域及SSR变异位点,为其后续的遗传多样性及种质资源评估提供参考。
1 材料与方法
1.1 材料采集、DNA提取和测序
供试用黑老虎采自江西省赣州市信丰县崇仙乡吐前(121°53′09″E,41°44′46″N),海拔267 m。为评估该地区的黑老虎群体内是否存在叶绿体基因组遗传变异,研究采集了两个黑老虎个体。每个个体采集1片幼嫩叶片,经蒸馏水清洗擦干后用变色硅胶干燥保藏,其凭证标本保存于中国科学院华南植物园标本馆。使用CTAB法(cyltrimethylammonium bromide,十六烷基三甲基溴化铵法)[19]提取干燥叶片总DNA。使用Qubit荧光计和凝胶电泳评估总DNA的含量、纯度及完整性。获取的高质量DNA交由深圳华大基因股份有限公司(武汉)开展基因组浅层测序(每个样品获得约2 Gbp的数据)。
此外,从GenBank下载6个南五味子属物种的叶绿体基因组序列。其中,2个为已发表的黑老虎叶绿体基因组(来自福建的个体MN480469[18]和广西的个体MT934443[17])及来自云南的中泰南五味子(K. ananosma,NC_057265[16])。另外3个为南五味子属其他物种[异形南五味子(K. heteroclita),NC_057266[20],NC_050348[21];南五味子(K. longipedunculata),MW801021[22]]。从GenBank下载了华中五味子(Schisandra sphenanthera)和五味子(S. chinensis)的叶绿体基因组序列(GenBank序号分别为MK193856[23]和NC_034908[24])作为系统发育分析的外类群。
1.2 叶绿体基因组组装和注释
使用GetOrganelle 1.7.7软件[25]进行叶绿体基因组组装。使用GeSeq注释基因[26]。结合已发表的黑老虎叶绿体基因组序列,利用Geneious "2019 软件[27]辅助检查并进行必要的手动调整。采用OGDRAW 1.3.1[28]绘制基因组序列物理图谱。
1.3 叶绿体基因组基本信息统计
利用Geneious 2019软件统计8个南五味子属样品的叶绿体基因组序列总长度、重复区与单拷贝区的边界等信息。通过Geneious软件的MAFFT插件[29]分别对8个南五味子属和5个黑老虎个体的叶绿体基因组序列进行比对,获得比对文件。利用DnaSP 6软件[30],采用滑动窗口分析方法计算黑老虎叶绿体基因组的核苷酸多态性,其中窗口长度设置为500 bp,步长为200 bp。
1.4 叶绿体基因组简单重复序列分析
为分析黑老虎的叶绿体SSR位点,利用在线软件MISA[31]检索了5个黑老虎个体的叶绿体基因组的单核苷酸(mononucleotide)SSR、二核苷酸(dinucleotide)SSR、三核苷酸(trinucleotide)SSR、四核苷酸(tetranucleotide)SSR、五核苷酸(pentanucleotide)SSR以及六核苷酸(hexanucleotide)SSR。最小重复参数设置分别为10、5、4、3、3、3;2个SSR间的最小距离设置为100 bp。使用Geneious 2019中的插件Phobos(http://www.rub.de/ecoevo/cm/cm_phobos.htm)分析黑老虎不同个体间的变异SSR位点(polymorphic SSRs)。
1.5 系统发育分析
将8个南五味子属物种的叶绿体基因组及2个外类群物种[五味子(NC_034908[24])和华中五味子(MK193856[23])],提取所有样品共有的79个蛋白编码基因。以上序列通过MAFFT 7软件[29]比对并组合成串联矩阵。最大似然树(Maximum likelihood tree, ML tree)采用RAxML 8.2.10软件[32]构建。设置GTRGAMMA为核苷酸替代模型;重复自举抽样(bootstrap)1 000次计算分支支持率。
2 结果与分析
2.1 黑老虎叶绿体基因组结构和基因组成
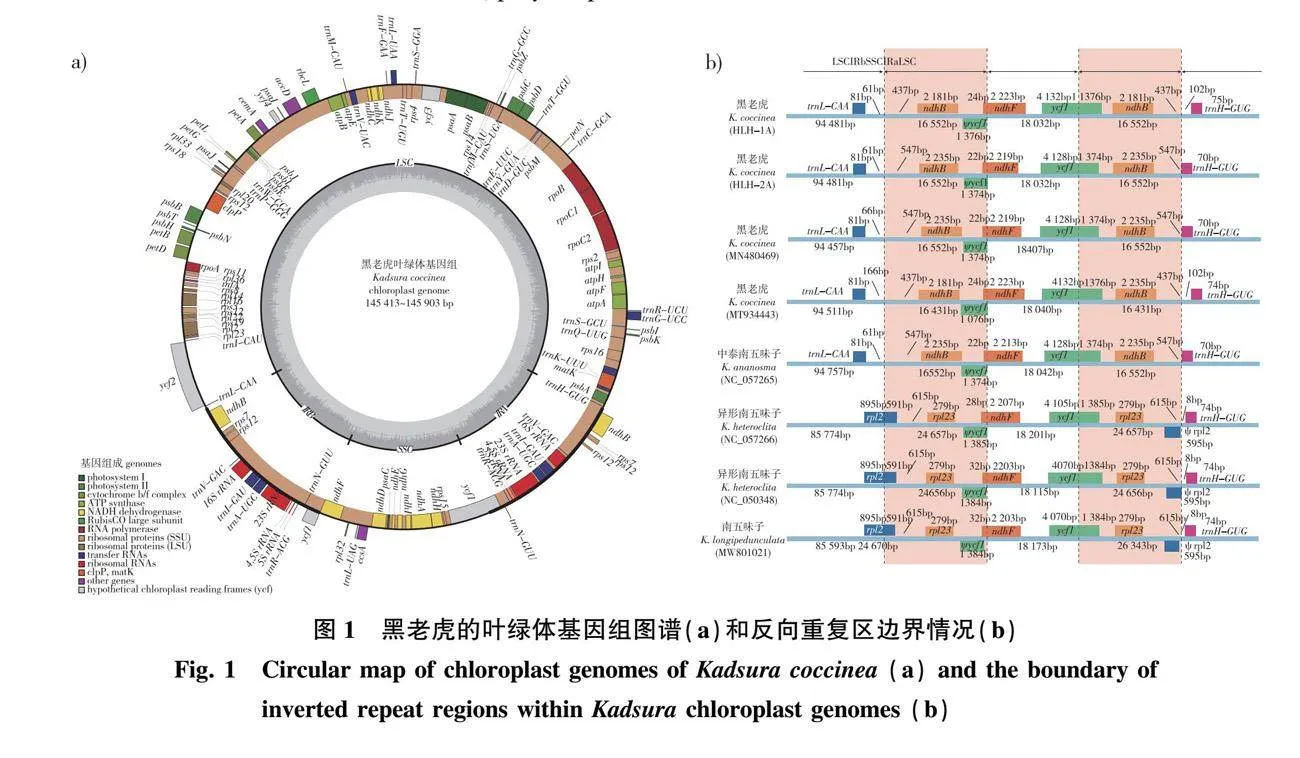
由于中泰南五味子在《中国植物志(英文版)》[1]中被归并到黑老虎中,并且本研究的系统发育结果也支持该分类修订。因此,本研究中有关黑老虎不同叶绿体基因组的比较分析均包含中泰南五味子的样品。黑老虎完整叶绿体基因组长度为145 413~145 903 bp,为典型的四分体环状结构(图1a)。
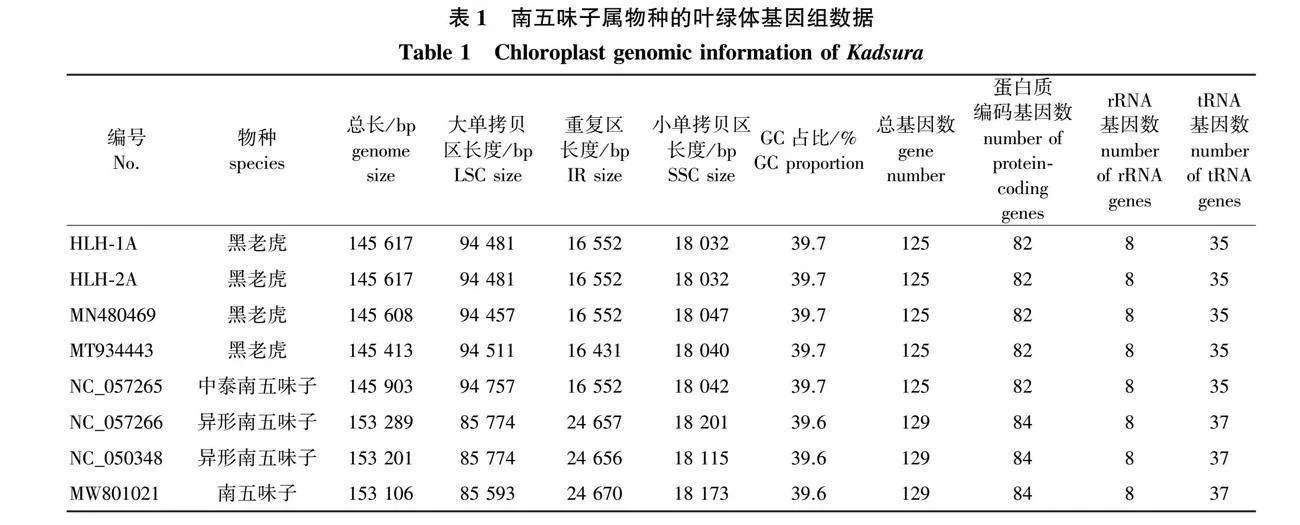
本研究新测序和组装了2个南五味子属黑老虎个体的叶绿体基因组,但2个基因组间不存在差异。为检验叶绿体基因组的组装结果是否准确,将原始测序数据比对到组装后的叶绿体基因组上,每个位点的测序深度平均为312.6次,说明这些叶绿体基因组序列是可靠的。将获得的两个黑老虎叶绿体基因组结合GenBank中6个已公开的南五味子属叶绿体基因组序列[16-18, 20-22]进行比对分析。其中,大单拷贝区(large single copy, LSC)的长度范围为94 457~94 757 bp,小单拷贝区(small single copy, SSC)的长度为18 032~18 047 bp,重复区(inverted repeat regions, IRs)长度为16 431~16 552 bp(表1)。黑老虎叶绿体基因组的总G(鸟嘌呤)和C(胞嘧啶)的碱基数占总碱基数的比例(GC占比)为39.7%(表1)。
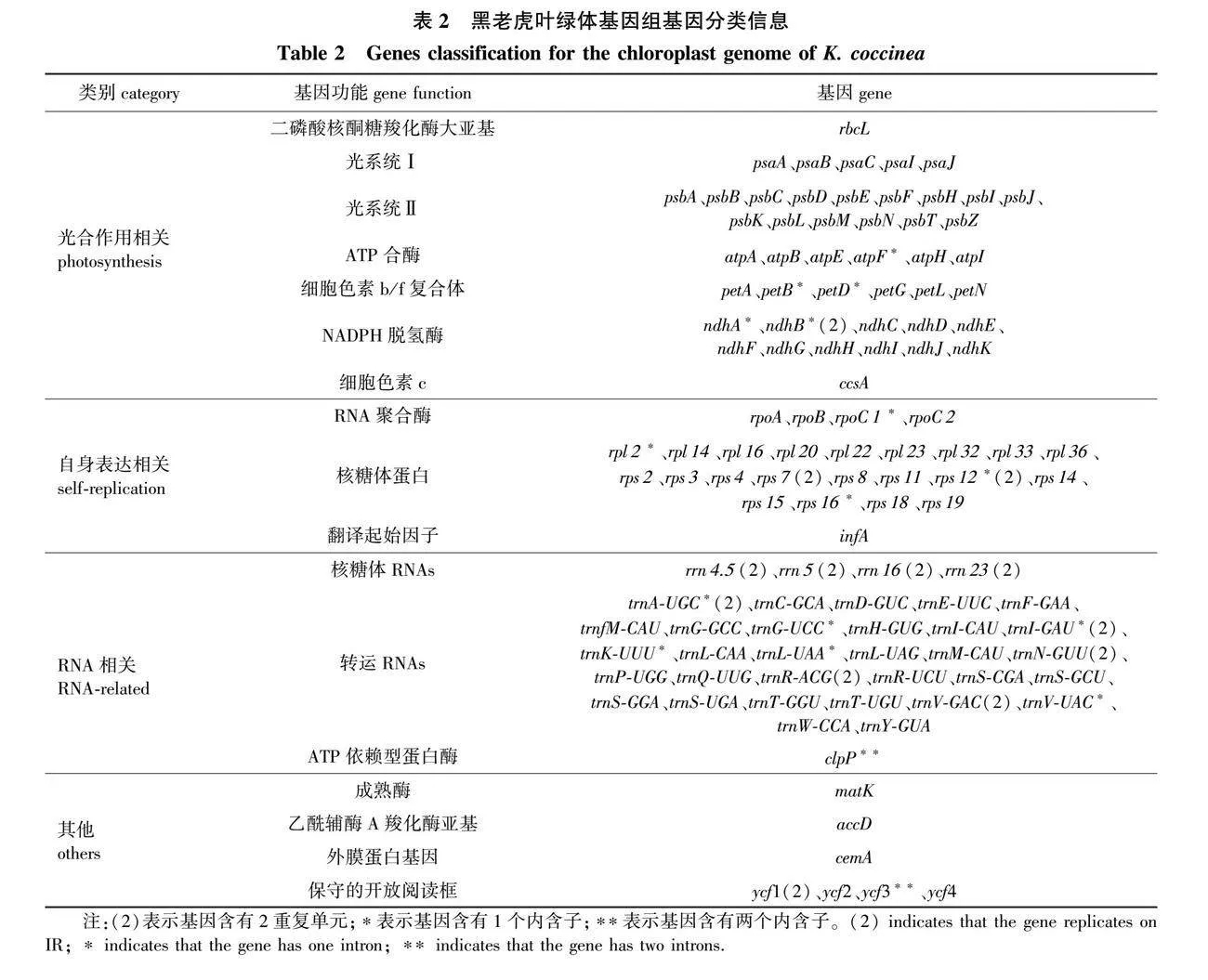
黑老虎叶绿体基因组编码125个基因,包含蛋白编码基因82个,tRNA基因和rRNA基因分别为35和8个(表1)。按照基因的功能分为4类(表2),其中15个基因含有1个内含子,分别是atpF、
petB、petD、ndhA、ndhB、rpoC1、rpl2、rps12、rps16、trnA-UGC、trnI-GAU、trnG-UCC、trnK-UUU、trnL-UAA、trnV-UAC。clpP和ycf3基因分别含有2个内含子。此外,有13个基因各有2个拷贝,分别分布在反向重复区上。叶绿体基因组重复区的边界分析表明,黑老虎5个叶绿体基因组(含中泰五味子)的重复区边界均较稳定(图1b)。重复区IRa与大单拷贝区的交界落在ndhB-trnH-GUG的间隔区内;与小单拷贝区的交界落在ycf1基因上。重复区IRb与大单拷贝区的交界落在trnL-CAA-ndhB的间隔区内,与小单拷贝区的交界落在ndhF基因上。
2.2 叶绿体基因组变异分析
采用滑动窗口分析方法计算南五味子属叶绿体基因组的核苷酸多态性,结果见图2,发现该属的核苷酸多态性(Pi)具有较大的变异范围:0~0.094 3,平均值为0.006 9。最高的Pi值出现在trnS-GCU-trnG-UCC基因间隔区内,为0.094 3。此外,psbI-trnG-UCC、trnG-UCC、petN-psbM均具有较高核苷酸多态性(Pi gt; 0.03)。本研究还对5个黑老虎的叶绿体基因组进行了核苷酸多态性(Pi)分析,发现petN-psbM和trnS-GCU-trnG-UCC具有很高的变异(Pi gt; 0.03),分别为:0.126 4和0.067 6。
2.3 叶绿体基因组SSR分析及系统发育分析
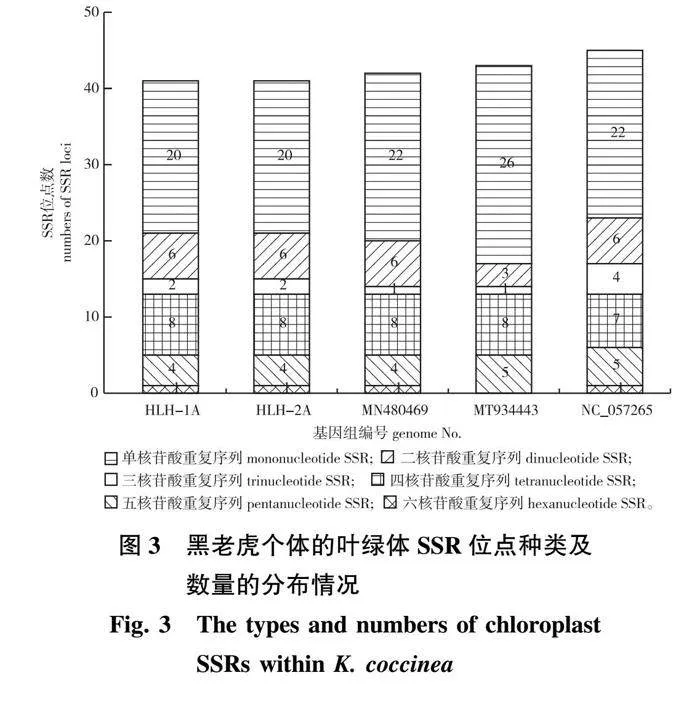
对5个黑老虎的叶绿体基因组简单重复序列(SSR)进行分析表明,黑老虎的叶绿体基因组共有212个SSR位点。其中,江西的两个黑老虎个体(HLH-1A和HLH-2A)分别有41个,福建的个体(MN480469)有42个,广西的个体(MT934443)有43个,云南的个体(NC_057265)有45个。每个样品的SSR种类的分布情况见图3。
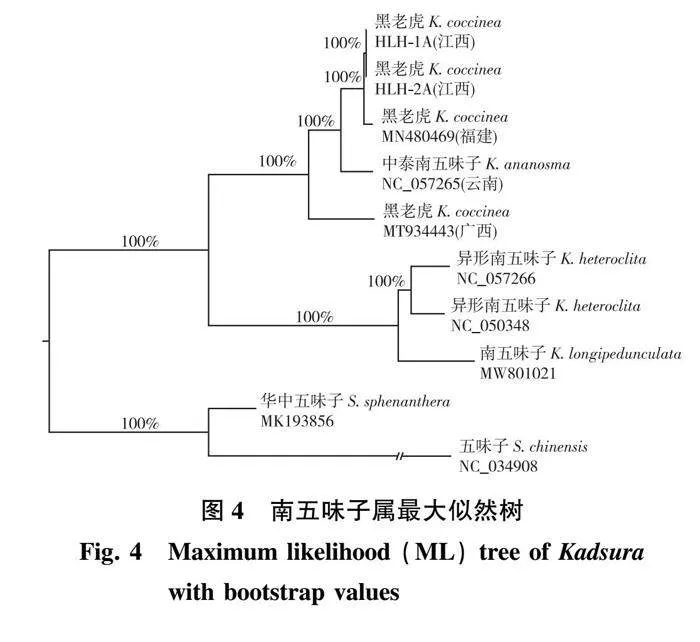
总体而言,单核苷酸重复序列最多,三碱基重复序列次之,五碱基重复序列最少(其中,广西的个体缺失了五碱基重复序列)。通过比对所有黑老虎叶绿体基因组间的SSR位点,共发现了24个SSR位点存在变异。选取8个南五味子属个体及2个外类群(五味子属)的叶绿体基因组序列进行系统发育分析。79个共有蛋白编码基因构建的最大似然树见图4,其所有分支节点的支持率均为100%。8个南五味子属的个体聚成一支,表明南五味子属是一单系群。4个黑老虎个体与中泰南五味子聚成一支,其中新增测的2个样品(来自江西赣州)聚在一起,再与来自福建的黑老虎个体聚成姐妹群,广西个体(MT934443)在黑老虎分支的最基部(图4)。
3 讨 论
叶绿体是植物细胞中非常重要的细胞器,对植物生长发育至关重要[33-34]。黑老虎的叶绿体基因组具典型的四分体环状结构,与绝大多数植物的叶绿体基因组结构一致[35]。此外,黑老虎叶绿体基因组的基因类型和数量也很稳定。通过系统比较南五味子属内及黑老虎的叶绿体基因组,得到以下4个发现:
1)叶绿体基因组数据表明中泰南五味子应归入黑老虎。南五味子属分为离蕊南五味子亚属(Kadsura subgen. Cosbaea)和南五味子亚属(Kadsura subgen. Kadsura)[36]。《中国植物志(中文版)》的离蕊南五味子亚属[37]包含中泰南五味子和黑老虎[30]。Smith[38]在1947年发表中泰南五味子的原文中承认该种与黑老虎形态上很相似,主要差别在于前者的叶片较薄、叶脉较密、雄蕊更多。然而,Saunders[36]对南五味子属进行了分类修订,发现中泰南五味子的形态均在黑老虎的形态变异范围内,因此在其1998年的专著中将中泰南五味子并入黑老虎。该观点与《中国植物志(英文版)》[1]及其他研究者的观点一致[39]。Liu等[40]的分子系统学研究虽包含中泰南五味子和黑老虎的样品,并证明彼此聚成一支,但由于各只有1个样品,故无法说明中泰南五味子与黑老虎间的依属关系。本研究证明中泰南五味子[15]与其他黑老虎多个个体的叶绿体基因组聚成一支,并“混在”黑老虎的分支内(图4),有力支持了Saunders[36]的分类处理观点。
2)黑老虎在南五味子属内具有比较独特的系统位置。黑老虎具有独特的雄蕊群特征,因此被认为是离蕊南五味子组(Sect. Cosbaea)或亚属(Subg. Cosbaea)的唯一成员[36]。本研究的叶绿体基因组系统发育关系也证明黑老虎与其他南五味子属类群分属两支,并与基于核基因ITS和叶绿体基因片段(如rbcL, matK, trnH-psbA)得到的系统发育关系一致[41-42]。此外,本研究通过叶绿体基因组比对,发现黑老虎具有独特的IR边界区的变异模式,与该属其他种存在明显区别,也显示出黑老虎具独特的系统地位。
3)黑老虎种下的叶绿体基因组具较高的遗传变异。一般而言,叶绿体基因组具有适中的核苷酸替换速率,且其序列演化速率在编码区和非编码区间存在较大差异,因此被用在不同分类阶元的系统发育研究中。例如:rbcL和atpB是早期分子系统学研究的主选基因片段[43-45],而非编码区(如trnL-trnF, trnH-psbA等)则因其较快的演化速率而被用于谱系地理学研究中。尽管已有大量高变异的叶绿体片段被开发出来[46-48],但不同类群的叶绿体基因组仍存在结构变异,故发掘出特定类群的特有高变异区域具有重要意义。与以往的研究类似[15],黑老虎叶绿体基因组的IR区域比大小单拷贝区保守。本研究进一步发现南五味子属及黑老虎叶绿体基因组上的热点变异出现在trnS-GCU-trnG-UCC和petN-psbM两个基因间隔区,为黑老虎的系统发育及谱系地理学研究奠定了基础。
4)SSR是遗传多样性、亲缘关系、遗传图谱构建等方面研究的重要分子标记[49]。尽管叶绿体SSR(chloroplast SSRs或cpSSRs)在数量及变异率等方面均小于核基因SSR,但仍具有独特优势。例如:叶绿体SSR缺乏重组,因而可用于谱系地理结构推断[49-50]。黑老虎叶绿体基因组共有212个SSR位点,并首次发现24个SSR位点在5个黑老虎个体间存在变异,这为后续遗传多样性分析提供了重要资料。
参考文献(reference):
[1]XIA N H, LIU Y H, SOUNDERS R M K. Schisandraceae[C]//WU Z Y, RAVEN P H, HONG D Y. Floral of China. Beijing: Science Press, 2008: 39-47.
[2]林祁,段林东,姚炳矾.南五味子属(五味子科)三种植物之补记[J].植物分类学报,2005,43(6):567-570.LIN Q,DUAN L D,YAO B F.Notes on three species of the genus Kadsura Juss.(Schisandraceae)[J].Acta Phytotaxon Sin,2005,43(6):567-570.
[3]黄珊珊,黄晓玲,宋卉,等.中药黑老虎的研究进展[J].海峡药学,2021,33(11):38-40.HUANG S S,HUANG X L,SONG H,et al.Advances in traditional Chinese medicine Kadsura coccinea[J].Strait Pharm J,2021,33(11):38-40.DOI: 10.3969/j.issn.1006-3765.2021.11.012.
[4]苏维,王欣悦,付港,等.南五味子属植物的化学成分、药理作用及临床应用研究进展[J].中国中药杂志,2024,49(1):26-38.SU W,WANG X Y,FU G,et al.Research progress on chemical constituents from Kadsura genus and its pharmacological activities and clinical application[J].China J Chin Mater Med,2024,49(1):26-38.DOI: 10.19540/j.cnki.cjcmm.20230718.201.
[5]杨赛男,戴斌,潘清平,等.黑老虎植物资源利用研究进展[J].湖南生态科学学报,2022,9(3):112-120.YANG S N,DAI B,PAN Q P,et al.Research progress on the application value and comprehensive utilization of Kadsura coccinea resources[J].J Hunan Ecol Sci,2022,9(3):112-120.DOI: 10.3969/j.issn.2095-7300.2022.03.015.
[6]杨芝干.地标奇果:通道“黑老虎”[J].生命世界,2019(9):66-69.YANG Z G.Landmark fruit-tongdao “Black Tiger”[J].Life World,2019(9):66-69.
[7]林旭俊,陆文,李善志,等.药用植物黑老虎的资源调查[J].热带林业,2019,47(2):34-36.LIN X J,LU W,LI S Z,et al.Investigation on the resources of medicinal plant Kadsura coccinea[J].Trop For,2019,47(2):34-36.DOI: 10.3969/j.issn.1672-0938.2019.02.009.
[8]韦霄,梁惠凌,唐辉,等.广西南五味子属植物的分布与利用[J].广西农业科学,2006,37(2):117-119.WEI X,LIANG H L,TANG H,et al.Distribution and utilization of Kadsura in Guangxi[J].Guangxi Agric Sci,2006,37(2):117-119.DOI: 10.3969/j.issn.2095-1191.2006.02.006.
[9]邹建文,罗先权,饶红欣,等.常绿木质藤本植物黑老虎基因组SSR特征分析及引物开发[J].中南林业科技大学学报,2021,41(4):130-138.ZOU J W,LUO X Q,RAO H X,et al.Characters of genomic SSRs and development of 28 SSR markers for Kadsura coccinea,an evergreen woody vine[J].J Cent South Univ For Technol,2021,41(4):130-138.DOI: 10.14067/j.cnki.1673-923x.2021.04.015.
[10]ZOU J W,HE R H,RAO H X,et al.Genetic diversity and population genetic structure in Kadsura coccinea (Schisandraceae),an evergreen woody vine from Hunan,China[J].J For Res,2023,28(5):364-373.DOI: 10.1080/13416979.2023.2220192.
[11]DONG Y Q,WEI X P,QIANG T Y,et al.RAD-Seq and ecological niche reveal genetic diversity,phylogeny,and geographic distribution of Kadsura interior and its closely related species[J].Front Plant Sci,2022,13:857016.DOI: 10.3389/fpls.2022.857016.
[12]赵儒楠,褚晓洁,刘维,等.鹅耳枥属树种叶绿体基因组结构及变异分析[J].南京林业大学学报(自然科学版),2021,45(2):25-34.ZHAO R N,CHU X J,LIU W,et al.Structure and variation analyses of chloroplast genomes in Carpinus[J].J Nanjing For Univ (Nat Sci Ed),2021,45(2):25-34.DOI: 10.12302/j.issn.1000-2006.202009007.
[13]袁钰晨,谢旭强,徐立清,等.不同年龄阶段胡桃楸天然更新幼树的光合生理特性[J].森林工程,2023,39(4):29-37. YUAN Y C, XIE X Q, XU L Q, et al. Photosynthetic physiological characteristics of naturally regenerated Juglans mandshurica saplings at different ages[J]. For Eng, 2023,39(4):29-37.
[14]WANG J,KAN S L,LIAO X Z,et al.Plant organellar genomes:much done,much more to do[J].Trends Plant Sci,2024,29(7):754-769.DOI: 10.1016/j.tplants.2023.12.014.
[15]邓叶,李翔,李平,等.黑老虎种质资源与分子生物学研究进展[J].湖南生态科学学报,2024,11(1):96-104.DENG Y,LI X,LI P,et al.Research progress on germplasm resources and molecular biology of Kadsura coccinea[J].J Hunan Ecol Sci,2024,11(1):96-104.DOI: 10.3969/j.issn.2095-7300.2024.01.012.
[16]LIU L Y,FU Y P,LI Y Q,et al.The complete chloroplast genome sequence of Kadsura ananosma[J].Mitochondrial DNA B Resour,2020,5(1):768-769.DOI: 10.1080/23802359.2020.1715866.
[17]QIN H Z,DENG L L,SHI Y C.Complete chloroplast genome of Kadsura coccinea (Lem.) A.C.Sm.(Schisandraceae):genome structure and evolution[J].Mitochondrial DNA B Resour,2021,6(3):1222-1223.DOI: 10.1080/23802359.2021.1904798.
[18]YANG J,WANG X A,GAO M Y,et al.The complete chloroplast genome of ‘black tiger 2’ (Kadsura coccinea (Lem.) A.C.Smith) in southeast of China and phylogenetic relationships[J].Mitochondrial DNA B Resour,2019,5(1):296-297.DOI: 10.1080/23802359.2019.1698328.
[19]DOYLE J J, DOYLE J L.A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J].Phytochem Bull,1987, 19:11-15
[20]WANG Q H,WANG H,FU Y P,et al.The complete chloroplast genome sequence of Kadsura heteroclita[J].Mitochondrial DNA B Resour,2020,5(3):2197-2198.DOI: 10.1080/23802359.2020.1768963.
[21]FU Y P,LI Y Q,CHEN W,et al.The complete chloroplast genome sequence of Kadsura interior[J].Mitochondrial DNA B Resour,2020,5(1):515-516.DOI: 10.1080/23802359.2019.1710297.
[22]JIN L,LIU J J,XIAO T W,et al.Plastome-based phylogeny improves community phylogenetics of subtropical forests in China[J].Mol Ecol Resour,2022,22(1):319-333.DOI: 10.1111/1755-0998.13462.
[23]WEI X P,LI H J,CHE P,et al.Comparing chloroplast genomes of traditional Chinese herbs Schisandra sphenanthera and S.chinensis[J].Chin Herb Med,2020,12(3):247-256.DOI: 10.1016/j.chmed.2019.09.009.
[24]GUO H J,LIU J S,LUO L,et al.Complete chloroplast genome sequences of Schisandra chinensis:genome structure,comparative analysis,and phylogenetic relationship of basal angiosperms[J].Sci China Life Sci,2017,60(11):1286-1290.DOI: 10.1007/s11427-017-9098-5.
[25]JIN J J,YU W B,YANG J B,et al.GetOrganelle:a fast and versatile toolkit for accurate de novo assembly of organelle genomes[J].Genome Biol,2020,21(1):241.DOI: 10.1186/s13059-020-02154-5.
[26]TILLICH M,LEHWARK P,PELLIZZER T,et al.GeSeq-versatile and accurate annotation of organelle genomes[J].Nucleic Acids Res,2017,45(W1):W6-W11.DOI: 10.1093/nar/gkx391.
[27]KEARSE M,MOIR R,WILSON A,et al.Geneious basic:an integrated and extendable desktop software platform for the organization and analysis of sequence data[J].Bioinformatics,2012,28(12):1647-1649.DOI: 10.1093/bioinformatics/bts199.
[28]LOHSE M,DRECHSEL O,BOCK R.OrganellarGenomeDRAW (OGDRAW):a tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes[J].Curr Genet,2007,52(5/6):267-274.DOI: 10.1007/s00294-007-0161-y.
[29]KATOH K,STANDLEY D M.MAFFT multiple sequence alignment software version 7:improvements in performance and usability[J].Mol Biol Evol,2013,30(4):772-780.DOI: 10.1093/molbev/mst010.
[30]ROZAS J,FERRER-MATA A,SNCHEZ-DELBARRIO J C, et al.DnaSP 6:DNA sequence polymorphism analysis of large data sets[J].Mol Biol Evol,2017,34(12):3299-3302.DOI: 10.1093/molbev/msx248.
[31]BEIER S,THIEL T,MNCH T,et al.MISA-web:a web server for microsatellite prediction[J].Bioinformatics,2017,33(16):2583-2585.DOI: 10.1093/bioinformatics/btx198.
[32]STAMATAKIS A.RAxML version 8:a tool for phylogenetic analysis and post-analysis of large phylogenies[J].Bioinformatics,2014,30(9):1312-1313.DOI: 10.1093/bioinformatics/btu033.
[33]GOULD S B, WALLER R R, MCFADDEN G I. Plastid evolution [J]. Annu Rev Plant Biol, 2008, 59: 491-517. DOI:10.1146/annurev.arplant.59.032607.092915
[34]杨圆圆,于世河,卜鹏图,等.不同培育模式下日本落叶松林灌草和土壤养分特征研究[J].森林工程,2023,39(6):12-25. YANG Y Y, YU S H, BU P T, et al. Study on shrub-grass and soil nutrient characteristics of Larix kaempferi forest under different cultivation modes[J]. For Eng, 2023, 39(6):12-25.
[35]JANSEN R K,RUHLMAN T A.Plastid genomes of seed plants[M]//BOCK R, KNOOP V. Advances in Photosynthesis and Respiration.Dordrecht:Springer Netherlands,2012:103-126.DOI: 10.1007/978-94-007-2920-9_5.
[36]SAUNDERS R M. Monograph of Kadsura (Schisandraceae) [C]// ANDERSON C. Systematic Botany Monographs. St. Louis: Missouri Botanical Garden Press, 1998.
[37]刘玉壶, 木兰科[C]//中国科学院中国植物志编辑委员会. 中国植物志. 北京: 科学出版社, 1996. LIU Y H.Magnoliaceae [C]//The Agenda of the Chinese Academy. Flora Reipublicae Popularis Sinicae. Beijing: Science Press,1996
[38]SMITH A C.The families Illiciaceae and Schisandraceae[J].Sargentia,1947,7:1-224.DOI: 10.5962/p.265318.
[39]毕海燕,林祁,刘长江,等.南五味子属(五味子科)的种子形态及其分类学意义[J].植物分类学报,2002,40(6):501-510.BI H Y,LIN Q,LIU C J,et al.Seed morphology of Kadsura Juss.(Schisandraceae) in relation to its taxonomic significance[J].Acta Phytotaxon Sin,2002,40(6):501-510.
[40]LIU Z,HAO G,LUO Y B,et al.Phylogeny and androecial evolution in Schisandraceae,inferred from sequences of nuclear ribosomal DNA ITS and chloroplast DNA trnL-F regions[J].Int J Plant Sci,2006,167(3):539-550.DOI: 10.1086/501476.
[41]GUO H J,LI X W,QI Y D,et al.Identification of Dian Ji Xue Teng (Kadsura interior) with DNA barcodes[J].World J Tradit Chin Med,2017,3(1):11-15.DOI: 10.15806/j.issn.2311-8571.2016.0017.
[42]HAO G,CHYE M L,SAUNDERS R M K.A phylogenetic analysis of the Schisandraceae based on morphology and nuclear ribosomal ITS sequences[J].Bot J Linn Soc,2001,135(4):401-411.DOI: 10.1006/bojl.2000.0420.
[43]CHASE M W,SOLTIS D E,OLMSTEAD R G,et al.Phylogenetics of seed plants:an analysis of nucleotide sequences from the plastid gene rbcL[J].Ann Mo Bot Gard,1993,80(3):528-580.DOI: 10.2307/2399846.
[44]QIU Y L,LEE J,BERNASCONI-QUADRONI F,et al.The earliest angiosperms:evidence from mitochondrial,plastid and nuclear genomes[J].Nature,1999,402(6760):404-407.DOI: 10.1038/46536.
[45]SOLTIS D E,SOLTIS P S,CHASE M W,et al.Angiosperm phylogeny inferred from 18S rDNA,rbcL,and atpB sequences[J].Bot J Linn Soc,2000,133(4):381-461.DOI: 10.1111/j.1095-8339.2000.tb01588.x.
[46]SHAW J,LICKEY E B,BECK J T,et al.The tortoise and the hare Ⅱ:relative utility of 21 noncoding chloroplast DNA sequences for phylogenetic analysis[J].Am J Bot,2005,92(1):142-166.DOI: 10.3732/ajb.92.1.142.
[47]SHAW J,LICKEY E B,SCHILLING E E,et al.Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms:the tortoise and the hare Ⅲ[J].Am J Bot,2007,94(3):275-288.DOI: 10.3732/ajb.94.3.275.
[48]SHAW J,SHAFER H L,LEONARD O R,et al.Chloroplast DNA sequence utility for the lowest phylogenetic and phylogeographic inferences in angiosperms:the tortoise and the hare Ⅳ[J].Am J Bot,2014,101(11):1987-2004.DOI: 10.3732/ajb.1400398.
[49]WEISING K.DNA fingerprinting in plants:principles,methods,and applications[M].2nd ed.Boca Raton,FL:Taylor amp; Francis Group,2005.
[50]YAN H F.Phylogeographic structure of Primula obconica (Primulaceae) inferred from chloroplast microsatellites (cpSSRs) markers[J].Acta Phytotaxon Sin,2007,45(4):488-496.DOI: 10.1360/aps06214.
(责任编辑 王国栋)

