基于改进U-Net的根系表型参数测量系统







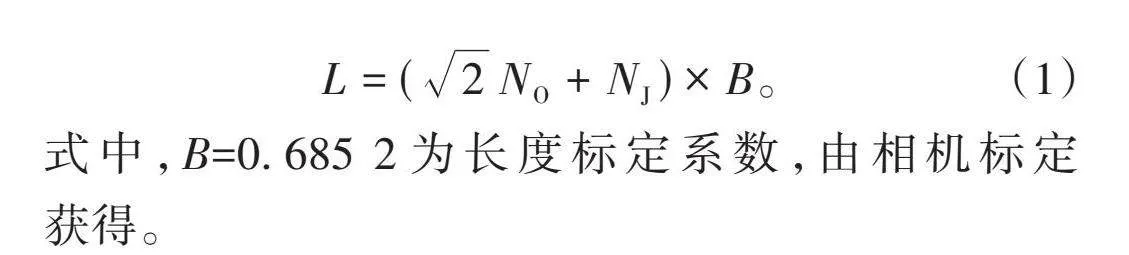



摘 要:为解决背景噪声干扰下,从微根管采集的原位根系图像中难以直接提取准确的表型参数问题,提出一种基于改进U-Net的微根管根系表型参数测量系统。在U-Net网络中引入优化后的空洞空间金字塔池化模块(Atrous Spatial Pyramid Pooling,ASPP)和高效通道注意力模块(Efficient Channel Attention,ECA),增大感受野,提升模型捕捉根系细节特征的能力,获取精确的根系分割图像。结果表明,改进的U-Net模型平均交并比和平均像素精度分别为87.07%和91.85%,相较原始U-Net分别提高了2.49%和2.3%。与WinRHIZO根系分析软件测量值相比,根长度和面积决定系数分别为0.951 8和0.984 9,Spearman相关系数分别为0.972 5和0.975 7,可以实现根系长度和面积的准确测量。
关键词:根系表型; 微根管; 图像分割; 参数测量; U-Net
中图分类号:TP391.4 文献标识码:A DOI:10.7525/j.issn.1006-8023.2024.04.014
Root Phenotypic Parameter Measurement System Based on Improved U-Net
ZHAO Yafeng, LIU Xiaolu, WANG Dongdong, WANG Mengxue, SONG Wenhua, HU Junfeng*
(College of Computer and Control Engineering, Northeast Forestry University, Harbin 150040, China)
Abstract: To address the challenge of accurately extracting phenotypic parameters from in situ root images collected from minirhizotrons amidst background noise interference, this paper proposes a minirhizotron root phenotypic parameter measurement system based on an improved U-Net model. In the U-Net network, optimized ASPP (Atrous Spatial Pyramid Pooling) and ECA (Efficient Channel Attention) modules are employed to increase the receptive field and enhance the ability to capture detailed features, thereby obtaining precise segmentation images. The experimental results show that the mean intersection over union and mean pixel accuracy of the improved U-Net model are 87.07% and 91.85%, which are 2.49% and 2.3% higher compared to the original U-Net, respectively. Compar3c7ba53c750c76d270ba8c53b6982a26ing with measurements obtained using WinRHIZO root analysis software, the determination coefficients for the root length and area are 0.951 8 and 0.984 9. respectively. The Spearman correlation coefficients are 0.972 5 for the root length and 0.975 7 for root area. This indicates the system′s capability to accurately measure the root length and area.
Keywords: Root phenotype; minirhizotron; image segmentation; parameter measurement; U-Net
0 引言
根系是植物的重要部分,不仅能够帮助植物抵抗自然灾害,同时也具有良好的土体锚固能力,在稳定土壤结构、防风固沙、防止水土流失方面具有重要作用[1-2]。研究根系表型参数对于深入了解植物的生长、改善农作物产量和进行生态研究等方面至关重要[3]。
由于根系生长环境的限制,根系表型无损检测一直是近年来的难点和重点。传统的方法主要依靠土壤挖掘法和洗根法观察根系结构,但是洗根法作为一种破坏性的取样方法,不仅费时费力,还可能导致侧根和根毛等精细的根系特征丢失,根系损失率较高[4]。气培法、水培法和凝胶培养法等无土培养方法也是一种高通量的植物根系表型观测方法,但这些无土培养方法影响了根毛的生长,对土壤中生长的植物的实际根系发育特征的表示有限[5]。并且上述方法仅适用于中小型植物,对于树木等具有复杂粗壮的根系植物来说操作难度大,且可实施性低。随着图像处理和计算机视觉技术的发展,研究者们开始探索利用数字图像获取和分析根系的信息。利用X射线[6]或计算机断层扫描(Computed Tomography,CT)技术[7]可以非破坏性地获取根系的内部结构信息,但实施困难、成本高。
微根管法是一种非破坏性且可以长期进行的观测方法,可以在植物的自然生长环境中进行。这种方法有助于保持植物根系的生理状态和环境适应能力[8],能够应用于包含林木在内的大多数植物的生长环境中,这有助于获得更真实和可靠的根系形态和功能信息,同时成本低,对根系生长影响更小,为研究植物根系的形态、功能、响应提供了有力的工具和手段,可实现高通量的根系表型无损检测。
目前关于高通量的根系表型信息提取的研究已经取得了一些进展。Smith等[9]证明了U-Net能够更精确地捕捉根系的轮廓和形状,为根系分割提供了一种有效的方法并实现了根长的测量。卢伟等[10]提出一种径向生长修复算法,解决了土壤遮挡导致的断根使表型参数获取不准确的问题。Narisetti等[11]提出在U-Net的每个卷积层后应用批量归一化并避免使用Dropout层,以及对卷积层的核大小进行调整,提高了根系图像分割的性能和连续性和根长、根表面积等参数提取的准确性。王传宇等[12]使用光盘作为标定物,在固定背景下结合像素分类背景分割方法实现玉米根系表型数据的提取。乌兰等[13]通过洗根法结合改进的DeepLabV3+网络采集并分割马铃薯根系图像,将直尺作为标定物实现了根系长度的测量。孙国祥等[14]使用阵列式相机结合根箱法采集番茄根系图像,通过引入多头自注意力机制的U-Net网络分割根系图像并实现番茄根系表型参数的测量。
为实现高通量的根系表型无损检测,采用微根管采集原位根系图像。由于植物根系分割过程繁琐且难度较大,提出一种改进的U-Net模型,引入高效通道注意力模块(Efficient Channel Attention,ECA)[15]以及优化后的空洞空间卷积池化金字塔模块(Atrous Spatial Pyramid Pooling,ASPP)[16],增强对细小根系的分割能力。为根长、根直径等根系表型参数提取提供更为有利的条件,有望推动植物根系研究领域的发展,并为解决重要的生物学和农业问题提供支持。
1 数据与方法
1.1 图像采集
本试验通过微根管、工业内窥镜、智能设备采集原位根系图像。选用直径为4 cm的透明PVC管作为微根管,并将其与被采集植物间隔25 cm,与地面呈45°的倾斜角度埋入地下,微根管两端使用黑色橡胶帽密封,防止灰尘的进入。相机选用最小焦距为2 cm的内窥镜,镜头直径为0.8 cm,配备有6颗可调节亮度的LED灯珠,方便相机在昏暗的地下环境工作。将镜头探入微根管内,利用智能设备上的inskam软件观察并获取原位根系图像,试验装置如图1所示。
通过原位根系图像采集装置采集了生长在多种土壤环境中的植物,包含生长在水淹土壤环境中的水稻,生长在黑钙土、砂壤土、黏壤土中的玉米,马齿苋、葱作为本试验的数据集,见表1。其中,水稻根系图像采集于黑龙江省哈尔滨市呼兰区,玉米根系图像采集于山东省济南市商河县,马齿苋和葱根系图像采集于山东省烟台市莱州市。每周采集一次根系数据,持续进行4周,图像分辨率为1 920×1 080像素。对数据集进行筛选及分类,去除模糊图像,最终保留220张原位根系图像,使用Labelme图像标记工具对获得的原位根系图像进行标注,根系像素标记为255,显示为白色;背景像素标记为0,显示为黑色,并将标注后的JAON源文件转换成png格式。
1.2 数据扩充与图像预处理
对采集到的根系图像进行筛选,去除不完整、模糊的图像,最终保留220张根系图像。为防止因数据集数量不足出现过拟合问题,本研究选取其中200张图像通过随机旋转、裁剪、加入高斯噪声的方法扩充数据集,最后得到2 000张根系图片,并按 9∶1的比例将其划分为训练集和验证集。剩余20张图片作为测试集。
由于根系生长环境的限制,采集到的根系图像往往存在亮度不足、对比度低等问题,同时,土壤中的根系背景复杂且存在与根系相似颜色的土壤颗粒,这些背景噪声的存在都对根系图像分割造成了干扰。为了减少以上背景噪声的干扰,本试验对扩充后的数据集进行预处理,其流程如图2所示。通过灰度变换、图像归一化、伽马校正、图像均衡化和限制对比度的自适应直方图均衡化(Contrast Limited Adaptive Histogram Equalization,CLAHE),有效提高了植物根系与土壤背景的对比度并减少了噪声干扰。
1.3 根系分割方法
1.3.1 改进的U-Net网络
作为语义分割领域的经典网络框架之一,U-Net首次提出便是为了解决医学领域中数据量匮乏这一问题,并通过数据扩充展示了其在小数据集训练方面的出众性能。考虑到植物根系数据集同样存在标注的样本数量较少的问题,本研究选择了在U-Net的基础上加以改进来实现植物原位根系图像分割,改进后的U-Net网络结构,如图3所示。U-Net网络包含编码器、解码器两大部分。编码器部分由卷积操作和下采样操作组成,在编码器末端,引入了空洞空间金字塔模块,增大感受野,捕获多尺度的上下文信息。解码器将深层的特征图与浅层的特征图进行上采样和融合,从而生成一个与原始输入图像相同大小的输出特征图,最后,将输出特征图通过高效通道注意力模块进行处理,以自适应地调整特征图的通道权重,增强对于根系特征的关注度,从而提高分割根系的性能。
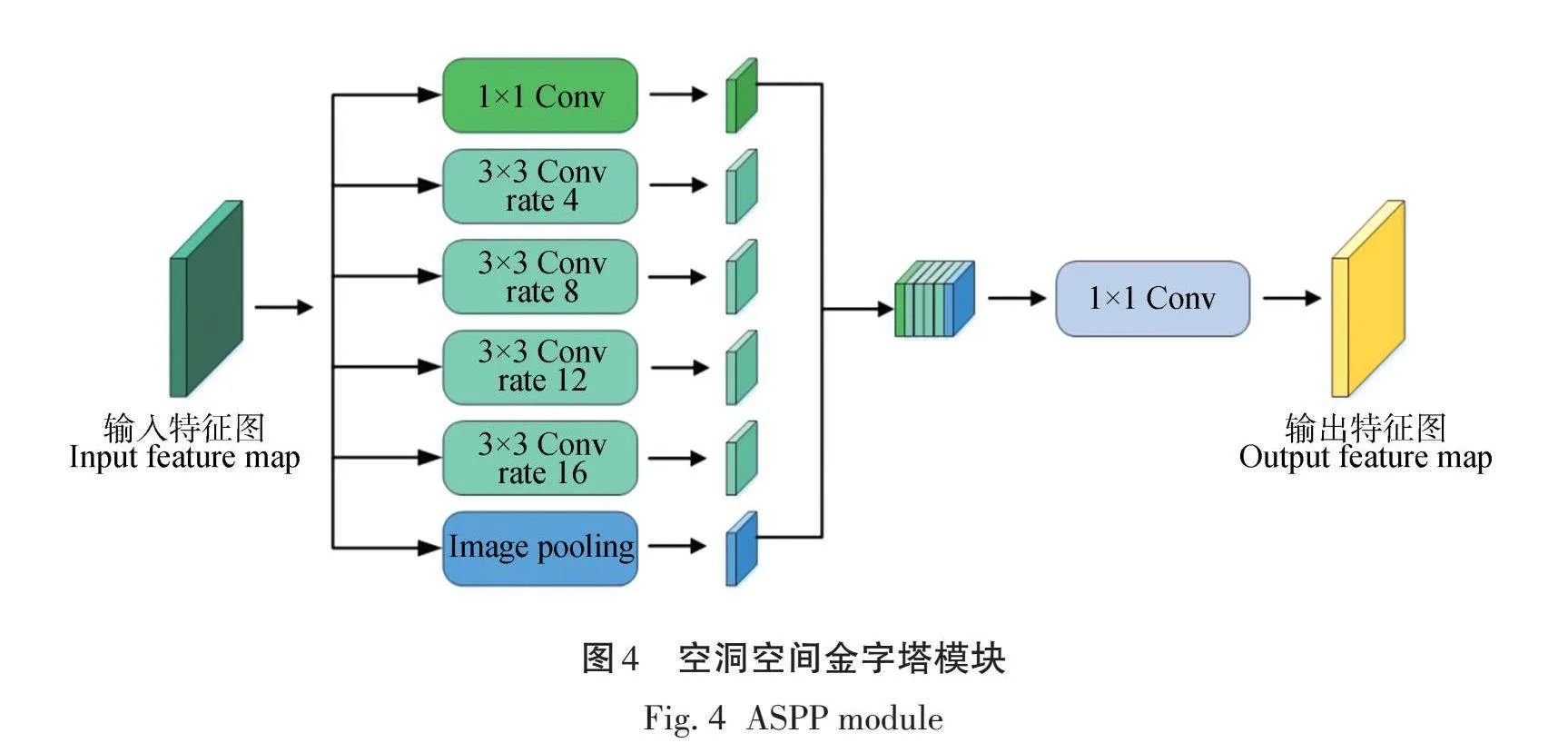
1.3.2 空洞空间金字塔模块
ASPP模块最初是在DeepLabv2中提出的,是空洞(Atrous)卷积和空间金字塔池化层(Spatial pyramid pooling)的结合。通过不同膨胀率的空洞卷积和池化,ASPP模块可以通过不同的感受野捕获特征,并避免出现传统卷积计算量大及分辨率损失的问题。植物根系拓扑结构复杂,根的粗细不一,且原位根系与土壤对比度较低。为更好地提取植物根系细节特征,增强模型对不同尺度根系的分割能力,本研究在ASPP模块上增加一层卷积层,并采用4、8、12、16作为膨胀系数。将优化后的ASPP模块嵌入到编码器末端,提高对不同尺度根系的感知能力,加强对根系细节特征的提取。ASPP结构如图4所示。
1.3.3 高效通道注意力模块
目前常用的注意力机制包含通道注意力、空间注意力、自注意力3种,通道注意力通过网络学习的方式自动获取每个特征通道的重要程度,然后再为每个通道赋予不同的权重系数,以此来达到强化重要特征、抑制非重要特征的目的。而ECA注意力模块去除了SE注意力机制(Squeeze and Excitation Networks)中的全连接层,将其改为1×1的卷积核,使用动态的卷积核来进行跨通道交互,卷积核的大小可通过函数自适应变化,解决输入特征图不同时提取不同范围的特征的问题。ECA模块可以不降维地实现局部交叉信道交互,在避免维度缩减的同时不增加模型的复杂度。本研究在U-Net模型的最终输出卷积层前引入了ECA模块,使模型在做出最后的分割决策前,更加关注植物根系中最为重要的特征,抑制无效的背景特征,减少土壤颗粒等背景噪声对根系分割的影响,提升模型的分割精度,增强其对不同图像的适应性。
1.3.4 评价指标
本研究使用准确性(Accuracy,ACC)、平均交并比(Mean intersection over union,MIou)、平均像素精度(Mean pixel accuracy,MPA)、F1分数这4个指标评估神经网络模型的性能。
1.4 原位根系表型参数测量
相机标定是图像测量中相当关键的环节,标定结果的精度直接影响了后续根系参数结果的准确度。本试验采用张正友棋盘格标定法对相机参数进行标定[17],这种标定方法介于传统标定法和自标定法之间,不仅克服了传统标定法需要的高精度标定物的缺点,也比自标定法具有更高的精度。选择行列数为6×8、每个方格长宽均为2.7 cm的棋盘格作为标定物,相机与棋盘格的间距为25 cm,相机姿态与实地采集根系图像时保持一致。使用根系图像采集设备完成相机标定,获得单位像素与实际距离之间的对应关系,即长度标定系数。
图像细化一般是二进制图像的骨架化(Image skeletonization)的一种操作运算,也是根系参数测量过程中不可或缺的步骤,本试验通过Zhang-Suen算法从分割后的根系图像中提取根系骨架。
Zhang-Suen算法的每一次迭代都要遍历所有不为零的像素,在对每个像素进行删除或保留的判断时都要关注其周围的8个像素的值,将图像线条从多像素宽度变为单像素宽度,从而在保留根系拓扑结构的基础上得到最接近根系中轴的骨架图,如图5所示。
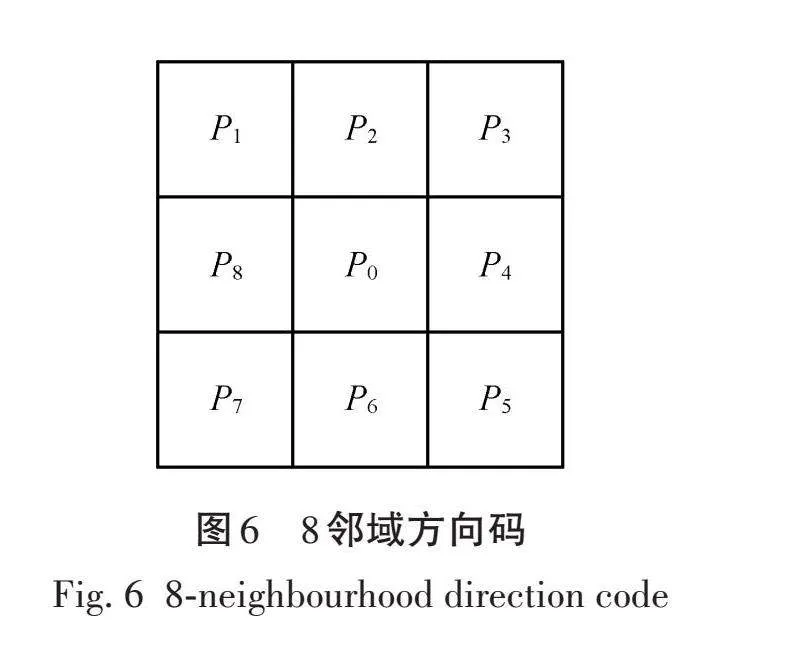
借助根系骨架图,在根上选取一点作为起始像素位置,对其周围8个邻域像素进行搜索并记录方向码,并不断重复这个过程。8邻域方向码如图6所示,假设中心像素P0与P2、P4、P6、P8之间的像素距离为1,与P1、P3、P5、P7之间的像素距离为√2,即若相邻两像素间的方向码为偶数,其像素距离为1;若相邻两像素间的方向码为奇数,其像素距离为√2。统计各像素间奇数方向码和偶数方向码出现的次数,并分别记作NO、NJ,由公式(1)计算得到根系长度L。
L=(√2 N_O+N_J)×B。 (1)
式中,B=0.685 2为长度标定系数,由相机标定获得。
分割后的根系图像上的每一个像素都代表根系的一部分,通过统计非零像素的数量可以得到根系面积为
S=N_1×B^2。 (2)
式中:S为根系面积,cm2;N1为二值图像中所有值为1的像素数;B为长度标定系数。
2 结果与分析
2.1 试验环境搭建
试验选用12核Intel(R)Xeon(R)Silver 4310 CPU,内存128 G,主频2.1 GHz,显卡为NVIDIA A100。使用Linux系统搭建基于Python3.7.16和Pytorch1.12.0的深度学习框架。在根系分割试验中,使用adam优化器,动量值为0.9,初始学习率设置为0.000 1,批次大小设置为8,训练迭代次数设置为100。
2.2 图像预处理结果与分析
将预处理操作前后的数据集分别记为D1和D2,改进后的U-Net模型在D1和D2数据集上的平均交并比曲线,如图7所示。对数据集进行预处理操作后,其平均交并比更高,根系分割效果更好。
2.3 根系分割试验结果与分析
使用自建的原位根系图像数据集对改进的U-Net模型进行训练和验证,为验证各模块对网络分割精度的影响,进行消融试验,并与经典网络进行对比。每5个epoch后进行一次评价指标和损失值的计算,经过100轮训练后,得到以下试验结果。
2.3.1 消融试验
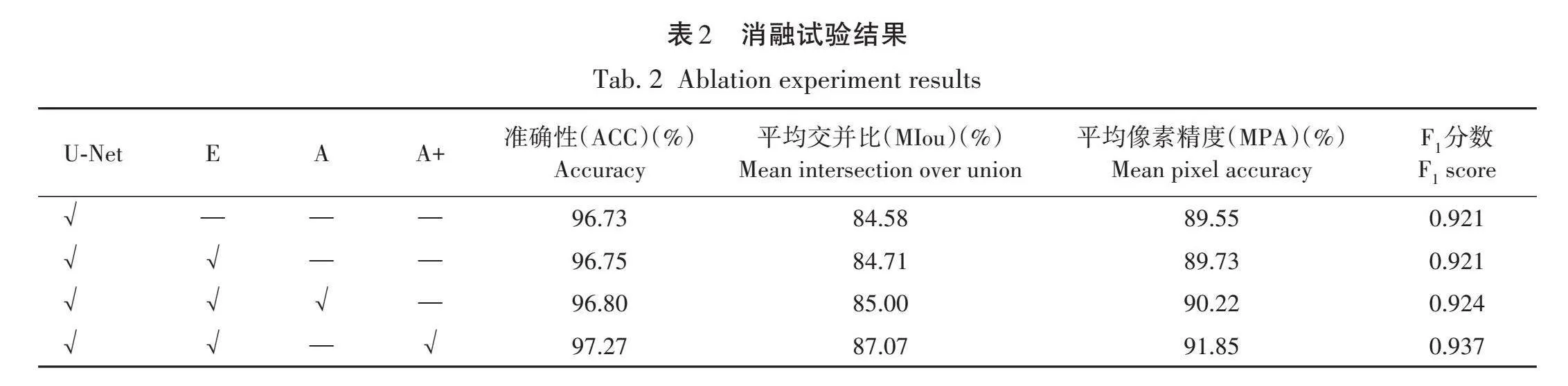
为验证引入的优化后ASPP模块和ECA模块对于改进的U-Net网络分割精度的影响,在本研究数据集上进行了消融试验。试验以原始U-Net网络作为基础网络,结果见表2。
表2中,√为添加的模块;E为在最后一层卷积前加入ECA注意力模块;A为在基础网络U-Net中引入原始ASPP模块;A+为引入优化的ASPP模块。试验结果表明,与U-Net网络进行对比,引入ECA注意力模块后,准确性、平均交并比、平均像素精度均有小幅度提升,说明ECA模块可以增强模型对更重要的根系特征的关注。在此基础上引入原始ASPP模块,除ACC、MIou、MPA这3项指标略有提升外,F1分数也有了提升;而引入优化的ASPP模块后,相较于引入原始ASPP模块,ACC、MIou、MPA以及F1分数分别提升了0.47%、2.07%、1.63%和0.013,说明引入优化后的ASPP模块增强了模型对多尺度根系的分割能力;改进后的U-Net模型相较于原始U-Net模型,ACC、MIou、MPA以及F1分数分别提升了0.54%、2.49%、2.3%和0.016,有了显著提升,这说明本研究引入的各个模块都能有效提高根系分割准确性和精度。
2.3.2 经典分割网络对比试验
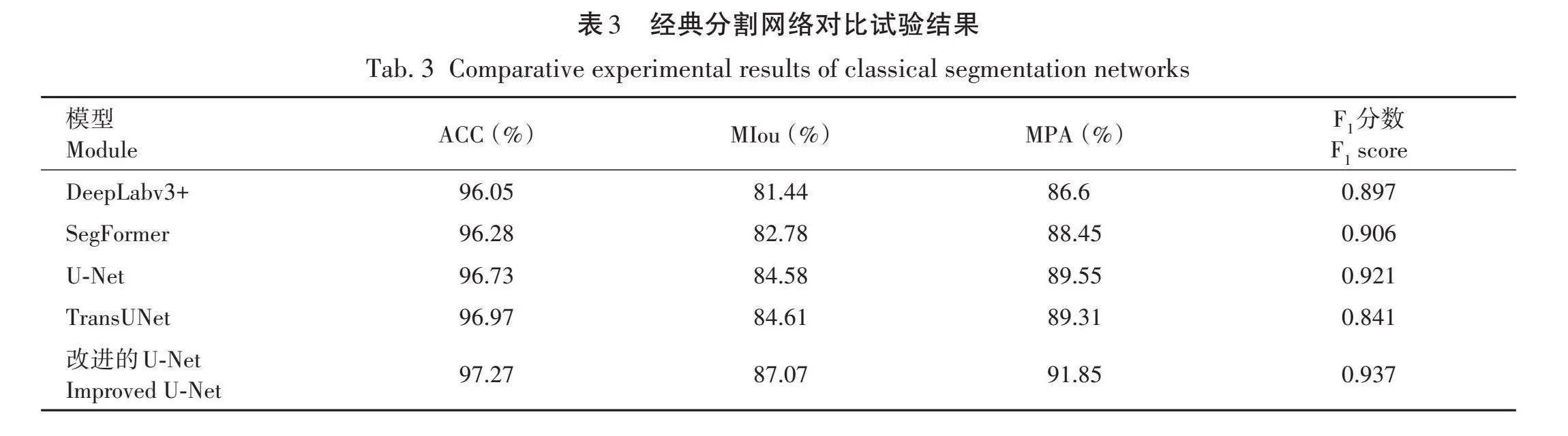
为进一步验证改进U-Net算法在根系图像分割中的效果,在本研究数据集上进行DeepLabv3+[18]、SegFormer[19]、U-Net[20]、TransUNet[21]和改进U-Net网络对比试验,试验结果见表3。
试验结果表明,改进U-Net模型的准确性、平均交并比、平均像素精度、F1分数均为最优值。改进的U-Net模型的ACC、MIou、MPA、F1分数分别达到97.27%、87.07%、91.85%、0.937,较原始U-Net模型均有提升,表明了改进U-Net模型具有较高的分割精度。
准确性和平均交并比较高的是TransUNet模型,分别为96.97%和84.61%,但TransUNet模型的平均像素精度以及F1分数没有U-Net模型高。为更清晰直观地比较各个经典语义分割模型以及改进U-Net模型的分割效果,将不同方法的分割效果进行可视化,如图8所示,红色虚线框标出了不同网络模型在根系分割结果中存在差异的区域。
由图8第1列图像可以看出,对于清晰明显的根系图像,改进U-Net、TransUNet以及U-Net分割效果较好,SegFormer和DeepLabV3+在根交叉处分割效果较差,无法被交叉遮挡的根系连通起来。由图8第2列图像和图8第3列图像可以看出,对于分根的分割效果,本研究提出的网络分割效果明显优于其他网络,不仅可以完整分割出主根和分根,而且能完成连通的根系末端分割,保留了根系的完整结构。图8第4列图像展示了本研究提出的网络对于细小根系的分割能力,ASPP模块和ECA注意力模块的加入增强了模型对根系特征的提取能力,相较其他4个网络(原始U-Net、DeeplabV3、SegFormer、TransUNet)能更好地分割出细小的根系,确保了根系分割的完整性和准确性。
2.3.3 根系分割网络对比
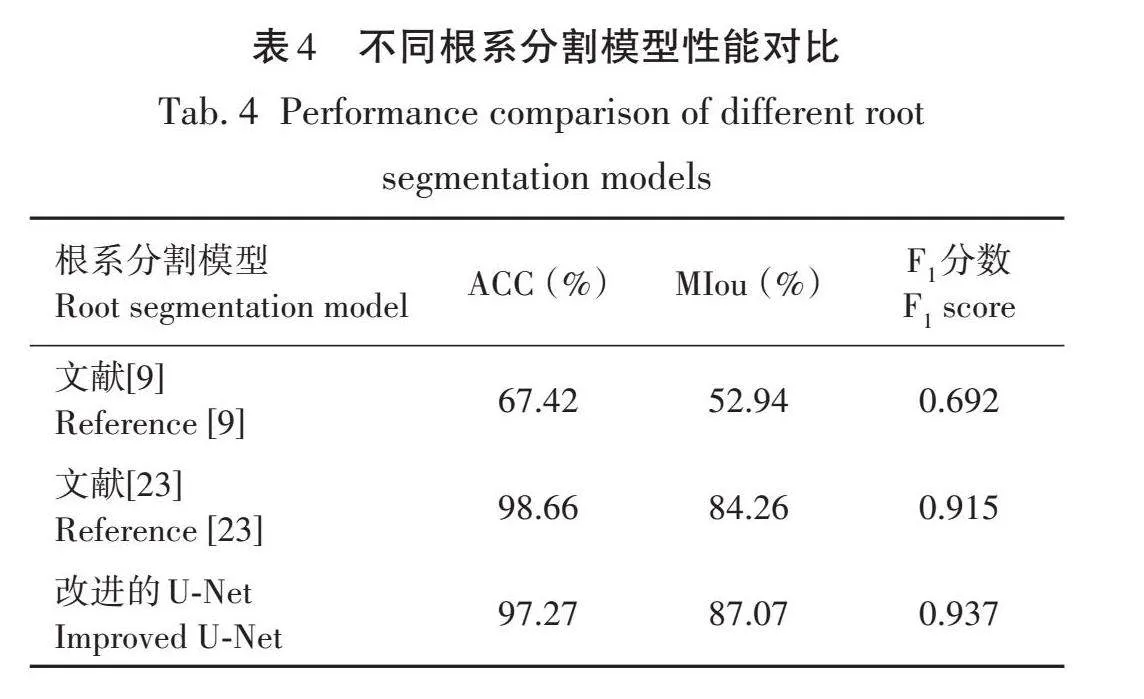
将改进U-Net模型与其他根系分割模型进行对比,各项性能指标见表4。
由表4可知,改进的U-Net模型,相比于文献[23],平均交并比和F1值分别提高了2.81%、0.022;相较于文献[9],准确性提升了29.85%,平均交并比提高了34.13%,F1分数提高了0.245,进一步证明了本模型具有较高的分割精度。
2.4 原位根系表型参数测量
为验证本研究提出的原位根系表型参数测量结果的准确性,使用WinRHIZO根系分析软件[22]提取根系参数作为对照组,使用Spearman相关系数,决定系数(R2)和均方根误差(RMSE)对试验结果进行评估。Spearman相关系数是利用单调方程衡量2个变量相关性的非参数指标,其绝对值越接近1,2个统计变量的相关性越强。R2越接近1,模型的拟合效果越好。RMSE表示本研究方法测量值和WinRHIZO软件测量值之间的偏离程度,RMSE值越小表示本研究方法的参数测量精度越高。
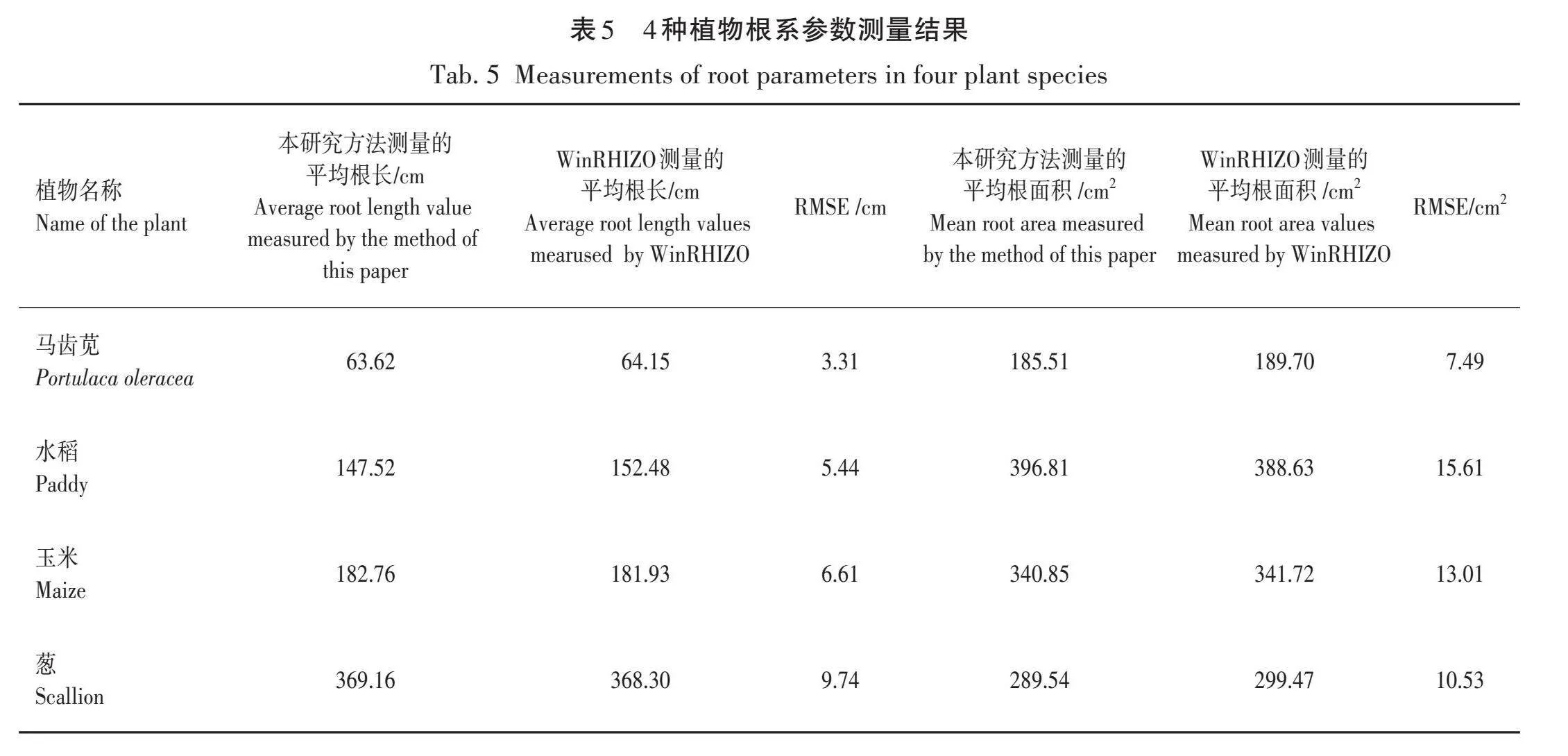
选取50张原位根系图像进行参数提取试验,利用回归分析方法对2种方法获得的测量结果进行线性回归分析,如图9所示,根系长度和根系面积的决定系数分别达到0.951 8和0.984 9,Spearman相关系数分别为0.972 5和0.975 7,表明本研究方法的测量结果与WinRHIZO软件的测量结果具有较好的相关性,证明了本系统的可信度。
对本试验中的4种植物各选择10张图片,通过对WinRHIZO根系分析软件得到的表型参数与本研究提出的测量方法得到的表型参数进行比较,测量结果见表5。由表5可以发现,根系长度和根系面积越小,2种方法得到的测量值之间的偏离程度越小,精确度越高。
3 结论
针对植物原位根系难以观察测量的问题fd731d3a7e2d20e99c3dff7b272f7dec,提出一种基于改进U-Net的微根管根系测量系统,主要结论如下。
1)在U-Net网络中引入优化后的ASPP模块和ECA模块后,有效提升了模型对粗细不一的根系的分割效果,同时加强了模型对根系细节特征的提取能力。改进U-Net模型的准确性、平均交并比、平均像素精度以及F1分数比改进前的U-Net模型分别提高了0.54%、2.49%、2.3%和0.016。试验结果表明,改进U-Net模型对根系分割的效果优于其他分割模型,特别是在细小根系的分割能力上表现优秀,可以完整准确地分割出植物根系。
2)利用根系分割图像,结合相机标定及图像细化算法,获得了原位根系长度和面积。分别使用本研究方法和WinRHIZO根系测量软件进行根系长度和面积测量,二者决定系数分别达到了0.951 8和0.984 9,Spearman相关系数分别为0.972 5和0.975 7,证明了本系统提取的根系表型参数具有较高可信度。
但由于植物根系易被土壤遮挡,完整的根系结构难以获取。此外,受限于植物根系图像的多样性和复杂性,部分植物根系分布较为离散,难以找到通用的最小长度参数。未来将进一步探索如何获得完整的植物根系,并计划探索最佳阈值,以便去除图像中的杂散点,提高根系表型参数提取的精确度。
【参 考 文 献】
[1] 刘定辉,李勇.植物根系提高土壤抗侵蚀性机理研究[J].水土保持学报,2003(3):34-37,117.
LIU D H,LI Y.Mechanism of plant roots improving resistance of soil to concentrated flow erosion[J].Journal of Soil and Water Conservation,2003(3):34-37,117.
[2] 李强,杨俊诚,张加琼,等.植物根系抗侵蚀指标及模型研究进展[J].农业资源与环境学报,2020,37(1):17-23.
LI Q,YANG J C,ZHANG J Q,et al.Progress of research on soil erosion resistance of plant roots and future prospects[J].Journal of Agriculture Resources and Environment,2020,37(1):17-23.
[3] LYNCH J P.Root biology in the 21st century:challenges and opportunities[J].Annals of Botany,2021,128(1):i-ii.
[4] LI A,ZHU L,XU W,et al.Recent advances in methods for in situ root phenotyping[J].PeerJ,2022,10:e13638.
[5] CAI J,ZENG Z,CONNOR J N,et al.RootGraph:a graphic optimization tool for automated image analysis of plant roots[J].Journal of Experimental Botany,2015,66(21):6551-6562.
[6] HEERAMAN D A,HOPMANS J W,CLAUSNITZER V.Three dimensional imaging of plant roots in situ with X-ray computed tomography[J].Plant and soil,1997,189:167-179.
[7] 李克新,李沐阳,薛瑞,等.林木幼苗根系CT序列图像分割[J].森林工程,2014,30(1):25-29.
LI K X,LI M Y,XUE R,et al.CT slice image segmentation of the seedling roots[J].Forest Engineering,2014,30(1):25-29.
[8] BAUER F M,LARM L,MORANDAGE S,et al.Development and validation of a deep learning based automated minirhizotron image analysis pipeline[J].Plant Phenomics,2022:9758532.
[9] SMITH A G,PETERSEN J,SELVAN R,et al.Segmentation of roots in soil with U-Net[J].Plant Methods,2020,16(1):1-15.
[10] 卢伟,邵昱宁,王玲,等.利用径向生长修复算法检测玉米根系表型[J].农业工程学报,2021,37(18):195-202.
LU W,SHAO Y N,WANG L,et al.Radial growth repair algorithm for maize root phenotype detection[J].Transactions of the Chinese Society of Agricultural Engineering,2021,37(18):195-202.
[11] NARISETTI N,HENKE M,SEILER C,et al.Fully-automated root image analysis(faRIA)[J].Scientific Reports,2021,11(1):16047.
[12] 王传宇,郭新宇,温维亮,等.基于玉米根系图像的表型指标获取方法[J].农业工程学报,2021,37(8):169-176.
WANG C Y,GUO X Y,WEN W L,et al.Phenotyping index acquisition method based on maize root images[J].Transactions of the Chinese Society of Agricultural Engineering,2021,37(8):169-176.
[13] 乌兰,苏力德,贾立国,等.基于改进DeepLabv3+网络的马铃薯根系图像分割方法[J].农业工程学报,2023,39(3):134-144.
WU L,SU L D,JIA L G,et al.Image segmentation of potato roots using an improved DeepLabv3+ network[J].Transactions of the Chinese Society of Agricultural Engineering,2023,39(3):134-144.
[14] 孙国祥,蔡嘉奇,周新竹,等.基于阵列式ESP32-CAM的番茄根系表型原位测量方法[J].农业工程学报,2023,39(18):172-182.
SUN G X,CAI J Q,ZHOU X Z,et al.In-situ measuring tomato root phenotype using array ESP32-CAM[J].Transactions of the Chinese Society of Agricultural Engineering,2023,39(18):172-182.
[15] WANG Q,WU B,ZHU P,et al.ECA-Net:Efficient channel attention for deep convolutional neural networks[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition.2020:11534-11542.
[16] CHEN L C,PAPANDREOU G,KOKKINOS I,et al.Deeplab:Semantic image segmentation with deep convolutional nets,atrous convolution,and fully connected crfs[J].IEEE Transactions on Pattern Analysis and Machine intelligence,2017,40(4):834-848.
[17] 王利华,李亮亮,郭恒林.基于目标的相机标定方法[J].北京测绘,2021,35(3):372-375.
WANG L H,LI L L,GUO H L.A target-based calibration method [J].Beijing Surveying and Mapping,2021,35(3):372-375.
[18] CHEN L C,ZHU Y,PAPANDREOU G,et al.Encoder-decoder with atrous separable convolution for semantic image segmentation[C]//Proceedings of the European Conference on Computer Vision(ECCV),2018:801-818.
[19] XIE E,WANG W,YU Z,et al.SegFormer:Simple and efficient design for semantic segmentation with transformers[J].Advances in Neural Information Processing Systems,2021,34:12077-12090.
[20] RONNEBERGER O,FISCHER P,BROX T.U-net:Convolutional networks for biomedical image segmentation[C]//Medical Image Computing and Computer-Assisted Intervention-MICCAI 2015:18th International Conference,Munich,Germany,October 5-9,2015,Proceedings,Part III 18.Springer International Publishing,2015:234-241.
[21] CHEN J,LU Y,YU Q,et al.Transunet:Transformers make strong encoders for medical image segmentation[J].arXiv preprint arXiv:2102.04306,2021.
[22] ARSENAULT J L,POULCUR S,MESSIER C,et al.WinRHlZO™,a root-measuring system with a unique overlap correction method[J].HortScience,1995,30(4):906D-906.
[23] HUANG Y,YAN J,ZHANG Y,et al.Automatic segmentation of cotton roots in high-resolution minirhizotron images based on improved OCRNet[J].Frontiers in Plant Science,2023,14:1147034.

