桑属植物叶绿体基因组特征比较及系统发育分析
张浩蓉 秃玉翔 李启少 曹正英 赵文植 沈伟祥 孙正海

张浩蓉,秃玉翔,李启少,等. 桑属植物叶绿体基因组特征比较及系统发育分析[J]. 江苏农业科学,2024,52(7):34-40.
doi:10.15889/j.issn.1002-1302.2024.07.005
=(西南林业大学园林园艺学院/国家林业和草原局西南风景园林工程技术研究中心,云南昆明 650224)
摘要:探究桑属叶绿体基因组特征及该属物种的系统发育进化关系。收集8种桑属植物叶绿体基因序列,基于最大似然法重建桑属植物系统发育关系,并运用生物信息学手段进行叶绿体基因组特征分析。桑树叶绿体基因组结构为典型的四段式结构,大单拷贝区长89 952 bp,小单拷贝区长 20 149 bp,2个反向重复区长25 751 bp。共检测到 661个SSR位点,包含480个单核苷酸位点、57个二核苷酸位点、25个三核苷酸位点、82个四核苷酸位点、16个五核苷酸位點、1个六核苷酸位点,碱基组成以A/T碱基类型为主,平均GC含量为36.2%。变异度在前3位的间隔区依次是[WTBX][STBX] psbl-trnS(GCU)-trnG(UCC)、 rps4-trnT(UGU)-trnL(UAA)、rp132-trnL(UAG)。[WTBZ][STBZ]桑属树种的叶绿体基因组相对较大,且序列稳定,变异主要发生在 LSC 区。相较于基因编码区,桑属植物的基因非编码区更容易积累突变。桑与蒙桑具有较近的亲缘关系,其次是华桑与吉隆桑。
关键词:桑属;叶绿体基因组;特征;系统发育
中图分类号:S888.2 文献标志码:A 文章编号:1002-1302(2024)07-0034-07
桑属(Morus L.)隶属于桑科(Moraceae),为灌木植物或乔木植物,速生且长寿。桑属植物分布于中国、朝鲜、日本、尼泊尔、印度等国海拔700 m左右的石灰岩山地及贫瘠土地[1],具有一定的观赏价值[2]和重要的经济价值[3-4]。[JP2]桑树韧皮纤维是纸张的重要来源,桑果既可熟食亦可生食,桑叶可以用来饲养桑蚕,树皮是提取桑色素的重要原料[5],茎干通直而结构细密,不易变形和开裂,是一种优良木材[6]。
叶绿体是普遍存在于陆地植物中的一种半自主型细胞器,能进行光合作用[7]。自 1986 年基于地钱和香烟植物的叶绿体基因组首次进行测序和分析后[8],学界开始探索分析叶绿体基因组的结构和特征。大多数植物的叶绿体基因组是2条链呈圈状的DNA分子,另外还有线形状、D环状、套索状及其他形状[9-10]。植物种类不同,其基因组的大小也不同,但总体上为120~160 kb[11]。叶绿体基因组具有相对较低的进化速率,结构简单,序列保守,遗传重组率低;而且,其独立的进化路线可以不依赖其他数据进行建树,这为探寻植物复杂的种间亲缘关系奠定了基础[12-13]。随着2代、3代测序技术的进步成熟和测序成本的降低,已有香花枇杷[14]、华山松[15]、杜梨[16]、蜡梅[17]、苹果[18]、莲[19]等许多植物的叶绿体基因组被报道,这为后期进一步对相关植物叶绿体基因组学基因功能及遗传改良的研究奠定了基础。目前对桑属植物叶绿体基因组方面的研究相对较少。李巧丽对鲁桑的叶绿体基因组结构及系统进化关系进行探索,发现鲁桑和蒙桑的亲缘关系较近[20]。武越基于 ITS 序列和4个叶绿体 DNA 片段(trnL-F、trnT-L、trnD-T、psbE-petG)分析桑属植物系统进化关系,发现在桑属中存在致同进化不完全现象[21]。然而他们并未对朴桑、非洲桑、鸡桑等其他桑属植物的叶绿体基因组特征和系统发育关系进行分析。
本研究基于生物信息学手段,利用桑属植物8个种的序列进行叶绿体基因组特征分析,同时利用叶绿体基因组重建桑属系统发育关系,解析桑属物种间的亲缘关系,旨在为后续开展桑属物种的分子标记开发、遗传背景分析、种质资源保护、系统发育进化等研究奠定基础。
1 材料与方法
1.1 材料
对NCBI 数据库中所有桑属物种的叶绿体全基因组序列进行筛选,收集已公布的桑(Morus alba)、蒙桑(M. mongolica)、吉隆桑(M. serrata)、华桑(M. cathayana)、印度桑(M. indica)、朴桑(M. celtidifolia)、非洲桑(M. mesozygia)、鸡桑(M. australis)等桑属植物 8 个种的叶绿体基因组序列用于分析。选择在桑科内与桑属植物亲缘关系较近的构树(Broussonetia papyrifera,MW465959)、无花果(Ficus carica,KY635880)作为构建系统发育树的外类群。
1.2 方法
1.2.1 叶绿体基因组序列比对 为在叶绿体基因组水平上判别桑属植物,上传桑属植物8 个种的叶绿体基因组序列比对工具 mVISTA(http://genome.lbl.gov/vista/index.shtml),运用该工具中的 LAGAN 程序进行叶绿体基因组解析[22],该程序可以直观地显示整体序列的相似性和变异区,参数设置默认值[23]。
1.2.2 简单重复序列 利用MISA perl脚本对8个桑属物种的叶绿体基因组中的简单重复序列(simple sequence repeat,SSR)进行计算。单核苷酸(mononucleotide,mono-)重复阈值设置为 10,二核苷酸(dinucleotide,di-)重复阈值设置为 5,三核苷酸(trinucleotide,tri-)重复阈值设置为 4,四核苷酸(tetranucleotide,tetra-)、五核苷酸(pentanucleotide,penta-)、六核苷酸(hexanucleotide)的重复阈值分别设置为 3、3、3[20]。
1.2.3 IR区边界的收缩与扩张 以朴桑的叶绿体基因序列为参考,于 GeSeq 在线网站(https://chlorobox.mpimp-golm.mpg.de/geseq.html)注释其余7个桑属植物叶绿体基因序列。注释好的叶绿体基因序列上传到IRscope(https://irscope.shinyapps.io/irapp/)在线软件,分析叶绿体结构的四大分区[24-25]。
1.2.4 高变区筛选 基于 MAFFT 软件对8个桑属叶绿体基因组序列进行比对,然后使用 DnaSP v6 软件[26]对桑属植物叶绿体基因组序列进行核苷酸多态性(Pi值)分析。参数窗口长度设置为600 bp,每步滑行大小设置为200 bp。
1.2.5 系统发育关系 将桑属植物 8 个种的叶绿体序列于MAFFT在线网站中比对后,以构树、无花果为外类群,构建系统发育树[21]。将比对后的序列矩阵使用Geneious软件转化成phy格式文件,然后将phy文件放入IQ-tree软件中计算核苷酸取代模型,计算得到最优模型:TVM+F+I+G4,在IQ-tree程序中使用最大似然法重建系统发育关系[27-28]。使用UFBoot2重复1 000个bootstrap折叠近零分支[29-30]。系统发育分析结果于Figtree v1.4.3 软件进行查看和美化[31]。当支持率大于 90% 时认为 ML 支持率较好,低于 50% 则认为是不可信数据[32]。
2 结果与分析
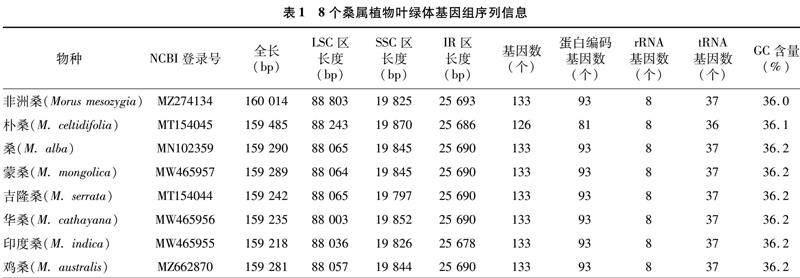
2.1 桑属8个种叶绿体基因组基本信息
由表1可知,桑属植物的叶绿体基因组具有典型的环状 DNA 分子特征,且为保守的四分体结构,基因组总长度在 159 218~160 014 bp,其中最大的为非洲桑,最小的为印度桑。大单拷贝区(LSC)为 88 003~88 803 bp,小单拷贝区(SSC)为 19 797~ 19 870 bp,反向重复区(IR)为 25 678~25 693 bp。8种桑属植物叶绿体基因组GC含量范围为 36.0%~36.2%。[JP]共包含126~133个基因,除去重复的蛋白编码基因外,其余物种蛋白编码基因数量一致;转录RNA的基因数量朴桑为36个,其余均为37个;核糖体RNA基因数量稳定在8个。
2.2 桑属植物序列差异性分析
运用mVISTA软件比对,结果表明,桑属叶绿体基因的变异程度较低,编码序列大多有很高的相似性(ycf 类基因不包括在内),且大部分突变在非编码区产生。 LSC、SSC 区的变异度明显高于 rRNA 类基因所在的 IR 区,且基因间隔区的变异度高于基因内含子区,如psbl-trnS(GCU)-trnG(UCC)、rps4-trnT(UGU)-trnL(UAA)、rp132-trnL(UAG)等(图1)。
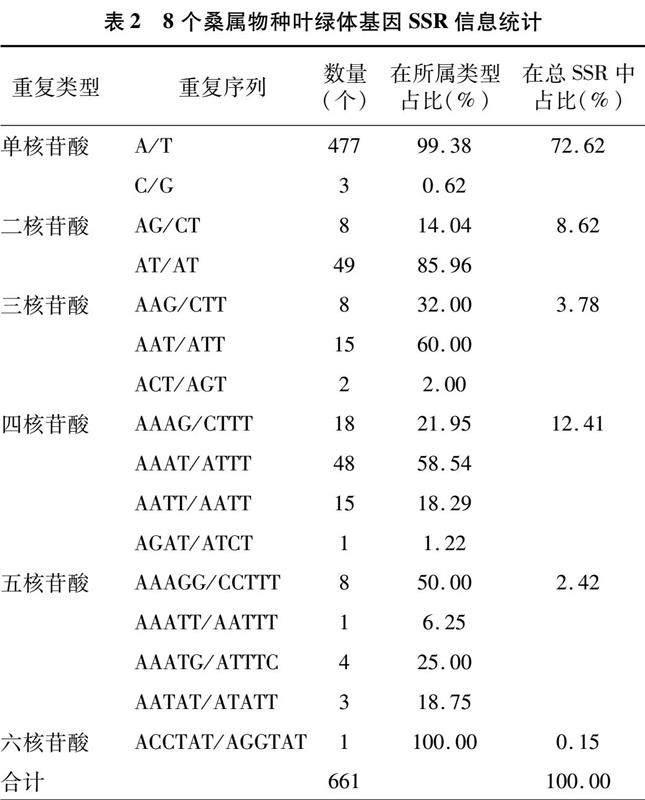
2.3 桑属植物简单重复序列分析
利用MISA 软件进行简单重复序列分析。共检测到 661 个潜在的 SSRs,其中单核核苷酸480个、二核苷酸57个、三核苷酸25个、四核苷酸82个、五核苷酸16个、六核苷酸1个,仅在朴桑中检测到。这些潜在的SSRs大部分为单核核苷酸,所占比例为72.62%,主要碱基为 A/T 堿基;其次是四核核苷酸,占比 12.41%,主要碱基为AAAT/ATTT;占比最少的是六核核苷酸,仅有 0.15%(表2、图2)。
2.4 桑属植物 IR 区边界分析
8个桑树 IR 区的长度在 25 678 bp(印度桑)~25 693 bp(非洲桑)之间(图3)。位于LSC与IRb边界处的基因为rps19,基因总长度为279 bp。在8个桑树叶绿体基因组中,基因均位于LSC区域,且距离IRb区域的长度仅有1 bp的差异。基因ycf1横跨 SSC/IRa 边界,IR 区的不完整拷贝使得ycf1基因成为一个假基因(ψ)。桑属不同种的ycf1基因一部分(5 600~5 639 bp)均延伸至 IRa 区域,基因长度基本一致。然而桑属不同物种ycf1基因横跨 SSC区、IRa区的长度不同,桑、蒙桑、华桑、吉隆桑、印度桑、鸡桑在SSC区、IRa区的长度一致,分别为4 638、1 001 bp,余下的非洲桑、朴桑在SSC区和IRa区的长度分别为4 604、4 639 bp、996~991 bp。 [JP+2]在SSC/IRb边界处, 8[JP]个桑属物种叶绿体基因组的ycf1基因均横跨SSC、IRb 区。但是树种不同,基因在SSC、IRb 区的长度存在差异。桑、蒙桑、华桑、吉隆桑、印度桑、鸡桑在 SSC、IRb 区的长度一致,分别为 80、1 002 bp,而非洲桑、朴桑在SSC和IRb区的长度分别为11、91 bp和996、991 bp。8个桑属物种之间ycf1基因在 IRa、IRb 区域的拷贝数不同。基因trnH与边界SSC/IRa距离,除了非洲桑为31 bp外,其余树种均为 22 bp。
2.5 桑属8个种核苷酸多态性分析
DnaSP 6软件筛选结果表明,桑属植物的核苷酸多态性值(Pi)在 0~0.020 95之间,平均值为 0.002 753(图4)。当Pi>0.01 时,检测到的3个高变区分别是psbl-trnS(GCU)-trnG(UCC)、rps4-trnT(UGU)-trnL(UAA)、rp132-trnL(UAG);其中,高变区 psbl-trnS(GCU)-trnG(UCC)、rps4-trnT(UGU)-trnL(UAA)位于 LSC 区,而rp132-trnL(UAG)位于 SSC 区。
2.6 系统发育关系分析
如图5所示,8个桑属植物很好地聚为一支(BS=100),第1个分化出来为国外种非洲桑(BS=100),其次为朴桑(BS=100),其余6个桑属物种则是在较短时间内分化成为2个亚支(BS=100);其中桑、蒙桑、鸡桑、吉隆桑、华桑聚成一支(BS=100),另一支只包含印度桑(BS=100)。桑、蒙桑、鸡桑形成一个单系进化支, 吉隆桑、华桑聚成一支,互为姐妹类群(BS=93),亲缘关系较近。绝大多数进化支的支持率都很高, 仅吉隆桑和华桑的进化支支持率较低(BS=60)。
3 讨论
对 8 个桑属物种进行叶绿体基因组特征分析,桑属植物均具有典型的四分体结构,基因组大小相近,介于 159 218~160 014 bp,总长收缩扩张不超过 1 kb,与之前报道的植物叶绿体基因组序列全长低至 114 914 bp、 高至 217 942 bp[33-34]相符合; 且基因个数和基因种类也大致相同,这再次证明叶绿体基因组具有高度保守的特性[35-36]。8个桑属植物IR区的差异整体较小,长度大多为 25 690 bp,与之前报道的绝大多数被子植物的IR/LSC范围较为保守,其IR区毗邻IR/LSC边界的位置往往会存在1个[WTBX][STBX]rpl22[WTBZ][STBZ]或者[WTBX][STBX]trnH-rps19[WTBZ][STBZ]基因簇[37-38]相符合。但非洲桑的IR区长了3 bp,朴桑、印度桑分别短了4、12 bp。IR区边界的收缩与扩张通常伴随着植物的进化,造成叶绿体基因组间的大小差异[39-40,41]。从系统发育树上可以看出,物种分化先后依次是非洲桑、朴桑、印度桑,但并不能得出“IR区越长,物种分化越靠前,约有可能为基部类群”的结论。物种间叶绿体基因组序列全长变化主要与IR区有关[42-43]。朴桑的IR区比桑的短4 bp,而朴桑的叶绿体基因组长度却比桑的长了至少195 bp,IR 区长度相同的5个桑属物种的叶绿体基因组长度也有差异。早在枣树和鸭跖草亚纲物种中就报道了由 IR 区收缩引起的叶绿体基因组长度的类似变化[44]。蒙桑、桑的叶绿体基因组 SSC、IR区大小一致,仅在LSC区相差1 bp,推测蒙桑可能是桑的变种[45]。
对桑属植物8个种的叶绿体基因组序列进行mVISTA 分析发现,该属植物叶绿体全基因组序列变异较小,这与李巧丽等用4个桑属树种探究的结果[20]一致,说明桑属不同物种的叶绿体基因组序列保守。核苷酸多态性分析结果与 mVISTA结果一致,在IR区的Pi值为0.004 58,其序列核苷酸的变异程度明显低于LSC区(Pi值为0.013 33)、SSC区(Pi值为0.020 95)。这与大多数植物叶绿体基因组序列分析结果[46-47]一致,突变主要发生在LSC区、SSC区。8个桑属树种叶绿体基因组中Pi>0.01,鉴定出3个可变区 psbl-trnS(GCU)-trnG(UCC)、ps4-trnT(UGU)-trnL(UAA)、rp132-trnL(UAG)。其中 trnS-trnG、rpl32-trnL 基因座曾被报道为种子植物中的高度可变区域[48-49],这一高度可变的区域也位于LSC 区、SSC 区。此外,8个桑属植物的叶绿体基因中共发现 661个SSRs标记。SSRs标记多分布在LSC区,主要由碱基A、T组成,与叶绿体基因组序列中SSRs主要由poly A、poly T所构成的结论[50-51]相互验证。然对mVISTA 分析、核苷酸多态性分析、简单重复序列分析共同得出结果突变主要发生在LSC区、SSC区之间的联系还需要进一步研究[52]。所检测到的重复序列可为桑属及桑科其他植物SSRs标记的开发提供理论依据,也可为桑属植物的分子遺传相关研究提供候选标记。
基于桑属叶绿体基因组,运用最大似然法,通过 IQ-tree 软件重建发育关系。非洲桑作为基部类群最先被分化出来,与甄攀等基于ITS序列构建系统发育树得出非洲桑是目前发现的最古老桑树种的结论[4]相符合。桑树种很好地形成了一个单系进化支,再次印证了Zhao等利用ITS、trnL-F序列进行系统发育分析得出的桑属的单系起源性[53]。同为野生种的华桑、吉隆桑互为姐妹类群的关系,也得到文言的研究[54]支持。
4 结论
本研究对桑属物种叶绿体基因组特征和系统发育关系进行探究。桑属物种叶绿体基因组序列长度为159 218~160 014 bp。桑、蒙桑叶绿体基因组的SSRs标记数量相同,且主要由A/T碱基组成。共检测到[WTBX][STBX]psbl-trnS(GCU)-trnG(UCC)、rps4-trnT(UGU)-trnL(UAA)、rp132-trnL(UAG) [WTBZ][STBZ]共3个高变区域,这些高变区域可作为桑属植物DNA条形码的候选片段。华桑与吉隆桑,蒙桑与桑、鸡桑分别互为姐妹类群。研究结果可为进一步对桑属植物叶绿体基因组基因的功能进行分析以及遗传改良等提供理论参考。
参考文献:
[1]李 鹏. 川桑CenH3基因的鉴定及着丝粒序列初探[D]. 重庆:西南大学,2020.
[2]孟钰程. 蒙桑种质资源多倍体诱导及加倍川桑抗性的初步评价[D]. 重庆:西南大学,2019.
[3]杨凤琼,李梅丽. 川桑葚中鞣酸的提取及纯化工艺研究[J]. 粮食与油脂,2018,31(5):92-96.
[4]甄 攀,倪 刚,陈晓光,等. 川桑的化学成分及其细胞毒作用[J]. 药学学报,2015,50(5):579-582.
[5]郭伟强,王 硕,李 军,等. 川桑中桑皮素、桑呋喃A和总黄酮的测定[J]. 中成药,2016,38(11):2506-2509.
[6]李巧丽,延 娜,宋 琼,等. 鲁桑叶绿体基因组序列及特征分析[J]. 植物学报,2018,53(1):94-103.
[7]李西文,胡志刚,林小涵,等. 基于454FLX高通量技术的厚朴叶绿体全基因组测序及应用研究[J]. 药学学报,2012,47(1):124-130.
[8]Shinozaki K,Ohme M,Tanaka M,et al. The complete nucleotide sequence of the tobacco chloroplast genome:its gene organization and expression[J]. The EMBO Journal,1986,5(9):2043-2049.
[9]Shen W X,Dong Z H,Zhao W Z,et al. Complete chloroplast genome sequence of Rosa lucieae and its characteristics[J]. Horticulturae,2022,8(9):788.
[10]Lilly J W,Havey M J,Jackson S A,et al. Cytogenomic analyses reveal the structural plasticity of the chloroplast genome in higher plants[J]. The Plant Cell,2001,13(2):245-254.
[11]Palmer J D.Comparative organization of chloroplast genomes[J]. Annual Review of Genetics,1985,19:325-354.
[12]楊琳懿,赵文植,辛 静,等. 光叶子花及紫茉莉科植物叶绿体基因组序列的比较分析[J]. 西部林业科学,2022,51(2):66-73,79.[JP]
[13]蔡苗苗,刘梦云,刘 红,等. 不同产地巴戟天叶绿体基因组比较研究[J]. 中药新药与临床药理,2021,32(5):709-715.
[14]屈亚亚,辛 静,冯发玉,等. 香花枇杷质体基因组序列密码子偏性分析[J]. 西北林学院学报,2021,36(4):138-144,158.
[15]鲁 华,董章宏,瞿绍宏,等. 华山松叶绿体基因组特征分析[J]. 分子植物育种,2021,19(10):3223-3234.
[16]辛雅萱,董章宏,瞿绍宏,等. 杜梨叶绿体基因组密码子偏好性分析[J]. 河北农业大学学报,2020,43(6):51-59.
[17]尚均忠. 蜡梅全基因组测序及其花香主成分合成相关基因功能解析[D]. 武汉:华中农业大学,2020.
[18]程 慧,葛春峰,张 红,等. 果树叶绿体基因组测序及系统发育研究进展[J]. 核农学报,2018,32(1):58-69.
[19]孙林鹤,常雅军,杜凤凤,等. 水生植物全基因组测序及其应用研究进展[J]. 分子植物育种,2024,227(7):2194-2204.
[20]李巧丽. 广东桑和鲁桑叶绿体基因组高通量测序及基因注释分析[D]. 杨凌:西北农林科技大学,2017.
[21]武 越. 基于桑树ITS和叶绿体DNA序列的系统发生分析[D]. 重庆:西南大学,2018.
[22]Frazer K A,Pachter L,Poliakov A,et al. VISTA:computational tools for comparative genomics[J]. Nucleic Acids Research,2004,32(issue suppl_2):W273-W279.
[23]Mayor C,Brudno M,Schwartz J R,et al. VISTA:visualizing global DNA sequence alignments of arbitrary length[J]. Bioinformatics,2000,16(11):1046-1047.
[24]杜致辉,杨 澜,张朝君,等. 黑喉石斛叶绿体基因组特征及比较分析[J]. 热带作物学报,2021,42(11):3111-3119.
[25]Amiryousefi A,Hyvnen J,Poczai P. IRscope:an online program to visualize the junction sites of chloroplast genomes[J]. Bioinformatics,2018,34(17):3030-3031.
[26]Librado P,Rozas J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics,2009,25(11):1451-1452.
[27]Nguyen L T,Schmidt H A,von Haeseler A,et al. IQ-TREE:a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Molecular Biology and Evolution,2015,32(1):268-274.
[28]Song Y,Yao X,Tan Y H,et al. Complete chloroplast genome sequence of the avocado:gene organization,comparative analysis,and phylogenetic relationships with other Lauraceae[J]. Canadian Journal of Forest Research,2016,46(11):1293-1301.
[29]Qu S H,Dong Z H,Gao L Y,et al. Complete plastome of a subtropical tree Eriobotrya malipoensis (Rosaceae) in Yunnan[J]. Mitochondrial DNA Part B Resources,2019,4(2):3534-3535.
[30]王 飞,辛雅萱,董章宏,等. 无刺龙舌兰叶绿体基因组特征及密码子偏好性分析[J]. 南方农业学报,2022,53(4):1030-1039.
[31]Cao Z Y,Zhao W Z,Xin Y X,et al. Characteristics of the complete chloroplast genome of Pourthiaea (Rosaceae) and its comparative analysis[J]. Horticulturae,2022,8(12):1144.
[32]Dong Z H,Zhang R L,Shi M,et al. The complete plastid genome of the endangered shrub Brassaiopsis angustifolia (Araliaceae):comparative genetic and phylogenetic analysis[J]. PLoS One,2022,17(6):e0269819.
[33]李 岩,吕光辉,张雪妮,等. 十字花科植物叶绿体基因组结构及变异分析[J]. 西北植物学报,2017,37(6):1090-1101.
[34]王瑞江. 中国植物分类及标本采集史简述[J]. 广西植物,2022,42(增刊1):62-70.
[35]赵文植,董章宏,辛 静,等. 高粱泡叶绿体基因组特征分析[J]. 云南农业大学学报(自然科学),2022,37(3):435-446.
[36]王海洋,龙 飞,沈伟祥,等. 滇黄精离体快繁体系建立及优化[J]. 黑龙江农业科学,2022(5):85-90.
[37]Wu F H,Chan M T,Liao D C,et al. Complete chloroplast genome of Oncidium Gower Ramsey and evaluation of molecular markers for identification and breeding in Oncidiinae[J]. BMC Plant Biology,2010,10:68.
[38]Luo J,Hou B W,Niu Z T,et al. Comparative chloroplast genomes of photosynthetic orchids:insights into evolution of the Orchidaceae and development of molecular markers for phylogenetic applications[J]. PLoS One,2014,9(6):e99016.
[39]Kim K J,Lee H L.Complete chloroplast genome sequences from Korean ginseng (Panax schinseng Nees) and comparative analysis of sequence evolution among 17 vascular plants[J]. DNA Research,2004,11(4):247-261.
[40]Wang W C,Chen S Y,Zhang X Z.Whole-genome comparison reveals divergent IR borders and mutation hotspots in chloroplast genomes of herbaceous bamboos (Bambusoideae:Olyreae)[J]. Molecules,2018,23(7):1537.
[41]Park S,An B,Park S.Reconfiguration of the plastid genome in Lamprocapnos spectabilis:IR boundary shifting,inversion,and intraspecific variation[J]. Scientific Reports,2018,8:13568.
[42]李 波. 让“维管植物生命之树”在祖国大地上枝繁叶茂:读《中国维管植物生命之树》有感[J]. 科学,2021,73(1):58-62.
[43]李显煌,杨生超,辛雅萱,等. 灯盏花叶绿体基因组密码子偏好性分析[J]. 云南农业大学学报(自然科学),2021,36(3):384-392.
[44]Song Y,Yao X,Tan Y H,et al. Comparative analysis of complete chloroplast genome sequences of two subtropical trees,Phoebe sheareri and Phoebe omeiensis (Lauraceae)[J]. Tree Genetics & Genomes,2017,13(6):120.
[45]刘 潮,韩利红,代小波,等. 辣椒属叶绿体基因组特征及进化[J]. 热带作物学报,2022,43(3):447-454.
[46]张明英,张雨曲,李依民,等. 北柴胡、紫花阔叶柴胡叶绿体全基因组解析及柴胡属药用植物叶绿体基因组比较与系统发育分析[J]. 药学学报,2021,56(2):618-629.
[47]辛雅萱,黎若竹,李 鑫,等. 杧果叶绿体基因组密码子使用偏好性分析[J]. 中南林业科技大学学报,2021,41(9):148-156,165.
[48]Dong W P,Liu J,Yu J,et al. Highly variable chloroplast markers for evaluating plant phylogeny at low taxonomic levels and for DNA barcoding[J]. PLoS One,2012,7(4):e35071.
[49]王 飛,赵文植,董章宏,等. 扁核木属植物叶绿体基因组特征分析[J]. 热带作物学报,2022,43(9):1759-1770.
[50]Yang R C,Juskiw P. Analysis of covariance in agronomy and crop research[J]. Canadian Journal of Plant Science,2011,91(4):621-641.
[51]李 凤,辛 静,辛雅萱,等. 楸树叶绿体基因组密码子偏性分析[J]. 南方农业学报,2021,52(10):2735-2743.
[52]李卫英,辛 静,赵文植,等. 朱蕉叶绿体基因组特征与系统发育分析[J]. 热带作物学报,2022,43(4):653-665.
[53]Zhao W,Pan Y,Zhang Z,et al. Phylogeny of the genus Morus (Urticales:Moraceae) inferred from ITS and trnL-F sequences[J]. African Journal of Biotechnology,2005,4(6):563-569.
[54]文 言. 川桑密码子使用模式与密码子分析工具的开发[D]. 重庆:西南大学,2017.
基金项目:云南省科技人才与平台计划(编号:202205AF150022);西南林业大学科研预研基金。
作者简介:张浩蓉(1998—),女,云南昆明人,硕士研究生,主要从事园林植物与观赏园艺研究。E-mail:zhr23122x@163.com。
通信作者:孙正海,博士,教授,主要从事园林植物遗传育种与分子生物学研究。E-mail:szh@swfu.edu.cn。

