南方番茄病毒在海南省的发生分布及全基因组序列分析
车海彦 林雅婷 罗大全 龙海波



关键词:南方番茄病毒;小RNA深度测序;RT-PCR检测;基因组
1984年,美国加利福尼亚因皮里尔河谷的番茄上出现叶片黄化、生长衰退和坐果少等症状[1]。2005年,墨西哥西南部和美国密西西比州东北部的番茄上也出现相似的症状,上述3地的病株体内均分离到一种基因组为3.5kb的dsRNA病毒,SABANADZOVIC等[2]将其命名为南方番茄病毒(Southerntomatovirus,STV)。目前,STV在亚洲(中国、孟加拉国、韩国、越南、日本、土耳其、泰国、巴基斯坦和以色列)[3-8]、欧洲(法国、意大利、西班牙、英国、瑞士、德国和塞尔维亚)[8,9-19]、非洲(留尼汪岛)[9]、北美洲(美国、墨西哥和加拿大)[1-2,8]、中美洲(多米尼加和巴拿马)[9]和南美洲(哥伦比亚和巴西)[8,20]的很多国家均有报道发生。2011年,我国首次在新疆加工番茄上发现STV感染的植株[3],随后在山东[21-22]、浙江[23]、江西[23]、四川[24]、重庆[25]、北京[26]、广东[27]、江苏[28]、宁夏[29]和贵州[8]等地的番茄上也相继检测到STV。但目前我国仅公布2条STV基因组全长序列[4,30],对STV的深入研究报道较少。
STV隶属于混合病毒科(Amalgaviridae)混合病毒属(Amalgavirus),尚未发现病毒粒子,其基因组由1条线性dsRNA组成,长约3.5kb,在正义链上有2个部分重叠的开放阅读框(openreadingframe,ORF),结构上与Totiviridae成员相似。ORF1长1134bp,编码由377个氨基酸组成的p42蛋白,推测为外壳蛋白(coatprotein,CP),分子量约为42.4kDa。ORF2长2289nt,编码由762个氨基酸组成的RNA依赖的RNA聚合酶(RNAdependentRNApolymerase,RdRp),RdRp可能通过程序性+1核糖体移码(programmed+1ribosomalframeshifting,+1PRF)表达为与ORF1的融合蛋白(类似于逆转录病毒的gag-pol融合蛋白),分子量约为121.5kDa[2,31]。尽管STV在基因组结构上与Totiviridae成员相似,但基于RdRp氨基酸的系统发育分析表明,STV与分体病毒科(Partitiviridae)成员的亲缘关系更近[2,31]。目前,尚未观察到STV的病毒粒子。STV除通过种子和花粉传播外,尚未发现其他传播途径[2,30-31]。仅在番茄(Solanumlycopersicum)、辣椒(Capsicumannuum)、龙葵(Solanumnigrum)和灯笼果(Physalisperuviana)4种茄科植物上检测到STV[8,10,20,32]。
番茄是海南冬季重要的北运瓜菜品种之一,主要种植于陵水、昌江、定安等地,至2019年,海南省种植面积达7822hm2,产量为28.15万t[33]。随着种植面积的增加和种植年限的延长,生产中病虫害发生为害日趋严重,其中病毒病是为害最严重的病害,部分田块病株率高达100%。笔者在利用小RNA深度测序技术鉴定海南番茄病毒病病原时发现,STV在海南多个市(县)的样品中均存在,而且发现1株STV单独侵染的样品。为进一步了解STV在海南的发生分布情况,利用RT-PCR技术检测了2015—2021年采集自海南的番茄样品,同时结合RACE和RT-PCR技术扩增了海南番茄样品上的STV全基因组序列,本研究结果有助于了解STV在海南的分布、发生趋势及遗传多样性,为病害防控提供科学依据。
1材料与方法
1.1材料
小RNA深度测序的番茄叶片样品(编号为T-DZ-2)于2019年采自海南儋州宝岛新村,田间症状表现为叶脉坏死、叶脉间不均匀褪绿,长势衰弱。STV检测的样品共987份,于2015—2021年采自海南陵水、定安、昌江、海口、琼海、三亚、儋州、文昌、临高、乐东共10个市(县)。
植物总RNA提取试剂盒和普通琼脂糖凝胶DNA回收试剂盒购自天根生化科技(北京)有限公司,TRIzolReagent购自美国Invitrogen公司,反转录试剂盒TransScriptFirst-StrandcDNASynthesisSuperMix购自北京全式金生物技术有限公司,2×RapidTaqMasterMix和2×PhantaFlashMasterMix购自南京诺唯赞生物科技股份有限公司,pMD18-TVectorCloningKit、E.coliCompetentCellDH5α、Poly(A)Polymerase和SMARTerRACE5′/3′Kit均购自宝日医生物技术(北京)有限公司。其他试剂均为国产或进口分析纯。
1.2方法
1.2.1小RNA深度测序和分析使用TRIzolReagent提取番茄叶片样品(编号为T-DZ-2)的总RNA。委托上海美吉生物医药科技有限公司构建小RNA文库,使用Hiseq2000平台进行SE50测序,原始数据经过质控分析后,获得cleansmallRNA序列,使用Velvet1.2.10软件[34]对其进行拼接组装。将拼接结果在NCBI数据库中进行Blastn和Blastx比对,将得到的病毒序列进行统计并注释。
1.2.2RT-PCR验证以1.2.1提取的总RNA为模板,采用反转录试剂盒TransScriptFirst-StrandcDNASynthesisSuperMix将总RNA反转录成cDNA。使用STV特異性检测引物STV-F/STV-R[23]对小RNA深度测序结果进行验证,引物序列见表1。PCR反应体系(25μL):12.5μL2×RapidTaqMasterMix、10μmol/L的上游引物和下游引物各1μL,1μLcDNA,9.5μL无菌ddH2O。PCR反应程序:98℃预变性30s;98℃变性10s,52℃退火5s,72℃延伸5s,循环35次;72℃延伸10min。PCR产物经1%琼脂糖凝胶电泳检测,使用普通琼脂糖凝胶DNA回收试剂盒回收特异片段,回收产物直接测序。本研究所有的引物合成和测序均委托北京六合华大基因科技有限公司完成。
1.2.3田间番茄样品的STV检测使用植物总RNA提取试剂盒提取田间采集的987份样品总RNA,cDNA合成和PCR检测反应参见1.2.2。
1.2.4STV全基因组序列扩增根据田间样品的检测结果,选取海南儋州(T-DZ-2)、陵水(T-LS-52)、定安(T-DA-23)和昌江(T-CJ-15)共4个样品进行全基因组扩增。STV全基因组扩增策略如图1所示。根据小RNA深度测序结果和GenBank已有序列,利用在线引物设计工具Primer3(https://bioinfo.ut.ee/primer3/)设计基因组扩增引物对STV-F1/STV-R1(表1),按照1.2.2方法对STV的基因组编码区进行扩增。5′和3′末端非编码区扩增:利用Poly(A)Polymerase将4个样品的RNA3′端加上poly(A)尾,使用SMARTerRACE5′/3′Kit进行cDNA合成。根据基因组编码区测序结果,设计特异性引物STV-GSP5和STV-GSP3(表1),分别对STV基因组的5′和3′末端序列进行扩增,获得的特异性片段产物经纯化回收后,与pMD18-Tvector连接,转化到E.coliCompetentCellDH5α中,利用PCR反应筛选阳性克隆。
1.2.5STV基因组结构分析使用VectorNTIAdvance11软件对测序结果进行校正、拼接。采用NCBI中BLAST程序进行相似性查找[35]。使用在线工具ORFFinder(https://www.ncbi.nlm.nih.gov/orffinder/)进行ORF预测。分别使用在线软件MAFFT(https://www.ebi.ac.uk/Tools/msa/mafft/)和ClustalOmega(https://www.ebi.ac.uk/Tools/msa/clustalo/)进行核苷酸和氨基酸序列一致性比对。
1.2.6STV基因组序列的系统发育分析利用MEGAX软件[36]的ClustalW法进行多序列比对,基于STV的全长基因组核苷酸序列,采用最大似然法(maximumlikelihood,ML)构建进化树,bootstrap值设置为1000。
1.2.7STV群体基因组序列相似性分析为进一步明确系统进化树分组中STV分离物在基因组水平上的差异,从组Ⅰ和组Ⅱ中选取29个代表性分离物,连同本研究获得的4个海南分离物,利用SDTv1.2软件[37]和Simplot3.5软件[38]进行相似性比对分析。
1.2.8重组分析使用RDP5软件[39]提供的7种重组检测算法(RDP、GENECONV、BootScan、MaxChi、Chimaera、SiScan和3Seq)对STV分离物的全基因组序列进行潜在重组事件分析,参数为软件默认。为了保证分析结果的可靠性,每个重组事件至少需要被上述7种算法中的5种所支持。
2结果与分析
2.1小RNA深度测序结果及RT-PCR验证
番茄样品经小RNA深度测序后获得22875457条rawreads,质控后得到9971315条cleanreads,将质控后获得的序列进行拼接组装,得到1509条contigs,将拼接结果与NCBI病毒库比对,获得3条源于病毒的contigs,均匹配到STV基因组,说明T-DZ-2样品中可能含有STV。
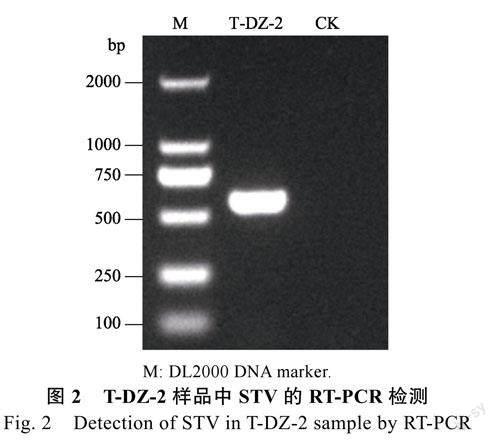
为了验证小RNA深度测序结果的准确性,利用STV特异性检测引物STV-F和STV-R对样品T-DZ-2进行RT-PCR检测(图2),扩增到582bp的目标条带,将PCR产物直接测序。Blastn比对分析发现,目标条带序列与已公布的STV基因组序列相似一致性为98.45%~99.83%,说明样品T-DZ-2中存在STV。
2.2STV在海南的发生与分布
利用STV特异性引物STV-F和STV-R对2015—2021年采集的987份番茄叶片样品进行检测,结果表明:在9个市(县)的142份样品中检测到STV(图3、表2)。2015年,仅在陵水和昌江样品中检测到STV。此后STV在海南的发生分布范围逐渐扩大,检出率逐年增加,到2021年,在陵水、文昌、昌江、三亚、临高、定安、乐东、儋州、琼海共9个市(县)检测到STV,检出率由2015年的8.82%上升到2021年的22.45%。上述结果说明,至少从2015年开始,海南番茄就已经被STV感染,而且STV在田间的发病率总体呈逐年上升趋势。
2.3STV全基因组序列分析
使用引物对STV-F1/STV-R1、STV-GSP5/UPM、STV-GSP3/UPM分别对海南儋州(T-DZ-2)、陵水(T-LS-52)、定安(T-DA-23)和昌江(T-CJ-15)的4个STV分离物基因组编码区、5′末端和3′末端非编码区进行扩增(图4),将测序获得的STV基因组编码区序列与5′和3′末端序列拼接,4个分离物的全基因组序列长度均为3446nt,不含3′-端poly(A)尾,GenBank登录号分别为OP484998、OP484999、OP485000、OP485001。STV基因組包含2个部分重叠的ORFs,ORF1位于基因组147~1280nt,编码377aa。ORF2位于基因组1048~3336nt,编码762aa。编码区A+T的含量为50.72%~50.75%。在993~999nt之间存在可能有助于核糖体移码的滑动序列GGGAAGA;5′UTR的长度为146nt,比已公布的STV分离物至少长8nt,A+T的含量为57.59%~58.12%。3′UTR的长度为110nt,A+T的含量为68.18%~69.09%。基因组非编码区比编码区富含更多的A+T。
本研究中的4个STV海南分离物间基因组核苷酸一致性为99.86%~100.00%,CP、RdRp的核苷酸一致性分别为99.82%~100.00%和99.91%~100.00%,氨基酸一致性分别为99.73%~100.00%和99.87%~100.00%;与我国已报道的2个STV分离物(KY228384,KT438549)的基因组核苷酸一致性分别为99.53%~99.65%和99.07%~99.19%,CP、RdRp的核苷酸一致性分别为99.47%~99.65%和99.26%~99.74%,氨基酸一致性分别为98.67%~99.73%和99.34%~99.61%;与GenBank中所有已公布的76个STV分离物(截至2022年9月28日)基因组核苷酸一致性为98.45%~99.94%,CP、RdRp的核苷酸序列一致性分别为98.68%~100.00%和98.38%~100.00%,氨基酸序列一致性分别为98.41%~100.00%和98.95%~100.00%。与所有已知的STV分离物全基因组核苷酸序列一致性为98.28%~100.00%,CP、RdRp的核苷酸一致性分别为98.41%~100.00%和97.99%~100.00%,氨基酸一致性分别为97.61%~100.00%和98.29%~100.00%。
2.4STV的系统发育和重组分析
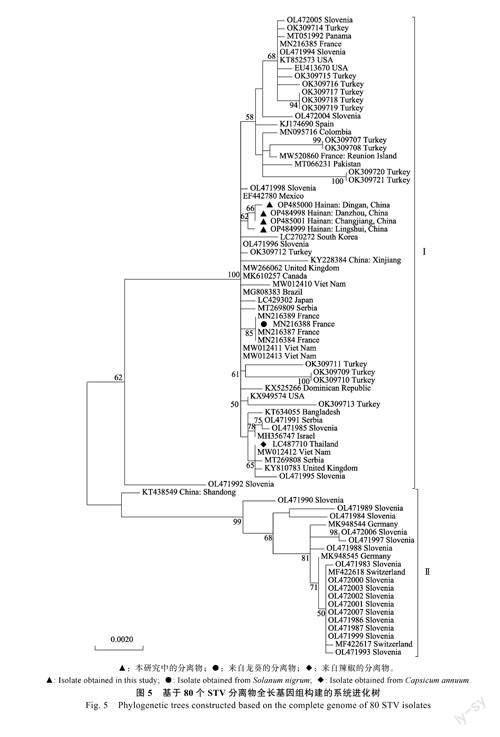
基于80个STV分离物的全基因组序列构建的系统进化树显示,STV分离物明显分为2个组,组Ⅰ包括亚洲、美洲、欧洲和非洲分离物;组Ⅱ中除1个亚洲分离物外,其他均为欧洲分离物;欧洲的斯洛文尼亚分离物在组Ⅰ和组Ⅱ均有分布。我国的6个STV分离物中,4个海南分离物在同一个小的进化分支上,与新疆分离物(KY228384)一起分在组Ⅰ,山东分离物(KT438549)分在组Ⅱ;泰国辣椒分离物(LC487710)和法国龙葵分离物(MN216388)分在组Ⅰ,与其他番茄分离物在小的进化支上,未单独分支(图5)。
全基因组重组分析结果表明,80个STV分离物基因组间未发现重组事件。
2.5STV群体基因组序列相似性分析
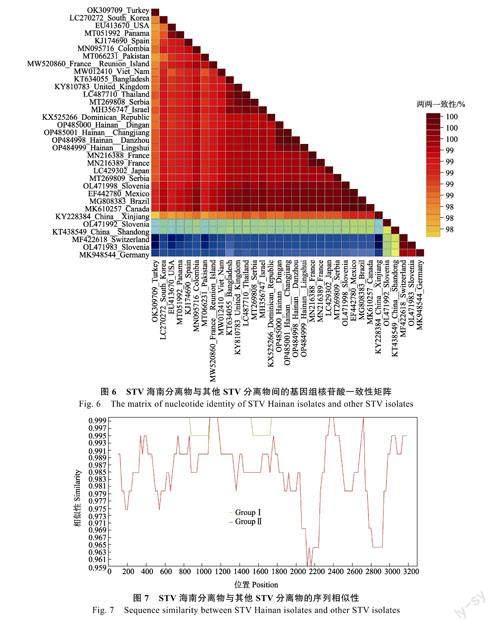
为明确系统进化树分组中STV分离物在基因组水平上的差异,从组Ⅰ和组Ⅱ中选取29个代表性分离物,与本研究获得的4个海南分离物,进一步利用SDT软件(图6)和Simplot软件(图7)进行相似性分析,发现4个STV海南分离物的核苷酸序列与组Ⅰ中用于分析的其他STV分离物间的相似性为99.65%~100.00%,基因组序列变异度比较高的区域位于881~1061nt和1521~1721nt;4个STV海南分离物的核苷酸序列与组Ⅱ中的分离物相似性为98.72%~99.07%,基因组序列变异度比较高的区域位于2041~2241nt和2761~2921nt。
3讨论
南方番茄病毒经常与其他病毒复合侵染番茄,如在我国北京房山,STV与番茄黄化曲叶病毒(Tomatoyellowleafcurlvirus,TYLCV)、番茄褪绿病毒(Tomatochlorosisvirus,ToCV)复合侵染番茄[26];在意大利,STV与凤果花叶病毒(Pepinomosaicvirus,PepMV)和马铃薯Y病毒(PotatovirusY,PVY)复合侵染番茄[11],故STV单独侵染引起的寄主症状尚不明确。但本研究通过小RNA深度测序发现1株单独侵染番茄的STV分离物,田间症状表现为叶脉坏死、叶脉间不均匀褪绿,这些症状与HARJU等[16]在英国南部发现的STV单独侵染番茄的表现症状极为相似,与美国加利福尼亚的因皮里尔河谷[1]、密西西比州东北部和墨西哥西南部[2]发现的STV引起的番茄感病症状存在明显不同。STV有时也会在健康植株上检测到[17,40],研究人员甚至发现感染STV的番茄植株不仅会结出更多的果实,而且结出的种子发芽率也高于无STV感染的植株[8]。根据上述研究结果推测:STV可能不仅存在致病性分化,而且同一毒株对同一寄主植物不同品种的致病性也有很大差异。
所有已知的STV分离物全基因组核苷酸序列一致性为98.28%~100.00%,说明不同地理来源的STV基因组序列变异率低,具有高度保守性。全基因组重组分析也未发现重组现象,与ELVIRAGONZÁLEZ等[14]的分析结果一致。
GenBank数据库(截至2022年9月28日)中共有119条STV序列,其中76条为全基因组序列[9]。STV的自然寄主均为茄科植物,除3个分离物的寄主为辣椒(LC487710)、龙葵(MN216388)和灯笼果(MN417999)外,其他分离物的寄主均为番茄[8]。基于全基因组序列构建的系统进化树显示,所有的STV分离物被明显分为2个组。除中国分离物(5个被分在组Ⅰ,1个被分在组Ⅱ)、塞尔维亚(4个被分在组Ⅰ,1个被分在组Ⅱ)和斯洛文尼亚分离物(6个被分在组Ⅰ,16个被分在组Ⅱ)外,同一國家的分离物均在同一组内,如5个法国分离物、15个土耳其分离物、2个英国分离物、3个美国分离物和4个越南分离物均被分在组Ⅰ,2个德国分离物和2个瑞士分离物均被分在组Ⅱ。泰国辣椒分离物(LC487710)和法国龙葵分离物(MN216388)被分在组Ⅰ(STV灯笼果分离物的序列片段不完整,未参与进化树构建),与其他番茄分离物在小的进化支上,未单独分支。说明STV分离物的分组与地域存在一定相关性,而与寄主植物之间不存在相关性。由于STV分离物主要来自番茄,而来自其他寄主植物的报道还较少,上述推论还需要更多的试验数据进行验证。
2011年,我国首次在新疆番茄上发现STV,目前该病毒至少已在10个省(区)的番茄植株上检测到[3,8,21-29],XU等[41]研究认为STV已经成为我国番茄上第四大流行病毒。本研究结果表明,早在2015年,海南陵水和昌江的田间番茄上就已存在STV。到2021年,STV已在陵水、文昌、昌江、三亚、临高、定安、乐东、儋州、琼海共9个市(县)检测到,STV在海南的发生分布范围逐渐扩大,发病率逐年上升。因为STV是严格的种传病毒,有必要开展制种田中番茄的抽样检测工作,对番茄种子实行严格检疫,同时加强STV的田间监测,及时清理早期感病植株,对STV的综合防控具有重要意义。

