质粒CRISPR/Cas9系统在马口鱼精原干细胞系基因编辑中的应用







摘要:【目的】检测质粒CRISPR/Cas9系统在马口鱼精原干细胞(ObSSCs)和斑马鱼胚胎中的基因编辑效果,为开展基因编辑的SSCs移植提供技术支撑,进而推动养殖鱼类基因编辑育种工作的快速发展。【方法】将靶向红色荧光蛋白(RFP)的gRNA整合至可用于体内外基因编辑的整合质粒pCas9-zU6sgRNA(带有支架序列的向导RNA,由来自斑马鱼的U6启动子驱动)和报告质粒pCVpf-gRNA(向导RNA)中,然后分别转染ObSSCs和显微注射斑马鱼胚胎,通过荧光显微镜观察和PCR检测质粒CRISPR/Cas9系统在ObSSCs及斑马鱼胚胎中的基因编辑效果。【结果】以整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA共转染正常ObSSCs及pCVpr质粒转染ObSSCs,在ObSSCs中能观察到绿色荧光,且在表达绿色荧光蛋白(GFP)的ObSSCs中观察到红色荧光信号明显减弱,而不转染质粒的ObSSCs未观察到绿色荧光信号;随着整合质粒pCas9-zU6sgRNA转染剂量由340 ng增加到410 ng,其基因编辑效率由0.10%增加到0.63%;此外,质粒CRISPR/Cas9系统在基因组中的编辑效率与在外源质粒中的编辑效率基本一致。为进一步检测质粒CRISPR/Cas9系统在体内的编辑效率,以整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA共注射斑马鱼胚胎,24 h后能观察到绿色荧光信号,而空白对照组和阴性对照组斑马鱼胚胎均未观察到绿色荧光信号。对ObSSCs和斑马鱼的基因编辑效果进行PCR验证,发现试验组均能检测到修复的GFP片段,而空白对照组未检测到修复的GFP片段。此外,整合质粒pCas9-zU6sgRNA在斑马鱼胚胎中的基因编辑效率显著高于ObSSCs(100% vs 0.63%)(Plt;0.05)。【结论】由整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA构成的质粒CRISPR/Cas9系统能在ObSSCs中直观评估基因编辑效率,且质粒CRISPR/Cas9系统在同为鲤科鱼类斑马鱼胚胎中的基因编辑效率高达100%。因此,质粒
CRISPR/Cas9系统可用于马口鱼和斑马鱼的sgRNA筛选,为创制养殖鱼类新品种(系)提供新思路。
关键词:马口鱼;精原干细胞(SSCs);基因编辑;质粒CRISPR/Cas9系统;斑马鱼
中图分类号:S917.4文献标志码:A 文章编号:2095-1191(2024)11-3381-11
Applications of plasmid CRISPR/Cas9 system in gene editing ofOpsariichthys bidens spermatogonial stem cell line
GU Kai-yan1,2, XU Hai-jing1,2, TAO Xin-ran1,2, WU Can1,2, WEI Jing3,GUI Lang1,2*, LI Ming-you1,2*
(1Key Laboratory of Integrated Rice-fish Farming, Ministry of Agriculture and Rural Affairs( Shanghai Ocean Univer‐sity), Shanghai 201306, China; 2Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources
(Shanghai Ocean University), Shanghai 201306, China; 3Integrative Science Center of Germplasm Creation in Western China Science City/Key Laboratory of Freshwater Fish Resources and Reproductive Development/School of Life Sciences, Southwest University, Chongqing 400715, China)
Abstract:【Objective】To test the gene editing effects of a plasmid CRISPR/Cas9 gene editing system in Opsariich‐thys bidens spermatogonial stem cells( ObSSCs) and zebrafish embryos, which could provide technical support for con‐ducting the transplantation of gene-edited SSCs and in turn, promote the rapid development of gene-edited breeding tech‐nology in farmed fish. 【Method】The gRNA targeting the red fluorescence protein fusion gene (RFP) was incorporated into the integrated plasmid pCas9-zU6sgRNA( a guide RNA with scaffold sequence, driven by the U6 promoter from ze‐brafish) and the reporter plasmid pCVpf-gRNA( guide RNA) for in vitro and in vivo gene editing, followed by transfec‐tion of ObSSCs and microinjection of zebrafish embryos. The gene editing effects of the plasmid CRISPR/Cas9 system in ObSSCs and zebrafish embryos were detected by fluorescence microscopy and PCR. 【Result】The integrated plasmid pCas9-zU6sgRNA and the reporter plasmid pCVpf-gRNA were co-transfected into ObSSCs and ObSSCs: pCVpr. Green fluorescence signals were observed in ObSSCs. A clear decrease in the red fluorescence signal was observed in cells ex‐pressing green fluorescence protein( GFP). No green fluorescence signal was observed in the non-transfected ObSSCs. As the dose of integrated plasmid pCas9-zU6sgRNA increased from 340 ng to 410 ng, the gene editing efficiency improved from 0.10% to 0.63%. The editing efficiency of the plasmid CRISPR/Cas9 system in genomes and exogenous plasmids was similar. To further test the editing efficiency of the plasmid CRISPR/Cas9 system in vivo, the integrated plasmid pCas9-zU6sgRNA and the reporter plasmid pCVpf-gRNA were co-injected into zebrafish embryos. Green fluorescence signals were observed after 24 h, while no green fluorescence signal was observed in the blank and negative control groups of zebrafish embryos. The gene editing effect in ObSSCs and zebrafish was detected by PCR. GFP fragments re‐paired were detected in all experimental groups but not in the blank control group. Compared to ObSSCs, the gene editing efficiency of the integrated plasmid pCas9-zU6sgRNA in zebrafish was significantly higher( 100% vs 0.63%)(Plt;0.05). 【Conclusion】The gene editing efficiency of the plasmid CRISPR/Cas9 system, consisting of the integrating plasmid pCas9-zU6sgRNA and the reporter plasmid pCVpf-gRNA, can be visually assessed in ObSSCs, which is up to 100% in zebrafish embryos of the same Cyprinidae family of fish. Consequently, the plasmid CRISPR/Cas9 system can be used to screen sgRNAs in Opsariichthys bidens and zebrafish, and provide a new way for the creation of new varieties of farmed
fish.
Key words: Opsariichthys bidens; spermatogonial stem cells( SSCs); gene editing; plasmid CRISPR/Cas9 system; Danio rerio
Foundation items: National Key Research and Development Program of China(2022YFD2401600); National Natu‐ral Science Foundation of China(32373130)
0 引言
【研究意义】精原干细胞(Spermatogonial stem cells,SSCs)是雄性体内唯一能将遗传信息传递给下一代的成体干细胞(Nagano and Yeh,2013;Valli et al.,2014),通过自我更新维持数量充足、分化成熟的功能性精子(de Kretser et al.,1998)。CRISPR/Cas9基因编辑技术已广泛应用于养殖鱼类的遗传改良,有效推进了鱼类新品种(系)的创制进程(Cui et al.,2017;Dong et al.,2023;Gan et al.,2023;Kuang et al.,2023)。然而,受限于鱼类漫长的发育周期,如青鱼(Mylopharyngodon piceus)性成熟需要6~7年,草鱼(Ctenopharyngodon idella)性成熟需要4年(Gur et al.,2000;欧琳,2021),导致直接利用CRISPR/Cas9系统进行养殖鱼类品种改良仍费时费力。借助质粒CRISPR/Cas9系统对SSCs进行基因编辑,能跨越鱼类漫长的发育周期,对促进基因编辑创制养殖鱼类新品种(系)具有重要意义。【前人研究进展】SSCs由原始生殖细胞分化而来(徐红艳等,2010;张国栋,2018),其中精子的发生是将遗传信息稳定传递给下一代的重要过程(Han et al.,2019;刘源壹等,2023)。目前,鱼类SSCs的研究进展相对较缓慢(陈秋宇和安立龙,2015),获取能长期稳定培养的养殖鱼类SSCs依然极具挑战性。在青鳉(Oryzias latipes)中,首次建立了能长期稳定培养的精原干细胞系SG3,且该细胞系在体外诱导后可完成减数分裂并产生精子(Hong et al.,2004)。随后,诸多学者开始在其他鱼类上进行探索,但发现其他鱼类培养的SSCs体外维系时间很难超过1个月,包括斑马鱼(Danio rerio)(Kawasaki et al.,2012)、罗非鱼(Oreochromis niloti‐cus)(dos Santos Nassif Lacerda et al.,2014)、牙鲆(Paralichthys olivaceus)(Zhou et al.,2021)、黄鳝(Monopterus albus)(Sun et al.,2022)及中华乌塘鳢(Bostrychus sinensis)(Zhang et al.,2022b)等。本研究团队通过无滋养层细胞培养技术突破了养殖鱼类SSCs无法长期培养的瓶颈,在马口鱼(Opsariichthys bidens)上建立了首个能体外长期稳定培养的马口鱼SSCs(ObSSCs),可在体外培养条件下诱导产生游动的精子(Chen et al.,2022)。此外,参照青鳉精原干细胞系SG3的方法,Zhong等(2022)、Gu等(2023)成功建立了能长期培养的石斑鱼(Epinephelus coioi‐des)SSCs和长江刀鲚(Coilia nasus)SSCs。Zhang等(2023)研究证实,通过质粒CRISPR/Cas9系统能简便、高效地基因编辑长期稳定培养的青鳉精原干细胞系SG3。可见,以质粒CRISPR/Cas9系统创制新品种(系)可不受鱼类繁殖周期的限制。【本研究切入点】基于长期稳定培养的鱼类SSCs研究体外分化产生基因编辑的功能性配子,能有效促进以基因编辑技术创制养殖鱼类新品种(系)技术的发展,但至今未见质粒CRISPR/Cas9系统应用于养殖鱼类SSCs的研究报道。【拟解决的关键问题】基于前期成功建立的ObSSCs,结合无缝克隆及细胞转染,筛选富集基因编辑的ObSSCs,为开展基因编辑的SSCs移植提供技术支撑,进而推动养殖鱼类基因编辑育种工作的快速发展。
1 材料与方法
1. 1 试验材料
ObSSCs及稳定表达红色荧光蛋白(RFP)的ObSSCs均由水产遗传资源发掘与利用教育部重点实验室提供。参照Chen等(2022)的研究方法,解剖并切碎7尾6月龄雄性马口鱼(体长约10 cm)的睾丸组织,采用胰蛋白酶和胶原酶消化分离睾丸组织以获得单个睾丸细胞,适当培养获得稳定的ObSSCs,在2年的培养过程中传代超过100代;将pCVpr质粒转染至ObSSCs中(Zhao et al.,2012),通过药物筛选出表达RFP的单细胞,转移至96孔细胞培养板中培养,以获得稳定表达RFP的单克隆ObSSCs。模式生物斑马鱼野生型AB品系暂养于28.5 ℃水循环系统中,光照周期为14 h光照/10 h黑暗,早晚投喂丰年虾,使用配种缸对亲鱼进行配对并收集胚胎。试验过程严格按照《世界医学协会赫尔辛基宣言》进行操作,且经上海海洋大学动物保育与使用委员会批准,批准号SHOU-2023-031。
1. 2 试验方法
1. 2. 1 设计靶向RFP的gRNA 根据pCVpr质粒的RFP序列设计gRNA靶向序列(Zhao et al.,2012),使用Centre for Organismal Studles网站(http://crispr.cos.uni-heidelberg.de/index.html)设计gRNA,物种选择与马口鱼同为理科的斑马鱼,根据评分筛选合适的CRISPR/Cas9靶位点;构建马口鱼基因组本地数据库(Xu et al.,2022),通过BLAST比对分析,筛选出最佳的CRISPR/Cas9靶向序列gRNA。
1. 2. 2 构建靶向RFP的CRISPR/Cas9系统质粒
在整合质粒pCas9-zU6sgRNA中,CMV启动子驱动Cas9表达,来自斑马鱼的U6启动子驱动sgRNA(带有支架序列的向导RNA)表达,SV40启动子驱动新霉素抗性基因(筛选富集基因编辑的细胞)表达(Zhang et al.,2023)。以整合质粒pCas9-zU6sgNtsf1为骨架,通过无缝克隆试剂盒(ABclonal MultiF Seamless Assembly,RK21020)将针对RFP的gRNA(向导RNA)序列连接至整合质粒中pCas9-zU6sgNtsf1(图1)。同时,以pCVpf为质粒骨架(Zhang et al.,2023)构建含针对RFP的gRNA靶序列报告质粒pCVpf-gRNA,用于监测整合质粒pCas9-zU6sgRNA的基因编辑效果(图1)。在质粒CRISPR/Cas9系统介导的基因组切割修复过程中,报告质粒pCVpf-gRNA通过同源重组修复绿色荧光蛋白(GFP)全编码序列,恢复GFP表达,而达到监测整合质粒pCas9-
zU6sgRNA基因编辑的效果。
以pCas9-zU6sgRNA/pCVpf为模板,采用含gRNA的引物(表1)对编码U6/PuroR序列和scaffoldSal/GFP序列的目的片段进行PCR扩增。PCR反应体系20.0 µL:DNA模板1.0 µL,上、下游引物(10 nmol/L)各0.5 µL,2×Taq酶混合液/高保真酶10.0 µL,去离子水8.0 µL。扩增程序:95 ℃预变性20 s;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 80 s,进行35个循环;72 ℃延伸7 min。PCR扩增产物通过1%或2%琼脂糖凝胶电泳进行检测(Chen et al.,2021)。
1. 2. 3 细胞培养 将ObSSCs冻存管取出,37 ℃水浴锅处理2 min使细胞迅速解冻,800×g离心4 min后以ESM4培养基重悬细胞,然后接种至由0.1%明胶包被的24孔细胞培养板中,置于28 ℃培养箱培养,每隔2 d传代1次(Chen et al.,2022)。参照Kan等(2022)、Gu等(2023)的方法配制ESM4培养基,其主要成分:DMEM 500.0 mL(pH 7.8),青鳉胚胎提取物(MEE)0.5 mL[MEE提取参照Hong等(1996,2004)的方法制备],100 ng/mL碱性成纤维细胞生长因子(bFGF)10 µL,澳洲胎牛血清(FBS)75.0 mL,2 µmol/L亚硒酸钠0.5 mL,海鲈血清5.0 mL,50 mmol/L 2-巯基乙醇2.0 mL,青链霉素混合液5.0 mL,L-谷氨酰胺5.0 mL,丙酮酸钠5.0 mL,非必需氨基酸5.0 mL。ESM4培养基经0.22 µm过滤器过滤后使用。
1. 2. 4 细胞转染 通过TransIT-X2转染试剂(美国Mirus Bio公司)将不同配比(340∶160或410∶190,ng/ng)的整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA共转染ObSSCs(整合质粒和报告质粒的摩尔比为1∶1),转染24~72 h后,在Nikon ECLIPSE Ti倒置显微镜(10×、20×和40×放大倍数)下进行观察。使用含1 mg/mL G418的ESM4培养基再培养7 d,富集成功转染的ObSSCs,在28 ℃下以胰蛋白酶-EDTA(美国Gibco公司)进行消化,经磷酸盐缓冲液(PBS)洗涤后收集ObSSCs(Gu et al.,2023),然后提取ObSSCs基因组DNA进行PCR鉴定。由于以整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA共转染正常ObSSCs,仅能检测sgRNA在外源质粒中的基因编辑效果。为进一步确认该系统在基因组中的编辑效率,还将整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA共转染基因组中整合了pCVpr序列的ObSSCs(Chen et al.,2022),转染与检测操作过程与正常ObSSCs一致。
1. 2. 5 显微注射 以整合质粒pCas9-zU6sgRNA(100 pg)与报告质粒pCVpf-gRNA(50 pg)同时显微注射斑马鱼1-细胞期胚胎卵黄(Porazinski et al.,2010;Shankaran et al.,2017),注射24 h后,在尼康AZ100M体视显微镜(10×和20×放大倍数)下连续观察胚胎绿色荧光信号。
1. 2. 6 测序分析 胚胎显微注射24 h后,提取胚胎基因组DNA。以20.0 μL NaOH(50 mmol/L)处理胚胎20 min(95 ℃,每10 min振荡1次),充分裂解胚胎细胞;使用2.0 μL Tris-HCl(pH 8.0)中和NaOH后,12000 r/min离心2 min,收集上清液。通过PCR检测整合质粒pCas9-zU6sgRNA的编辑效果:95 ℃预变性20 s;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 80 s,进行35个循环;72 ℃延伸7 min。使用2%琼脂糖凝胶电泳检测PCR扩增产物,以胶回收试剂盒(TaKaRa)回收目的片段,通过TA克隆将目的片段插入pGEM-T Easy载体,然后转化大肠杆菌(Escherichia coli)DH5α感受态细胞,37 ℃培养14~16 h后挑取单菌落进行菌落PCR鉴定。将阳性菌液和引物GFP Xba I-R送至生工生物工程(上海)股份有限公司测序,并使用Vector NTI对测序结果进行比对分析。
1. 3 统计分析
试验数据采用GraphPad Prism 8.0.1进行统计分析并制图。
2 结果与分析
2. 1 整合质粒pCas9-zU6sgRNA在ObSSCs中的剂量评估及优化
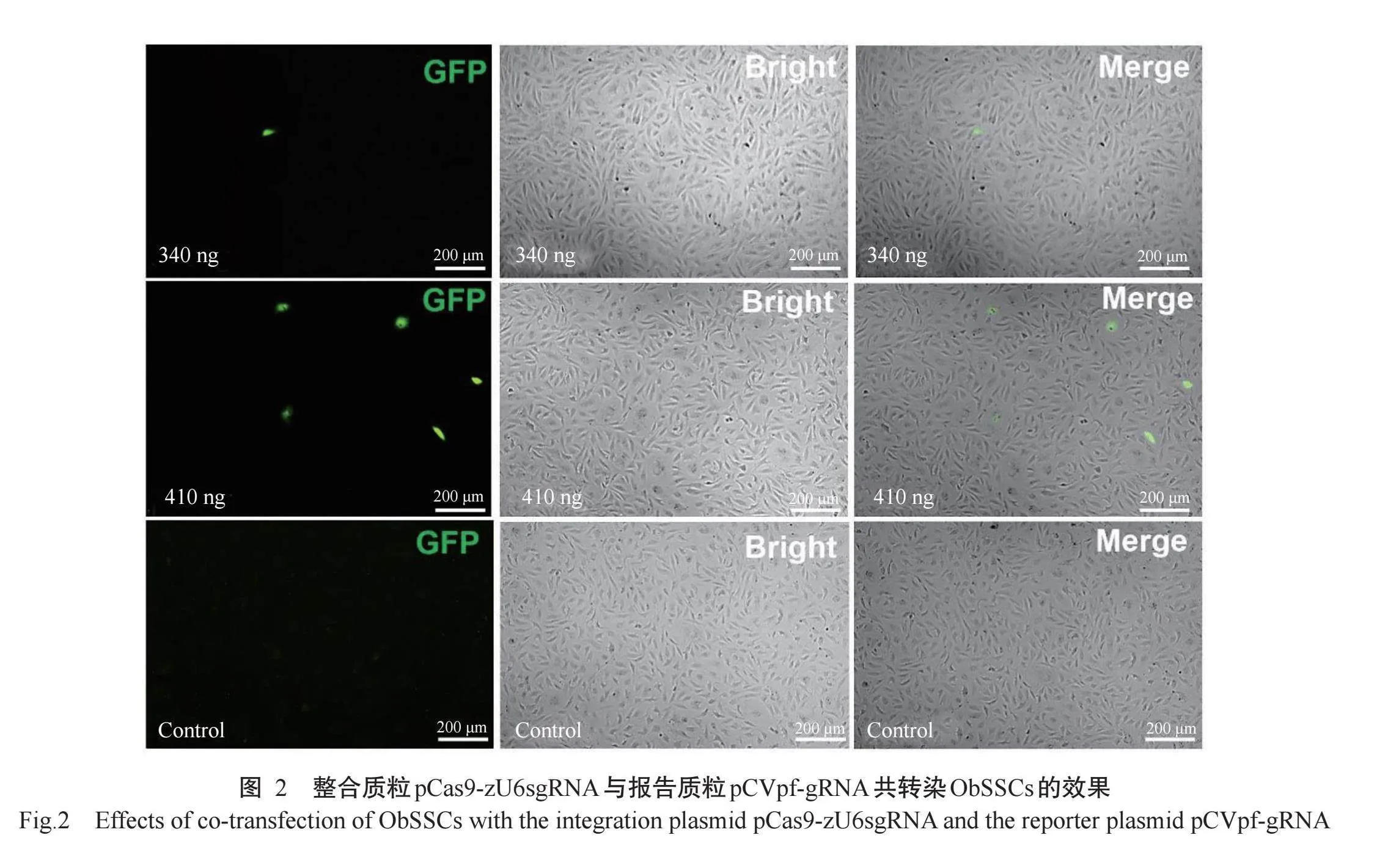
待24孔细胞培养板中的ObSSCs培养至汇合度达70%时,以不同配比的整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA共转染ObSSCs,结果显示,转染后的ObSSCs能正常生长,细胞形态、生长状态与对照组的ObSSCs无明显差异。ObSSCs在28 ℃培养箱中培养24~72 h后观察转染效率,结果在荧光显微镜下可观察到少量表达GFP而显示绿色荧光的ObSSCs,对照组则未观察到具有绿色荧光信号的ObSSCs(图2)。此外,以不同配比的整合质粒与报告质粒共转染ObSSCs,发现GFP阳性细胞数量存在明显差异(图3),当整合质粒pCas9-zU6sgRNA的转染剂量由340 ng增加到410 ng时,GFP阳性细胞百分比由0.10%显著增加到0.63%(Plt;0.05,下同)。
2. 2 整合质粒pCas9-zU6sgRNA体外基因敲除与同源重组修复效果
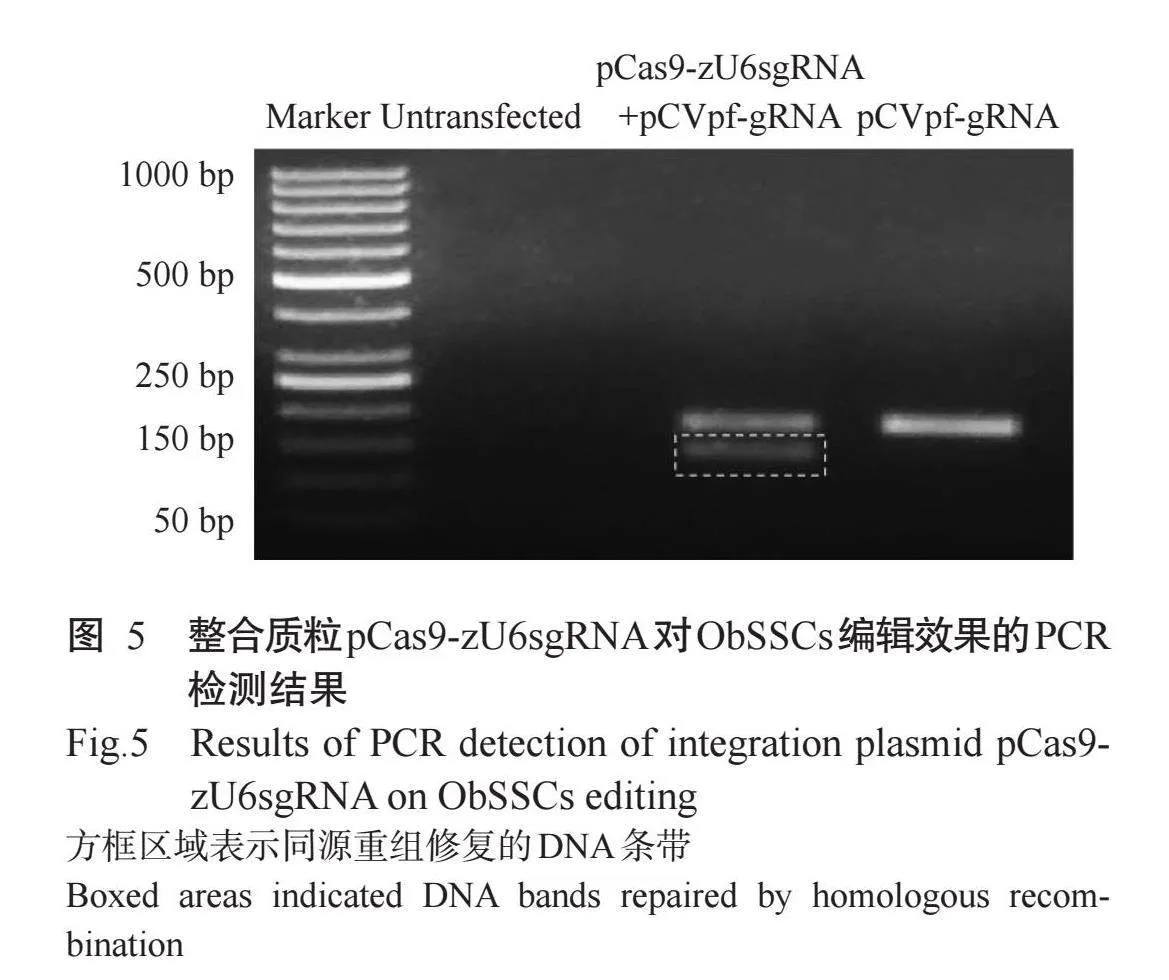
为了从基因组DNA层面评估整合质粒pCas9-zU6sgRNA对ObSSCs的编辑效果,于转染72 h后,使用1 mg/mL G418富集表达GFP的ObSSCs,结果显示,表达GFP的ObSSCs数量明显增多(图4)。同时提取ObSSCs基因组DNA,通过PCR检测整合质粒pCas9-zU6sgRNA的编辑效果,结果表明,空白对照组的ObSSCs未检测到GFP相关片段,仅转染报告质粒pCVpf-gRNA阴性对照组的ObSSCs检测到未经修复的GFP片段,在共转染整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA的ObSSCs中GFP全编码序列被修复,GFP正常表达而检测到对应的目的条带(图5)。
2. 3 整合质粒pCas9-zU6sgRNA在ObSSCs中的基因编辑效果
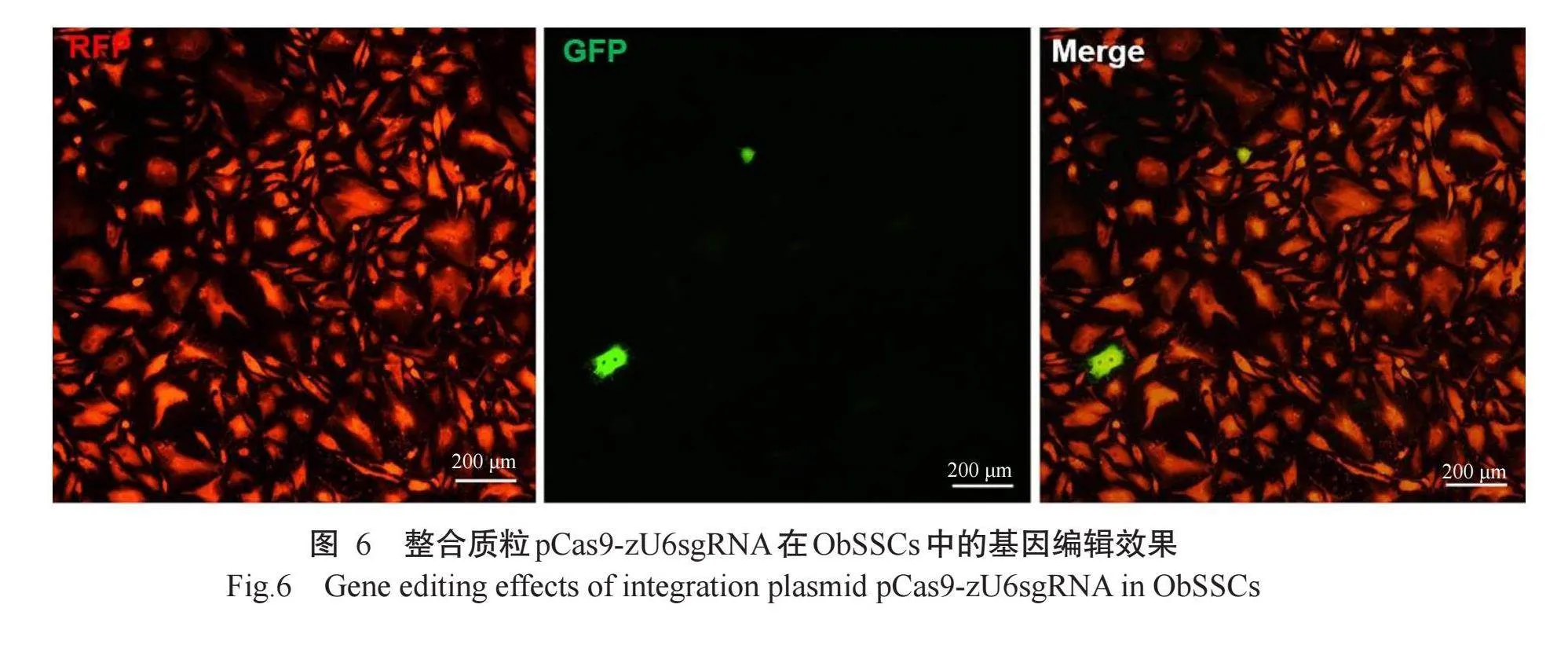
为进一步验证质粒CRISPR/Cas9系统在基因组中的编辑效率及其在外源质粒中的编辑效率差异,以410 ng整合质粒pCas9-zU6sgRNA与190 ng报告质粒pCVpf-gRNA共转染稳定表达RFP的ObSSCs,转染24~72 h后观察到表达GFP的ObSSCs;相对于未表达GFP的ObSSCs,表达GFP的ObSSCs红色荧光信号明显减弱(图6),进一步证实在共转染整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA的ObSSCs中GFP全编码序列被修复,GFP得以正常表达,即整合质粒pCas9-zU6sgRNA在ObSSCs中有明显的编辑效果。此外,对比整合质粒pCas9-zU6sgRNA在pCVpr质粒转染ObSSCs和正常ObSSCs中的基因编辑效率,结果(图7)显示,质粒CRISPR/Cas9系统在基因组中的编辑效率与在外源质粒中的编辑效率基本一致。
2. 4 整合质粒pCas9-zU6sgRNA体内基因编辑效率
为测试质粒CRISPR/Cas9系统在体内的基因编辑效率,选择与马口鱼同为鲤科的斑马鱼胚胎进行体内验证。以共注射整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA的斑马鱼胚胎为试验组,不注射质粒的胚胎为空白对照组,只注射报告质粒pCVpf-gRNA(50 pg)的胚胎为阴性对照组,检测整合质粒pCas9-zU6sgRNA的体内基因编辑效果,结果(图8)显示,注射24 h后,在空白对照组和阴性对照组斑马鱼胚胎均未观察到绿色荧光信号,而在试验组斑马鱼胚胎的动物极区观察到明显的绿色荧光信号。
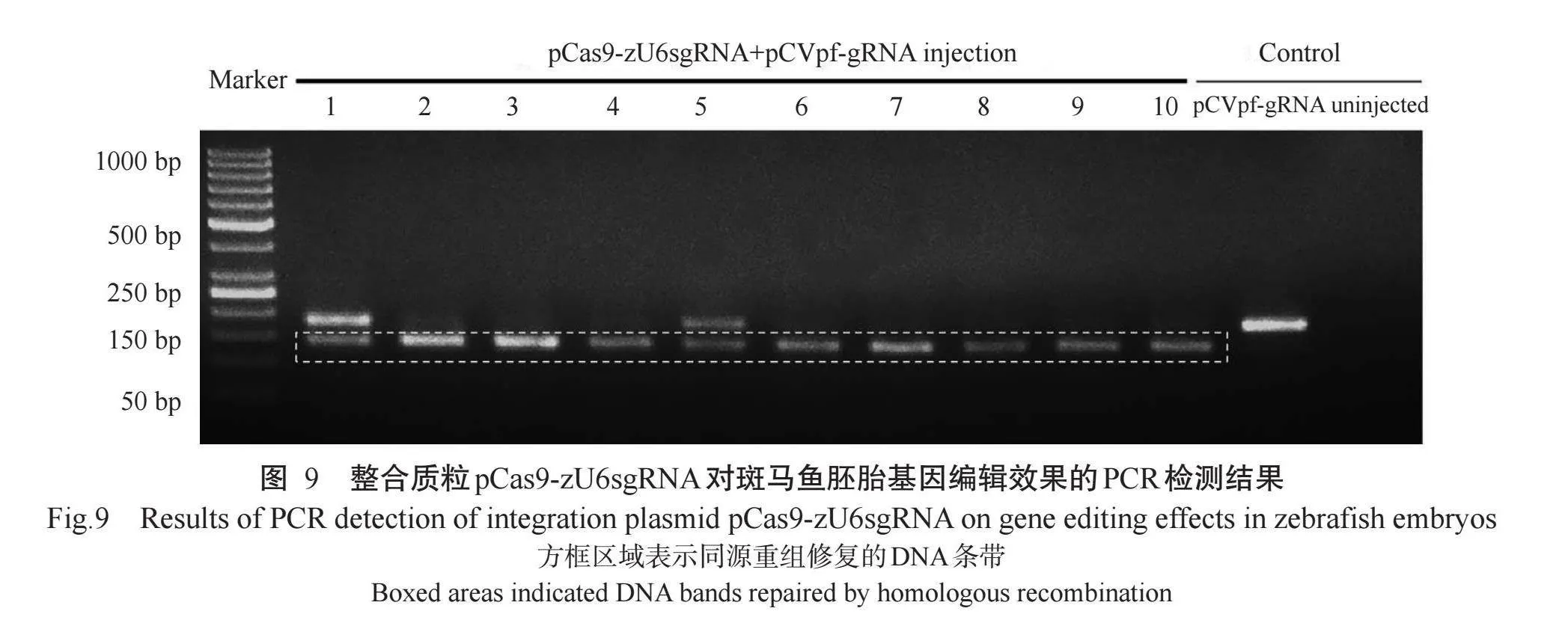
为进一步确认GFP序列是否在动物体内被成功修复,提取斑马鱼胚胎基因组DNA进行PCR鉴定,结果(图9)显示:空白对照组斑马鱼胚胎未检测到GFP片段;只注射报告质粒pCVpf-gRNA阴性对照组斑马鱼胚胎检测到未修复的GFP片段,但不存在经基因编辑修复的GFP片段;在10枚共注射整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA的斑马鱼胚胎中均检测到同源重组修复的双链DNA条带,表明报告质粒pCVpf-gRNA在斑马鱼胚胎内发生了同源重组修复,GFP全编码序列被修复,GFP正常表达,且整合质粒pCas9-zU6sgRNA在斑马鱼胚胎中的编辑效率达100%。为了直观评估报告质粒pCVpf-gRNA在斑马鱼胚胎内发生同源重组修复的效率,统计表达GFP全编码序列的斑马鱼胚胎比例,结果发现,空白对照组和阴性对照组均未观测到表达GFP全编码序列的斑马鱼胚胎,而共注射整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA试验组斑马鱼胚胎表达GFP全编码序列的百分比达100%。此外,对比整合质粒pCas9-zU6sgRNA在马口鱼SSCs和斑马鱼胚胎中的基因编辑效率,发现整合质粒pCas9-zU6sgRNA在斑马鱼胚胎中的基因编辑效率显著高于ObSSCs(100% vs 0.63%)。
同时进一步确认GFP序列是否在动物体内被正确修复,通过菌落PCR鉴定及送至生工生物工程(上海)股份有限公司测序分析,以检验报告质粒pCVpf-gRNA是否完成同源重组修复,结果(图10)显示,在空白对照组和阴性对照组斑马鱼胚胎中均检测到gRNA和同源臂序列,而在共注射整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA的斑马鱼胚胎(2#)中未检测到gRNA和同源臂序列,表明报告质粒pCVpf-gRNA在斑马鱼胚胎内已发生同源重组修复,修复了GFP全编码序列。
3 讨论
目前,基因工程育种已成为水产种质创新的重要途径,但受限于养殖鱼类的繁育周期,致使基因工程育种的进展缓慢。借助体外基因编辑,将SSCs的基因编辑技术与体外诱导技术相结合,有利于基因编辑精子的获取,进而加速养殖鱼类基因编辑新品种(系)的创制。本研究构建了用于体外和体内基因编辑的整合质粒pCas9-zU6sgRNA和报告质粒pCVpf-gRNA,为鱼类SSCs基因编辑技术及诱导SSCs产生基因编辑精子提供了技术支持。在实现CRISPR/Cas9系统的几种方案中,制备Cas9/sgRNA核糖核蛋白(RNP)复合物需要特定的设备和技术,以及成本高昂的试剂(Liu et al.,2018;Hamar and Kültz,2021;Strømsnes et al.,2022),且体外合成的sgRNA易降解(Dehler et al.,2016);体外合成mRNA注射则需要成本昂贵的体外转录试剂,且合成的mRNA极易降解(Pan et al.,2022;陈若雪等,2024)。相比之下,整合质粒pCas9-zU6sgRNA和报告质粒pCVpf-gRNA通过替换gRNA及细胞转染即可获得编辑的ObSSCs,且通过PCR或显微镜观察GFP信号可直接检测编辑效率;整合质粒pCas9-zU6sgRNA还可有效编辑斑马鱼胚胎。相对于使用RNP复合物进行基因编辑,质粒CRISPR/Cas9系统具有经济、简单等优点,可作为未来获取基因编辑配子的主要途径,以加快创制养殖鱼类新品种(系)。
U6启动子作为一种聚合酶III启动子,已广泛应用于shRNA或sgRNA的表达(Kim et al.,2012;Ma et al.,2014)。哺乳动物的U6启动子可基因编辑鲤科鱼类细胞系,如鲤(Cyprinus carpio)尾鳍细胞系(KF-1)(Zhao et al.,2016)和草鱼肾细胞系CIK(Ma et al.,2018),但其基因编辑效率尚未得到评估。斑马鱼的U6启动子可基因编辑罗非鱼细胞(Boonanuntanasarn et al.,2009)和鲑(Salmo salar)细胞(Escobar-Aguirre et al.,2019)。为进一步提高基因编辑效率,本研究选择与马口鱼同为鲤科的斑马鱼U6启动子进行试验,以不同比例的整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA共转染ObSSCs后,观察到较弱的绿色荧光信号。G418富集编辑的ObSSCs通过PCR可检测到报告质粒的同源重组修复。此外,质粒CRISPR/Cas9系统在正常ObSSCs和pCVpr质粒转染ObSSCs中的基因编辑效果基本一致,表明质粒CRISPR/Cas9系统在基因组中的编辑效率与在外源质粒中的编辑效率相似,提示质粒CRISPR/Cas9系统也可应用于其他养殖鱼类细胞系,并根据报告质粒的编辑效率确认所设计sgRNA在该养殖鱼类SSCs中的基因编辑效率。细胞内mRNA和蛋白的代谢与降解需要一定时间,虽然质粒CRISPR/Cas9系统在pCVpr质粒转染ObSSCs基因组上已完成编辑,但其细胞内仍存在具备活性的RFP及具备转录活性的RFP基因编码序列,因此仅能观察到未新增转录本而导致红色荧光信号减弱及恢复编码GFP序列而显示绿色荧光信号的ObSSCs,但未观察到红色荧光完全消失只显示绿色荧光的ObSSCs。
为进一步验证质粒CRISPR/Cas9系统在体内的基因编辑效果,以整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA共注射斑马鱼胚胎,在斑马鱼胚胎中同样可观察到明显的绿色荧光信号。本研究发现,整合质粒pCas9-zU6sgRNA在斑马鱼胚胎的基因编辑效率显著高于ObSSCs(100% vs 0.63%),可能是多种因素所致:(1)细胞转染试剂仍无法保证整合质粒在个性化干细胞系中具备较高的转染效率。使用TransIT-X2转染试剂对ObSSCs进行单质粒转染时的转染效率约20.00%,2个质粒同时转染同一细胞的概率为4.00%;其中0.63%的ObSSCs发生基因编辑,故基因编辑效率约15.75%。(2)U6启动子在异源物种中的活性同样无法保证(Das et al.,1987),因此本研究选择与马口鱼同为鲤科的斑马鱼U6启动子。(3)在胚胎注射时,注射后在单个胚胎中的少量细胞完成基因编辑即可显示绿色荧光,在细胞中则需单个细胞完成全部重组修复过程后才能显示绿色荧光。综上所述,质粒CRISPR/Cas9系统在ObSSCs和斑马鱼胚胎均实现了有效的基因编辑,为后续应用该系统实现SSCs基因编辑及借助体外诱导技术实现养殖鱼类基因编辑新品种(系)的快速创制打下了基础。
在养殖鱼类基因编辑育种研究中,设计的gRNA编辑效率低下或无效则会导致新品种(系)创制失败,而重新设计gRNA进行试验又需要遵循养殖鱼类的繁育周期。为直观、快速地评估ObSSCs中sgRNA的基因编辑效率,本研究检测了报告质粒pCVpf-gRNA在ObSSCs和斑马鱼胚胎中经同源重组修复后GFP正常表达的效果。报告质粒pCVpf-gRNA是将gRNA序列与编码GFP的同源臂序列同时插入GFP编码序列,在质粒CRISPR/Cas9系统介导的基因组切割修复过程中,报告质粒pCVpf-gRNA通过同源重组修复GFP全编码序列,恢复GFP正常表达。因此,可通过观察绿色荧光信号直接快速评估sgRNA的基因编辑效率,而节省时间、经济和人力成本。
近年来,SSCs已广泛应用于鱼类生殖细胞移植(Zhang et al.,2022a)和体外诱导(Zhang et al.,2022b)。将鱼类生殖细胞系的基因编辑技术与移植技术或体外诱导技术相结合,可缩短创制养殖鱼类新品种(系)的时间(Jin et al.,2021)。在稀有鮈鲫(Gobiocy‐pris rarus)中,通过将SSCs移植到斑马鱼性腺中,可获得CRISPR/Cas9编辑的功能性精子(Zhang et al.,2022a);在马口鱼精巢原代细胞的诱导下,ObSSCs能发生减数分裂而产生游动的精子(Chen et al.,2022)。可见,将SSCs的基因编辑技术与移植技术或体外诱导技术相结合,有利于基因编辑精子的获得。本研究成功富集了基因编辑的ObSSCs,可作为供体应用于移植或体外诱导技术研究,为获得马口鱼基因编辑的功能性精子及加快基因编辑创制养殖鱼类新品种(系)提供新的见解。
4 结论
由整合质粒pCas9-zU6sgRNA与报告质粒pCVpf-gRNA构成的质粒CRISPR/Cas9系统能在ObSSCs中直观评估基因编辑效率,且质粒CRISPR/Cas9系统在同为鲤科鱼类斑马鱼胚胎中的基因编辑效率高达100%。因此,质粒CRISPR/Cas9系统可用于马口鱼和斑马鱼的sgRNA筛选,为创制养殖鱼类新品种(系)提供新思路。
参考文献((References)):
陈秋宇,安立龙. 2015. 鱼类精原干细胞的研究进展[J]. 广东农业科学,42(6):105-110.[ Chen Q Y,An L L. 2015. Advances in fish spermatogonial stem cells[J]. Guang‐dong Agricultural Sciences,42(6):105-110.] doi:10.3969/
j.issn.1004-874X.2015.06.020.
陈若雪,蒋月雯,王梦洋,许朝然,梁晶婕,陈天圣. 2024. 基于密码子优化mSaCas9蛋白的重组表达与应用[J]. 广东海洋大学学报,44(4):19-26.[ Chen R X,Jiang Y W,Wang M Y,Xu Z R,Liang J J,Chen T S. 2024. Recombinant expression and application of mSaCas9 protein based on codon-optimization[J]. Journal of Guangdong Ocean Uni‐versity,44(4):19-26.] doi:10.3969/j.issn.1673-9159.2024.04.003.
刘源壹,李昕俞,张磊,芒来,杜明. 2023. 动物精原干细胞体外培养研究进展[J]. 饲料研究,46(14):144-150.[ Liu Y Y,Li X Y,Zhang L,Mang L,Du M. 2023. Research prog‐ress on in vitro culture of animal spermatogonial stem cells[J]. Feed Research,46(14):144-150.] doi:10.13557/j.cnki. issn1002-2813.2023.14.028.
欧琳. 2021. 初探草鱼与赤眼鳟性成熟[J]. 水产养殖,(5):56-58.[ Ou L. 2021. A preliminary study of sexual matura‐tion in Ctenopharyngodon idellus and Squaliobarbus cur‐riculu[sJ]. Journal of Aquaculture,(5):56-58.] doi:10.3969/j.issn.1004-2091.2021.05.018.
徐红艳,李名友,桂建芳,洪云汉. 2010. 鱼类生殖细胞[J]. 中国科学:生命科学,40(2):124-138.[ Xu H Y,Li M Y,Gui J F,Hong Y H. 2010. Fish germ cells[J]. Science China:Life Sciences,40(2):124-138.] doi:10.1007/s11427- 010-0058-8.
张国栋. 2018. 哺乳动物原始生殖细胞的体内特化和体外培养[J]. 中国生物化学与分子生物学报,34(5):467-472. [Zhang G D. 2018. In vivo specialisation and in vitro cul‐ture of mammalian primordial germ cells[J]. Chinese Jour‐nal of Biochemistry and Molecular Biology,34(5):467-472.] doi:10.13865/j.cnki.cjbmb.2018.05.02.
Boonanuntanasarn S,Panyim S,Yoshizaki G. 2009. Usage of putative zebrafish U6 promoters to express shRNA in Nile tilapia and shrimp cell extracts[J]. Transgenic Research,18:323-325. doi:10.1007/s11248-009-9249-0.
Chen X,Kan Y T,Zhong Y,Jawad M,Wei W B,Gu K Y,Gui L,Li M Y. 2022. Generation of a normal long-term-cultured Chinese hook snout carp spermatogonial stem cell line capable of sperm production in vitro[J]. Biology,11(7):1069. doi:10.3390/biology11071069.
Chen X,Song P,Xia J,Guo J,Shi Y H,Zhong Y,Li M Y. 2021. Evolutionarily conserved boule and dazl identify germ cells of Coilia nasus[J]. Aquaculture and Fisheries,8(3):244-251. doi:10.1016/j.aaf.2021.10.001.
Cui Z K,Liu Y,Wang W W,Wang Q,Zhang N,Lin F,Wang N,Shao C W,Dong Z D,Li Y Z,Yang Y M,Hu M Z,Li H L,Gao F T,Wei Z F,Meng L,Liu Y,Wei M,Zhu Y,Guo H,Cheng C H K,Schartl M,Chen S L. 2017. Genome edi-ting reveals dmrt1 as an essential male sex-determining gene in Chinese tongue sole( Cynoglossus semilaevis)[J]. Scientific Reports,7:42213. doi:10.1038/srep42213.
Das G,Henning D,Reddy R. 1987. Structure,organization,and transcription of Drosophila U6 small nuclear RNA genes[J]. Journal of Biological Chemistry,262(3):1187-1193. doi:10.1016/s0021-9258(19)75769-x.
de Kretser D M,Loveland K L,Meinhardt A,Simorangkir D,Wreford N. 1998. Spermatogenesis[J]. Human Reproduc‐tion,13(S1):1-8. doi:10.1093/humrep/13.suppl_1.1.
Dehler C E,Boudinot P,Martin S A M,Collet B. 2016. Deve-lopment of an efficient genome editing method by CRISPR/Cas9 in a fish cell line[J]. Marine Biotechnology,18:449-452. doi:10.1007/s10126-016-9708-6.
Dong Q,Nie C H,Wu Y M,Zhang D Y,Wang X D,Tu T,Jin J,Tian Z Y,Liu J Q,Xiao Z Y,Wan S M,Gao Z X. 2023. Generation of blunt snout bream without intermuscular bones by runx2b gene mutation[J]. Aquaculture,567:739263. doi:10.1007/s10126-016-9708-6.
dos Santos Nassif Lacerda S M,Costa G M J,de Franca L R. 2014. Biology and identity of fish spermatogonial stem cell[J]. General and Comparative Endocrinology,207:56-65. doi:10.1016/j.ygcen.2014.06.018.
Escobar-Aguirre S,Arancibia D,Escorza A,Bravo C,Andrés M E,Zamorano P,Martínez V. 2019. Development of a bicistronic vector for the expression of a CRISPR/Cas9-mCherry system in fish cell lines[J]. Cells,8(1):75. doi:10.3390/cells8010075.
Gan R H,Li Z,Wang Z W,Li X Y,Wang Y,Zhang X J,Tong J F,Wu Y,Xia L Y,Gao Z X,Zhou L,Gui J F. 2023. Crea-tion of intermuscular bone-free mutants in amphitriploid gibel carp by editing two duplicated runx2b homeologs[J]. Aquaculture,567:739300. doi:10.1016/j.aquaculture. 2023.739300.
Gu K Y,Zhang Y,Zhong Y,Kan Y T,Jawad M,Gui L,Ren M C,Xu G C,Liu D,Li M Y. 2023. Establishment of a Coilia nasus spermatogonial stem cell line capable of spermato‐genesis in vitro[J]. Biology,12(9):1175. doi:10.3390/bio-logy12091175.
Gur G,Melamed P,Gissis A,Yaron Z. 2000. Changes along the pituitary-gonadal axis during maturation of the black carp,Mylopharyngodon piceus[J]. The Journal of Experimental Zoology,286(4):405-413.
Hamar J,Kültz D. 2021. An efficient vector-based CRISPR/Cas9 system in an Oreochromis mossambicus cell line using endogenous promoters[J]. Scientific Reports,11:7854. doi:10.1101/2020.08.04.237065.
Han J Y,Miao Y L,Hua J L,Li Y,Zhang X,Zhou J L,Li N,Zhang Y,Zhang J Y,Liu Z H. 2019. Porcine pluripotent stem cells:Progress,challenges and prospects[J]. Fron‐tiers of Agricultural Science and Engineering,6(1):8-27. doi:10.15302/j-fase-2018233.
Hong Y H,Liu T M,Zhao H B,Xu H Y,Wang W J,Liu R,Chen T S,Deng J R,Gui J F. 2004. Establishment of a nor‐mal medakafish spermatogonial cell line capable of sperm production in vitro[J]. Proceedings of the National Aca-demy of Sciences of the United States of America,101(21):8011-8016. doi:10.1073/pnas.0308668101.
Hong Y H,Winkler C,Schartl M. 1996. Pluripotency and diffe-rentiation of embryonic stem cell lines from the medaka-fish( Oryzias latipes)[J]. Mechanisms of Development,60(1):33-44. doi:10.1016/s0925-4773(96)00596-5.
Jin Y H,Robledo D,Hickey J M,McGrew M J,Houston R D. 2021. Surrogate broodstock to enhance biotechnology research and applications in aquaculture[J]. Biotechnology Advances,49:107756. doi:10.1016/j.biotechadv.2021.10 7756.
Kan Y T,Zhong Y,Jawad M,Chen X,Liu D,Ren M C,Xu G C,Gui L,Li M Y. 2022. Establishment of a Coilia nasus gonadal somatic cell line capable of sperm induction in vitro[J]. Biology,11(7):1049. doi:10.3390/biology1107 1049.
Kawasaki T,Saito K,Sakai C,Shinya M,Sakai N. 2012. Pro‐duction of zebrafish offspring from cultured spermatogo‐nial stem cells[J]. Genes to Cells,17(4):316-325. doi:10. 1111/j.1365-2443.2012.01589.x.
Kim N Y,Baek J Y,Choi H S,Chung I S,Shin S H,Lee J I,Choi J Y,Yang J M. 2012. Short-hairpin RNA-mediated gene expression interference in trichoplusiani cells[J]. Jour‐nal of Microbiology and Biotechnology,22(2):190-198. doi:10.4014/jmb.1108.08045.
Kuang Y Y,Zheng X H,Cao D C,Sun Z P,Tong G X,Xu H,Yan T,Tang S Z,Chen Z X,Zhang T T,Zhang T,Dong L,Yang X X,Zhou H J,Guo W L,Sun X W. 2023. Generate a new crucian carp (Carassius auratus) strain without intermuscular bones by knocking out bmp6[J]. Aquacul‐ture,569:739407. doi:10.1016/j.aquaculture.2023.739407.
Liu Q Z,Yuan Y M,Zhu F,Hong Y H,Ge R W. 2018. Efficient genome editing using CRISPR/Cas9 ribonucleoprotein approach in cultured medaka fish cells[J]. Biology Open,7(8):bio035170. doi:10.1242/bio.035170.
Ma H M,Wu Y G,Dang Y,Choi J G,Zhang J L,Wu H Q. 2014. Pol III promoters to express small RNAs:Deli-neation of transcription initiation[J]. Molecular Therapy-Nucleic Acids,3(5):e161. doi:10.1038/mtna.2014.12.
Ma J,Fan Y D,Zhou Y,Liu W Z,Jiang N,Zhang J M,Zeng L B. 2018. Efficient resistance to grass carp reovirus infec‐tion in JAM-A knockout cells using CRISPR/Cas9[J]. Fish amp; Shellfish Immunology,76:206-215. doi:10.1016/j.fsi. 2018.02.039.
Nagano M C,Yeh J R. 2013. Chapter Three—The identity and fate decision control of spermatogonial stem cells:Where is the point of no return?[J]. Current Topics in Developmen‐tal Biology,102:61-95. doi:10.1016/B978-0-12-416024-8.00003-9.
Pan Q H,Luo J Z,Jiang Y W,Wang Z,Lu K,Chen T S. 2022. Efficient gene editing in a medaka (Oryzias latipes) cell line and embryos by SpCas9/tRNA-gRNA[J]. Journal of Zhejiang University-SCIENCE B,23:74-83. doi:10.1631/jzus.B2100343.
Porazinski S R,Wang H J,Furutani-Seiki M. 2010. Microinjec‐tion of medaka embryos for use as a model genetic orga-nism[J]. Journal of Visualized Experiments,46:e1937.doi:10.3791/1937-v.
Shankaran S S,Dahlem T J,Bisgrove B W,Joseph Yost H,Tristani-Firouzi M. 2017. CRISPR/Cas9-directed gene edi-ting for the generation of loss-of-function mutants in high-throughput zebrafish F0 screens[J]. Current Protocols in Molecular Biology,119:31. doi:10.1002/cpmb.42.
Strømsnes T A H,Schmidke S E,Azad M,Singstad Ø,Grønsberg I M,Dalmo R A,Okoli A S. 2022. CRISPR/Cas9-mediated gene editing in salmonids cells and effi‐cient establishment of edited clonal cell lines[J]. Interna‐tional Journal of Molecular Sciences,23(24):16218. doi:10.3390/ijms232416218.
Sun X Y,Tao B B,Wang Y X,Hu W,Sun Y H. 2022. Isolation and characterization of germline stem cells in protogynous hermaphroditic Monopterus albu[sJ]. International Journal of Molecular Sciences,23(11):5861. doi:10.3390/ijms 23115861.
Valli H,Phillips B T,Shetty G,Byrne J A,Clark A T,Meistrich M L,Orwig K E. 2014. Germline stem cells:Toward the regeneration of spermatogenesis[J]. Fertility and Sterility,101(1):3-13. doi:10.1016/j.fertnstert.2013.10.052.
Xu X J,Guan W Z,Niu B L,Guo D D,Xie Q P,Zhan W,Yi S K,Lou B. 2022. Chromosome-level assembly of the Chi‐nese hooksnout carp( Opsariichthys bidens) genome using PacBio sequencing and Hi-C technology[J]. Frontiers in Genetics,12:788547. doi:10.3389/fgene.2021.788547.
Zhang F H,Hao Y K,Li X M,Li Y,Ye D,Zhang R,Wang X S,He M D,Wang H P,Zhu Z Y,Sun Y H. 2022a. Surrogate production of genome-edited sperm from a different sub‐family by spermatogonial stem cell transplantation[J]. Science China Life Sciences,65:969-987. doi:10.1007/s11427-021-1989-9.
Zhang H,Zhang W W,Mo C Y,Dong M D,Jia K T,Liu W,Yi M S. 2022b. Production of functional sperm from in vitro-cultured premeiotic spermatogonia in a marine fish[J]. Zoological Research,43(4):537-551. doi:10.24272/j.issn.2095-8137.2022.058.
Zhang Z M,Wang J,Li J N,Liu X,Liu L,Zhao C L,Tao W J,Wang D S,Wei J. 2023. Establishment of an integrated CRISPR/Cas9 plasmid system for simple and efficient genome editing in medaka in vitro and in vivo[J]. Bio-logy,12(2):336. doi:10.3390/biology12020336.
Zhao H B,Hong N,Lu W Q,Zeng H Q,Song J X,Hong Y H. 2012. Fusion gene vectors allowing for simultaneous drug selection,cell labeling,and reporter assay in vitro and in vivo[J]. Analytical Chemistry,84:987-993. doi:10.1021/ac202541t.
Zhao Y C,Wang T D,Yu Z,Wang H M,Liu B,Wu C Y,Teng C B. 2016. Inhibiting cyprinid herpesvirus-3 replication with CRISPR/Cas9[J]. Biotechnology Letters,38:573-578. doi:10.1007/s10529-015-2020-0.
Zhong C Y,Tao Y H,Liu M F,Wu X,Yang Y,Wang T,Meng Z N,Xu H Y,Liu X C. 2022. Establishment of a spermato‐gonial stem cell line with potential of meiosis in a her‐maphroditic fish,Epinephelus coioides[J]. Cells,11(18):2868. doi:10.3390/cells11182868.
Zhou L,Wang X Y,Liu Q H,Yang J K,Xu S H,Wu Z H,Wang Y F,You F,Song Z C,Li J. 2021. Successful sper‐matogonial stem cells transplantation within Pleuronecti‐formes:First breakthrough at inter-family level in marine fish[J]. International Journal of Biological Sciences,17:4426. doi:10.1101/2021.01.29.428910.
(责任编辑 兰宗宝)

