E2F表达与肝癌患者预后及免疫浸润的相关性分析
刘程豪 卓怡呈 杨晨 乔世源 卜媛媛 吴向未

摘要:目的 探討E2F基因与肝癌患者的预后关系及其作为肝癌标志物的可能性。方法 GENT2 数据库用于评估 E2F基因在肝癌中的表达。生存分析和 Cox 分析提示 E2F基因的预后价值。使用 STRING 数据库构建包含 40 个最相似基因和 E2F1-8 的基因网络。TIMER数据库用于分析 E2F基因表达与免疫细胞浸润之间的相关性。cBioPortal数据库分析E2F1-8的基因突变情况。GEO数据集用于验证 E2F5 的表达差异。UALCAN、MEXPRESS、GEO和TCGA数据库中甲基化信息用于分析E2F5的表达与启动子甲基化之间的相关性,并通过人类肝癌细胞系进行验证。结果 E2F1-8在肝癌组织中均高表达。晚期肝癌患者E2F1-3 和E2F5-8的转录水平与总生存率(OS)呈正相关。多因素分析显示,高水平的E2F5 表达是肝癌患者OS缩短的独立预后因素。E2F5/7在肝癌中的表达与肿瘤诱导的免疫反应激活和免疫浸润有关。E2F5在肿瘤组织中高表达与基因突变及启动子甲基化水平相关。结论 E2F5是肝癌患者生存的独立预后生物标志物。E2F家族成员在肝癌中的作用可能与其在细胞周期调节、免疫细胞浸润或基因突变等方面的功能有关。
关键词:肝癌;E2F基因家族;免疫浸润;预后
中图分类号:中图分类号R285.5文献标志码:A文献标识码
Correlation between E2F expression and immune infiltrates and prognosis
of hepatocellular carcinoma
LIU Chenghao1,ZHUO Yicheng1,YANG Chen1,QIAO Shiyuan1,BU Yuanyuan1,WU Xiangwei1,2*
(1 School of Medicine,Shihezi University/NHC Key Laboratory of Prevention and Treatment of Central Asia High Incidence Diseases, Shihezi,Xinjiang 832000, China; 2 The First Affiliated Hospital, School of Medicine, Shihezi University,
Shihezi,Xinjiang 832000, China)
Abstract: Objective To explore the relationship between E2F gene and prognosis of patients with liver cancer and its potential as a marker of liver cancer. Methods The GENT2 databases were used to assess the expression of E2F1-8 in hepatocellular carcinoma and the association with individual cancer staging and tumor grading. Survival analysis and Cox analysis suggested the prognostic value of E2F. The STRING database was used to construct a gene network containing the 40 most similar genes and E2F1-8. The TIMER database was used to analyze the correlation between E2F expression and immune infiltrating cells. The cBioPortal database was analyzed for mutations in E2F. The GEO dataset was used to validate the expression differences of E2F5. Methylation information from the UALCAN, MEXPRESS, GEO and TCGA databases was used to analyze the correlation between E2F5 expression and promoter methylation, and validated by human hepatocellular carcinoma cell lines. Results E2F1-8 was highly expressed in all hepatocellular carcinoma tissues and correlated with tumor grade and pathological stage. The transcript levels of E2F1-3 and E2F5-8 were positively correlated with overall survival (OS) in hepatocellular carcinoma patients. Multifactorial analysis showed that high level of E2F5 mRNA was an independent prognostic factor for shortened OS in hepatocellular carcinoma patients. E2F5/7 expression in hepatocellular carcinoma was associated with tumor-induced activation of immune response and immune infiltration. High expression of E2F5 mRNA in tumor tissues correlated with gene mutation and promoter methylation levels. Conclusion E2F5 is an independent prognostic biomarker for survival in patients with hepatocellular carcinoma. The role of E2F family members in hepatocellular carcinoma may be related to their functions in cell cycle regulation, immune cell infiltration or gene mutation.
Key words: hepatocellular carcinoma;E2F gene family;immune infiltration;prognosis
肝癌,主要是肝细胞癌(Hepatocellular Carcinoma,HCC),是世界范围内高发的一种预后较差的恶性消耗性疾病[1-2]。肝癌的特点是复发及转移率较高,在可治愈期的检出率极低,且治疗无效[3]。因此,明确参与肝癌发生发展的分子机制和识别准确的预后生物标志物,有助于为肝癌的早期诊断和治疗提供可靠的理论依据以及治疗靶点。
E2F蛋白最初被认定是启动腺病毒 E2 基因的激活因子,可诱导宿主细胞发生增殖[4-5]。目前已确定的E2F转录因子家族成员共有8名,即E2F1-8[6]。家族中各成员的生物学功能不尽相同,主要通过激活特定的下游靶基因来调控细胞增殖和细胞周期进程[4]。目前在不同类型的癌症中可普遍观察到 E2F 家族基因表达水平的异常升高。例如,研究发现E2F1 和 E2F3 在肝癌中表达升高,而 E2F1/E2F3 的过表达可促进小鼠自发性肝癌的恶性进展[7]。由于其结构和功能的复杂性,先前对 E2F 家族基因的研究仅局限在单个 E2F 基因与肝癌之间的相关性。
本研究全面分析了E2F家族成员在肝癌不同病理阶段中的表达水平。通过Cox 分析探索E2F基因表达水平在肝癌中的预后价值 。通过评估E2F表达与免疫微环境表型的相关性来挖掘潜在的抗肿瘤机制。本研究中的大部分数据来自TCGA和GEO数据库,有助于更好的理解E2F家族基因在肝癌中的功能。最终表明 E2F 家族基因可作为肝癌治疗研究的潜在生物标志物。
1 材料与方法
1.1 主要试剂
E2F5引物(F:5′-TGATACTTTGGCTGTGAGGCA-3′;R:5′-CAGCACCTACACCTTTCCACTG-3′ 购自上海生工生物有限公司),内参引物(F:5′-AACCGCGAGAAGATGACCCAG-3′;R:5′-GGATAGCACAGCCTGGATAGCAA-3′ 购自上海生工生物有限公司),特异性DNA甲基化转移酶抑制剂地西他滨(北京索莱宝科技有限公司)。
1.2 实验方法
1.2.1 GENT2数据库
GENT2数据库(http://gent2.appex.kr/gent2/)是一个可对公共基因表达数据集中正常和肿瘤组织的基因表达模式进行编译的搜索平台,可用于研究基于肿瘤亚型的差异表达及预后意义[8]。本研究以P<0.05作为纳入标准。通过two-sample t 检验比较表达的差异。
1.2.2 TCGA数据库
癌症基因组图谱(The cancer genome atlas,TCGA)数据库是一个包括30多种人类癌症的测序和临床信息的数据分析平台[9]。TCGA-LIHC数据集中下载的mRNA表达数据及与之配对的临床参数可通过R软件中 Limma 软件包进行预处理,剔除缺乏随访数据或E2F基因表达缺失的样本,分析肝癌组织中E2F1-8的mRNA表达及与临床参数的关系。使用Survival软件包分析目的基因表达量与患者OS之间的相关性。通过Students t检验比较目的基因表达差异,Cox分析比较生存相关性。
1.2.3 UALCAN数据库
UALCAN(http://ualcan.path.uab.edu)可对TCGA中的RNA-seq数据和31种癌症类型的临床数据进行深入分析[10]。在本研究中,UALCAN用于分析E2F5在肝癌组织中的甲基化程度。通过Students t检验比较转录表达的差异。
1.2.4 cBioPortal数据库
cBioPortal (www.cbioportal.org) 数据库可从多维度对癌症基因组学数据进行分析和可视化[11]。本实验中分析了E2F1-8的基因组图谱,包括来自GISTIC和mRNA表达z评分(RNASeq V2 RSEM)的突变,z评分阈值±1.8。E2F的基因突变及其与OS,肝癌患者的无病生存期 (DFS)、无进展生存期(PFS)和疾病特异性生存期(DSS)之间的相关性并通过KM图展示。通过Log-rank test统计生存曲线之间的差异。
1.2.5 免疫评分
ESTIMATE算法可通过基因表达特征来预测肿瘤样本中基质细胞和免疫细胞的比率。TCGA数据库中肝癌患者的免疫评分可从http://bioinformatics.mdanderson.org/estimate下载[12]。本实验中根据免疫评分的中位值将肝癌病例分为高低两组,判断目的基因在各组中的表达情况。
肿瘤免疫评估资源(Tumor immune estimation resource,TIMER)数据库是一个综合分析肿瘤浸润免疫细胞的网络资源(https://cistrome.shinyapps.io/timer/)[13]。免疫细胞浸润程度可通过基因表达谱进行统计评估,并通过病理学评估进一步验证。通过“Correlation”模块评估肝癌中 E2F的表达与免疫浸润细胞基因标志物之间的Spearman 相关性和统计学意义。
1.2.6 GEPIA2和STRING数据库
基因表达谱交互分析2(Gene Expression Profiling Interactive Analysis 2,GEPIA2)网络服务器是基于 TCGA 和 GTEx 数据库中肿瘤和正常样本的基因表达分析平台[14]。通过 “Similar Gene Detection” 模块(http://gepia2.cancer-pku.cn/#similar)來识别与E2F1-8最相似的5个基因。STRING 数据库可以收集、评分和整合各蛋白质之间相互作用关系[15]。使用蛋白质相互作用模块,构建E2F1-8 及40个相似基因的相互作用网络,通过本地工具Cytoscape(version 3.8.2)按degree算法评分对该网络进行可视化。
1.2.7 GO富集分析
通过基因本体论(Gene Ontology,GO)富集分析工具分析E2F潜在的生物学功能。GO富集分析主要包括细胞成分(Cellular Component,CC)、分子功能(Molecular Function,MF)和生物过程(Biological Process,BP),由工具包(WebGestalt, http://www.webgestalt.org/)[16]通过在线基因集进行分析,设定各组间差异统计学意义显著的标准为P和FDR值均<0.05。
1.2.8 GEO数据集
数据集 GSE14520、GSE62232和GSE76427均由 Gene Expression Omnibus (GEO, https://www.ncbi.nlm.nih.gov/geo/) 获得。
1.2.9 细胞培养
MHCC97H和HuH-7细胞系均由中国科学院细胞库购买。细胞在添加10%胎牛血清(Gibco)的高糖DMEM培养基中培养,培养条件为37℃,5% CO2,实验所用细胞均无支原体污染。
1.2.10 RNA的提取和qRT-PCR分析
使用RNA提取试剂盒提取经预先处理好细胞的总RNA。利用RevertAid First Strand cDNA提取试剂盒(Thermo Fisher Scientifi)将提取的总RNA中逆转录成cDNA。基因表达在CFX96触控实时PCR检测系统(Bio-Rad)上进行定量。
1.2.11 统计学处理
通过R 3.6.1 软件用 Cox 回归分析对 E2F 基因的表达与患者存活率之间的关联性进行评估。使用非配对 t 检验比较高、低免疫评分组之间E2F基因的表达差异,多个GEO数据集中正常和肿瘤组织之间 E2F5 的表达差异以及细胞系中处理组与对照组之间目的基因的表达差异。GraphPad Prism (v. 7.0) 用于统计分析和图形创建。P<0.05被认为具有统计学意义。
2 结果
2.1 肝癌患者中E2F1-8 表达水平升高
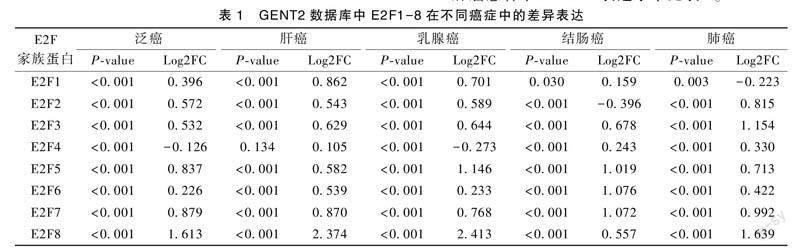
肝癌患者中E2F1-8表达水平见表1。
通过GENT2数据库在GPL570平台的多个肿瘤芯片数据中比较肿瘤和正常组织样本之间 E2F1-8 的表达。肿瘤患者中E2F1-8的表达水平均上调。在肝癌芯片数据集中,与正常组织相比,肝癌组织中E2F8表达水平增加了2.374倍(P<0.001)。此外,肝癌组织中E2F1(Log2FC=0.8621,P<0.001)、E2F7 (Log2FC=0.870,P<0.001)在肝癌组织中的表达也均高于正常组织(表1)。
2.2 晚期肝癌患者中E2F1-8 表达的预后价值
由于E2F1-8 在晚期肝癌患者中表达更显著,因此通过生存分析进一步探讨 E2F1-8 表达与 Ⅲ 期 肝癌患者生存率之间的关系。结果显示,在肝癌Stage Ⅲ 期患者中较高的 E2F3/5表达与较差的 OS 相关(P<0.05,图1C,图1E);同样在Grade Ⅲ 期患者中,E2F2/4/5/6的高表达对 OS 更不利(P<0.05,图1J,图1L,图1M,图1N)。表明E2F1/2/3/5/6/7/8的表达与不同分期肝癌患者的预后显著相关,由此初步判断E2F基因可作为预测肝癌患者存活率的有效生物标志物,且E2F5对于晚期肝癌患者更为敏感。
2.3 E2F1-8 表达水平对肝癌患者OS的独立预后价值
利用TCGA数据库中373例肝癌患者的mRNA表达和临床资料进行Cox分析。单因素分析显示在肝癌中病理分期、T分期、M分期、E2F1-3和E2F5-8高表达与显著缩短的OS相关。多因素分析显示,仅E2F5高表达与OS的显著缩短独立相关(表2),表明E2F5是所有肝癌患者OS的独立预后因素。
2.4 肝癌中E2F表达与免疫浸润的关系
利用ESTIMATE算法计算TCGA数据库中肝癌患者样本的免疫评分,来预测肿瘤组织中是否存在浸润性的免疫细胞。将肝癌患者按免疫评分的中位数分为高、低评分组,比较E2F1-8的表达差异(图2A~图2H)。结果显示,E2F5在高评分组表达略显升高而E2F7在高评分组表达显著降低(P<0.05,图2E,图2G),提示E2F5/7可能参与了肝癌的免疫浸润。
通过ssGSEA算法分析TCGA数据库肝癌样本中E2F5/7分别与各免疫浸润细胞之间的相关性。结果显示,E2F5/7均与Th2细胞呈正相关(图2I,图2J)。使用TIMER数据库评估E2F1-8的表达与肿瘤纯度和免疫浸润细胞水平的关系。结果表明,E2F5/7的表达与肝癌的肿瘤纯度及肿瘤细胞中的免疫浸润水平相关(P<0.05,图2K,图2L)。
此外,利用TIMER数据库进一步分析E2F5/7表达与免疫细胞标志物之间的相关性(表3)。经过肿瘤纯度矫正后,E2F5/7的表达与不同功能T细胞、B细胞、DCs、中性粒细胞、NKs等大部分免疫细胞的基因标志物显著相关。由此表明,E2F5/7与肝癌中免疫细胞的浸润有关。
2.5 肝癌患者中E2F基因突变及与生存的相关性
肝癌患者中E2F基因的突变率较高,在379例肝癌患者样本中有177份样本發生了突变,且E2F5的突变率最高,为22%(图3A)。
生存分析和Log- rank检验结果显示,肝癌患者中E2F基因的遗传改变均与更短的OS、PFS、DFS和DSS相关(图3B~图3E,P<0.05)。这些结果提示,E2F基因突变可显著影响肝癌患者的预后。
2.6 肝癌患者中E2F及其相关基因变化对功能和通路的影响
通过GEPIA2 数据库探索与E2F1-8密切程度最高的5个基因。应用 STRING 数据库构建包含E2F1-8及40个已识别基因的相互作用网络(图3F)。GO富集分析发现,E2F家族成员在肝癌中主要参与有丝分裂和细胞周期的转录调控等功能相关(图3G)。同样的,与E2F基因密切相关的MCM2、EXO1、CCNF、DTL、MELK、MKI67、FBXO5和KIFC1等基因也多是调控染色体或驱动细胞有丝分裂的关键基因。MCM2可能是E2F基因在肝癌中的关键节点。根据STRING结果,E2F基因高表达可能与染色体有丝分裂有关。
2.7 E2F5可作为肝癌预后的潜在标志物
GEO数据集中,E2F5在肿瘤组织中的表达量高于正常组织(图4A~图4C,P<0.05)。
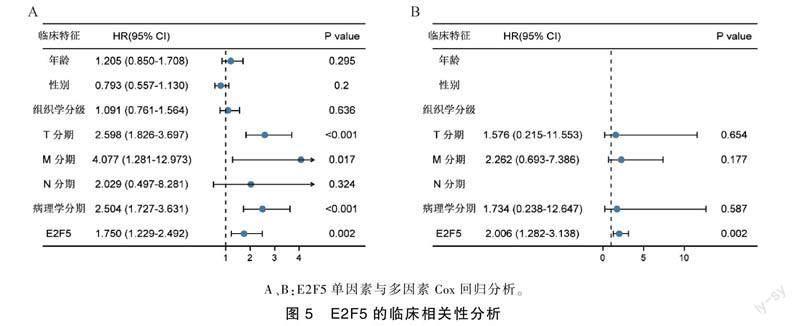
为了解E2F5在肝癌组织中异常上调的机制,通过分析UALCAN、MEXPRESS数据库,以及GEO14520和TCGA-LIHC数据集中的甲基化位点数据,发现E2F5的表达水平与启动子DNA序列中多个位点的甲基化状态呈负相关(图4D~图4G,P<0.05)。此外,使用1 μmol·L-1甲基化抑制剂地西他滨处理细胞48 h后,通过qRT-PCR检测肝癌细胞中E2F5的表达情况。结果显示,MHCC97H细胞中与对照组(1.000±0.221)相比,药物刺激组(2.323±0.342)E2F5表达水平升高(P<0.01);HuH-7细胞中与对照组(1.000±0.197)相比,药物刺激组(1.908±0.240)E2F5表达水平升高(图4H,P<0.01)。将TCGA-LIHC项目中的临床数据校正年龄、性别、肿瘤分级、病理分期、T分期等多项临床参数变量后,进行单因素和多因素Cox分析。表明在肝癌中E2F5是一种可预测患者总生存的独立预后因素(图5A,图5B)。
3 讨论
虽然E2F家族中的部分成员在肿瘤发生和预后中的功能已得到部分证实[17-19],但尚未进行生物信息学分析来证明这些因子在肝癌中的作用。本研究基于公共数据库的高质量数据集,对肝癌患者中所有可获得的E2F1-8 基因表达、基因突变、预后价值、与免疫浸润的关系和潜在功能进行了深入分析。在肝癌患者中观察到E2F基因的突变率高达47%,并与较短的生存期相关。并且为E2F基因及其40个最密切相关的基因构建了一个相互作用网络。此外,E2F基因在晚期肝癌中的预后价值已被确定。E2F5的稳定表达和生存价值在多个数据集中得到验证。本研究通过对E2F1-8全面分析,为提高肝癌患者的预后准确性及肝癌的治疗做出贡献。
E2F家族成员中,E2F1在细胞中功能已被不断证实。研究发现,E2F1通过介导LOC100130075激活MDM2的转录使宫颈癌的恶性程度进一步加深[20];E2F1可通过调节STMN1的过表达促进卵巢癌的增殖和迁移[21];DTX3通过调控E2F1及其下游基因CDC2和Cyclin D3表达来调控结直肠癌细胞的生长[22]。此外,E2F1还是真核细胞中细胞周期的主要调控因子,在细胞有丝分裂G1-S期的转化中起到促进作用[23-24]。与此相同的,本研究发现肝癌组织中E2F1的表达显著高于正常组织,且E2F1表达与肝癌患者的分期和分级密切相关。E2F1表达较高的肝癌患者总体生存率普遍较低。
到目前为止,E2F2在人类癌症中的表达和功能仍然相对空白。生理状态下,在细胞增殖、分化和DNA损伤应答等方面发挥关键作用[25]。据文献报道,E2F2可通过调控细胞周期阻滞、DNA损伤和细胞增殖抑制等方式参与内皮细胞的衰老过程[26]。在肝癌中,E2F2可作为LncRNA AC026401.3调控的下游靶向转录因子。AC026401.3通过与OCT1作用来促进其对E2F2启动子区的募集,从而增强肝癌患者对索拉非尼和乐伐替尼的耐药性[27]。本实验中,E2F2在肝癌组织中的表达量随肿瘤恶性程度分级的加深而不断增加,且总体生存率更加恶化。
E2F3位点转录表达时有E2F3a和E2F3b两种替代的亞型蛋白[28]。其过表达对肿瘤细胞所产生的恶性生物学行为目前已被普遍认可。E2F3在儿童的成神经细胞瘤中具有和E2F1相似的下游转录特征。E2F1和E2F3作为成神经细胞瘤独立的预后因素,高表达时患者的预后普遍较差,可能与其异常表达可导致成神经细胞瘤细胞周期和DNA复制失控有关[29]。在肝癌中也有相关miRNA被证明可通过调控E2F3来影响细胞增殖和抑制G0/G1期。据报道,miR-15a-5p可通过靶向E2F3调控肝癌细胞的迁移、凋亡和生长[30]。同样的,与正常对照组相比,肝癌样本中E2F3表达上调且与肿瘤分期显著相关。E2F3过表达的肝癌患者OS普遍较差。综上所述,认为E2F3在肝癌中可能是一种较强的致癌基因。
目前已有报道指出,E2F4在鼻鳞癌肿瘤组织中高表达且与预后不良有关。E2F4在细胞核中作为转录因子可调节免疫细胞,被认为是诊断和治疗鼻咽癌的重要分子靶点,是鼻咽癌潜在的预后因素[31]。除此之外,E2F4在结直肠癌中也普遍被认为是潜在的致癌基因。在人结直肠癌细胞中,E2F4及其靶细胞周期蛋白A的表达均上调。E2F4的表达受抑制时可减缓细胞周期中G1/S相的转化和降低结肠癌细胞的增殖率[32]。不仅如此,E2F4还可以刺激AGAP2-AS1通过调节miR-182-5p/CFL1 轴促进结直肠癌的发展[33]。与先前报道相符,本研究发现E2F4在肝癌细胞中与正常细胞相比表达上调。然而,E2F4的表达上调与较短的OS之间并无明显相关,表明其在肝癌中并未起到主要的致癌作用。
E2F5的高表达和致癌能力目前已在多种肿瘤中得到证实,然而在肝癌中的研究还相对较少。胰腺癌中,METTL3可通过调控E2F5的m6A甲基化修饰来促进肿瘤细胞的恶性进展[34];视网膜母细胞瘤中,circ_0084811通过吸附miR-18a(18b)-5p对E2F5的表达进行调控来促进细胞增殖[35];miR-32、miR-34a和miR-181a-5p可以通过靶向E2F5调控结直肠癌细胞系的增殖、迁移和侵袭[32]。本研究发现E2F5在肝癌组织中的表达高于正常组织。在肿瘤分级及个别肿瘤分期中,E2F5的高表达与不利的OS显著相关,提示是OS缩短的独立因素。此外,E2F5与肝癌中CD4+/CD8+ T细胞、巨噬细胞和DC的浸润呈显著正相关,提示E2F5可能通过参与免疫浸润细胞的招募和调节来影响肝癌患者的预后。E2F5的表达主要与Th细胞不同亚群标记物的表达相关,包括Th1细胞(STAT1)、调节性T细胞(CCR8、CD25)。表明E2F5在调节Th细胞肿瘤浸润中发挥重要作用。由于调节性T细胞在肿瘤免疫抑制中发挥重要作用,这可能从侧面解释了为什么E2F5是一种致癌基因。8个E2F基因中E2F5的基因突变率最高,E2F5的高基因突变可能通过破坏Th2/Th1的相对平衡状态来促进肿瘤细胞的生长。综上所述,我们的研究结果表明,E2F5可能通过调节肿瘤细胞中免疫细胞的浸润,在肝癌的发生和恶性进展中起着重要作用。
E2F6在多種肿瘤中均有表达上调的报道,但在肝癌中的作用却未涉及较深入的研究。E2F6在肝癌的恶性增殖中并未起到主要作用,其作为辅助因子可与CENPU发生物理作用来促进其泛素化降解,通过影响E2F1的转录水平来促进肝癌细胞的增殖[36]。在本研究中,E2F6在肝癌中的表达水平升高,且与OS之间具有显著相关性。因此,E2F6在肝癌中致癌的潜在机制有待进一步探究。
在肿瘤中E2F7既是转录抑制因子,也可作为激活因子[37-38]。有文献提到,透明细胞肾细胞癌中RP11-478C19.2/E2F7轴参与了预后和免疫浸润水平,可作为治疗的新生物标志物[37]。目前有研究表明,E2F7作为E2F家族中一个特殊成员,对其下游基因的功能具有一定抑制作用,并且其在癌症中所发挥的功能目前颇有争议[38]。E2F7是在肝癌中普遍上调的一个抑制性转录因子,其高表达与患者的临床病理特征不良和生存率差相关。E2F7可通过抑制miR-383-5p转录和依次激活SP1/SOX4/Anillin轴促进肝癌细胞的生长[38]。在本研究中,E2F7在肝癌组织中比在正常组织中表达更高,且与患者的个体癌症分期和肿瘤分级相关。免疫相关性分析发现,E2F7在肝癌高评分组中表达显著降低,并且与包括Th1/Th2细胞、耗竭性T细胞和调节性T细胞在内的T细胞亚群标记物密切相关。由此推断,E2F7可能通过免疫浸润改善肝癌患者的预后。
家族最后一个成员E2F8可通过调节E2F1的活性来控制DNA损伤相关的细胞周期过程[29,32]。在包括肝脏在内的一些组织中,细胞增殖的有丝分裂过程中可被转录抑制因子E2F7和E2F8抑制,导致多倍体细胞的形成[39]。此外,E2F8在结直肠癌患者中上调,并且是miR-1258.72的特异性靶点。一项研究报道,敲低E2F8会减少细胞周期调控因子的表达,并且可通过调节NF-κB通路影响结肠癌细胞的生长[32]。同样,本研究显示,所有肝癌患者中E2F8表达上调,且提示OS不良。但其在肝癌中的预后作用尚不清楚,有待进一步研究。
如上所述,本研究证实E2F基因的高突变率(48%)及遗传改变与较短的生存期相关。进一步发现,E2F5高表达是所有肝癌患者OS缩短的独立预后因素且对晚期肝癌患者的预测更为敏感。通过免疫相关性分析,发现E2F5与肿瘤诱导的免疫反应激活和免疫浸润相关。这可能是E2F5影响肝癌进展的潜在机制。此外,通过分析数据库的甲基化样本以及甲基化抑制实验,证实启动子低甲基化是导致其异常表达的重要原因之一。本次研究结果希望将为拓宽E2F基因作为肝癌中重要标志物的认识提供帮助,同时为肝癌的免疫治疗提供新的思路。然而,需要进一步的研究来验证我们的发现。
参考文献(References)
[1]MENG J, WANG L H, HOU J, et al. CCL23 suppresses liver cancer progression through the CCR1/AKT/ESR1 feedback loop[J]. Cancer Sci, 2021, 112(8): 3099-3110.
[2]ZHU H Z, ZHOU W J, WAN Y F, et al. Downregulation of orosomucoid 2 acts as a prognostic factor associated with cancer-promoting pathways in liver cancer[J]. World J Gastroenterol, 2020, 26(8): 804-817.
[3]SHI Y, ZHANG D D, LIU J B, et al. Comprehensive analysis to identify DLEU2L/TAOK1 axis as a prognostic biomarker in hepatocellular carcinoma[J]. Molecular therapy Nucleic acids, 2021, 23: 702-718.
[4]XIE D, PEI Q, LI J, et al. Emerging role of E2F family in cancer stem cells[J]. Front Oncol, 2021, 11: 723137.
[5]KENT L, LEONE G. The broken cycle: E2F dysfunction in cancer[J]. Nature Reviews Cancer, 2019, 19(6): 326-338.
[6]SUN C C, LI S J, HU W, et al. Comprehensive analysis of the expression and prognosis for E2Fs in human breast cancer[J]. Molecular Therapy: The Journal of the American Society of Gene Therapy, 2019, 27(6): 1153-1165.
[7]WANG H W, WANG X R, XU L P, et al. Integrated analysis of the E2F transcription factors across cancer types[J]. Oncol Rep, 2020, 43(4): 1133-1146.
[8]PARK S, YOON B, KIM S, et al. GENT2: an updated gene expression database for normal and tumor tissues[J]. BMC Med Genomics, 2019, 12(suppl5): 101.
[9]TOMCZAK K, CZERWIN'SKA P, WIZNEROWICZ M. The cancer genome atlas (TCGA): an immeasurable source of knowledge[J]. Contemporary Oncology (Poznan, Poland), 2015, 19(1A): A68-77.
[10]CHANDRASHEKAR D, BASHEL B, BALASUBRAMANYA S, et al. UALCAN: a portal for facilitating tumor subgroup gene expression and survival analyses[J]. Neoplasia (New York, NY), 2017, 19(8): 649-658.
[11]CERAMI E, GAO J, DOGRUSOZ U, et al. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data[J]. Cancer Discov, 2012, 2(5): 401-404.
[12]YOSHIHARA K, SHAHMORADGOLI M, MARTíNEZ E, et al. Inferring tumour purity and stromal and immune cell admixture from expression data[J]. Nature Communications, 2013, 4: 2612.
[13]LI T W, FAN J Y, WANG B B, et al. TIMER: a web server for comprehensive analysis of tumor-infiltrating immune cells[J]. Cancer Res, 2017, 77(21): e108-e110.
[14]TANG Z F, KANG B X, LI C W, et al. GEPIA2: an enhanced web server for large-scale expression profiling and interactive analysis[J]. Nucleic Acids Res, 2019, 47(W1): W556-W560.
[15]SZKLARCZYK D, GABLE A, LYON D, et al. STRING v11: protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Res, 2019, 47(D1): D607-D613.
[16]LIAO Y X, WANG J, JAEHNIG E, et al. WebGestalt 2019: gene set analysis toolkit with revamped UIs and APIs[J]. Nucleic Acids Res, 2019, 47(W1): W199-W205.
[17]TAN Z B, CHEN M, PENG F, et al. E2F1 as a potential prognostic and therapeutic biomarker by affecting tumor development and immune microenvironment in hepatocellular carcinoma[J]. Translational Cancer Research, 2022, 11(8): 2713-2732.
[18]LIU N, LIU Z G, ZHANG W D, et al.[Retracted]MicroRNA-433 reduces cell proliferation and invasion in non-small cell lung cancer via directly targeting E2F transcription factor 3[J]. Mol Med Report, 2022, 26(5): 341.
[19]CHEN M, ZHAO Z Z, WU L Q, et al. E2F1/CKS2/PTEN signaling axis regulates malignant phenotypes in pediatric retinoblastoma[J]. Cell Death Dis, 2022, 13(9): 784.
[20]XU Y, LIU Y D, HUANG W, et al. LOC100130075 promotes cervical cancer progression by activating MDM2 transcription through E2F1[J]. Reproductive Sciences (Thousand Oaks, Calif), 2022, 29(5): 1439-1448.
[21]NIE L K, ZHANG C, SONG H Y, et al. Overexpression of stathmin 1 predicts poor prognosis and promotes cancer cell proliferation and migration in ovarian cancer[J]. Dis Markers, 2022, 2022: 3554100.
[22]XU H L, LIANG S N, HU J J, et al. Deltex E3 ubiquitin ligase 3 inhibits colorectal cancer cell growth and regulates cell cycle progression via upregulating E2F transcription factor 1[J]. Mol Biol Rep, 2022, 49(3): 1661-1668.
[23]WANG C F, YANG Y, ZHANG G, et al. Long noncoding RNA EMS connects c-Myc to cell cycle control and tumorigenesis[J]. Proc Natl Acad Sci U S A, 2019, 116(29): 14620-14629.
[24]VREB J, BRADLEY-GILL M, ZIELKE N, et al. E2f1Translational control of regulates the cell cycle[J]. Proc Natl Acad Sci U S A, 2022, 119(4): e2113704119.
[25]LI L W, WANG S G, ZHANG Y H, et al. The E2F transcription factor 2: What do we know?[J]. Biosci Trends, 2021, 15(2): 83-92.
[26]LIU H F, CHEN L P, XIAO W L, et al. Alteration of E2F2 expression in governing endothelial cell senescence[J]. Genes, 2022, 13(9): 1522.
[27]WANG Y, TAN K, HU W, et al. LncRNA AC026401.3 interacts with OCT1 to intensify sorafenib and lenvatinib resistance by activating E2F2 signaling in hepatocellular carcinoma[J]. Exp Cell Res, 2022, 420(1): 113335.
[28]GAO C, LIU S G, LU W T, et al. Downregulating CREBBP inhibits proliferation and cell cycle progression and induces daunorubicin resistance in leukemia cells[J]. Mol Med Report, 2020, 22(4): 2905-2915.
[29]WANG H W, WANG X R, XU L P, et al. Prognostic analysis of E2F transcription factors E2F1 and E2F3 in four independent pediatric neuroblastoma cohorts[J]. BMC Pediatr, 2022, 22(1): 376.
[30]LI Y L, LI D, YANG Y, et al. miR-15a-5p regulates liver cancer cell migration, apoptosis and cell cycle progression by targeting transcription factor E2F3[J]. Crit Rev Eukaryot Gene Expr, 2022, 32(6): 1-10.
[31]QI L, REN Z H, LI W. E2F4 transcription factor is a prognostic biomarker related to immune infiltration of head and neck squamous cell carcinoma[J]. Sci Rep, 2022, 12(1): 12132.
[32]XU Z H, QU H, REN Y Y, et al. Systematic analysis of e2f expression and its relation in colorectal cancer prognosis[J]. Int J Gen Med, 2022, 15: 4849-4870.
[33]GUO Z, LIU X Z, SHAO H J. E2F4-induced AGAP2-AS1 up-regulation accelerates the progression of colorectal cancer via miR-182-5p/CFL1 axis[J]. Digestive and Liver Disease: Official Journal of the Italian Society of Gastroenterology and the Italian Association for the Study of the Liver, 2022, 54(7): 878-889.
[34]TANG Y, GAO G, XIA W W, et al. METTL3 promotes the growth and metastasis of pancreatic cancer by regulating the m6A modification and stability of E2F5[J]. Cell Signal, 2022, 99: 110440.
[35]JIANG G W, QU M X, KONG L L, et al. hsa_circ_0084811 regulates cell proliferation and apoptosis in retinoblastoma through miR-18a-5p/miR-18b-5p/E2F5 axis[J]. BioMed Research International, 2022, 2022: 6918396.
[36]LIU Y Y, YAO Y, LIAO B, et al. A positive feedback loop of CENPU/E2F6/E2F1 facilitates proliferation and metastasis via ubiquitination of E2F6 in hepatocellular carcinoma[J]. Int J Biol Sci, 2022, 18(10): 4071-4087.
[37]ZENG K, SONG G, CHEN B L, et al. Comprehensive analysis to identify the RP11-478C19.2/E2F7 axis as a novel biomarker for treatment decisions in clear cell renal cell carcinoma[J]. Transl Oncol, 2022, 25: 101525.
[38]HAO F J, WANG N, ZHANG Y F, et al. E2F7 enhances hepatocellular carcinoma growth by preserving the SP1/SOX4/Anillin axis via repressing miRNA-383-5p transcription[J]. Mol Carcinog, 2022, 61(11): 975-988.
[39]JIN Y H, ANBARCHIAN T, WU P, et al. Wnt signaling regulates hepatocyte cell division by a transcriptional repressor cascade[J]. Proc Natl Acad Sci U S A, 2022, 119(30): e2203849119.
(責任编辑:编辑唐慧)
收稿日期:中文收稿日期2022-11-17
基金项目:国家自然科学基金项目(81760570),中国医学科学院中央级公益性科研院所基本科研业务费专项资金项目(2020-PT330-003),新疆生产建设兵团财政科技计划项目(2021BB006),新疆生产建设兵团中青年领军人才项目(2018CB017)
作者简介:刘程豪(1995—),男,硕士研究生,专业方向为外科学。
*通信作者:吴向未(1973—),男,教授,博士生导师,从事干细胞与再生医学、肝胆外科良恶性疾病的临床与基础研究,e-mail:wxwshz@126.com。

