玉米籽粒发育突变体emp35 的表型分析与基因定位




摘要 为解析玉米籽粒形成的遗传基础,探究Emp35 基因在玉米籽粒发育中的作用,对籽粒缺陷突变体empty pericarp35(emp35)进行表型鉴定、胚乳细胞显微观察、胚乳贮藏物质含量测定及图位克隆。结果显示:突变体籽粒发育缓慢,明显小于同期发育的正常籽粒,成熟籽粒干瘪呈空皮状;胚乳细胞显微观察发现emp35 的胚和胚乳发育严重滞后,胚乳细胞中线粒体结构异常;淀粉和蛋白质积累减少;F2代分离果穗上正常籽粒与发育缺陷籽粒呈3∶1 分离,表明籽粒缺陷表型由单个隐性核基因突变所致。采用集团分离分析法(bulked segregantanalysis, BSA) 将Emp35 定位于第8 染色体 127.90~163.36 Mb 区间,在该区间内开发了4 个InDel 标记,连锁作图将Emp35 精细定位于139 571 117~146 176 858 区间。
关键词 玉米(Zea mays L.); 籽粒发育; 集团分离分析法; 基因定位; 表型鉴定
中图分类号 S513.3 文献标识码 A 文章编号 1000-2421(2024)02-0085-08
玉米籽粒产量由单位面积穗数、每穗粒数和籽粒质量3 个因素构成。籽粒质量由籽粒库容和胚乳充实程度所决定[1],籽粒发育与充实程度影响籽粒产量。玉米籽粒发育包括胚胎发育、胚乳细胞分化和贮藏物质积累3 个关键过程,每个发育过程都受众多基因调控。目前,研究者已经克隆了百余个控制籽粒发育进程的基因[2]。
胚是玉米授粉后籽粒中最早开始发育的器官,也是决定种子发育的关键组织。胚发育缺陷突变体主要涉及能量供应和重要蛋白质合成相关功能,如Lem1 和Emb14 控制质体核糖体的形成与组装[3-4];Emb8522 参与叶绿体RNA 的转录后加工[5];Bige1编码转运体蛋白,参与信号通路的反馈调节[6]。一方面,玉米胚乳的发育和分化决定籽粒大小和籽粒质量,影响胚乳发育的基因多与淀粉和蛋白质的合成加工相关。如Opaque2 编码转录因子,调控相关醇溶蛋白合成基因的表达[7];Sh2 和Bt2 分别编码腺苷二磷酸葡萄糖焦磷酸化酶的大、小亚基,共同构成淀粉合成系统中重要的酶[8];Fl2、Opaque1、Opaque10 基因参与贮藏蛋白质加工并形成蛋白质体的生理过程[9-11]。另一方面,胚乳细胞在发育过程中会分化出功能特异的细胞群,如基底胚乳传递层、糊粉层细胞和胚周围组织细胞,这些细胞可以从植株母体组织向籽粒运输营养物质,与籽粒的发育和充实有着密切的关系[12]。如INCW2(Mn1)、Cr4 等基因变异引起胚乳细胞异常分化,导致胚和胚乳发育不良[13-14]。玉米籽粒发育和充实的调控途径仍有待深入解析。
本研究以1 个从γ 射线诱变B73 的突变体库中筛选得到的空种皮(empty pericarp, emp)籽粒表型突变体emp35 为材料,分析该突变体籽粒中胚和胚乳的动态发育和主要贮藏物质的含量变化,并开展Emp35基因定位,以期从发育生物学和遗传学角度解析突变体的籽粒表型,进而全面解析籽粒质量形成的遗传基础,为玉米籽粒质量的遗传改良提供科学依据。
1 材料与方法
1.1 试验材料
本研究所用的emp35 突变体由长江大学杜何为教授提供。将B73 种子经γ 射线诱变处理后种植于大田,植株自交。突变体emp35 是从自交果穗中分离出来的籽粒发育缺陷突变体。由于纯合emp35/emp35 无法繁殖,以emp35 杂合体植株(+/emp35)自交繁殖保存突变体基因型。通过+/emp35 与Mo17杂交,然后自交,选择表型分离的自交果穗用于遗传分析和基因定位。
1.2 籽粒发育表型观察与遗传分析
种植+/emp35 杂合单株,授粉后分别选取授粉后10、12、14、18 d 自交果穗上的幼嫩籽粒,纵向分割,保留籽粒中部厚2~3 mm 的组织用于包埋。石蜡包埋、染色以及切片参考Ren 等[15]的方法。切片样片用体视显微镜观察拍照。
取授粉后9、14 d 的幼嫩籽粒,从籽粒中部靠近顶端的位置进行横切,切片厚约2 mm,浸泡于2.5%戊二醛中固定。扫描电镜制样参照文献[15]的方法进行。样品于华中农业大学电镜平台的JSM-6390LV 扫描电镜(NTC, 日本)下观察拍照。
取授粉后10 d 的野生型和突变体籽粒,从籽粒上部取微量胚乳组织固定于2.5% 戊二醛溶液中。透射电镜制样参考Cai 等[16]的方法。制样后在华中农业大学电镜平台Hitachi-H7650 透射电子显微镜(Hitachi,日本)下观察拍照。
收获+/emp35 自交成熟果穗,选取21 个正常发育且籽粒数目较多的果穗,统计干瘪皱缩籽粒和饱满圆润籽粒数,计算野生型籽粒与突变籽粒的分离比,进行χ2检验。
1.3 籽粒淀粉含量和蛋白组分含量测定
从+/emp35 植株自交果穗上随机挑选野生型和突变体成熟籽粒,使用淀粉测定试剂盒(K-TSTA-100A/K, Megazyme, Ireland) 提取并测定玉米籽粒总淀粉含量,3 次生物学重复,每个生物学重复包括3次技术重复。具体步骤见操作手册。
采用硼酸钠浸取法提取籽粒总蛋白质,再用无水乙醇萃取总蛋白溶液分离醇溶蛋白和非醇溶蛋白[17]。采用BCA( bicinchoninic acid)-标准曲线试剂盒(KTP3006, Abbkine, China)测定野生型和突变体籽粒总蛋白、醇溶蛋白和非醇溶蛋白的含量,3 次生物学重复,每个生物学重复包括3 次技术重复。具体步骤见操作手册。
1.4 BSA测序及数据分析
将+/emp35 单株与Mo17 杂交获得F1,F1 单株分别自交,选取籽粒表型发生分离的授粉后14 d 的果穗。在果穗上挑选30 粒正常籽粒和30 粒发育缺陷籽粒,分别混合构建野生型池(bulk-WT, Bwt)和突变型池(bulk-Mut, Bmt)。采用CTAB 法提取基因组DNA[18]。取亲本B73、Mo17、Bwt 和Bmt 基因组DNA 各200 ng,按照Illumina Nextera DNA LibraryPrep Kit (FC-121-1030, Illumina, America)说明书构建测序文库,构建好的DNA 文库使用IlluminaHiSeq4000 测序仪(Illumina, USA)分别进行双端150 bp (PE150)测序。B73 和Mo17 测序深度为100×,每个混池的测序深度为30×。
对于测序原始数据,首先使用Cutadapt [19](version1.13)和Trimmomatic [20](version 0.36)软件进行筛选,过滤得到高质量的clean data, 后用FastQC(https://www. bioinformatics. babraham. ac. uk/projects/fastqc/)对测序长度、GC 含量、测序质量参数等数据进行统计。用BWA[21]( version 0.7.15-r1140)软件的MEM 算法将质量合格的PE reads (pairedendreads, 双端测序片段)比对到B73 参考基因组AGPv4(http://plants. ensembl. org/Zea_mays/ Info/Index)上,然后使用软件Samtools[22] (version1.3.1)将文件转化为BAM格式。接着使用 Picard( version1.9.1)工具中的 SortSam 对 BAM 文件中的 reads 进行排序,得到最终可以用于变异分析(variant calling)的 BAM 文件。使用 GATK[23]( version 3.7)的 HaplotypeCaller模块和GenotypeGVCFs 模块进行变异检测,包括单核苷酸多态性 (single nucleotide polymorphisms,SNPs)和小片段(lt;10 bp)插入和缺失(insertion/deletion, InDel)。
完成变异检测后,分别计算Bwt 和Bmt 基因组上各变异位点的突变reads 与总reads 的比值,即SNP-index。再计算SNP-index (WT)与SNP-index(Mut)的差值,即Δ(SNP-index)。Δ(SNP-index)值高于阈值(在Plt;0.01 水平下进行10 000 次计算)的基因区间即可认为可能为目的基因所在区段。
1.5 候选基因的分子标记定位
根据上述分析结果,选择目标基因区间在B73和Mo17、Bwt 和Bmt 之间的InDel 变异,进一步将B73 序列与Mo17 参考基因组(http://maize.plantbiology.msu.edu/)进行比对,确定InDel 变异的真实性,并设计InDel 标记引物(表1)。用所设计的InDel标记检测+/emp35 × Mo17 构建的F2群体中的突变体籽粒的基因型。将基因型数据导入QTL lciMapping3.0软件中,按照规定格式将Mo17 特有带型记作“2”,B73 特有带型记作“0”,杂合带型记作“1”。 构建遗传连锁图谱,计算重组率和遗传图距。
2 结果与分析
2.1 突变体籽粒表型
从γ 射线诱变B73 的突变体库中筛选到1 个籽粒发育缺陷突变体,突变体籽粒干瘪,只有空种皮,没有或只有少量内容物,命名为empty pericarp 35(emp35), 该突变体只能以杂合体形式保存。选取+/emp35 杂合体植株自花授粉后不同时间的果穗,观察籽粒发育的动态变化。授粉后12 d,籽粒颜色都为乳白色,野生型和突变体籽粒大小已有区别,突变体种皮皱缩(图1A)。授粉后18 d 野生型籽粒呈现黄色,而突变体籽粒呈乳白色、胚乳不充实、籽粒大小明显小于野生型 (图1B)。成熟果穗上的野生型籽粒圆润饱满、呈现金黄色,而突变体籽粒干瘪皱缩或成片状、籽粒无胚乳或仅有少量胚乳(图1C、D)。
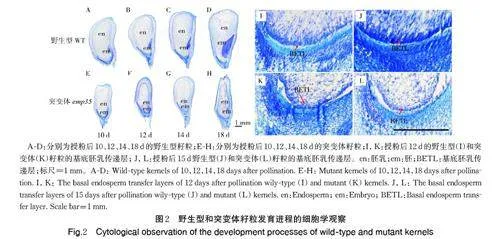
从不同发育时期的+/emp35 自交果穗上,选取野生型和突变体籽粒进行石蜡切片和显微观察。授粉后10 和12 d 野生型籽粒胚分化产生胚芽鞘,胚乳细胞分化、数目多且分布致密( 图2A、B);同时期突变体籽粒的胚为未分化的胚体,胚乳细胞数量少且分布松散,胚乳与种皮之间存在较大空隙(图2E、F)。授粉后14 d,野生型籽粒胚分化出多片叶原基(图2C),而突变体胚的分化仍停留在原胚阶段(图2G)。授粉后18 d,野生型籽粒胚发育接近完成,胚乳充实(图2D);而突变体籽粒的胚仅分化出盾片,胚乳中的基底胚乳传递层(basal endosperm transfer layer,BETL)细胞和糊粉层细胞没有正常分化,籽粒外种皮皱缩(图2H)。可见,e mp35 籽粒的胚发育迟缓,胚乳细胞的发育与分化异常,胚乳干物质积累减少。
BETL 是营养物质从母体向籽粒运输的组织。显微观察发现,授粉后12 和15 d 野生型籽粒已形成成熟的BETL 细胞和多层的传递层细胞,BETL 细胞沿垂直方向伸长,次生细胞壁向胞内交错生长(图2I、J);而突变籽粒中仅有1 层分化异常的BETL 细胞(图2K、L)。这些结果表明,Emp35 基因突变会导致BETL 细胞的发育和分化异常,影响营养物质从母体向籽粒运输。
扫描电子显微镜观察发现,授粉后9 d 的野生型籽粒胚乳细胞中已有较多成熟淀粉粒,淀粉粒分布集中( 图3A),授粉后15 d的野生型胚乳细胞中的淀粉粒充实、堆积紧密(图3B)。授粉后9 d 的emp35 籽粒胚乳细胞中几乎无淀粉粒存在(图3C),授粉后15d 胚乳细胞中存在少量淀粉粒且分布零散(图3D)。这些结果表明,emp35 籽粒胚乳细胞干物质积累明显落后于正常籽粒。
通过透射电子显微镜观察发现,授粉后10 d 野生型胚乳细胞中的线粒体形态完整,线粒体内膜堆积排列规律整齐、内嵴清晰可见、无明显空泡(图3E)。而突变体胚乳细胞中的线粒体结构异常、体积缩小,线粒体内嵴结构松散且有明显空泡化现象(图3F)。线粒体结构缺陷可能导致供给细胞生长的能量不足,对胚乳充实乃至整个籽粒发育过程产生不良影响。
2.2 突变体籽粒干物质积累
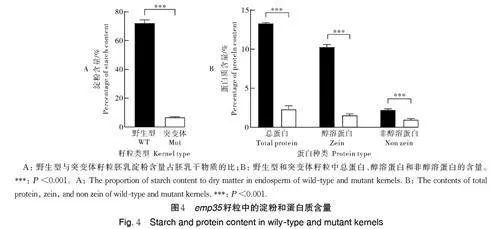
胚乳细胞中淀粉和蛋白质定量检测结果显示,平均每颗野生型籽粒中胚乳淀粉含量占胚乳干物质的72.17%,而平均每颗emp35 籽粒中胚乳淀粉含量则仅占胚乳干物质比的6.72%(图4A),显著低于野生型(Plt;0.001)。这与扫描电镜观察到的淀粉粒积累受阻现象吻合。
野生型和突变体籽粒中总蛋白、醇溶蛋白和非醇溶蛋白的含量测定结果显示,emp35 籽粒总蛋白占籽粒质量的2.29%,显著低于野生型籽粒的13.30%(Plt;0.001)(图4B)。同时,emp35 籽粒醇溶蛋白和非醇溶蛋白的质量比分别为1.53% 和0.96%,显著低于野生型籽粒的10.26%和2.21%( Plt;0.001),表明emp35 基因突变严重影响籽粒总蛋白、醇溶蛋白及非醇溶蛋白的积累。
2.3 emp35 基因的定位
以+/emp35 杂合植株与Mo17 杂交,F1 代植株自交,部分F2 植株上的果穗存在正常籽粒与干瘪籽粒的分离。在21 个分离果穗中,共有野生型籽粒4 675 粒、突变体籽粒1 545 粒,野生型与突变型分离比= 3.02∶1。χ2检验表明,分离果穗上正常籽粒与突变体籽粒符合3∶1 的分离比(χ2=0.085, Plt;0.05),表明籽粒缺陷表型是由单个核基因隐性突变所致。
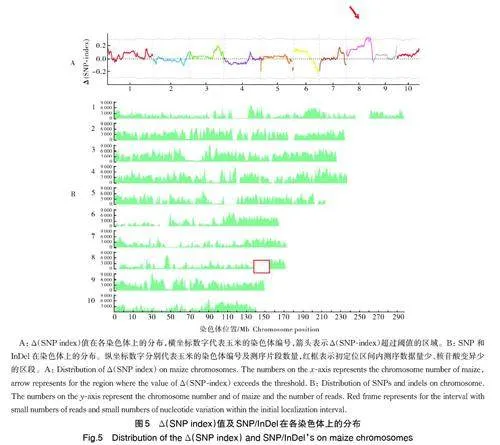
从表型分离的果穗上分别挑选30 粒野生型和30粒突变体籽粒,构建野生型(Bwt)和突变体(Bmt)混池,对亲本B73 和Mo17 及混池进行重测序,结果显示,B73 与Mo17 分别获得422 989 871 和377 327 339个reads,分别可覆盖参考基因组的95.03% 和79.90%,覆盖深度达36.87×和30.76×。Bwt 和Bmt混池则分别获得330 257 568 和270 665 372 个reads,可覆盖B73 参考基因组的90.90% 和90.03%,覆盖深度为25.82×和21.67×。将所有样品的reads 序列比对到B73 参考基因组上,在2 个混池间共筛选到5 114 337个SNP 位点和681 377个InDel 位点。使用Δ(SNP-index)值进行候选基因定位,Δ(SNP-index)值超过设定阈值的reads 集中在玉米第8 号染色体上127.90~163.26 Mb的物理区间内,该区间的物理距离为35.36 Mb(图5A),Emp35基因可能位于该区间内。
BSA-seq 分析结果显示,第8 染色体146~163Mb 区间测序数据量低、核苷酸变异少,难以开发多态性标记(图5B)。因而,我们仅在第8 染色体128~146 Mb 区间内每隔6 Mb 选取1 个InDel 变异,开发了4 个InDel 标记(ID128、ID134、ID140 和ID146)。检测发现这4 个标记在B73 和Mo17、Bwt 及Bmt 之间均存在多态性,表明4 个InDel 标记都与emp35 连锁。选取授粉后14 d 的F2果穗,共挑选421 粒突变体种子,提取胚乳DNA,进行基因型分析,构建该染色体区段的分子标记遗传连锁图,将Emp35 基因定位在139 571 147~146 176 858 Mb(ID140 的物理位置为chr8: 139 571 147~139 571 148,ID146 的物理位置为chr8: 146 176 858~146 176 859),与ID140 和ID146 的遗传距离分别为4.17 和0.24 cM,对应物理距离约为6.25 Mb 和359 kb(图6)。
3 讨论
玉米籽粒发育是决定产量的重要因素,而玉米籽粒发育缺陷突变体是研究籽粒发育机制的重要资源。空种皮突变体是一类胚和胚乳发育异常、籽粒皱缩、种子败育,对产量产生极大影响的籽粒发育缺陷突变体。已克隆的Emp 基因大多数为编码35 肽重复蛋白家族 (pentatricopepetide repeat protein,PPR)。PPR 蛋白家族是一类由多个串联重复基序组成的蛋白质,重复数为2~30,每个基序由35 个低保守度的氨基酸组成[24]。PPR 蛋白可以特异性识别并结合线粒体与叶绿体基因的转录本,参与线粒体和叶绿体基因转录本的RNA 剪接、RNA 编辑、RNA翻译、RNA 稳定化和RNA 的成熟等转录后加工过程[25]。PPR 家族基因突变会直接影响线粒体成熟和正常功能,使呼吸作用受阻,进而导致籽粒发育供能不足,引起籽粒变小和皱缩等严重缺陷表型。例如:Emp8 蛋白参与线粒体基因nad1 第四内含子的反式剪接和nad4 第一内含子的顺式剪接,emp8 突变体胚与胚乳发育缓慢,籽粒变小[26]。EMP9 参与线粒体ccmB-43 和 rps4-335 位点的 C-to-U 编辑,emp9 突变体籽粒干瘪皱缩[27]。本研究中的籽粒发育缺陷突变体emp35 具有胚发育滞后、胚乳细胞发育不良、胚乳干物质积累严重不足、种皮干瘪塌陷、籽粒无生活力等表型。透射电镜观察发现emp35 胚乳细胞的线粒体内嵴排列松散,线粒体腔存在较多空泡。线粒体结构的异常可能导致细胞代谢水平降低,细胞生长发育和细胞内生化反应所需的能量供应短缺,致使胚和胚乳发育异常。emp35 突变体展现的籽粒发育表型、生理生化指标、微观细胞结构与PPR 基因的突变体类似,因此,我们推测Emp35 可能编码1 个线粒体结构或功能相关蛋白,并可能是PPR 家族基因。
本研究中,F2群体的遗传分析表明,emp35 的突变表型是由单个核基因隐性突变所致。通过BSAseq将Emp35 基因定位在8 号染色体上127.90~163.26 Mb 的区间内。用4 个InDel 标记分析F2群体中的突变体表型籽粒的基因型并作出遗传连锁图,将Emp35 精细定位于139 571 147~146 176 858 区间内。目前该定位区间内没有已克隆的玉米籽粒发育基因,表明Emp35 是一个新发现的籽粒发育相关基因。在后续研究中,将开展Emp35 的精细定位与基因克隆,探究Emp35 基因的表达部位,研究线粒体基因的转录翻译过程以验证Emp35 突变基因对线粒体合成的影响,解析Emp35 基因在籽粒发育过程中的生理功能及作用机制,为玉米籽粒质量的改良与稳产品种的选育提供理论依据。
参考文献References
[1] 王汉宁,王晓明. 从源库观点看玉米籽粒产量的形成[J]. 甘
肃农业大学学报,1992,27(1):86-92.WANG H N,WANG
X M. Viewing the formation of maize grain yield from the
source-sink theory[J].Journal of Gansu Agricultural University,
1992,27(1):86-92( in Chinese with English abstract).
[2] DAI D W,MA Z Y,SONG R T.Maize kernel development
[J/OL]. Molecular breeding,2021,41(1):2[2023-04-26].
https://doi.org/10.1007/s11032-020-01195-9.
[3] MA Z R,DOONER H K.A mutation in the nuclear-encoded
plastid ribosomal protein S9 leads to early embryo lethality in
maize[J].The plant journal,2004,37(1):92-103.
[4] LI C L,SHEN Y,MEELEY R,et al.Embryo defective 14 encodes
a plastid-targeted cGTPase essential for embryogenesis
in maize[J].The plant journal,2015,84(4):785-799.
[5] SOSSO D,CANUT M,GENDROT G,et al. PPR8522 encodes
a chloroplast-targeted pentatricopeptide repeat protein
necessary for maize embryogenesis and vegetative development
[J].Journal of experimental botany,2012,63(16):5843-5857.
[6] SUZUKI M,SATO Y,WU S,et al. Conserved functions of
the MATE transporter BIG EMBRYO1 in regulation of lateral
organ size and initiation rate[J].The plant cell,2015,27(8):
2288-2300.
[7] SCHMIDT R J,KETUDAT M,AUKERMAN M J,et al.
Opaque-2 is a transcriptional activator that recognizes a specific
target site in 22-kD zein genes[J]. The plant cell,1992,4
(6):689-700.
[8] GIROUX M J,HANNAH L C.ADP-glucose pyrophosphorylase
in shrunken-2 and brittle-2 mutants of maize[J].Molecular
amp; general genetics,1994,243(4):400-408.
[9] COLEMAN C E,CLORE A M,RANCH J P,et al.Expression
of a mutant alpha-zein creates the floury2 phenotype in
transgenic maize[J].PNAS,1997,94(13):7094-7097.
[10] WANG G F,WANG F,WANG G,et al.Opaque1 encodes a
myosin XI motor protein that is required for endoplasmic reticulum
motility and protein body formation in maize endosperm
[J].The plant cell,2012,24(8):3447-3462.
[11] YAO D S,QI W W,LI X,et al.Maize opaque10 encodes a cereal-
specific protein that is essential for the proper distribution
of zeins in endosperm protein bodies[J/OL]. PLoS genetics,
2016,12(8):e1006270 [2023-04-26]. https://doi. org/
10.1371/journal.pgen.1006270.
[12] BOMMERT P,WERR W. Gene expression patterns in the
maize caryopsis:clues to decisions in embryo and endosperm
development[J].Gene,2001,271(2):131-142.
[13] MILLER M E,CHOUREY P S. The maize invertase-deficient
miniature-1 seed mutation is associated with aberrant
pedicel and endosperm development[J].The plant cell,1992,4
(3):297-305.
[14] BECRAFT P W,STINARD P S,MCCARTY D R.CRINKLY4:
a TNFR-like receptor kinase involved in maize epidermal
differentiation[J].Science,1996,273(5280):1406-1409.
[15] REN X M,PAN Z Y,ZHAO H L,et al. EMPTY PERICARP11
serves as a factor for splicing of mitochondrial nad1 intron
and is required to ensure proper seed development in maize
[J].Journal of experimental botany,2017,68(16):4571-4581.
[16] CAI M J,LI S Z,SUN F,et al.Emp10 encodes a mitochondrial
PPR protein that affects the cis-splicing of nad2 intron 1 and
seed development in maize[J].The plant journal,2017,91(1):
132-144.
[17] CHEN X Z, YAO D S, SONG R T.Maize endosperm protein
extraction and analysis[J/OL]. Bio-protocol, 2013,3
(14): e832[2023-04-26]. https://doi.bio-protocol.org/e832.
[18] DOYLE J. DNA protocols for plants[M]//HEWITT G M,
JOHNSTON A W B,YOUNG J P W.Molecular techniques
in taxonomy.Berlin:Springer,1991:283-293.
[19] MARTIN M. Cutadapt removes adapter sequences from highthroughput
sequencing reads[J/OL]. EMBnet.journal, 2011,
17 (1) : 10 [2023-04-26]. https://doi. org/10.14806/
ej.17.1.200.
[20] BOLGER A M,LOHSE M,USADEL B. Trimmomatic:a
flexible trimmer for Illumina sequence data[J].Bioinformatics,
2014,30(15):2114-2120.
[21] LI H,DURBIN R.Fast and accurate short read alignment with
Burrows-Wheeler transform[J].Bioinformatics,2009,25(14):
1754-1760.
[22] LI H,HANDSAKER B,WYSOKER A,et al.The sequence
alignment/map format and SAMtools[J]. Bioinformatics,
2009,25(16):2078-2079.
[23] MCKENNA A, HANNA M, BANKS E, et al. The genome
analysis Toolkit: a MapReduce framework for analyzing next
generation DNA sequencing data [J]. Genome research,
2010, 20(9): 1297-1303.
[24] SMALL I, PEETERS N. The PPR motif : a TPR-related
motif prevalent in plant organellar proteins[J]. Trends in biochemical
sciences, 2000, 25(2):45-47.
[25] SCHMITZ-LINNEWEBER C,SMALL I. Pentatricopeptide
repeat proteins: a socket set for organelle gene expression[J].
Trends in plant science, 2008, 13(12): 663-670.
[26] SUN F, ZHANG X Y, SHEN Y, et al. The pentatricopeptide
repeat protein EMPTY PERICARP8 is required for the
splicing of three mitochondrial introns and seed development
in maize[J]. The plant journal, 2018, 95:919-932.
[27] YANG Y Z, DING S, WANG H C, et al. The pentatricopeptide
repeat protein EMP9 is required for mitochondrial cc⁃
mB and rps4 transcript editing, mitochondrial complex biogenesis
and seed development in maize[J]. New phytologist,
2017, 214(2):782-795.
(责任编辑:张志钰)

