基于InDel标记的杏鲍菇遗传多样性评价
姜晓红 朱家漘, 孙忠刚 曲绍轩 马 林 李辉平*
(1. 江苏省农业科学院蔬菜研究所 江苏省高效园艺作物遗传改良重点实验室,江苏 南京 210014;2. 江苏大学生命科学学院,江苏 镇江 212013;3. 江苏淮香食用菌有限公司,江苏 淮安 223009)
杏鲍菇(Pleurotuseryngii),学名刺芹侧耳,隶属侧耳科、侧耳属,是我国三大重要工厂化栽培珍稀食用菌之一,其野生种质资源主要集中在南欧、北非、中亚等地[1]。杏鲍菇含有多种膳食纤维、维生素、人体必需氨基酸及功能性多糖等,具有药食兼用的功能,深受消费者的欢迎。
分子标记的开发对开展种质资源收集与评价,培育优质新品种具有重要意义。杏鲍菇子实体形态特征较少且极易受外界环境因素影响,单从形态水平鉴定杏鲍菇菌株的亲缘关系比较困难。DNA分子标记技术作为一种快速鉴定样品间遗传多样性的方法被广泛应用于种质资源鉴定、基因定位、分子辅助育种等多方面。目前在杏鲍菇上得到应用的分子标记主要包括有限制性片段长度多态性(RFLP)[2]、随即扩增多态性DNA(RAPD)[3]、简单重复序列多态性(SSR)[4]、简单重复序列间区(ISSR)[5]和多核苷酸多态性(MNP)[6]标记技术等。
插入/缺失(insertion-deletion,InDel)是指在不同个体之间基因组同一位点的序列发生了数个碱基的插入或缺失,在基因组中的分布密度仅次于SNP,适用于全基因组分子标记的开发。InDel标记目前在医学、动植物遗传学都得到广泛应用,在食用菌遗传多样性中也逐步得到应用[7]。Xiang等筛选了68对InDel引物,分析了88个香菇栽培群体的遗传多样性[8]。沈秀芬等通过对5个香菇菌株重测序,开发并利用InDel标记对44份香菇菌株进行遗传多样性分析,并将其分为4个亚群[9]。
本研究通过InDel分子标记技术,对搜集的杏鲍菇菌株进行遗传多样性评价,为杏鲍菇种质资源评价及开发利用提供技术支撑。
1 材料与方法
1.1 供试菌株
65份杏鲍菇菌株由江苏省农业科学院蔬菜研究所提供(表1),其中栽培菌株主要来自国内企业和市场,CCMSSC菌株来自国家食用菌标准菌株库。

表1 供试菌株编号信息
1.2 菌丝培养
采用常规的马铃薯葡萄糖琼脂培养基(PDA)进行菌丝活化3 d后,打孔接入250 mL马铃薯葡萄糖液体培养基(PDB)摇瓶进行菌丝扩大培养,25 ℃下150 r/min培养5 d,接真空泵抽滤收集菌丝,用纯净水洗涤2次。
1.3 DNA纯化
取10 mg的菌丝体,研钵中用液氮将其迅速研磨成粉状,用DNA提取试剂盒(上海生工)按操作手册纯化DNA。检测OD260/OD280比值,范围在1.7~1.9为合格,并将核酸浓度统一为50 ng/μL后放入 - 20 ℃冰箱备用。
1.4 InDel位点筛选与引物设计
依据NCBI网站公布的杏鲍菇基因组数(GCA_015484515.1和GCA_001717165.1),通过Minimap2进行基因组比对[10],以GCA_001717165.1为参考基因组,筛选30~200 bp碱基差异的InDel位点,随机选择36个位点,在其上下游各150 bp内利用Primer 3设计(参数默认)引物(表2),引物由北京擎科生物科技股份有限公司合成。
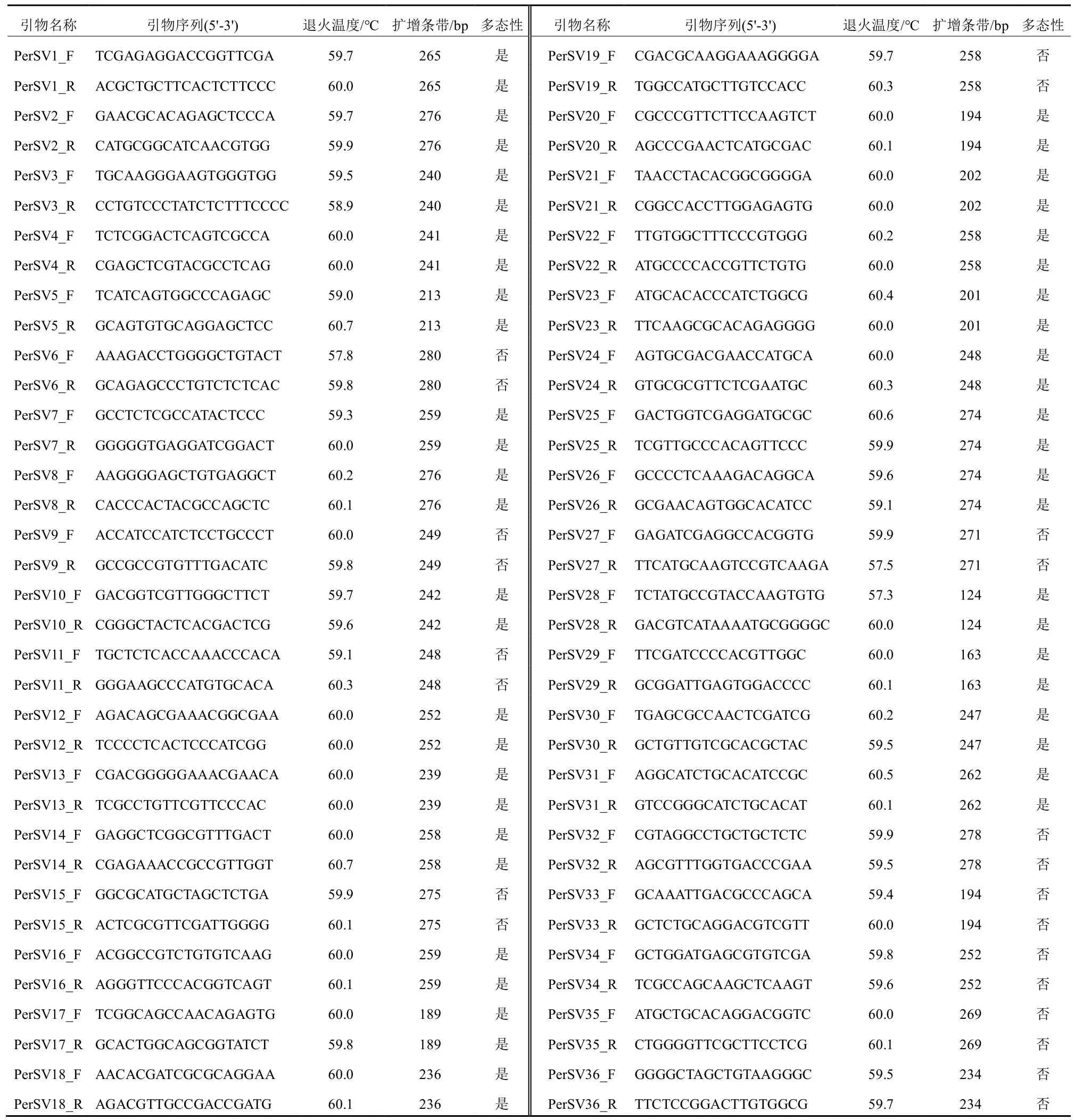
表2 引物序列及多态性
1.5 PCR扩增和电泳分析
PCR反应体系:2 × PCRmix 10 μL,正反向引物(10 μmol/L)各1μL,模板DNA 1μL,补dd H2O至20 μL。PCR反应条件:98 ℃ 4 min预变性;95 ℃10 s,63 ℃10 s,72 ℃ 10 s,30个循环;72 ℃ 5 min延伸后4 ℃保存。扩增产物用2%琼脂糖凝胶电泳进行检测,200 V电泳20 min,凝胶成像系统拍照并记录结果。
1.6 数据处理
在Excel 2016中用1或0标记泳道间相同位移上条带的有或无,再导入NTSYSpc 2.10进行分析,计算遗传相似系数并用UPGMA法进行聚类分析。
2 结果与分析
2.1 InDel标记开发
经比对发现,GCA_015484515.1和GCA_001717165.1间30~200 bp InDel位点共135个,其中插入45个,缺失90个,分布在64条Contig上,每条Contig上的目标InDel位点少于5个。位点之间平均距离大于20 kb,50个位点之间距离大于10 kb,只有4个位点距离小于1 kb。随机选取的36对InDel引物中筛选出条带清晰、重复性好的多态性引物25对,对扩增出的63个位点进行统计分析(表2,图1)。
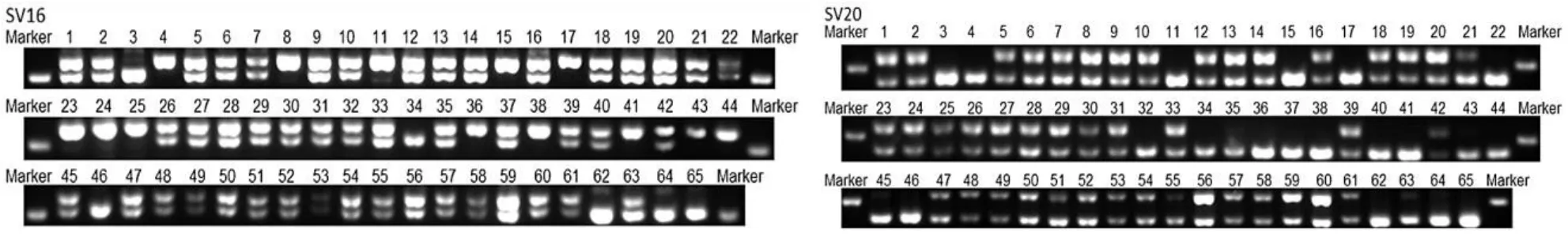
图1 PerSV16和PerSV20 InDel标记多态性标记检测结果
2.2 供试菌株遗传多样性分析
利用NTSYSpc 2.10软件进行矩阵分析并计算65份材料间遗传相似系数,供试菌株遗传相似系数最低值是0.362,表明存在较大的遗传差异,有丰富的遗传多态性,具有很大的开发潜力。其中菌株Per22与其他菌株间的遗传相似系数均很低,与Per18之间的相似遗传系数仅为0.362;而Per53、Per54、Per55、Per56两两之间的相似系数为1,表明这四者之间的亲缘关系极近,很有可能属于同株异名。
通过UPGMA聚类分析法构建的杏鲍菇菌株遗传亲缘聚类树(图2),在遗传相似系数为0.616的水平上将其分为5大类,第1类群数量最多,包括Per1、Per2、Per14等51个菌株,包含了大量国内栽培菌株;第2类群6个菌株,分别是Per4、Per37、Per39、Per42、Per17、Per46;第3类群6个菌株,分别是Per3、Per48、Per68、Per70、Per67、Per69。Per45和Per22与其他菌株之间的亲缘关系都很远。
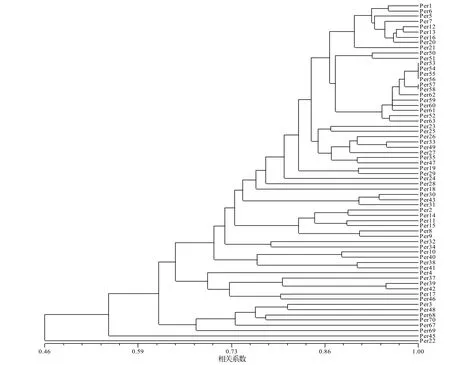
图2 65株杏鲍菇菌株遗传亲缘聚类树
3 结论与讨论
InDel在基因组中分布广泛、数目众多,分布密度仅次于SNP,远高于SSR,是人类和植物中第二丰富的基因变异形式[11-13],而且大片段InDel标记可以直接利用琼脂糖凝胶电泳进行分型检测,对检测设备要求低、简单易行,成本大大低于SNP和SSR检测成本[14,15],更因其共显性遗传、变异稳定、准确性高、重复性好等特点,在群体遗传分析、基因定位及遗传图谱构建等领域被广泛应用[9,11,16-19]。本研究专门针对30~200 bp的InDel位点进行开发,虽然得到的候选标记位点数量有限,但是符合易于检测的出发点,且在首批36个位点中就筛选得到25个多态性候选标记,都可用琼脂糖凝胶直接电泳检测,成功比例较高。
InDel标记开发一般利用不同材料重测序后与参考基因组进行比对分析获得[9,20-22],这就需要该物种有公开的参考基因组,同时对不同材料进行重测序,需要一定的资金和时间成本。对于SNP和小InDel,重测序数据更多的测序深度带来了更高的准确度。但随着测序技术和组装算法的不断进步,大InDel在基因组组装过程中假阳性越来越低。本研究通过比对已公开的杏鲍菇基因组数据,简单快捷且成本低,后续可利用更多的基因组组装数据进一步扩充。
杏鲍菇野生种质资源主要分布于南欧、北非、中亚等地区,虽我国四川、青海、新疆也有分布报道[23],但关于野生种质资源采集的报道较少。国内工厂化栽培菌株很多是引进自日本,并得到推广[24]。此间经过系统选育和杂交育种,我国栽培菌株具有一定的遗传多样性。刘晓红等对来自中国不同地区的杏鲍菇品种进行RAPD分析,发现23份材料遗传多样性很丰富,可分为5大类[25]。马红等用RAPD和ISSR标记将34个中国工厂化栽培杏鲍菇菌株分为4~5大类[26]。杨和川等基于ISSR标记对27株杏鲍菇种质资源评价也得到类似结果[27]。这与本研究结果较为类似。但是魏传正等利用基因二代测序技术的MNP标记对国内56份工厂化栽培菌株分析后,发现相似度均接近100.00%,认为国内杏鲍菇栽培品种高度一致[6]。对国内栽培菌株的遗传多样性分析还值得进一步研究探讨。

