毛红椿TcNAC2基因克隆及在干旱胁迫下的表达分析
钟秋蔚,马际凯,贾 婷,程强强,杜宣瑾,聂资旋,朱 源,张 露
(江西农业大学 江西省森林培育重点实验室∕江西特色林木资源培育与利用2011 协同创新中心∕林学院,江西 南昌 330045)
【研究意义】NAC(NAM,ATAF1∕2,CUC2)是植物中广泛存在且特有的一类转录因子,由矮牵牛(Petunia hybrida)NAM(No apical neristem)[1]与拟南芥(Arabidopsis thaliana)ATAF1/2(Arabidopsis transcription activation factor)[2]和CUC2(Cup-shaped cotyledon)基因的首字母组合而成,广泛存在于陆生植物中,是植物最大的转录因子家族之一[3]。1996 年研究人员从矮牵牛中首次获得第一个NAC 转录因子NAM,并研究出其与胚发育相关。在拟南芥nam突变体中,幼胚无法正常分化出根尖分生组织,幼苗生长停滞,不能分化出根和叶,子叶呈现融合现象[1]。而后研究人员在拟南芥[4]、水稻(Oryza sativa)[5]、马铃薯(Solanum tuberosum)[6]、玉米(Zea mays)[7]、棉花(Gossypium spp)[8]等多种植物中相继发现了数量不等的NAC 类转录因子。研究发现,NAC 转录因子家族参与调控植物器官生长发育进程,包括种子萌发及幼苗生长[9]、根的生长和侧根形成[10-11]、植株茎的伸长[12]、茎尖器官边界和分生组织的形成[13]、叶片衰老[14]、花的形成[15]、果实成熟[16]、细胞分裂和次生壁及木质部形成[17]、植物细胞程序性死亡[18]等。另一方面,在植物胁迫应答进程中,NAC 至关重要。在植株受到逆境胁迫时,NAC 家族的相关基因表达能够使植株的耐旱[19]、耐盐[20]及耐低温[21]等能力得到提升。如拟南芥中ATAF1/2基因负调控植株抗灰霉病,ATAF2负调控抗枯萎病等[22]。基因的功能通常情况下与其编码的蛋白结构是密切相关的,NAC 转录因子的N 端有约150 个氨基酸残基组成了高度保守特征NAC 结构域,C 端为高度多样化的转录调控区,可能具有转录激活或转录抑制活性[23]。NAC 转录因子因其结构的多样性使其具有多种功能,在植物器官生长发育和逆境胁迫响应中至关重要。【前人研究进展】毛红椿(Toona ciliatavar.Pubescens)是楝科(Meliaceae)香椿属的高大落叶乔木,现已并入红椿,1991年被列入第一批国家珍稀濒危Ⅱ级保护树种,分布于我国江西、湖南、广西等省份。毛红椿主干圆满通直,心材呈淡红至赭红色,纹理美丽整齐,速生性好,被誉为“中国桃花心木”,具有很高的经济价值和应用前景[24]。现因生境碎片化以及自然更新速度缓慢等原因,天然林资源仅存零星分布。此外,近年来全球气候变化异常,干旱时有发生,导致毛红椿濒危形势日益严峻。刘文剑等[25]的研究表明,土壤水分对毛红椿苗木生长有一定限制,干旱对毛红椿生长和发育有严重影响。孙洪刚等[26]通过模拟不同水分胁迫研究发现,一年生毛红椿在轻度水分胁迫时,苗木水分先供给毛红椿茎和叶片并限制根系生长,提高苗木抗旱性;随着水分胁迫程度的逐渐增大,苗木会通过落叶及根系的二次生长来优先供给根系对水分的需求和提高根系的水分吸收效率,从而缓解水分胁迫对生存的不利。【本研究切入点】前期已有研究表明NAC 转录因子可以通过清除植物体内的过氧化物或调节清除氧化物相关的基因的表达以增强植株的耐旱性[27]。【拟解决的关键问题】为探究毛红椿抗旱机制,为毛红椿种群保护提供理论基础,该研究聚焦NAC 转录因子家族一个成员,从毛红椿中首次克隆获得TcNAC2基因,对其编码蛋白进行蛋白理化性质、空间结构等生物信息学分析,利用qRT-PCR(实时荧光定量PCR)技术分析TcNAC2基因在不同组织及不同程度干旱胁迫下的表达水平,为深入研究Tc-NAC2基因的结构以及组织表达特异性等特点提供理论依据。
1 材料和方法
1.1 试验材料
以江西农业大学校园内(28°45′45″N,115°49′36″E)毛红椿成年树上的根、茎、叶、叶芽和叶柄等组织用于组织特异性表达分析;以一年生毛红椿实生苗为材料模拟不同程度干旱胁迫以探讨TcNAC2基因在毛红椿受到干旱胁迫时的表达特性。植物材料取样后用自来水清洗表面杂质,置于液氮中迅速转移至实验室,置于-80 ℃冰箱保存,待用。
1.2 试验设计
盆栽模拟干旱胁迫试验设计:选择生长较一致的毛红椿一年生实生苗移栽至塑料花盆,置玻璃大棚培育,7 月1 日生长稳定后进行干旱胁迫处理,设计4 种处理即对照组(土壤相对含水量80%)、轻度胁迫(土壤相对含水量60%)、中度胁迫(土壤相对含水量40%)和重度胁迫(土壤相对含水量20%)。采用盆栽控水法模拟不同干旱胁迫程度,每日傍晚18:00对试验盆苗称重,计算质量差及时补充损失水分。15 d后每处理取幼叶保存以用于TcNAC2基因在干旱胁迫下的表达分析,3次生物学重复。
1.3 总RNA提取和cDNA的合成
根据植物总RNA 提取试剂盒(天根生化科技试剂公司)说明书分别提取毛红椿成年树根、茎、叶、叶芽和叶柄等组织及4种干旱胁迫程度下毛红椿叶片的总RNA,通过核酸紫外分光光度计和10 g∕L琼脂糖凝胶电泳(120 V,15 min)分别检测RNA 的浓度、纯度和质量。使用EasyScript®First-Strand cDNA Synthesis SuperMix 反转录试剂盒(全式金生物技术有限公司)将检测合格的RNA 反转录为cDNA,于-20 ℃冷冻保存。
1.4 引物设计和PCR扩增
NAC2原始序列选自课题组毛红椿转录组测序数据库,利用Oligo 7软件设计特异性引物(表1),选择评分较高,特异性好的引物,由生工生物工程股份有限公司(上海)合成用于PCR扩增目的基因。PCR扩增体系采用50 µL 扩增体系,体系参照2×TransStart®FastPfu Fly PCR SuperMix 说明书,采用50 µL 扩增体系:cDNA 模板1 µL,2×TransStart®FastPfu Fly PCR SuperMix 25 µL,Forward primer 和Reverse primer(10 µmol∕L)各1~2 µL,其余加Nuclease-free Water 补齐体系。扩增目的基因反应程序为预变性98 ℃1 min;变性98 ℃ 10 s,退火 58℃ 5 s,延伸72 ℃ 10 s,30 个循环;彻底延伸72 ℃ 1 min。通过1.5%琼脂糖凝胶电泳初步检测PCR产物大小及质量。
1.5 PCR产物连接转化
按DNA 凝胶快速回收试剂盒(全式金生物技术有限公司)说明书将目的片段切胶回收并与Blunt 克隆载体连接,连接产物转入DH5α 大肠杆菌感受态细胞中,涂板,37 ℃过夜培养,筛选蓝白斑后挑取白色阳性单一菌落置于配有抗生素的LB 培养液中,摇床培养6 h(37 ℃,180 r∕min),菌落PCR 鉴定后将初步检测正确的阳性克隆菌液送至上海生工生物工程股份有限公司进行测序验证。
1.6 TcNAC2蛋白序列的生物信息学分析
使用多种生物信息学软件克隆测序得到的基因序列进行蛋白质理化性质、磷酸化位点预测等生物信息学分析。利用ORF Finder(www.ncbi.nlm.nih.gov∕projects∕gorf∕)获取开放阅读框(open reading frame),并翻译出氨基酸序列;利用ProtParam 网站(web.expasy.org∕protparam∕)在线分析TcNAC2 蛋白质理化特性;利用Protscale(web.expasy.org∕protscale∕)在线分析TcNAC2蛋白质的疏水性;利用NetPhos网站(services.healthtech.dtu.dk∕service.php?NetPhos-3.1)在线预测TcNAC2 蛋白磷酸化位点;利用TMHMM 程序(services.healthtech.dtu.dk∕service.php?TMHMM-2.0)在线分析TcNAC2 蛋白质跨膜结构;利用PSORT 在线网站(www.genscript.com∕psort.html)对TcNAC2 蛋白的亚细胞定位进行预测;利用SOPMA 在线网站(npsaprabi.ibcp.fr∕cgi-bin∕npsa_automat.pl?page=npsa%20_sopma.html)预测TcNAC2 蛋白的二维结构,并用SWISS-MODEL(swissmodel.expasy.org)在线预测目的基因编码TcNAC2蛋白的三维结构;利用NCBI网站在线进行序列同源性比对(blast.Ncbi.Nlm.Nih.gov),并使用MEGA 11软件(邻位相连法)构建系统进化树分析进化关系。
1.7 TcNAC2基因的表达分析
根据毛红椿TcNAC2基因测序结果,设计一对特异性高的荧光定量引物(表1),用于分析TcNAC2基因的表达水平差异,试验以Actin基因为内参基因,设置3次生物学重复和3次技术重复。采用2-ΔΔCT法计算相对表达量。使用SPSS 24.0软件进行数据处理和分析,不同组织及不同干旱胁迫处理下的相对表达量差异图使用Origin 2021软件绘制完成。
2 结果与分析
2.1 毛红椿TcNAC2基因克隆及序列分析
通过总RNA 提取试剂盒获得了不同器官和组织的高质量的RNA。根据毛红椿转录组数据库筛选得到的NAC2基因序列,设计特异性引物,以毛红椿RNA 反转录得到的cDNA 为PCR 模板扩增NAC2基因。PCR产物检测凝胶电泳获得一条大小为700 bp左右的特异性条带(图1)。测序结果分析表明,TcNAC2基因编码完整的开放阅读框561 bp,能够编码186 个氨基酸。将得到的氨基酸序列通过NCBI 数据库中进行BLASTP 比对,发现与其他物种的NAC domain-containing protein 2 相似度高,因此命名为TcNAC2基因。
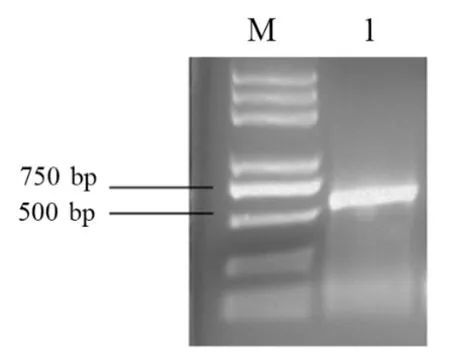
图1 TcNAC2基因扩增电泳分析Fig.1 TcNAC2 gene amplification electropherogram
2.2 TcNAC2蛋白理化性质、亲疏水性及磷酸化位点分析
对TcNAC2基因编码蛋白进行保守结构域分析和理化性质分析发现,TcNAC2蛋白含有NAM(No apical meristem)结构域(图2),说明TcNAC2是一个带有NAM 保守结构域的蛋白,属于NAC 转录因子家族,证明了该研究克隆的准确性。通过分析其理化性质发现TcNAC2蛋白分子式为C988H1511N271O257S10,分子量为21.618 13 ku,pI(理论等电点)为9.66,属于强碱性蛋白。TcNAC2 蛋白不稳定系数为36.45,因此该蛋白属于稳定蛋白。预测蛋白的亲疏水性说明TcNAC2属于亲水蛋白,其亲水氨基酸残基数多于疏水氨基酸残基数。用NetPhos 软件预测TcNAC2磷酸化位点,结果显示(图3)TcNAC2氨基酸序列中占比最多的是Ser位点有4个被磷酸化,Thr位点和Tyr位点分别是3个和2个被磷酸化。在线跨膜区分析结果表明,TcNAC2蛋白属于非跨膜蛋白,不存在跨膜结构。对TcNAC2蛋白的亚细胞定位预测,结果表明其最有可能定位于细胞质,可能性为65.2%;定位于细胞核的可能性为17.4%;定位于线粒体的可能性为8.7%;定位于细胞质膜可能性较低,为4.3%。

图2 毛红椿TcNAC2保守结构域分析Fig.2 TcNAC2 conserved domain analysis of T.ciliata var.pubescens
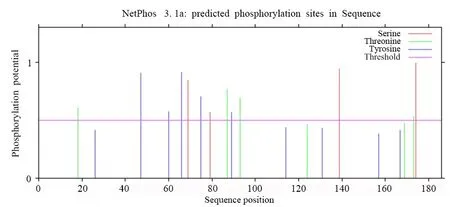
图3 毛红椿TcNAC2蛋白磷酸化位点预测分析Fig.3 Analysis of phosphorylation site of TcNAC2 protein in T.ciliata var.pubescens
2.3 TcNAC2蛋白质高级结构预测分析
对TcNAC2 蛋白二级结构进行预测分析,该研究使用SOPMA 软件完成。结果(图4A)表明,TcNAC2蛋白二级结构中含有13.98% α-螺旋(26个)、4.3% β-螺旋(8个)、18.28%延伸链(34个)及63.44%无规则卷曲(118个)。对TcNAC2基因所编码的蛋白质进行三维结构分析结果如图4B。
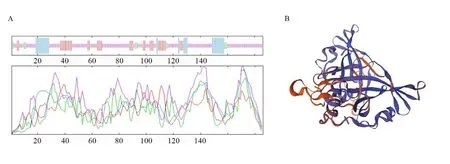
图4 毛红椿TcNAC2蛋白二级结构分析及三维结构预测Fig.4 Secondary structure analysis and three-dimensional structure prediction of TcNAC2 protein
2.4 TcNAC2蛋白的系统进化分析
将TcNAC2基因编码的氨基酸序列在NCBI 数据库中进行BLASTP 比对,选择较高同源性的其他15 个物种的同源氨基酸序列,包括:克里曼丁橘(Citrus clementina)、甜橙(Citrus sinensis)、木薯(Manihot esculenta)、杧果(Mangifera indica)、阿月浑子(Pistacia vera)、胡桃(Juglans regia)、蓖麻(Ricinus communis)、橡胶树(Hevea brasiliensis)、白桦(Betula platyphylla)、番木瓜(Carica papaya)、欧洲栓皮栎(Quercus suber)、棉花(Gossypium hirsutum)、美国山核桃(Carya illinoinensis)、香樱桃咖啡(Coffea eugenioides)、土沉香(Aquilaria sinensis)。使用DNAMAN 9.0 对毛红椿TcNAC2 蛋白及其他15 个物种的同源蛋白氨基酸序列进行多重序列比对分析(图5)。结果显示TcNAC2与其他植物NAC转录因子一样,N-端氨基酸序列呈高度保守,可进一步划分为A~E 5 个保守的亚结构域,这说明NAC 转录因子在进化过程中保守程度较高。使用MEGA 11 软件邻接法(NJ)构建系统进化树,自引导值为500 次,结果表明毛红椿TcNAC2 与克里曼丁橘、甜橙、芒果和阿月浑子植物进化关系较近(图6)。
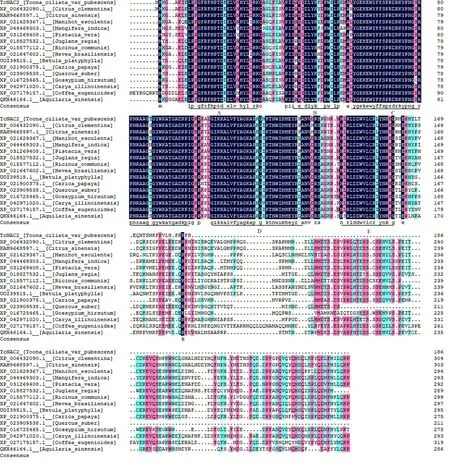
图5 毛红椿TcNAC2氨基酸序列比对Fig.5 Alignment of TcNAC2 amino acid sequence of T.ciliata var.pubescens

图6 TcNAC2蛋白与其他植物同源蛋白的系统进化树分析Fig.6 Phylogenetic tree analysis of TcNAC2 protein and other plant homologous proteins
2.5 TcNAC2基因在毛红椿不同组织中的表达分析
为了解TcNAC2基因在毛红椿不同组织中的表达模式,以前人报道的Actin基因为内参基因,设计特异性引物,采用qRT-PCR 技术检测TcNAC2基因的相对表达量。TcNAC2基因在多个组织中表达,但其表达量具有组织特异性(图7)。TcNAC2基因在毛红椿根中表达量最高,叶其次,在毛红椿茎、叶芽和叶柄中表达量较低。在根中的相对表达量约为叶的2倍,叶芽和叶柄的6倍,茎的9倍。
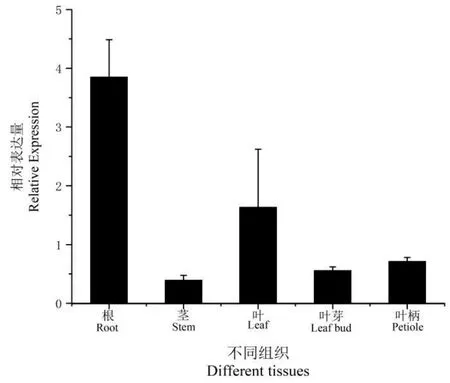
图7 TcNAC2基因在毛红椿不同组织中的表达特异性分析Fig.7 Specific expression analysis of TcNAC2 gene in different tissues of T.ciliata var.pubescens
2.6 毛红椿TcNAC2基因在干旱胁迫下的表达模式分析
为了解毛红椿TcNAC2基因在干旱胁迫下的表达模式,在土壤相对含水量80%(无胁迫)、60%(轻度胁迫)、40%(中度胁迫)、20%(重度胁迫)4 种处理中分别采集叶片样品检测TcNAC2基因的表达情况。以Actin基因为内参基因,设计特异性引物,采用qRT-PCR 技术检测TcNAC2基因在无胁迫、轻度胁迫、中度胁迫、重度胁迫4 种不同程度胁迫下的基因相对表达量。结果显示,TcNAC2在重度胁迫处理时表达量最高,显著高于无胁迫时TcNAC2基因表达量,在轻度胁迫中表达量次之,无胁迫处理中几乎不表达(图8)。

图8 TcNAC2基因在毛红椿不同干旱胁迫程度中的表达分析Fig.8 Analysis of the expression of TcNAC2 gene in different degrees of drought stress of T.ciliata var.pubescens
3 讨论与结论
本研究从毛红椿中克隆获得TcNAC2基因,TcNAC2蛋白在N 端具有NAC 转录因子典型且特有NAM结构域,含有A、B、C、D、E 5 个亚结构域,C 端保守性较低,符合NAC 转录因子家族的特征,故命名其为TcNAC2。
对TcNAC2基因编码蛋白特性分析发现,TcNAC2蛋白与其他植物同源蛋白序列具有较高的相似性,说明了NAC2蛋白在不同物种中相对保守,也证明了该研究基因克隆的正确性。TcNAC2蛋白与克里曼丁橘、香橙等芸香科植物和芒果、阿月浑子等漆树科植物氨基酸序列一致性较高,通过构建进化树分析其与克里曼丁橘、香橙等芸香科植物处于同一分支,亲缘关系较近。将TcNAC2蛋白结构与其他植物已知同源蛋白结构相比较发现,毛红椿TcNAC2蛋白与白木香(Aquilaria sinensis)AsNAC2蛋白[28]具有相同的结构特征:蛋白结构稳定,均属于亲水蛋白且不存在跨膜结构;TcNAC2蛋白二级结构与紫花苜蓿(Medicago sativa)MsNAC2[29]、甘蔗(Saccharum officinarum)ScNAC2[30]相似,均以无规则卷曲为主。亚细胞定位预测结果表明TcNAC2蛋白定位于细胞质,而白木香AsNAC2蛋白[28]和甘蔗ScNAC2[30]预测结果则表明其定位于细胞核的可能性最大,这说明NAC2蛋白在不同的植物中的重组蛋白原核表达条件有所不同。
基因表达调控在植物生长发育过程中非常重要,而转录因子是基因表达功能调控中最基本的调控元件。NAC 转录因子C 端区域通过调节NAC 转录因子与各种靶蛋白的相互作用,赋予NAC 蛋白多种功能[31-32],这使得NAC 转录因子几乎在植物的每个生长发育阶段及多种逆境响应过程都发挥着重要作用[33]。植物中NAC2基因在不同组织的表达水平具有特异性,通过基因表达的组织特异性可以初步预测该基因调控的功能。
该研究中,TcNAC2在毛红椿根中高表达,说明TcNAC2可能参与调控根发育进程。白木香AsNAC2和沙冬青(Ammopiptanthus mongolicus)AmNAC2主要在根中表达[28,34],与本结果基本一致。已有研究表明NAC 转录因子参与调控拟南芥、大豆等植物的根发育进程。例如拟南芥AtNAC1基因表达受到miR164的负调控,可以转导生长素信号进而促进侧根的形成[10]。大豆(Glycine max)GmNAC109基因通过调控生长素相关基因的表达促进侧根生长,过表达GmNAC109使转基因拟南芥的侧根数显著增多[35]等。
本研究发现毛红椿受到重度干旱胁迫时TcNAC2基因高表达,无胁迫时几乎不表达。TcNAC2表达随着不同处理水分梯度下降呈现先升高后下降最后再升高的趋势,在重度胁迫时TcNAC2表达量达到巅峰,说明干旱条件会诱导TcNAC2基因的表达。前人研究发现,NAC 转录因子参与干旱胁迫应答[36],是调控植物抗旱性的关键基因。在大豆中与野生型(WT)植物相比,GmNAC8过表达和GmNAC8敲除株系分别表现出较高的抗旱性和较低的抗旱性[37]。过表达AhNAC3烟草能够上调超氧化物清除相关基因以及与干旱胁迫相关的基因表达,进而提高转基因烟草抗旱性[38]。结合本试验结果,笔者推测TcNAC2基因参与正向调控毛红椿的抗旱性。
综上,本研究从珍稀植物毛红椿中克隆获得一个NAC 转录因子基因TcNAC2,并推测该基因可能与毛红椿根发育及植物抗旱性有关,研究结果为进一步研究TcNAC2基因的结构以及组织表达特异性等特点及其在毛红椿分子育种中的应用提供理论基础。
致谢:江西省教育厅科学研究项目(GJJ200447)同时对本研究给予了资助,谨致谢意!

