融合多头注意力的轻量级作物病虫害识别
赵法川,徐晓辉,宋 涛,郝淼淼,汪 曙,朱伟龙
(河北工业大学 电子信息工程学院,天津 300401)
农作物病虫害种类多、密度大,极易造成作物大量减产,严重制约农业生产,而快速高效地识别病虫害是防治的关键。随着智慧农业的兴起与发展,利用深度学习技术对病虫害进行智能识别以辅助农业生产,减少不必要的农药喷施,对保护生态环境、提高农作物的品质,有着十分重要的作用。
随着数据量的增长和算力的提升,深度学习发展迅猛,诞生出CNN、Transformer等特征提取器,催生出一系列模型。比较经典的卷积网络如VGGNets[1]、ResNets[2]具有识别准确率高的优点,但也存在参数量大、性能差、难以广泛应用于移动端的问题,因此越来越多的学者将目光转向轻量级网络的研究。李静等[3]通过迁移学习对GoogLeNet的Inception-v4网络结构进行优化,对玉米螟虫害识别任务达到96.44%的准确率。刘洋等[4]对轻量级网络MobileNet和Inception V3进行优化,在PlantVillage数据集上分别达到95.02%和95.62%的识别准确率。陆健强等[5]提出一种基于Mixup算法和卷积神经网络的柑橘黄龙病果实识别模型,对柑橘黄龙病数据集的识别准确率达到94.29%。邱文杰等[6]通过知识蒸馏得到压缩模型Distilled-MobileNet,该模型在38种常见病害中达到了97.62%的分类准确率,且模型仅为19.83M。轻量级卷积网络在作物病虫害识别中的应用研究已经颇有成效,但其在模型参数和性能方面仍有继续提升的空间,也许多头注意力机制将是一个突破点。
近两年,在自然语言处理领域大火的Transformer也被成功应用到计算机视觉领域。Dosovitskiy等[7]提出了直接应用于图像块序列的视觉Transformer (Vision transformer,ViT),在ImageNet-1K上取得了88.55%的准确率,刷新了该榜单纪录。相较于CNN(如ResNet),ViT依靠多头注意力机制捕获图像块之间的长距离依赖关系,因此拥有更大的感受野,能获取全局信息,但长程的多头注意力也使得ViT很容易忽略图像的局部性质,而刚好CNN能弥补这一点。相较于ViT网络,CNN的卷积核大多尺寸较小,具有局部特征提取能力,且在现实的工业部署场景中,执行CNN比大多数现有的ViT都要高效。由此可见,将卷积和多头注意力混合设计,有效结合CNN和ViT的优点,可进一步提升轻量级作物病虫害识别模型的性能。
为了将卷积与多头注意力有效结合,设计出高效的作物病虫害识别方法,本研究提出了一个全新的架构M2CNet (Multi-head attention to convolutional neural network)。M2CNet基于层级金字塔结构,并引入深度可分离卷积和循环全连接层进行局部特征提取,同时设计轻量级的全局特征捕捉块,既提高了性能,也节省了计算开销,以期为病虫害精准识别提供新的思路,为后续的边缘平台部署和作物病害检测系统的开发提供新的解决思路和方案。
1 材料与方法
1.1 数据集及处理
1.1.1 CIFAR100数据集 CIFAR100由Krizhevsky等[8]收集,图片主要来自Google和各类搜索引擎。CIFAR100数据集有100个类别,每个类别有600张大小为32像素 × 32像素的彩色图像,其中500张作为训练集,100张作为测试集。这100类被分为20个超类,每个图像都带有一个“精细”标签(它所属的类)和一个“粗略”标签(它所属的超类)。将M2CNet应用于CIFAR100数据集,并与其他模型进行效果比较。
1.1.2 PlantVillage数据集 PlantVillage由Hughes等[9]创建,在植物病理学专家的辅助下完成标注,目的是帮助解决传染病导致的作物产量损失问题。该数据集包含54 309张图像,涵盖南瓜白粉病、桃细菌性斑点病、樱桃白粉病、柑橘黄龙病、玉米枯叶病、玉米灰斑病、玉米锈病、番茄二斑叶螨病、番茄叶霉病、番茄斑枯病、番茄早疫病、番茄晚疫病、番茄细菌性斑点病、番茄花叶病、番茄轮斑病、番茄黄曲叶病、苹果疮痂病、苹果锈病、苹果黑腐病、苹果叶焦病、葡萄叶枯病、葡萄黑痘病、葡萄黑腐病、辣椒细菌性斑点病、马铃薯早疫病、马铃薯晚疫病共计26种作物疾病。试验中按数量8∶2的比例划分训练集和测试集,PlantVillage将用于检验M2CNet在作物病害识别任务中的表现。
1.1.3 IP102数据集 IP102是用于作物害虫识别的野外构建的大规模数据集[10],共有75 222张图像,涵盖了102种常见的害虫,平均每种害虫737个样本,这些图像呈现出自然的长尾分布。病虫害生命周期有不同阶段,例如稻纵卷叶螟在幼虫时期呈现翠绿色的长条节状,而在成虫时期呈现棕灰色的飞蛾形态,与水稻二化螟类似,因此IP102呈现出类间差异小和类内差异大的特点。试验中同样按数量8∶2的比例来划分训练集和测试集,各类具体害虫的训练集、测试集包含的图像数量汇总如表1所示。IP102用于检验M2CNet在作物虫害识别任务中的表现。

表1 IP102 数据集害虫分级分类体系Table 1 Taxonomy of the IP102 dataset on different class levels
1.2 构建病虫害识别网络
本文构建了一种识别作物病虫害的轻量模型-M2CNet,该模型采用金字塔结构,降低空间分辨率的同时能够在不同阶段扩展通道数。M2CNet主要开发了2个重要组件,首先构建了局部捕获块(Local capture block,LCB),该组件主要由深度可分离卷积和多层循环全连接构成,用来捕捉病虫害图片的短距离和细粒度信息;其次构建了轻量级全局捕获块(Lightweight global capture block,LGCB),该组件由全局子采样注意力(Global subsampling attention,GSA)和轻量级前馈网络(Lightweight feedforward network)构成,用来捕捉病虫害图片的长距离和高维信息。模型总体组成如图1所示,下面将分别介绍局部捕获块和轻量级全局捕获块。

图1 M2CNet网络总体组成Fig.1 Overall structure of the M2CNet network
1.2.1 局部捕获块 局部捕捉块的结构如图2所示,其中引入了残差学习[11]的思想,主要由2个连续的深度可分离卷积[12]和1个多层循环全连接[13]构成。深度可分离卷积由1个深度卷积和1个逐点卷积构成,每层卷积后跟随一个批规范化[14],由于频繁地做非线性投影会有害于模型特征的信息传递[15],因此这里减少了激活层。深度可分离卷积先从空间维度获取局部信息,再将获取的局部信息向通道维度传递,最大程度地降低特征的损失;多层循环全连接由2个伪核为 1×3 和 3×1 的循环全连接层构成,其中也使用了残差学习以避免模型加深时出现的退化现象。多层循环全连接层通过阶梯状采样来增大其感受野以更好地集成上下文特征,相比通道全连接有着一步操作就可以同时提取局部信息和融合通道信息的优势。
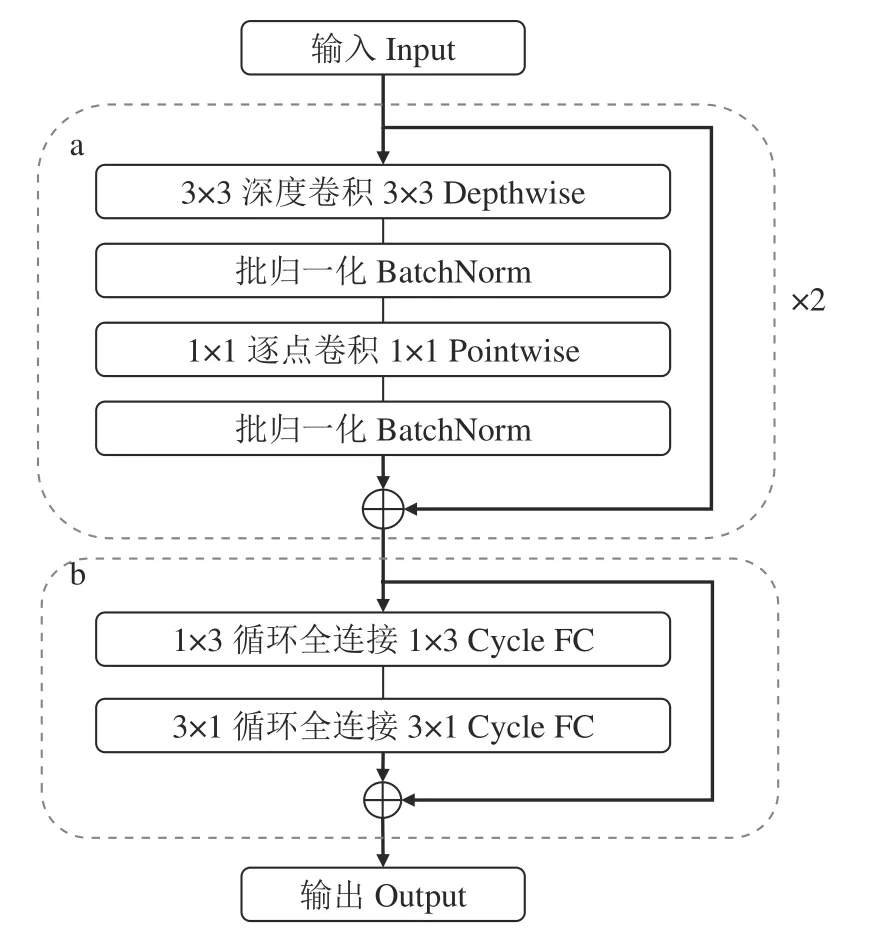
图2 局部捕捉块结构图Fig.2 Structure diagram of a local snap block
深度可分离卷积和多层循环全连接的感受野基本相当,都可以关注局部信息,但深度可分离卷积更侧重于空间维度,多层循环全连接更侧重于通道维度。由于图片数据的纹理在空间维度表现更加明显,因此在局部捕捉块中采取先空间后通道的思想,深度可分离卷积在前,多层循环全连接在后,避免特征提取过程中图片纹理被过度压缩。
1.2.2 轻量级全局捕获块 轻量级全局捕获块由多个轻量结构组成,旨在通过更少的参数来学习更鲁棒的表征。LGCB最核心的部分是一种特殊的多头注意力:全局子采样注意力[16]。图3是标准多头注意力与全局子采样注意力的对比,可以看到全局子采样注意力多出一个次采样(Subsampling)结构,该结构把特征图分为多个不重叠的子窗口(s×s),在子窗口上提取代表键(K)和值(V),但由于查询(Q)是全局的,因此注意力仍可以恢复到全局,这种做法显著减少了计算量。
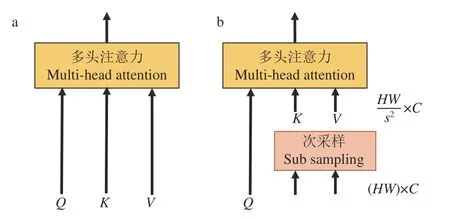
图3 标准多头注意力(a)与全局子采样注意力(b)的对比Fig.3 Comparison of standard multi-head attention and global subsampling attention
轻量级全局捕获块的整体结构如图4所示,LGCB首先对输入特征图做条件位置编码[17](Conditional position encoding,CPE),将输入向量H×W×di映射到高维空间,然后在空间维度展平成向量HW×di,过程中得到了输入特征图的位置信息。在全局子采样注意力阶段,输入特征尺寸为HW×di,次采样的输出尺寸为HW/s2×di,其中di为通道维数,s为子窗口的大小,得到Q=HW×di,K=V=HW/s2×di/h,h为多头注意力头的数量,将QKV共同送入多头注意力。最后经过轻量级前馈网络[18]将输入从di降维到di/r,再从di/r升维到di,其中r为降维因子,通常取r=4,该操作用于提升模型容量。简单地,轻量级全局捕获块可以表述如下:

图4 轻量级全局捕获块Fig.4 Lightweight global capture block
式中,Xin表示输入张量,Norm是层归一化操作,CPE是条件位置编码,GSA是全局子采样注意力,Lightweight FFN是轻量级前馈网络。所有这些操作都可以在标准深度学习平台通过常用和高度优化的操作来实现。
1.2.3 M2CNet模型架构 为满足不同的边缘部署需求,本研究提出了3个典型的变体,即M2CNet-S/B/L。架构规范如表2所示,对于归一化,在局部捕捉块中使用批归一化,在轻量级全局捕获块中使用层归一化,对于激活函数均使用ReLU。
2 结果与分析
2.1 试验环境与评价指标
本研究在Ubuntu 20.04系统展开,该系统搭载GeForce RTX 3 090图形处理器并通过并行计算架构CUDA 11.4和CUDNN 8.2.4驱动,深度学习框架选择PyTorch 1.10.1,编程语言为Python 3.8.5。训练时CIFAR100和IP102的迭代次数设为300,PlantVillage的迭代次数设为60,批次均为64。学习率选择余弦衰减[19]策略,PlantVillage和CIFAR-100的初始学习率设为0.000 5,IP102的初始学习率设为0.005,前10个迭代次数学习率均使用线性启动。优化器选择Adamw[20],并将权重衰减设置为0.05,在训练中还使用了标签平滑[21]和Mixup[22]数据增强来进一步探索模型性能。训练时图像使用224像素×244像素的随机裁剪,测试时使用224像素×244像素的中心裁剪。
评价指标采用Top1准确率、Top5准确率和损失值,Top1准确率指预测概率排名第1的类别与实际结果相符的准确率,Top5 准确率是指预测概率排名前5的类别与实际结果相符的准确率。准确率(Accuracy)和损失值(Loss)的计算公式如下:
式中,TP为真正类,TN为真负类,FP假正类,FN假负类;p(xi)代表真实的标签,q(xi)代表预测的概率。
2.2 模型性能对比
本研究将M2CNet应用于CIFAR100,并与多种模型进行了比较,包括许多经典的计算量(Floating point operations,FLOPs)小于1G的轻量级卷积网络,例如ShuffleNets[23-24]、SqueezeNet[25]、MobileNetV2[26]、MobileNetV3[27]、MnasNet[28]、EfficientNet[29],还包括ViT模型MobileViT[30]和大型模型VGG,M2CNet-S/B/L的训练过程见图5,从图5可以直观地看到随着300次迭代的收敛,M2CNet-S/B/L在训练集和测试集的损失值逐渐降低,直至趋于平稳。

图5 M2CNet-S/B/L在CIFAR100数据集的训练过程Fig.5 M2CNet-S/B/L training process in the CIFAR100 dataset
表3是对比结果,在参数量和计算量相似的情况下,M2CNet-S/B/L占据一定优势,且M2CNet-L参数量和准确率最优。与ShuffleNet系列相比,本研究的M2CNet-S/B比ShuffleNet-V2 1.5/2.0分别在Top1准确率上实现了4.53、2.53个百分点的提升。与MnasNet系列相比,M2CNet-S/B/L比MnasNet 0.75/1.0/1.3分别在Top1的准确率上实现了1.89、2.62和1.75个百分点的提升。与MobileNet系列相比,M2CNet-S/B/L分别在Top1准确率上实现了9.35、6.16和5.12个百分点的提升。由此可见将多头注意力机制与卷积结合可以有效提升卷积模型的性能,例如M2CNet-S与MobileNet-V2、MobileNet-V3-Large参数量和计算量相似,但其识别精度却更优。在与MobileViT系列的对比中,M2CNet-S/B/L同样在识别精度上展现出明显优势。本研究也将M2CNet与大型模型做对比,可以看到M2CNet-L比VGG系列、ResNet 18准确率更高,而参数量仅为ResNet 18的一半,是VGG系列的1/20。由此可见M2CNet可以在模型参数量和准确率之间保持平衡。
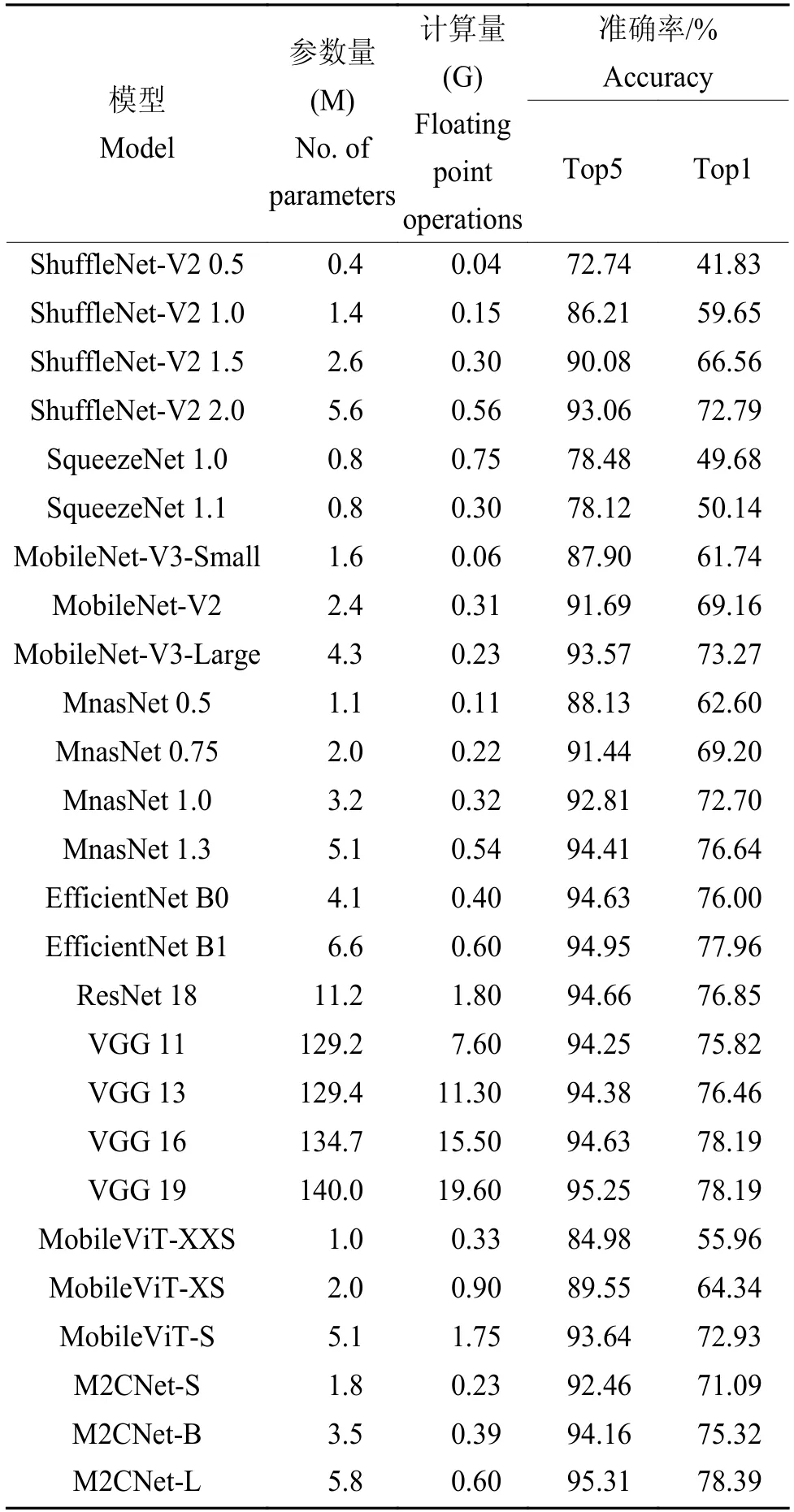
表3 CIFAR100数据集模型对比结果Table 3 Comparison results of CIFAR100 dataset model
2.3 病虫害识别效果
为了更好地比较M2CNet-S/B/L的效果,本研究针对每一种变体找到了在参数量和计算量上相似的对照,即M2CNet-S对应MobileViT-XS、MobileViT-XXS、MnasNet 0.75、MobileNet-V2;M2CNet-B对应MobileNet-V3-Large、EfficientNet B0、MnasNet 1.0;M2CNet-L对应EfficientNet B1、MobileViT-S、MnasNet 1.3。将以上网络分别在PlantVillage病害数据集和IP102虫害数据集上展开试验,试验结果见图6。

图6 病虫害数据集识别结果Fig.6 Identification results of pest data sets
图6a是PlantVillage数据集识别结果,可以看到在各组对照中M2CNet-S/B/L分别取得了95.92%、96.82%、97.15%的最大Top1识别准确率,在参数量相似的情况下取得了最优的结果。图6b是IP102数据集识别结果,M2CNet-S/L依然延续了在PlantVillage上的表现,分别取得了67.08%、71.0%的最大Top1准确率和88.49%、90.50%的最大Top5准确率。在M2CNet-B对照中MnasNet 1.0取得了69.46%的最大Top1准确率,超出M2CNet-B 0.47个百分点,不过从整体来看,M2CNet变体在作物病虫害识别任务中依然表现出色。M2CNet变体能在对照试验中取得比其他轻量级网络更有竞争力的结果,分析原因在于融合多头注意力的M2CNet不仅关注局部信息,也关注全局信息,因此能够灵活应对不同特征尺度的变化。
2.4 热力图可视化
为了进一步解释融合多头注意力后M2CNet-S/B/L关注的区域,这里使用Grad-CAM[31]方法在病害和虫害的部分数据集上抽取特征图进行可视化,其可视化结果如图7所示。可以看到,由M2CNet-S到M2CNet-B再到M2CNet-L,模型对于分类识别任务中更有判别性的特征区域给予了更高的关注,在一定程度上降低了背景特征的干扰,进而提升了模型识别精度。

图7 网络关注区域热力图Fig.7 Thermal map of the network focus area
3 结论
本研究为设计出轻量级作物病虫害识别方法,将多头注意力机制捕捉长距离依赖关系的能力与卷积神经网络的局部特征提取能力相结合,设计出满足不同边缘部署需求的3个变体:M2CNet-S/B/L。
为了验证M2CNet-S/B/L的特征提取能力,在CIFAR100数据集上将其与其他轻量级网络展开对比,在参数量和计算量相似的情况下,M2CNet 3个变体均表现出良好的性能。在与经典的轻量级网络MobileNet系列比较中,M2CNet-S/B/L在Top1准确率上分别实现了9.35、6.16和5.12个百分点的增益。
在作物病虫害数据集的试验中,M2CNet-S/B/L在PlantVillage病害数据集上取得了大于99.70%的Top5准确率和大于95.92%的Top1准确率,在IP102虫害数据集上取得了大于88.4%的Top5准确率和大于67.0%的Top1准确率,且在同级别网络的对比中均占有优势,证明M2CNet能够胜任作物病虫害识别任务。
M2CNet网络有着参数量少的优点,以M2CNet-S为例,其参数内存仅占用1.8M,对硬件性能(FLOPs)要求仅为0.23G,这极大降低了对硬件平台的要求,有利于后续的边缘平台部署和作物病害检测系统的开发和普及。

