小麦高质量SNP对产量及根系表型的潜在影响分析与在线数据库的构建
胡尊铠,朱志伟,刘子辉,赵 鹏,王晓明,许盛宝
(西北农林科技大学 农学院,陕西杨凌 712100)
小麦(TriticumaestivumL.)是全球种植范围最广泛的作物之一,为人类提供了20%的食物来源,其产量与人类的生存息息相关,而根系作为小麦扎根地下的器官,对小麦吸收土壤中的水分和营养物质起到至关重要的作用[1-2]。随着下一代测序技术(Next-generation sequencing,NGS)的快速发展,分子标记辅助育种已经成为较为前沿的手段。分子标记种类有很多,例如简单重复序列(SSR)、限制性片段长度多态性(RFLP)、随机扩增多态性DNA(RAPD)、任意引物PCR(AP-PCR)以及单核苷酸多态性(SNP)等[3-6]。相比于其他分子标记,SNP标记有着遗传稳定性较高、位点丰富、分布较为广泛以及代表性强等特点,已经被广泛用于各种生物学研究[7-9]。但是将原始测序数据转化为储存变异信息的SNP数据需要使用大量的命令行工具以及编程语言脚本,这对于几乎没有生物信息学经验的生物学家以及育种家来说,是一项及其耗时耗力的工作。随着计算机科学以及生物信息学的不断发展,由专门的生物信息学研究人员将原始测序数据转化为储存变异信息的结果文件,利用在线数据库将这些信息通过网页展示并提供查询、分析以及下载,使研究人员不需要专门学习生物信息学技能便可以对需要的信息进行检索和利用。
其他植物在线数据库的应用度要高于小麦,比如拟南芥的PHOSPHAT磷酸化位点数据库、GENEVESTIGATOR微阵列数据库、SUBA亚细胞数据库、ARACYC生化途径数据库以及DATF转录因子数据库等[10-14];水稻的RAP-DB基因组数据库、ORYZABASE基因组数据库、RICEVARMAP基因组数据库以及RMD突变体数据库等[15-18]。小麦由于其基因组较为庞大和复杂,导致参考基因组测序完成的时间较晚,因此小麦可供查询和使用的SNP数据库屈指可数,这严重制约了小麦研究的推进。因此利用小麦苗期根系转录组数据开发的原始SNP过滤得到高质量SNP,研究其对小麦产量及根系等相关性状存在的潜在影响,并以此为基础构建数据库,为分子标记辅助育种提供参考。
本研究利用413份小麦自然材料苗期根系转录组测序数据开发高质量SNP标记,根据该群体产量、根系等相关性状的表型观测数据,鉴定这些SNP标记对产量以及根部表型存在的潜在影响,并将过滤得到的高质量SNP标记构建在线数据库,为后续的分子标记辅助育种提供参考。
1 材料与方法
1.1 试验材料
413份收集自世界各地的小麦材料构成的自然群体。所有材料的详细信息见数据库首页的Variety information部分(https://iwheat.net/links/)。
1.2 试验设计
1.2.1 田间试验 2018-2019、2019-2020和2020-2021年,在陕西省咸阳市杨陵区曹新庄试验农场进行两个播期的播种,分别为当年10月初的正常播种和次年1月中旬的晚播;2018-2019年和2020-2021年,在四川省崇州市四川农业大学现代农业研究基地当年10月末进行正常播种。采取随机区组设计进行试验,每个地点均设计3个重复,品种间均以行长1 m、行距20 cm种植,每行点播10粒种子,并根据当地的气候和地质状况进行田间管理。
1.2.2 根系培养试验 对每份供试小麦材料分别选取籽粒饱满且大小均匀一致的种子16 粒。将种子分别装入15 mL离心管,注入无菌蒸馏水常温浸泡6 h。用消毒液对种子表面消毒10 min,使用蒸馏水冲洗干净,注入1 mL无菌蒸馏水于4 ℃冰箱低温处理2 d。在发芽盒中铺入滤纸,并将种子移入,注入5 mL无菌蒸馏水后用保鲜膜封口,并戳6~9 个小孔通气。将发芽盒放入培养箱培养14 d(温度:光照条件下24 ℃,黑暗条件下20 ℃;湿度:50%;光照度:2 000 lx;光照 16 h;黑暗8 h),期间定期补充无菌蒸馏水,保证滤纸处于湿润状态。
1.3 表型测定
1.3.1 产量表型测定 对产量性状使用下述标准进行调查:有效分蘖数(Productive tiller number,PTN):人工统计小麦能够抽穗并结实的分蘖;穗粒数(Kernel number per spike,KNS):人工统计每个小穗的籽粒数;千粒质量(Thousand kernel mass,TKM):使用SC-G谷粒外观质量快速图像分析系统分析1 000粒籽粒的质量。
1.3.2 根系表型测定 将培养箱中生长14 d的小麦初生根于贴近种子处剪下,擦干表面水分并称重,获得根鲜质量(Root fresh mass,RFM),利用万深LA-S植物根系扫描仪进行根系表型扫描。通过万深LA-S根系分析系统(V.2.6.4.4)分析每个小麦材料根表面积(Root surface area,RS)和根体积(Root volume,RV)数据。
1.4 表型数据处理
使用Excel 2019(Microsoft Office Home and Student 2019)和Python(3.7.4)程序语言处理产量以及根部表型的观测数值,利用R语言(3.6.4)的R包Lme4计算产量相关的多年多点表型观测值的最佳线性无偏估计值(Best linear unbiased estimate,BLUE)。
1.5 高质量SNP标记过滤
对西北农林科技大学小麦非生物胁迫耐受机理实验室前期开发的原始SNP文件,使用以下标准进行过滤:只保留纯合基因型,并且每个样本的支持reads必须大于等于30。根据上述参数,未通过的SNP被认定为缺失,通过的SNP认定为高质量SNP。
1.6 显著性检验
按照SNP的基因型将小麦群体分为两个基因型亚群,对两个亚群的表型进行显著性检验,结合随机抽样的置换检验。使用Python语言的Scipy模块中的stats功能进行显著性检验,首先检验两组试验数据的方差齐性,若方差齐性通过检验,参数equal_var设置为True,否则该参数设置为Fasle。为去除偶然性带来的影响,使用Python的random函数,对需要显著性检验的数据进行随机抽样,每次抽取80%的数据,抽样1 000次,取超过800次显著的结果为通过置换检验的结果。显著性检验阈值参考Bonferroni矫正,取0.05/SNP总数=1×10-7。
1.7 在线数据库搭建
利用R语言Shiny框架对数据库进行构建。整体的数据库构建流程如图 1所示,使用R语言shiny包搭建web框架,分别编写UI模块以及Server模块,UI模块用于设计web网页的页面前端布局以及页面,本数据库对UI模块使用R包shinydashboard对前端页面进行设计,Server模块的代码负责后端程序的功能实现并且控制输出,将上述结果整合到CSV文件后,借助R包DT将数据在网页数据库的前端界面中输出,并且在Server模块中结合CSS语言代码块对数据库表格进行外观属性配置。

图1 在线数据库搭建流程Fig.1 Processfor constructionof online database
2 结果与分析
2.1 表型分析
通过对413份供试材料的产量(有效分蘖数、千粒质量和穗粒数)和根系(根体积、根表面积和根鲜质量)相关性状的表型观测值进行统计分析,结果表明,产量与根系相关性状的表型变异系数为10%~22%,品种之间存在广泛的表型变异;表型的偏度系数的绝对值都<1,除穗粒数表型之外,其他表型的峰度系数的绝对值也都<1,这表明本研究所使用群体表型数据均呈现正态分布,符合数量性状的特征,说明本研究的目标性状为多基因控制的数量性状(表 1)。相关性分析表明根系表型之间呈现极显著的强烈正相关,而产量性状之间呈现显著的负相关关系;千粒质量与根部表型之间呈现显著正相关关系,有效分蘖数与根部表型呈现显著的负相关关系,穗粒数与根部表型之间不存在相关关系(表 2)。
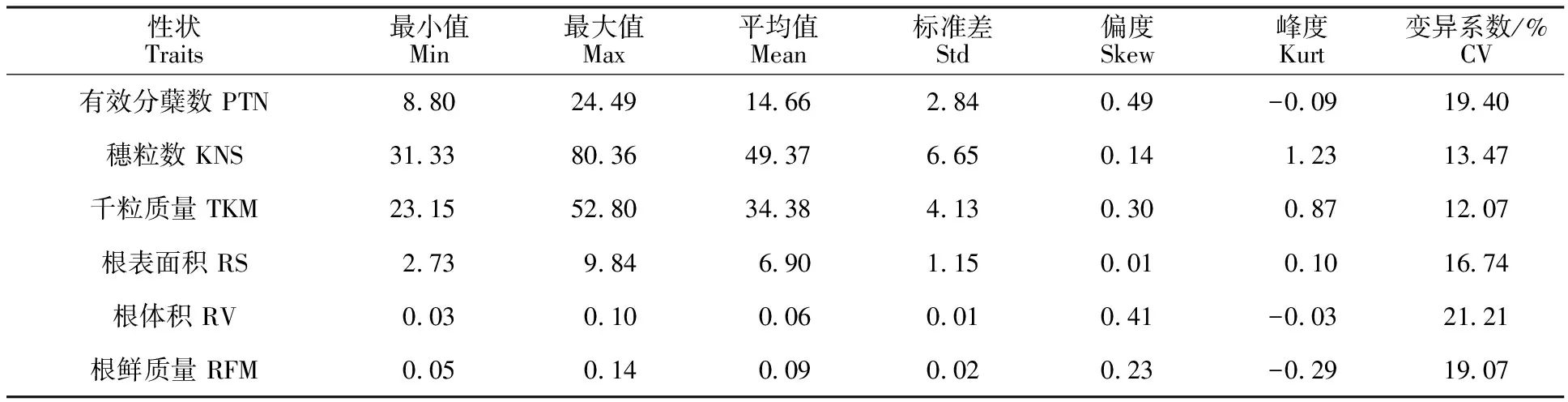
表1 产量及根部相关性状的统计分析Table 1 Statistical analysis of yield and root-related traits
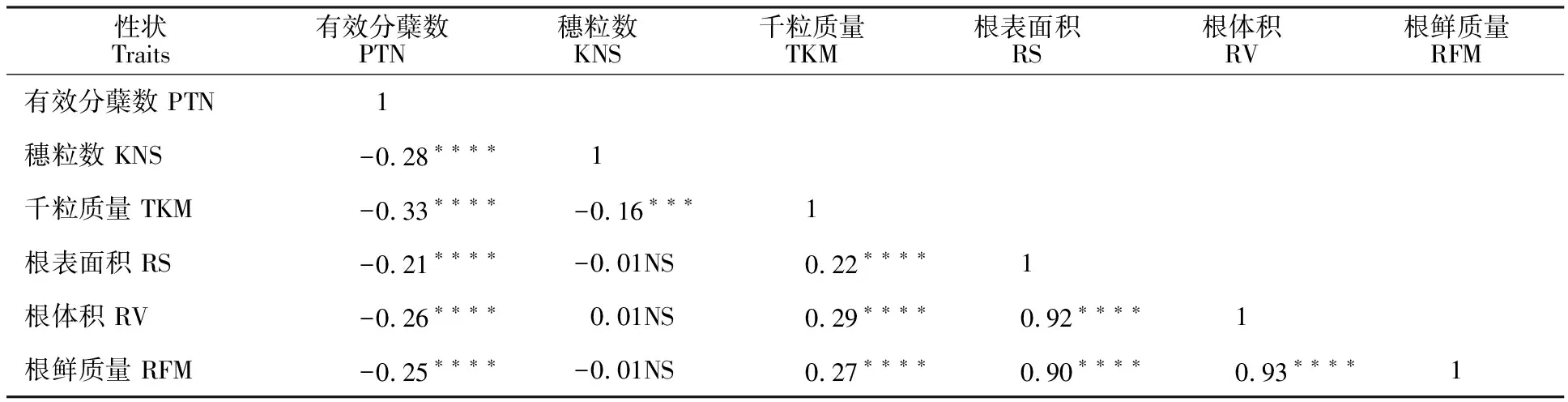
表2 产量及根部相关性状的相关性分析Table 2 Correlation analysis of yield and root-related traits
2.2 SNP分布特征
通过对小麦苗期根系转录组数据的过滤以及质量控制,在其中筛选出高质量SNP标记 45 898个,在小麦A、B和D亚基因组上鉴定到的SNP数目分别为19 381、22 444和4 073,其中B亚基因组含有的SNP最多,而D亚基因组上鉴定到的SNP数目远小于A亚基因组和B亚基因组。从染色体的分布上看,2B染色体上存在的SNP最多(3 973个),4D染色体上存在的SNP最少(173个)(表 3)。
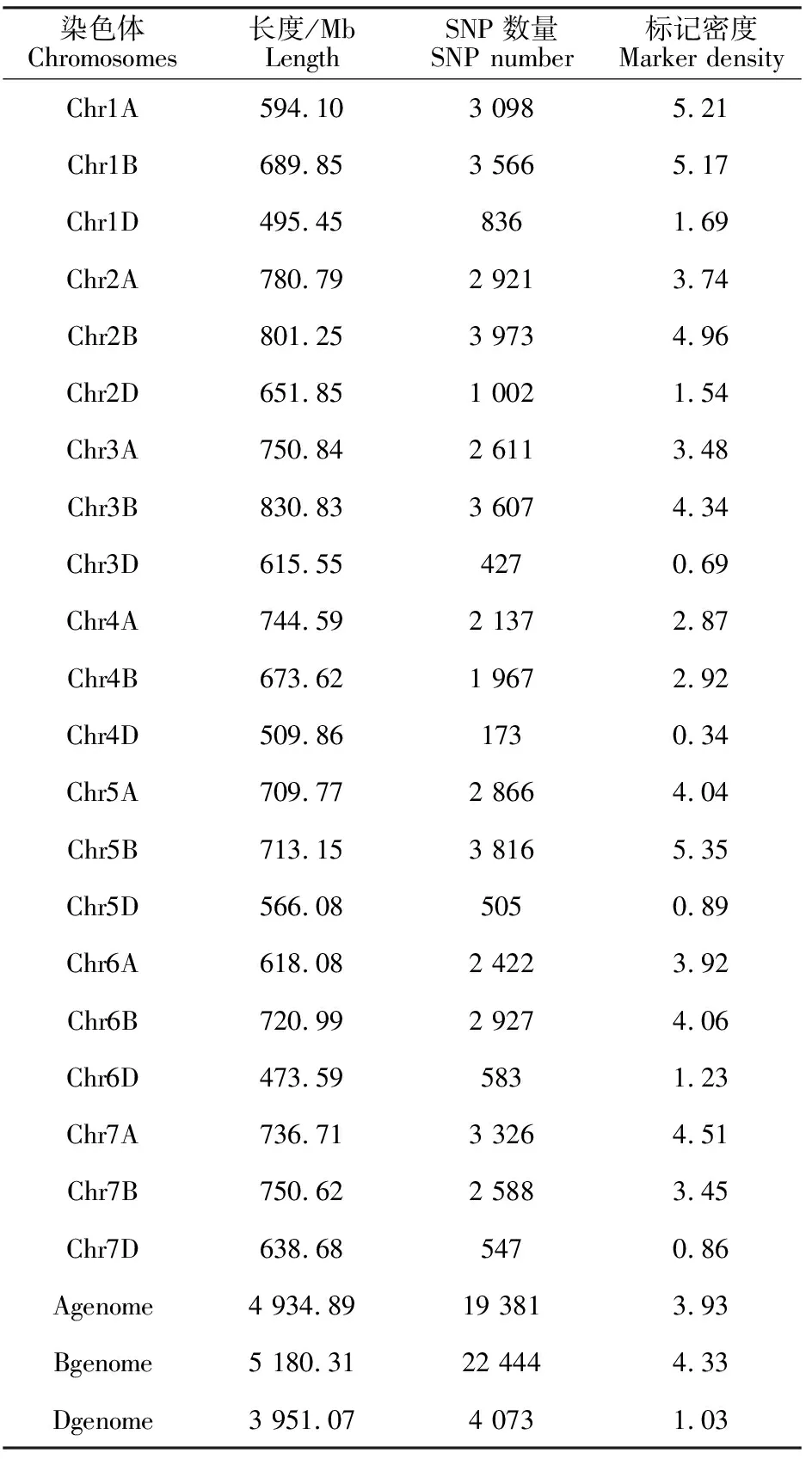
表3 小麦自然群体SNP标记基本统计分析Table 3 Basic statistical analysis of SNP markers in natural populations of wheat
2.3 SNP对表型的影响
为检验本试验群体过滤得到的高质量SNP标记对小麦产量性状的影响,选取所有高质量SNP,根据其基因型对群体进行分组,并采用显著性检验分析不同基因型对产量性状的影响。结果表明,在产量相关性状上,有123个SNP的2种基因型间千粒质量存在显著差异,其中87%的SNP突变后会增加千粒质量;有299个SNP的2种基因型间穗粒数存在显著差异,其中92%的SNP突变后会减少穗粒数;有1 660个SNP的2种基因型间有效分蘖数存在显著差异,其中41%的SNP突变后会增加有效分蘖数。在根系相关性状上,有51个SNP的2种基因型间根表面积存在显著差异,这些SNP突变后全部增加根表面积;有121个SNP的2种基因型间根体积存在显著差异,其中97%的SNP突变后增加根体积;有97个SNP的2种基因型间根鲜质量存在显著差异,其中98%的SNP突变后增加根鲜质量 (表 4)。

表4 SNP对表型的潜在影响Table 4 Potential effect of SNPs on phenotype
2.4 数据库使用
本数据库包含3个部分,分别为小麦群体材料信息部分(Variety Information)、高质量SNP查询部分(Population SNP Database)以及小麦群体标记数据下载部分(SNP Data Download)。
小麦群体材料信息包含413份小麦材料的编号、名称、类型(农家种或现代栽培种)以及种植地区。该模块内容见图 2。
高质量SNP标记信息包含SNP物理位置、碱基突变类型、对产量和根部表型的影响情况以及在每个材料的变异情况,Chr列与Pos列可以得到SNP标记的物理位置信息,REF列与ALT列得到SNP标记的碱基类型,Down列与Up列得到SNP标记对产量和根系表型的影响信息(Down为降低表型数值,Up为增加表型数值);群体标记信息栏可以随下方滑块拖动,该信息栏可以得到413份小麦材料的SNP信息(“0/0”代表该位置碱基类型与中国春参考基因组一致, “1/1”代表该位置碱基类型与中国春参考基因组不一致,“./.”代表该位置没有测得高质量的SNP)。该模块的内容如图 3所示,研究人员可以通过对感兴趣的表型进行搜索,得到与该表型相关的SNP标记信息以及这些标记在群体材料中的分布情况信息。
小麦群体标记数据下载部分包含供试群体所有高质量SNP标记数据以及每条染色体的SNP标记数据。该模块的内容见图 4。
图2 小麦群体材料信息部分Fig.2 Information material of wheat population
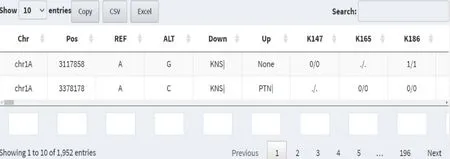
图3 高质量SNP查询部分Fig.3 High-quality SNP query
图4 小麦群体标记数据下载部分Fig.4 Download links of wheat population marker data
综上,研究人员通过本数据库获得的信息,可以为后续的育种工作提供参考。例如,查询群体中对千粒质量存在影响的SNP标记,并且根据SNP变异后对千粒质量表型的效应(增加或减少),在育种工作中对相应的SNP进行有目标的选择使用。
3 讨 论
小麦是世界三大作物之一,其种植面积占粮食总种植面积的五分之一,小麦产量的变化将会对粮食安全产生重大影响[19]。SNP标记具有位点多、分布广、稳定性高和代表性强的优点,已广泛用于小麦育种研究[7]。前人研究表明,在亚基因组层面,小麦SNP标记在B亚基因组最多,D亚基因组最少[20]。本研究也得到了相同结果,B亚基因组存在的SNP最多(48.9%),其次是A亚基因组(42.2%),在D亚基因组上存在的SNP最少(8.9%),可能是D亚基因组在小麦进化过程中较为保守所致[21]。
产量以及根系相关性状的相关性分析表明,千粒质量与根系相关性状呈显著正相关,刘佳熠等[22]发现苗期根系性状与千粒质量性状呈现显著正相关,与穗粒数性状相关性不显著。本研究结果与其相同,可能是根系发育较好的小麦,能够更好地吸收利用土壤中的水分和养料,促进籽粒灌浆,增加千粒质量[23]。此外,本研究还发现有效分蘖数与根系相关性状之间呈显著负相关,此现象鲜有报道,以后可继续进行研究。
目前已经有一些小麦SNP数据库公布,用来辅助育种工作者,以便于他们使用分子标记进行育种工作,如WGVD数据库、Triticeae-GeneTribe数据库以及Snphub数据库[24-26]。本研究基于小麦苗期根系转录组测序数据过滤得到的高质量SNP标记构建在线数据库,与其他小麦数据库相同的是,本数据库可以对标记信息进行查询和下载,不同的是,本研究的高质量SNP大多数位于编码区。此外,本数据库还提供了原始标记数据的下载方式,研究人员可以下载原始标记数据进行分析。相较于其他数据库,本数据库也存在一些不足,比如未提供数据可视化接口,后续会针对此不足对数据库进行维护和更新。

