基于RoBERTa-CRF的肝癌电子病历实体识别研究*
邓嘉乐 胡振生 连万民 华赟鹏 周 毅
(1中山大学中山医学院 广州 510080 2广东省第二人民医院 广州 510317
3 中山大学附属第一医院 广州 510080)
1 引言
肝癌患者在医院就诊时产生大量电子病历数据,记录患者入院检查、治疗以及出院全过程,蕴含大量医学专业数据和专业知识。然而这些数据多是非结构化文本数据。对其进行结构化处理,将相关知识自动提取出来,有助于后续医疗辅助决策系统、医疗知识图谱的构建以及精准医学研究。此外,结构化数据有助于患者和医生直观获取所需信息。如何将这些信息从无结构化文本中自动抽取出来成为研究的重点问题。而应用自然语言处理技术中的命名实体识别技术可以完成电子病历自动化知识抽取任务。
2 研究意义及背景
2.1 研究意义
肝癌又名肝脏恶性肿瘤,分为原发性肝癌和继发性肝癌。世界卫生组织国际癌症研究机构发布的2020年最新全球癌症负担数据显示,中国肝癌新发病例数约占全球45.3%,死亡例数约占全球47.1%,是目前我国第4位常见恶性肿瘤及第2位致死肿瘤。在实体识别领域,国内肝癌电子病历实体识别研究较少。运用机器学习算法对肝癌电子病历进行实体识别有利于肝癌电子病历规范化、结构化数据存储,以及相关潜在医学知识挖掘。
2.2 国内外研究现状
命名实体识别[1]是自然语言处理任务中的重要一环,对关系抽取[2]等任务有重要影响。其目的是从非结构化文本中抽取实体,医疗实体包括症状、疗效以及疾病名称等。在机器翻译[3]任务中,实体翻译有特殊规则或固定表达。精准找出实体并通过预先规则或字典库翻译,可提升机器翻译的效果和流畅性。Wang Q等[4]将命名实体识别技术应用于中文实体链接方法,实现了将中文诊断和程序术语规范化为国际疾病分类(international classification of diseases,ICD)代码的应用。实体识别相关技术在生物医学[5]、专病库建设[6]和健康医疗大数据[7]等多个领域得到较好应用。
命名实体识别发展经历3个阶段。第1阶段是基于词典和规则的方法,需要不同领域专家制定词典与规则模板,在文本中进行匹配。第2阶段是基于统计机器学习的方法,基于序列化标注方式并最大化联合概率进行求解。第3阶段是深度学习方法,代表模型有长短期记忆人工神经网络(long-short term memory,LSTM)[8]、双向编码器表征(bidirectional encoder representations from transformers,BERT)[9]等。
电子病历研究在国外已开展多年,Clark C等[10]提出基于最大熵模型和条件随机场的方法。Jiang M等[11]采用支持向量机方法,F1值达0.848。Bruijn B等[12]则采取统一医学语言系统(unified medical language system,UMLS)资源半监督隐马尔科夫模型,F1值达0.852 3。Gligic L等[13]使用基于注意力的序列到序列模型。英文电子病历实体识别经过数年发展完善,已形成标准处理流程。
中文医疗实体识别[14]难度更大,边界定义模糊,存在嵌套关系,缺少统一公开的大规模语料库。用统计学习方法进行实体识别[15]时,特征选择影响结果。Lei J等[16]根据出入院记录,分别采用条件随机场(conditional random field,CRF)、支持向量机(support vector machine,SVM)、最大熵等模型对4类医疗实体进行识别。Wang Y等[17]在自标注中医文本语料上,采用分句形式,F1值达0.95。李博等[18]采用Transformer[19]模型,使数据中长依赖关系进一步提升。杨红梅等[20]对肝癌电子病历采用BiLSTM-CRF模型取得较好效果;马欢欢等[21]利用CNN-BiLSTM-CRF模型对癫痫电子病历进行实验,达到较好效果。
近年来,研究者提出BERT算法,通过注意力学习字符间上下文关系。核心创新点在于两个训练任务:掩码语言模型训练和下句话预测任务。掩码策略下,模型在训练中随机掩盖单词再进行预测。下句话预测任务目的是服务问答、句主题关系等任务。针对BERT的缺点,研究人员提出RoBERTa模型[22]。该模型有更大参数量、更大批大小、更多训练数据。在掩码策略上,修改关键超参数,实现动态掩码策略,删除下句话预测任务,使模型能更好推广到下游任务。
3 构建RoBERTa-CRF实体识别模型
3.1 RoBERTa层
该层输入向量由词嵌入向量、位置嵌入向量、段嵌入向量组成。词嵌入向量词表为50 000比特级别的文本编码词汇。位置嵌入向量可以记录词位置,弥补自注意力模型无法感知词位置的缺点。段嵌入向量记录句子位置关系。
该层使用编码器双向编码,使词可以无视前后远近被其他词参与编码。编码器中包含多头自注意力,以全连接前馈网络方式连接。多头自注意力将关注句子不同信息的注意力融合计算。以3个向量为例,多头自注意力计算方式如下:
MultijheadAttention(X,Y,Z)=
Concat(head1,head2,...,headh)WO
(1)
(2)
自注意力对X,Y,Z3个向量进行向量运算,编码器输入的字向量在整个输入中点积和加权求和,得到此位置的自注意力,计算方式如下:
(3)
训练方法上,去掉下一句预测任务,实现动态掩码策略。每次输入序列随机选择15%的词,其中80%被[Mask]标签代替,10%用另一个词代替,10%不改变。数据分多序列输入过程中,不同掩码模式生成,模型在不同掩码模式中预测被掩码覆盖或者被替换的原词汇。
3.2 CRF层
CRF对类之间的决策边界进行建模。命名实体识别可看成多分类问题,因此该层任务是由输入序列对输出序列预测,形式为对数线性模型,学习方法是极大似然估计或正则化极大似然估计。假定句子长度为n,句子序列为X=(x1,x2,...,xn),对应预测标签序列为Y=(y1,y2,...,yn),该预测序列总分数如下:
(4)
其中,T表示标签间的转移分数,Pi,yi表示每个字到对应yi标签的分数。预测序列有多种可能性,只有一种正确,应对所有可能序列进行全局归一化。学习方向是调整参数将模型正确预测的概率提升。归一化预测序列概率如下:
(5)
3.3 RoBERTa-CRF实体识别模型
将CRF的输入变为含有更准确语言表征的词向量,组合模型预测能力会得到提升。因此将CRF层作为第2层,在CRF层之前加入RoBERTa层。用分词工具对原始语料句子分词后,RoBERTa层可学习到更准确的字符向量表示。再将字符向量输入到CRF层中,可得到更好预测结果,见图1。
4 实验及结果分析
4.1 数据集介绍
所用数据集为500位肝癌患者的电子病历数据,已进行脱敏处理。电子病历中包含入院记录、出院记录和超声检查3部分。结合相关医学知识,本文定义23类肝癌数据相关实体(不包含无标签实体),并对数据进行BIO方法标注(实体第1个字符标签为“B-实体类型”,后续字符为“I-实体类型”,无标签字符为“O”)。为验证语料标注一致性,3名标注人员分别标注同样5份数据,计算标注一致性达88%。全部标注后得到有效标注242份,见表1。

表1 实体类型
将标注后的数据按1∶1∶3划分为测试集、验证集和训练集。训练集实体类型频数分布,见图2。

图2 训练集实体类型频数分布
训练集各实体类别数量存在较大差异,分布极不均衡,如疾病、体位等实体数量大约在35 000个左右,性别、年龄等实体数目只有几百个。对每份电子病历数据的文本长度进行分析统计,主要集中在2 000个字符,同时也存在包含8 000~10 000个字符的超长文本,文本长度过长会导致模型难以捕获到文本间的长依赖关系,因此在模型训练前需要对数据进行截断切分。
4.2 数据平移
数据平移是将原始电子病历数据从不同切分点开始切分,然后将切分后的数据分别进行训练,最终对多个模型进行投票。本实验将每份电子病历数据拆分成最大长度为256个字符的样本,第1种切分方式为从起始位置累计达到256个字符进行切分;第2种切分方式是首先将数据中前两个句子作为一个样本,然后再依次切分。
4.3 交叉验证
采用5折交叉验证,将训练集划分为5部分,每次以其中4部分作为训练集剩余部分组成验证集,最终训练得到5个模型参数。对于实体识别任务,可以对5个模型结果进行投票,也可以将5个模型参数平均后再进行解码预测。本文采用投票方式。
4.4 参数设置
本实验中RoBERTa预训练模型使用RoBERTa-zh-base版本,模型层数为24层,使用30G原始文本,近3亿个句子,100亿个中文字,产生2.5亿个训练数据。覆盖新闻、社区问答、多个百科数据等。RoBERTa训练时使用批大小为12,学习率为1e-4,优化器为Adam优化器,CRF层学习率为1e-2。
4.5 评价指标
实验使用精确率P=TP/(TP+FP),召回率R=TP/(TP+FN)以及F1=2×P×R/(P+R)值对模型效果进行评价。其中,真阳性(true positive,TP)是指预测的实体类型与原本实体类型一致的观测样本数量,假阳性(false positive,FP)是指模型将本不属于该实体类型的观测预测为该实体类型的样本数量,假阴性(false negative,FN)是指模型将原本属于该实体类型的观测预测为非该实体类型的数量。
4.6 结果分析
4.6.1 多模型性能对比 实验使用RoBERTa-CRF命名实体算法模型,并将命名实体识别中传统的隐马尔科夫(hidden markov model,HMM)模型、条件随机场、BiLSTM-CRF、BERT-CRF算法模型作为基线模型进行对比,见表2。
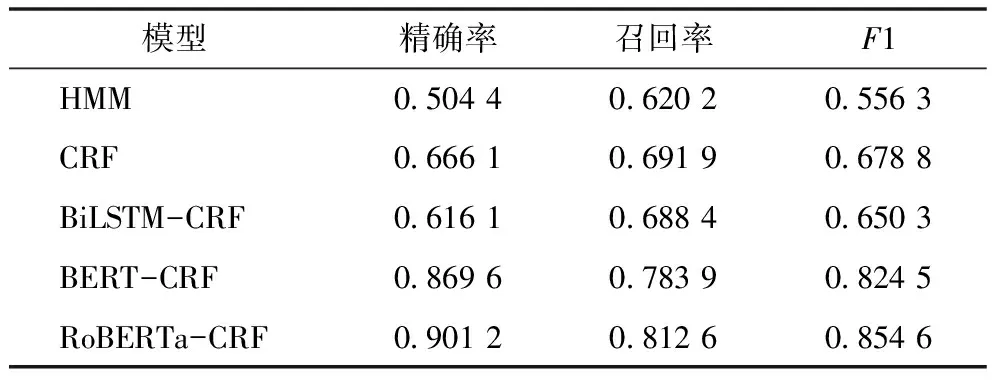
表2 各模型测试集性能对比
RoBERTa-CRF算法模型在肝癌电子病例数据上各指标均达最高,表现最好。BiLSTM-CRF模型F1值相对CRF模型略低,主要是因为肝癌电子病历数据中存在很多短句和缩略词,其中缩略词记录患者病情或者身体状况,短句间没有很强语义关系,模型未学得合理的上下文语义表示。BERT-CRF在3个指标上都低于RoBERTa-CRF,说明RoBERTa层学习到的词向量有更加准确的语义表示。
4.6.2 RoBERTa-CRF模型实验结果分析 本实验中RoBERTa-CRF模型在各标签上的F1值、精确率、召回率结果,见图3。
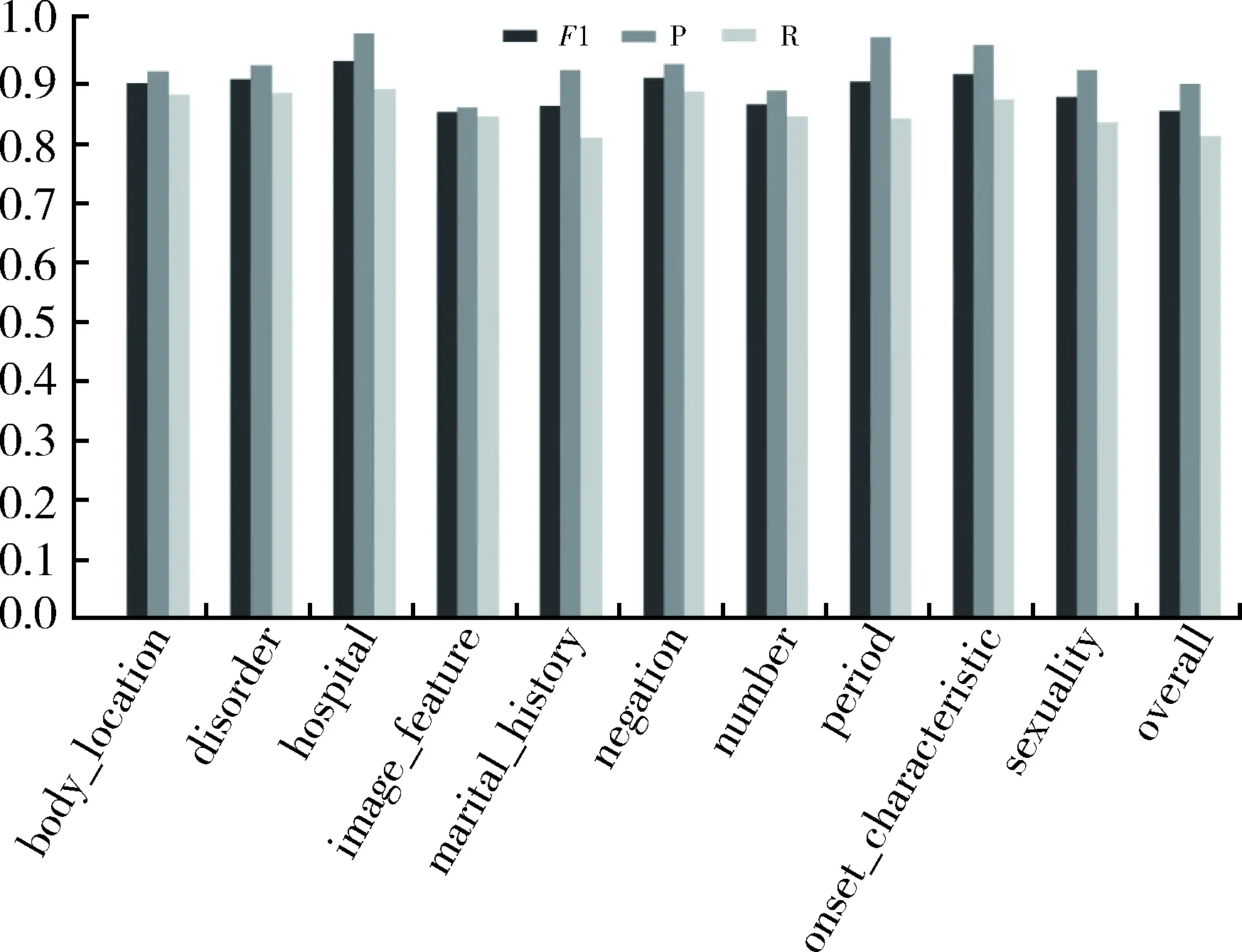
图3 RoBERTa-CRF模型识别结果
RoBERTa-CRF模型在全部实体上的精确率达到0.901 2,召回率达到0.812 6。精确率较高代表模型实体识别正确率高,部分未正确识别可能是由于模型对标签之间的边界没有正确界定,例如number标签原意是病变区域个数,但是其数字表达形式可能会使模型误识别为period;召回率相比之下较低,有部分实体被模型判断为非实体。通过学习更多高质量肝癌电子病历语料集可以提高模型性能。具体到各实体标签的识别结果,可以看到大部分实体类别有较高的精确率、召回率、F1值。RoBERTa-CRF模型在这类标签中达到较好学习效果。然而在某些标签中并没有达到理想效果。例如pathology_feature、condition_change等,模型由于该类标签定义比较模糊并且没有充足的样本训练,没有达到很好效果。针对该问题,可以扩大该类训练样本容量来达到更好效果。而在test、test_result标签上,在没有关系定义的前提下,模型很难将test和对应的test_result相联系。针对该问题,将关系抽取与实体识别相结合,会产生更加全面准确的判别能力。在drug标签上,模型没有在专业医药库中训练过,因此专业药物名称识别能力较差。针对该问题,可以将模型在专业医药语料中进行训练,以增强模型医药识别能力。
5 结语
预训练模型在通用语料库各项任务上都表现出较好性能,但是针对肝癌电子病历数据某些语句较短语义联系不强的特点,RoBERTa-CRF模型、BERT-CRF模型表现较好,BiLSTM-CRF表现相对于CRF模型F1值略差。其原因是肝癌电子病历数据中存在大量短句和缩略词,导致基于LSTM的深度学习模型未能学到合理的上下文表示,反而通过CRF定义的局部特征效果较好。相较于BERT-CRF模型,RoBERTa-CRF模型中,RoBERTa有更高鲁棒性、更优训练策略以及更大规模预训练数据,因此模型性能表现更好。
针对这一问题,可以从肝癌电子病历数据本身着手,规范记录形式或者提高数据标签质量。此外,本实验中使用的RoBERTa预训练模型是应用通用领域数据训练的预模型,如果能够使用大量医学文本数据对模型参数进行训练调整,训练出医学领域专用RoBERTa预训练模型,将会在该任务上有更好表现。而针对具体标签识别能力差的问题,应分析具体原因,采取相应措施,弥补模型短板。

