土壤微生物对长雄野生稻及其化感潜力后代抑草作用的影响
徐高峰 申时才 张付斗, 杨韶松 金桂梅 郑凤萍 温丽娜 张 云3, 吴冉迪
土壤微生物对长雄野生稻及其化感潜力后代抑草作用的影响
徐高峰1,2申时才1,2张付斗1,2,*杨韶松1,2金桂梅1,2郑凤萍1,2温丽娜1,2张 云3,*吴冉迪1,4
1云南省农业科学院农业环境资源研究所, 云南昆明 650205;2共建澜湄农业生物安全联合研究中心, 云南昆明 650205;3云南省农业科学院生物技术与种质资源研究所, 云南昆明 650205;4昆明学院农学与生命科学学院, 云南昆明 650205
明确土壤微生物对化感作物田间抑草作用的影响及其机制, 对农田杂草绿色防控具有重要意义。长雄野生稻()是培育化感水稻的优良抗原, 然而目前尚不清楚土壤微生物对该野生稻及其后代化感抑草作用的影响。本试验以非化感亚洲栽培稻(RD23)作为参照, 通过温室盆栽试验对比研究了土壤微生物对长雄野生稻及其中化感潜力后代(RL169)的抑草效应, 并分析了与其共培稗草的根际土壤微生物群落结构特征和对稗草养分吸收与利用的影响。结果表明: 1) 土壤微生物显著增强了长雄野生稻及其化感后代(RL169)的抑草效应(<0.05), 而对非化感亚洲栽培稻(RD23)则无明显影响; 方差分析表明, 土壤微生物、根系分泌物和不同化感潜力水稻互作(土壤微生物×根系分泌物×不同化感潜力水稻)对稗草的株高、根长和生物量具有显著影响(<0.05)。2) 长雄野生稻及其化感后代(RL169)改变了稗草根际土壤微生物的群落结构, 降低了稗草根际土壤细菌的多样性水平和丰富度, 与其共培稗草的根际细菌在科、属和种水平的数量显著低于非化感亚洲栽培稻(RD23)处理组(<0.05)。3) 长雄野生稻及其化感后代(RL169)显著降低了共培稗草对N、P和K营养元素的吸收与利用, 且在土壤微生物作用下, 长雄野生稻处理组稗草对N和P的吸收和中化感潜力水稻(RL169)处理组稗草对N的吸收显著降低; 方差分析也显示, 土壤微生物和不同化感潜力水稻极显著或显著影响稗草对N元素(<0.01)或K元素(<0.05)的吸收, 但对P元素的吸收却无显著影响; 另外, 稗草养分的利用仅受到不同化感潜力水稻的显著影响(<0.05)。综上所述, 长雄野生稻及其化感后代(RL169)改变了稗草根际土壤细菌的群落结构, 降低了其对营养元素N、P和K的吸收和利用, 其化感抑草效应在土壤微生物的作用下被显著提高。本研究结果对进一步认清土壤微生物对水稻化感作用的影响以及对野生种质资源的开发利用均具有重要意义。
水稻化感作用; 根际土壤; 土壤微生物多样性; 化感抑草效应; 养分的吸收与利用
稻田杂草严重影响水稻的产量和品质, 化学除草剂是目前世界控制稻田杂草的主要措施。然而, 化学除草剂长期大量施用导致了环境污染、杂草抗药性增强和残留药害等, 引起了社会各界和政府的广泛关注[1-2]。水稻化感作用(Allelopathy)是指化感水稻通过适当途径向环境(如根际土壤)释放化学物质, 从而直接或间接影响邻近或后续杂草种子萌发和生长的现象[3]。由于未向环境中引入难降解的人工合成化学物质, 利用水稻化感作用控制稻田杂草是国内外公认的杂草绿色治理技术之一[4-6]。
现有研究表明, 化感水稻常通过其根系分泌苯甲酸、香豆酸和环己烯酮等化感物质来控制杂草[7-8]。土壤微生物作为土壤的第二基因, 其在化感水稻的田间抑草作用过程中常扮演重要的角色[9]。研究发现土壤微生物可能通过分解、转化作物根系分泌的化感物质, 直接影响其田间抑草效应[10]。如孔垂华等研究表明, 化感水稻根系分泌的无活性麦黄酮、稻壳酮内酯, 其在土壤微生物下能转变为具有抑草活性的苷元, 从而对杂草种子的萌发和生长产生抑制作用[6,11]。另外, 土壤微生物也可能与根系分泌物互作, 改变受体根际微生物的群落结构, 从而影响受体的养分吸收, 间接影响其抑草效应[12-13]。因此, 掌握土壤微生物对水稻化感抑草效应的影响及其机制, 对稻田杂草的绿色生态治理具有重要意义。
长雄野生稻具有AA基因组, 是向亚洲栽培转移抗性基因的重要基因库[14-15]。前期研究发现, 长雄野生稻通过根系释放酚酸类物质, 具有较强的化感抑(抗)草特性, 其在2~3叶期对稗草的化感作用与国际公认的化感水稻PI312777差异不显著[16]。作者前期通过构建近等基因系, 已将长雄野生稻的化感抑草性状转移至亚洲栽培稻[17-18], 为我国化感水稻新材料的培育和应用奠定了基础。化感水稻的田间抑草效应可能受到土壤微生物的调控, 然而目前尚不清楚土壤微生物对长雄野生稻及其化感潜力后代抑草作用的影响。
本研究以稗草作为受体, 非化感亚洲栽培稻(RD23)作为参照, 通过温室盆栽试验和室内生理生化分析, 对比研究土壤微生物对长雄野生稻及其中化感潜力后代(RL169)的抑草效应、受体根际土壤微生物的群落结构以及其对养分吸收与利用的影响, 探讨土壤微生物对长雄野生稻及其后代化感作用的影响及其机制; 相关研究结果对进一步认清土壤微生物对水稻化感作用的影响以及对野生种质资源的开发利用均具有重要意义。
1 材料与方法
1.1 试验材料
供试水稻材料分别为强化感潜力的长雄野生稻(OL)及其中化感潜力后代(RL169)和非化感亚洲栽培稻(RD23), 相关试验材料由项目组前期筛选、培育获得, 且前期已对上述水稻的化感潜力和农艺性状进行了测定[18]。供试稗草()种子在2020年收集于云南省农业科学院嵩明试验基地水稻田, 常温晾干保存, 试验时种子发芽率大于95%。
1.2 试验方法
1.2.1 土壤微生物对不同化感潜力水稻抑草效应的影响 为测试土壤微生物对不同化感潜力水稻抑草效应的影响, 试验通过土壤是否灭菌和是否添加活性炭来调控土壤微生物和水稻根系分泌物。其中土壤灭菌表示无土壤微生物, 土壤未灭菌则表示有土壤微生物, 土壤添加活性炭处理表示无根系分泌物, 未添加活性炭处理则表示有根系分泌物。
2021年5月, 在温室控制条件下采用盆栽共培法, 对比研究了土壤灭菌未加活性炭、土壤灭菌加活性炭、土壤未灭菌未加活性炭、土壤未灭菌加活性炭4种条件下, 不同化感潜力水稻对受体的抑草效应。供试塑料盆的长×宽×深为40 cm×15 cm×15 cm,试验前所有塑料盆用84消毒液(消毒液∶水 = 1∶80)消毒处理24 h, 后用无菌水冲洗干净, 晾干后备用。供试土壤取自云南省农业科学院嵩明实验基地水稻田, 试验前把土壤晾干、敲碎。然后, 将供试土壤一半采用Υ射线进行灭菌处理, 另一半不做灭菌处理; 随后, 再分别将灭菌的土壤和未灭菌的土壤一半加入2% (按质量百分比)的活性炭, 另一半不加活性炭。最后将每种土壤搅拌均匀后, 每盆装15 kg处理的各种土壤。后用水将土壤润湿, 其中灭菌土壤处理组用无菌水润湿至饱和状态, 未灭菌土壤处理组则用自来水润湿至饱和状态。
选择2~3叶期大小一致、生长健壮的不同化感潜力水稻秧苗, 按照每行9株每盆移栽3行, 每盆共计种植27株, 行间距为5 cm。缓苗1周后, 在每行水稻之间分别播入经催芽露白的稗草种子各50粒(每盆100粒), 将其轻轻压入松软的稀泥表层后再盖1~3 mm细土。试验分别设置了未灭菌且未加活性炭的单播稗草空白对照和未灭菌但添加活性炭的单播稗草活性炭对照, 每处理重复8次(种8盆), 共计112盆。处理完成后, 将塑料盆置于20~35℃的温室条件下培养, 期间无菌土壤处理组浇无菌水, 其他土壤处理组浇自来水。稗草播后21 d, 将每盆稗草全部挖出, 用水清洗干净后, 随机抽取15株稗草, 测量稗草的株高和根长, 后将其装入纸袋于80℃烘至恒重后称其生物量。
1.2.2 不同化感潜力水稻对稗草根际土壤微生物群落的影响 温室条件下, 在每盆未灭菌土壤中移栽2~3叶期大小一致、生长健壮的不同化感潜力水稻秧苗27株, 每盆播入经催芽露白的稗草种子100粒(塑料盆规格以及水稻移栽和稗草播种的方法同1.2.1)。稗草播后21 d, 采用抖落法收集各处理稗草根际的土壤。土壤中微生物群落结构采用16S rDNA测序分析。采用EZNA Soil DNA Kit (OMEGA, 美国)试剂盒提取土壤微生物总DNA; 以各土壤样品微生物总DNA为模板, 采用细菌341F (5'-CCTACGGGN GGCWGCAG-3')和805R (5'-GACTACHVGGGTATC TAATCC-3')对16S rDNA基因V3和V4区域进行PCR扩增。采用Illumina Mi Seq测序平台对PCR扩增产物进行双端测序分析, 委托上海派森诺生物科技有限公司完成测序。
1.2.3 土壤微生物对不同化感潜力水稻对稗草的养分吸收与利用的影响 通过实验室化学分析对比研究了土壤微生物对不同化感潜力水稻对稗草N、P和K营养元素吸收与利用的影响。首先, 在温室条件下, 在每盆灭菌和未灭菌的土壤中播入经催芽露白的稗草种子100粒, 21 d后每盆随机拔出30株稗草, 后分别移栽2~3叶期大小一致、生长健壮的不同化感潜力水稻秧苗, 每盆移栽27株(塑料盆、土壤灭菌的方法和水稻移栽的方法同1.2.1), 同时将拔出的稗草秧苗晾干称取生物量后粉碎备用。秧苗移栽14 d后, 每盆再次随机拔出30株稗草, 晾干称取生物量后粉碎备用。分别取处理前、后的稗草植株干粉各0.3 g于消煮管中, 加入10 mL HNO3和5 mL H2SO4, 然后将其置于消化炉中消煮至溶液澄清。将消煮液转移至100 mL 容量瓶中, 用双蒸水定容。后参照刘春生等[19]《农业化学分析》中的方法分别测定稗草组织氮、磷、钾的含量。
1.3 数据分析

2 结果与分析
2.1 土壤微生物对不同化感潜力水稻化感作用的影响
2.1.1 对共生稗草生长的影响 在水稻/稗草共生系统, 长雄野生稻及其化感后代(RL169)显著降低了共生稗草的株高、根长和生物量, 其中未灭菌土壤条件下其抑草效应显著强于灭菌土壤; 显示土壤微生物显著提高了长雄野生稻及其化感后代(RL169)的化感抑草效应。研究也发现, 与亚洲栽培稻(RD23)共生的稗草, 其在不同土壤条件下, 稗草的株高、根长和生物量间无显著差异, 显示土壤微生物对非化感亚洲栽培稻的抑草效应无明显影响。另外, 当稗草单种时, 土壤是否添加活性炭对稗草的苗高、根长和生物量无显著影响, 表明土壤添加活性炭对稗草生长无显著影响(图1-A~C)。
2.1.2 土壤处理对稗草生长的多因素方差分析
根系分泌物、土壤微生物、不同化感潜力水稻及其互作对稗草的株高、根长和生物量的多因素方差分析显示, 根系分泌物、不同化感潜力水稻以及二者互作(根系分泌物×不同化感潜力水稻)对稗草的株高、根长和生物量均具有极显著影响(<0.01), 土壤微生物对稗草的株高、根长和生物量均无显著影响(>0.05)。但土壤微生物与根系分泌物和不同化感潜力水稻互作(土壤微生物×根系分泌物×不同化感潜力水稻)对稗草的株高、根长和生物量则具有显著影响(<0.05), 显示土壤微生物与水稻化感抑草效应密切相关(表1)。
2.1.3 对共生稗草的抑制效应 当土壤未添加活性炭时, 长雄野生稻(OL)在未灭菌土壤中对稗草的综合抑制率显著高于灭菌土壤, 而中化感潜力水稻 (RL169)和非化感亚洲栽培稻(RD23)二者则无显著差异; 表明土壤微生物显著提高了强化感水稻的化感抑草效应, 而对中化感水稻和非化感水稻则无显著影响。另外, 当土壤添加活性炭后, 土壤是否灭菌对不同化感潜力水稻对稗草的生长抑制作用均无显著影响, 表明在无根系分泌物作用时, 单一的土壤微生物不影响水稻化感作用(表2)。上述研究结果表明, 水稻的化感潜力越强, 土壤微生物对其化感抑草效应提高的越显著。
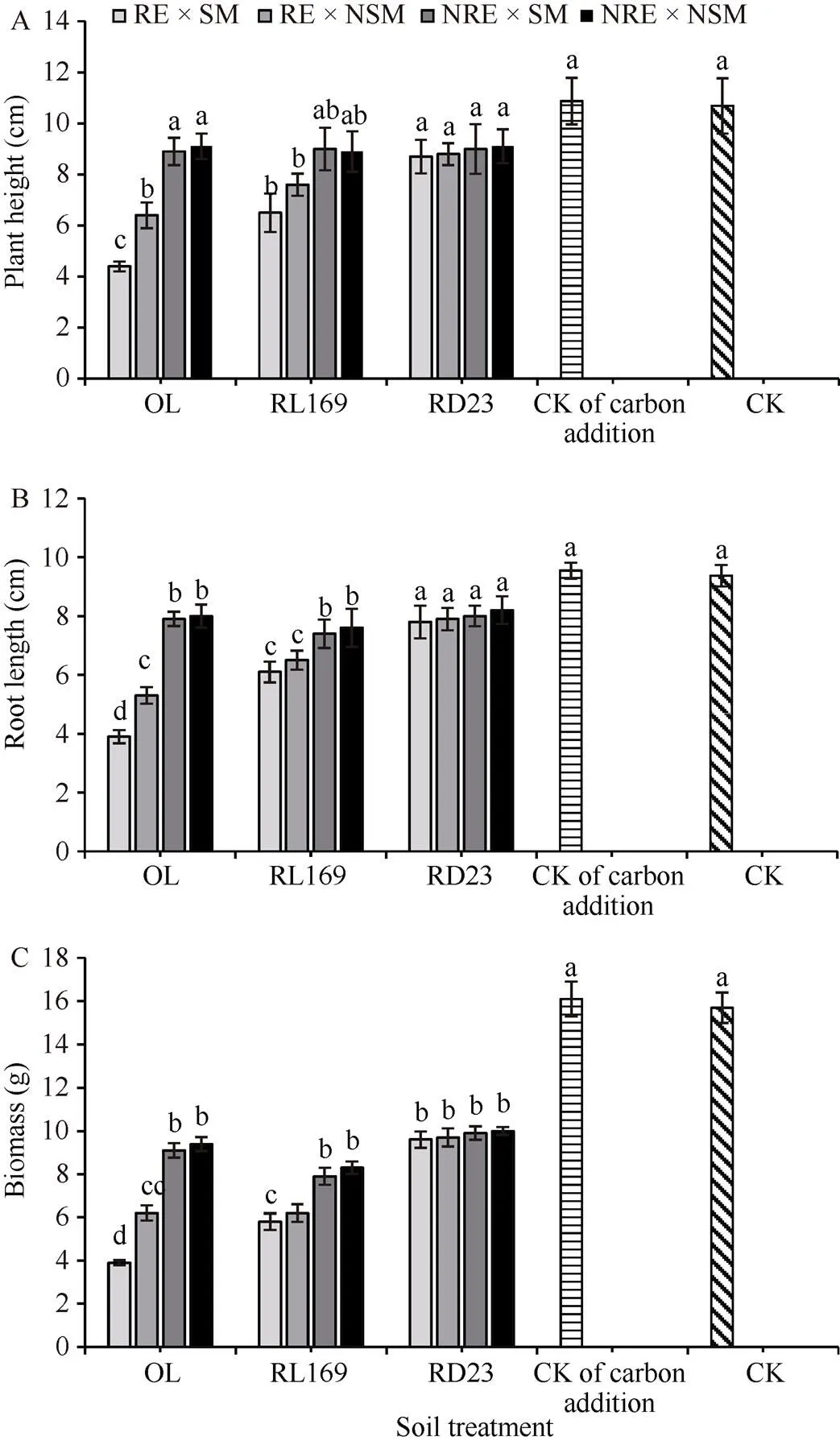
图1 不同化感潜力水稻在不同土壤处理条件下对稗草的株高、根长和生物量的影响
图A、B和C分别表示稗草在不同条件下的株高、根长和生物量; 图中小写字母表示在0.05概率水平显著性比较, 小写字母不同表示差异显著; 图中RE × SM表示根系分泌物 × 土壤微生物; RE × NSM表示仅有根系分泌物; NRE × SM表示仅有土壤微生物; NRE × NSM表示即无根系分泌物, 又无土壤微生物。
Fig. A, B, and C show plant height, root length, and biomass of barnyardgrass, respectively. Lowercase letters indicate significant difference at the 0.05 probability level. RE × SM: root exudates × soil microbes; RE × NSM: only root exudates; NRE × SM: only soil microbes; NRE × NSM: no root exudates and no soil microbes.
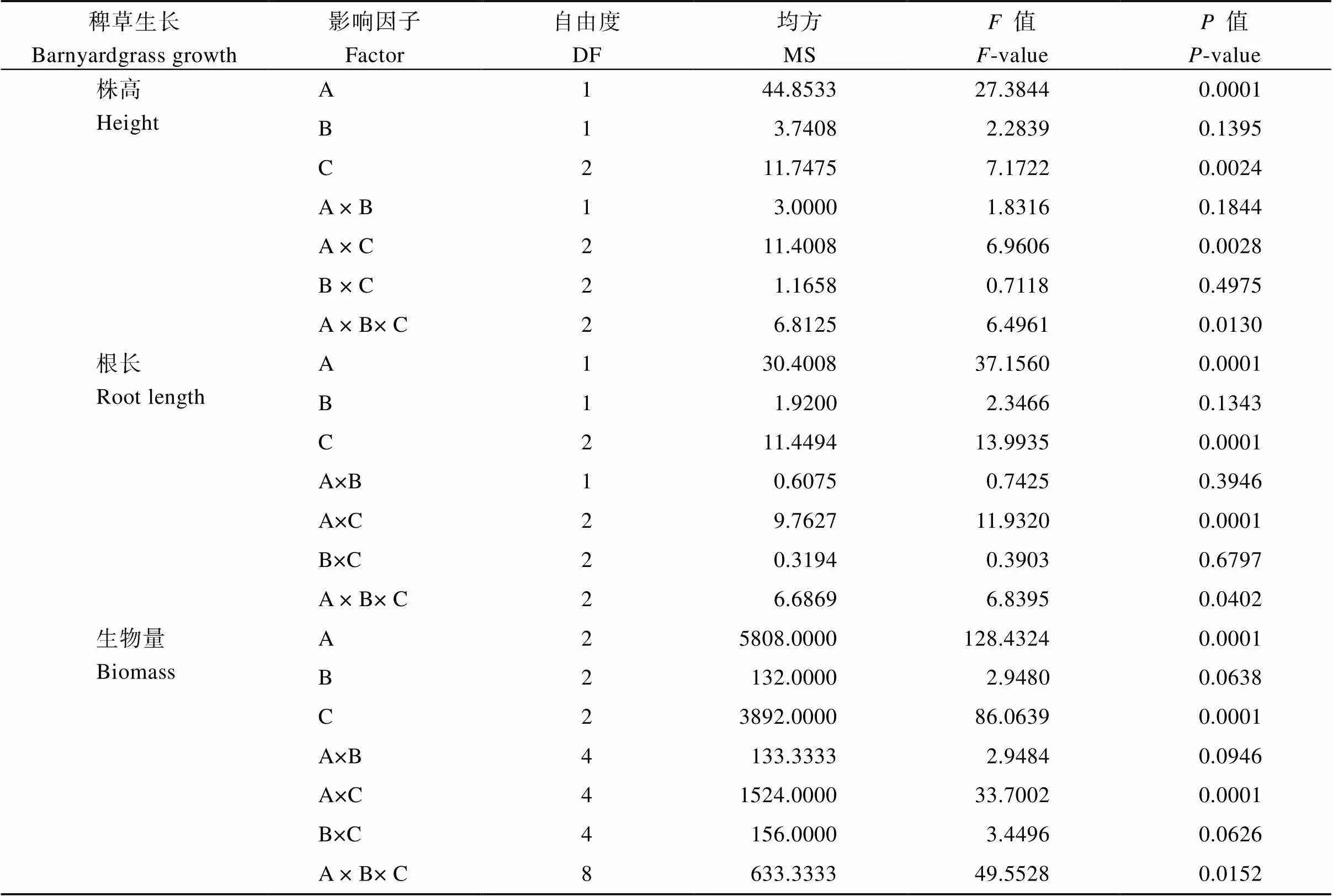
表1 不同因子对稗草生长的影响多因素方差分析
表中A、B和C分别表示根系分泌物、土壤微生物和不同化感潜力水稻。
Uppercase letters A, B, and C indicate root exudates, soil microbes, and different allelopathic potential rice genotypes, respectively.

表2 不同化感潜力水稻在不同土壤条件下的抑草能力
表中数据为平均值±标准误; 表中小写字母为同列数据在0.05概率水平差异显著性比较, 小写字母不同, 差异显著, 反之, 则差异不显著。RE × SM, RE × NSM, NRE × SM, NRE × NSM表示的意思与图1相同。
The value in the table is mean ± standard error. Lowercase letters indicate significant comparison of the data in the same column at< 0.05. Abbreviations of RE × SM, RE × NSM, NRE × SM, NRE × of NSM are the same as those given in Fig. 1.
2.2 稗草根际土壤微生物群落结构变化特征
长雄野生稻(OL)及其化感后代(RL169)改变了稗草根际土壤微生物的群落结构, 降低了稗草根际土壤细菌的种群数量, 与其共培的稗草, 其根际细菌在科、属和种水平的数量显著低于非化感亚洲栽培稻(RD23)处理组。另外, 稗草在3种水稻作用下, 其根际土壤细菌的多样性指数表现为长雄野生稻处理组的Shannon指数、Simpson指数和 Pielou_e指数均低于亚洲栽培稻(RD23), 化感后代(RL169)处理组的Chao 1值、Faith_pd值和Observed_species值最低, 且与非化感亚洲栽培稻(RD23)处理组差异显著。另外,研究也发现, 与长雄野生稻(OL)共培的稗草,其放线菌门(Actinobacteria)明显高于其他2种水稻, 但厚壁菌门(Firmicutes)和酸杆菌门(Acidobacteria)则明显低于另外2种水稻(图2-A, B和图3)。
2.3 土壤微生物对不同化感潜力水稻对稗草的养分吸收与利用的影响
研究结果表明, 尽管不同水稻材料混种均导致了共培稗草对N、P和K营养元素吸收与利用的降低, 但是, 强化感潜力水稻(OL)和中化感潜力水稻(RL169)导致了共培稗草对N、P和K营养元素吸收和利用的显著降低, 而非化感亚洲栽培稻(RD23)对稗草营养元素的吸收和利用无显著改变。研究也发现, 土壤微生物显著降低了强化感潜力水稻(OL)处理组稗草对N和P的吸收以及中化感潜力水稻(RL169)处理组稗草对N的吸收, 而对非化感亚洲栽培稻(RD23)处理组N、P和K营养元素吸收和利用无显著影响。另外, 研究也显示, 土壤微生物对不同化感潜力处理组共培稗草对营养元素N、P和K的利用率没有显著影响(表3)。
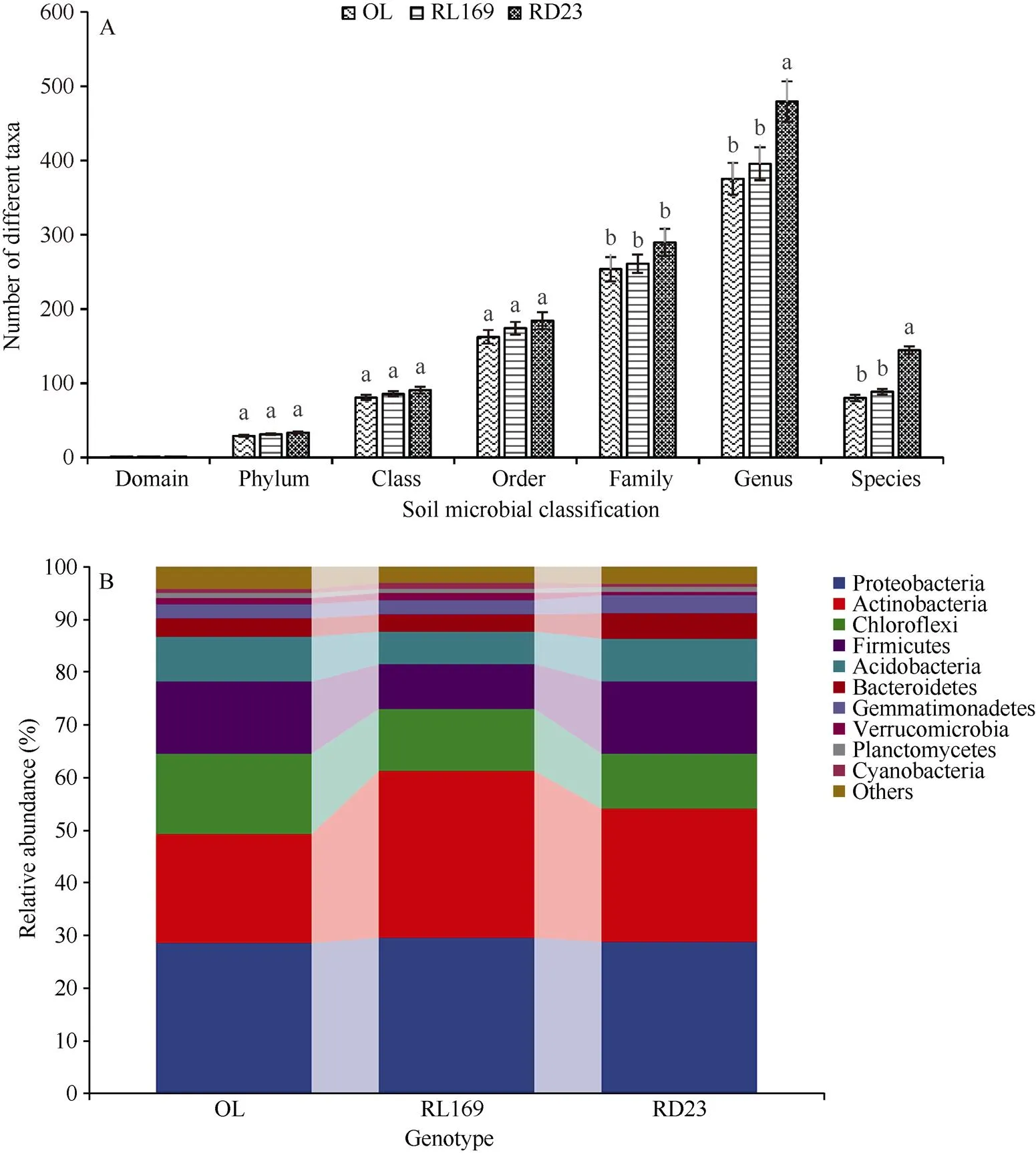
图2 不同化感潜力水稻对稗草根际土壤微生物群落结构的影响
图A和图B分别表示不同化感潜力水稻对稗草根际不同分类单元细菌的数量和土壤微生物群落结构的影响。
Fig. A and Fig. B show the number of bacteria in different taxa and their community structure character of barnyardgrass rhizosphere soil under different allelopathic rice genotypes.
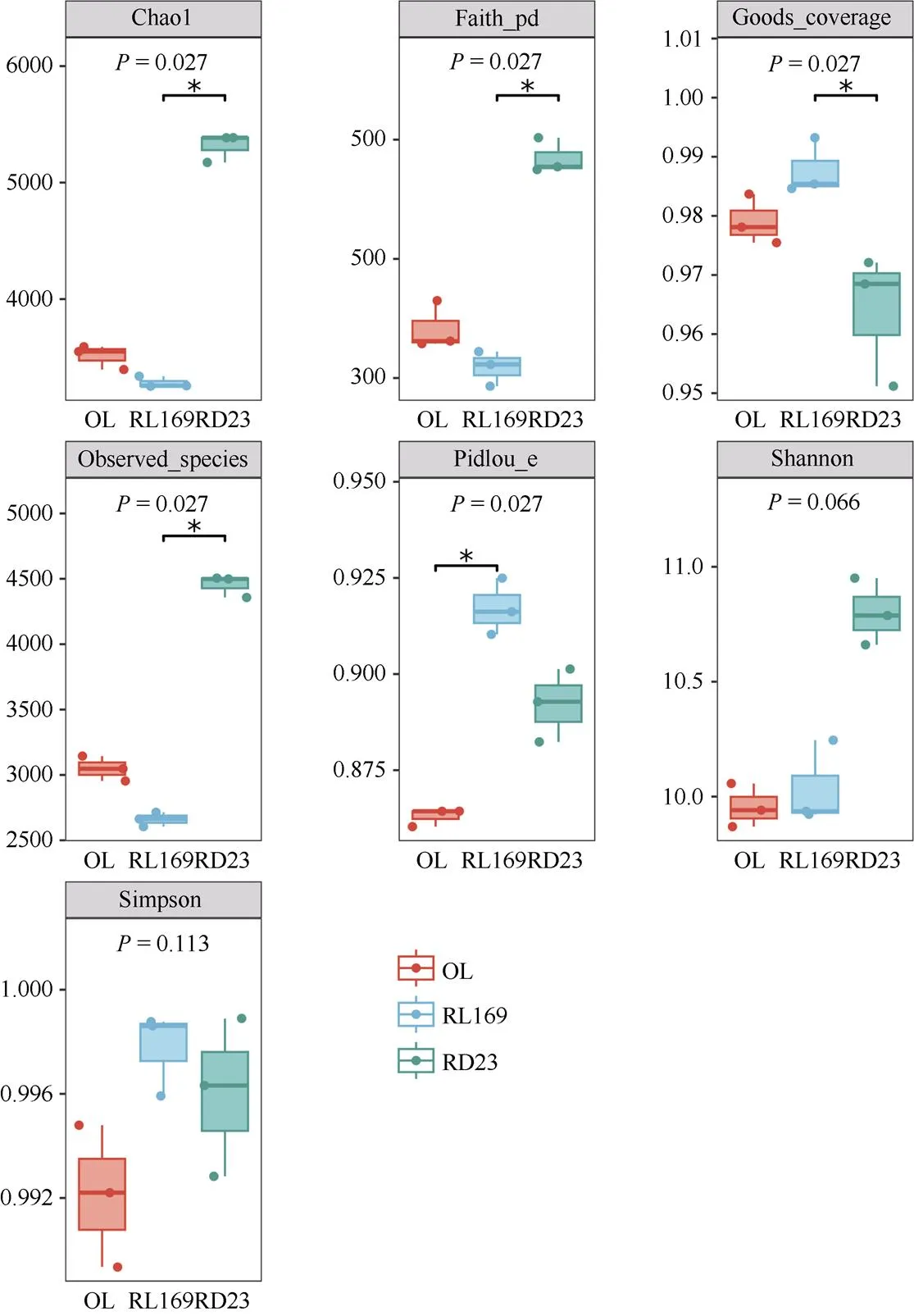
图3 不同化感潜力水稻对稗草根际土壤微生物生物多样性指数的影响
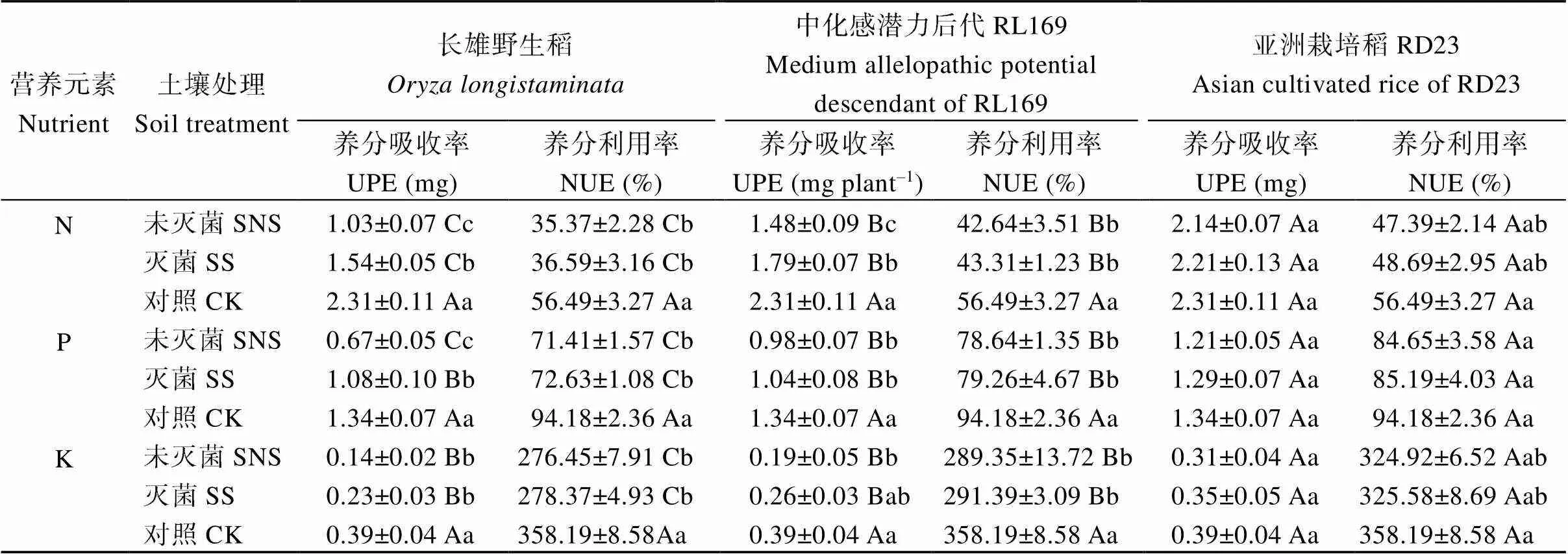
表3 不同化感潜力水稻在不同土壤条件下对稗草养分吸收和利用的影响
表中UPE和NUE分别表示稗草对营养元素的吸收率和利用效率; 大写字母表示不同化感潜力水稻在同等土壤条件下稗草的养分吸收或利用间的比较, 小写字母表示土壤是否灭菌对同一化感潜力水稻处理组稗草的养分吸收或利用间的比较; 字母不同表示在0.05概率水平差异显著。
UPE and NUE represent nutrient absorption efficiency and utilization efficiency of barnyardgrass, respectively. Uppercase letters indicate the comparison of nutrient absorption efficiency and utilization efficiency of barnyard grass of different rice genotypes under the same soil conditions and lowercase letters indicate the comparison of the same rice varieties under different soil conditions. Different letters indicate significant difference at< 0.05. SNS: soil not sterilized; SS: soil sterilized.
多因素方差分析显示, 土壤微生物、不同化感潜力以及二者互作(土壤微生物×不同化感潜力水稻)对共生稗草N元素的吸收具有显著影响, 除不同化感潜力水稻对K元素的吸收以及对N、P和K元素的利用具有显著影响外(<0.01), 其余因子对P和K元素的吸收以及对N、P和K元素的利用均无显著影响(表4)。
3 讨论
长期以来, 人们一直认为释放化感物质是化感植物抑草作用的本质[23]。然而, 在田间条件下, 研究者们发现无论是植物分泌较低浓度的萜类和黄酮类物质, 还是作用较高浓度的酚酸类物质, 其对受体的化感作用能力并不与土壤中实际化感物质浓度呈正相关[12,24]。因此, 这一结论长期以来也受到广泛的质疑。本研究结果表明, 土壤微生物显著提高了长雄野生稻及其化感后代的化感抑草效应, 但对非化感亚洲栽培稻的抑草效应则无明显影响。这一研究结果暗示了化感水稻的田间抑草效应可能是其根系分泌物与土壤微生物共同作用的结果, 同时也为解析植物化感作用机理和化感作物的筛选、培育和应用提供了理论基础。

表4 不同因子对稗草养分吸收和利用的影响多因素分析
表中A、B分别表示土壤微生物和不同化感潜力水稻; UPE和NUE分别表示养分吸收率和利用效率。
Uppercase letters A and B represent soil microbes and different allelopathic potential rice, respectively. UPE and NUE indicate nutrient absorption efficiency and utilization efficiency, respectively.
作物释放化感物质进入土壤, 可能引起土壤微生物群落结构的改变, 而植物根际土壤微生物的群落大小和结构特征与植物生长密切相关[25]。细菌占土壤微生物总量的70%~90%, 是土壤中含量最多的一类微生物, 因此, 探讨化感作物对土壤细菌群落结构的影响一直是国内外研究的热点。国内外诸多研究显示, 化感作物通常提高根际土壤细菌的多样性水平和丰富度[26-27]。如林瑞余等研究表明化感水稻PI312777显著增加了根际土壤细菌的多样性水平和丰富度[28]。本研究结果表明, 长雄野生稻(OL)及其化感后代(RL169)降低了受体稗草根际土壤细菌的Shannon指数、Simpson指数、Pielou_e指数和种群数量, 与其共培稗草的根际细菌在科、属和种水平的数量显著低于非化感亚洲栽培稻(RD23)处理组。本研究结果与国内外诸多研究存在明显不同, 暗示了以长雄野生稻为抗原的化感水稻材料, 其化感抑草作用机理可能与以化感水稻PI312777为抗原的化感水稻材料存在差异。
土壤微生物与土壤肥力密切相关, 化感作物释放化感物质进入土壤, 可能改变土壤微生物的群落结构, 进而影响受体对养分的吸收与利用[12]。如研究表明, 植物根系分泌的化感物质能够改变根际区域土壤微生物的群落结构和功能, 促进有利于其养分吸收的共生菌的生长, 同时改变土壤的物理和化学特性, 抑制与其竞争的植物对养分的吸收, 进而实现其化感抑草效应[12-13]。本研究显示长雄野生稻(OL)及其化感后代(RL169)显著降低稗草对营养元素N、P和K吸收与利用。这一结果表明, 长雄野生稻及其化感后代通过释放的化感物质, 降低稗草根际土壤细菌的多样性水平和丰富度, 导致稗草对养分吸收与利用的降低, 进而抑制稗草的生长, 这可能是长雄野生稻及其后代化感抑草的原因之一。同时, 本研究也暗示土壤微生物在长雄野生稻及其后代化感抑草作用过程中扮演了重要的角色。
土壤微生物如何影响化感作物的田间抑草表现一直是国内外研究的热点。本研究在温室盆栽条件下首先通过土壤灭菌和添加活性炭试验, 明确了土壤微生物显著增强了长雄野生稻及其化感后代的化感抑草效应。后通过室内生理生化分析研究了受体根际土壤细菌群落结构特征以及其对养分吸收与利用, 显示长雄野生稻及其化感后代降低了稗草根际土壤细菌的多样性水平和丰富度, 显著减弱了稗草对对营养元素N、P和K的吸收与利用。本研究结果首次阐明了以长雄野生稻为抗原的化感水稻抑草作用机理, 且研究结果与目前国内外常用抗原材料PI312777的相关研究报道明显不同。因此, 本研究对进一步认清水稻化感作用机理和野生种质资源的开发利用等均具有重要意义。
4 结论
长雄野生稻及其化感后代改变稗草根际土壤微生物的群落结构, 降低了稗草根际土壤细菌的群落大小, 并显著降低了与化感水稻共生的稗草对N、P和K养分的吸收与利用, 土壤微生物显著提高了长雄野生稻及其化感潜力后代的化感抑草效应。本研究为应用化感水稻实施稻田杂草生态控制提供了理论基础。
[1] Heap I. Global perspective of herbicide-resistant weeds., 2014, 70: 1306–1315.
[2] Chen G Q, Wang Q, Yao Z W, Zhu L F, Dong L Y. Penoxsulam-resistant barnyardgrass () in rice fields in China., 2016,16: 16–23.
[3] Kong C H, Hu F, Wang P, Wu J L. Effects of allelopathic rice varieties combined with cultural management options on paddy field weeds., 2008, 64: 276–282.
[4] 郭怡卿, 张付斗, 陶大云, 余柳青. 野生稻化感抗(耐)稗草种质资源的初步研究. 西南农业学报, 2014, 17(3): 295–298. Guo Y Q, Zhang F D, Tao D Y, Yu L Q. A preliminary study on the allelopathic activity of wild rice germplasm., 2014, 17(3): 295–298 (in Chinese with English abstract).
[5] Khanh T D, Xuan T D, Chung M I. Rice allelopathy and the possibility for weed management., 2007, 151: 325–339.
[6] Kong C H, Xu X H, Wang P, Zhao H, Gu Y. Activity and allelopathy of soil of flavone O-glycosides from rice., 2007, 55: 6007–6012.
[7] 林文雄. 水稻化感抑草作用的根际生物学特性与研究展望. 作物学报, 2013, 39: 951–960. Lin W X. Rhizobiological properties of allelopathic rice in suppression of weeds and its research prospect., 2013, 39: 951–960 (in Chinese with English abstract).
[8] Wu L K, Wang H B, Zhang Z X, Lin R, Zhang Z Y, Lin W X. Comparative metaproteomic analysis on consecutively-monocultured rhizosphere soil., 2011, 6: e20611.
[9] 李秋玲, 肖辉林. 土壤性质及生物化学因素与植物化感作用的相互影响. 生态环境学报, 2012, 21: 2013–2036. Li Q L, Xiao H L. The interactions of soil properties and biochemical factors with plant allelopathy., 2012, 21: 2031–2036 (in Chinese with English abstract).
[10] Zhu X, Zhang J, Ma K. Soil biota reduce allelopathic effects of the invasive., 2011, 6: e25393.
[11] 孔垂华, 徐效华, 梁文举, 周勇军, 胡飞. 水稻化感品种根分泌物中非酚酸类化感物质的鉴定与抑草活性. 生态学报, 2004, 24: 1317–1322. Kong C H, Xu X H, Liang W J, Zhou Y J, Hu F. Non-phenolic allelochemicals in root exudates of an allelopathic rice variety and their identification and weed-suppressive activity., 2004, 24: 1317–1322 (in Chinese with English abstract).
[12] Kong C H, Zhang S Z, Li Y H, Xia Z C, Yang X F, Meiners S J, Wang P. Plant neighbor detection and allelochemical response are driven by root-secreted signaling chemicals., 2018, 9: 3867.
[13] 蒋婧, 宋明华. 植物与土壤微生物在调控生态系统养分循环中的作用. 植物生态学报, 2010, 34: 979–988. Jiang Q, Song M H. Review of the roles of plants and soil microorganisms in regulating ecosystem nutrient cycling., 2010, 34: 979–988 (in Chinese with English abstract).
[14] Guo Y Q, Zhang F D, Tao D Y, Yu L Q, David G. Preliminary studies on the allelopathic potential of wild rice () germplasm., 2005, 15: 13–19.
[15] 张付斗, 郭怡卿, 余柳青, 陶大云. 野生稻和非洲栽培稻抗稗草种质资源筛选和评价. 作物学报, 2014, 30: 1144–1148. Zhang F D, Guo Y Q, Yu L Q, Tao D Y. Evaluation and screening of resistance to barnyardgrass in germplasm of wild rice () and African cultivar., 2014, 30: 1144–1148 (in Chinese with English abstract).
[16] 张付斗, 郭怡卿, 余柳青. 水稻对稗草化感作用的生物测定. 华中农业大学学报, 2004, 23(2): 203–207. Zhang F D, Guo Y Q, Yu L Q. The study on bioassay for rice allelopathy to barnyardgrass., 2004, 23(2): 203–207.
[17] 徐高峰, 申时才, 张付斗, 张玉华. 长雄野生稻及其后代抑草效果与化感潜力和农艺性状的关系. 中国生态农业学报, 2014, 22: 1348–1356. Xu G F, Shen S C, Zhang F D, Zhang Y H. Relationships among weed suppression effect, allelopathy and agronomic characteristics ofand related descendants., 2014, 22: 1348–1356 (in Chinese with English abstract).
[18] Xu G F, Shen S C, Zhang F D, Zhang Y, Kato-Noguchi H, David R C. Relationship between allelopathic effects and functional traits of different allelopathic potential rice accessions at different growth stages., 2018, 25: 32–41.
[19] 刘春生, 杨守祥. 农业化学分析. 北京: 中国农业出版社, 1996, pp 8–60. Liu C S, Yang S X. The Methods of Agrochemical Analysis. Beijing: China Agriculture Press1996. pp 8–60 (in Chinese).
[20] Rice E L. Allelopathy. Orlando, FL, USA: Academic Press, 1984.
[21] Williamson G B. Richardson D. Bioassays for allelopathy: measuring treatment responses within independent controls., 1989, 14: 181–187.
[22] 徐高峰, 申时才, 张付斗, 张玉华. 温度对不同叶龄长雄野生稻及其后代化感作用的影响. 中国水稻科学, 2016, 30: 559–566. Xu G F, Shen S C, Zhang F D, Zhang Y H. Allelopathic response to different temperature conditions of wild rice () and its descendants., 2016, 30: 559–566 (in Chinese with English abstract).
[23] Chandramohan D, Purushothaman D, Kothandaraman R. Soil phenolics and plant growth inhibition., 1973, 39: 303–308.
[24] Putnam A R, Duke W B. Biological suppression of weeds: evidence for allelopathy in accessions of cucumber., 1974, 185: 370–372.
[25] 俞慎, 何振立, 黄昌勇. 重金属胁迫下土壤微生物和微生物过程研究进展. 应用生态学报, 2003, 14: 618–622. Yu S, He Z L, Huang C Y. Advances in the research of soil microorganisms and their mediated processes under heavy metal stress., 2003, 14: 618–622 (in Chinese with English abstract).
[26] 陈良生. 低磷对化感水稻根际微生物多样性的影响. 福建农林大学硕士学位论文, 福建福州, 2011. Chen L S. Impact of Lower Phosphorus Supplies on Rhizospheric Microbial Diversity of Different Allelopathic Rice. MS Thesis of Fujian A&F University, Fuzhou, Fujian, China, 2011 (in Chinese with English abstract).
[27] 熊君, 林辉锋, 李振方, 方长旬, 韩庆典, 林文雄. 旱直播条件下强弱化感潜力水稻根际微生物的群落结构. 生态学报, 2012, 32: 6100–6109. Xiong J, Lin H F, Li Z F, Fang C X, Han Q D, Lin W X. Analysis of rhizosphere microbial community structure of weak and strong allelopathic rice varieties under dry paddy field., 2012, 32: 6100–6109 (in Chinese with English abstract).
Effects ofsoil microbes on rice allelopathy and its mechanism of wild rice () and its descendants
XU Gao-Feng1,2, SHEN Shi-Cai1,2, ZHANG Fu-Dou1,2,*, YANG Shao-Song1,2, JIN Gui-Mei1,2, ZHENG Feng-Ping1,2, WEN Li-Na1,2, ZHANG Yun3,*, and WU Ran-Di1,4
1Institute of Agricultural Environment and Resources Research, Yunnan Academy of Agricultural Sciences, Kunming 650205, Yunnan, China;2Yunnan Lancang-Mekong Agricultural Bio-Security International Science and Technology Cooperation Joint Research Center, Kunming 650205, Yunnan, China;3Biotechnology and Germplasm Resources Institute, Yunnan Academy of Agricultural Sciences, Kunming 650205, Yunnan, China;4College of Agronomy and Life Sciences, Kunming University, Kunming 650205, Yunnan, China
Soil microbes may affect weed inhibitory effects of allelopathic crops that it is great significant to understand their causes and mechanisms for green ecological management of weeds in paddy fields.(OL), a wild rice with strong allelopathic potential, is excellent cultivars for breeding allelopathic rice. However, the effect of soil microbes on allelopathy of this wild rice and its descendants is still unclear. In this study, the allelopathic effects of two allelopathic rice genotypes (OL and its descendant-RL169) and non-allelopathic Asian cultivated rice cultivar (RD23) affected by soil microbes on barnyardgrass were studied, and characteristics of rhizosphere soil microbes, and absorption and utilization of nutrients of barnyardgrass were analyzed. The results showed that: 1) Soil microbes significantly increased weed suppression of wild rice (OL) and its descendants (RL169) (< 0.05) and had no significant effect on RD23. Multivariate analysis of variance showed that plant height, root length and biomass of barnyardgrass were significantly increased with interaction of soil microbes, root exudates and different allelopathic rice genotypes (< 0.05). 2) Wild rice (OL) and its descendants (RL169) changed soil microbe community structure, reduced diversity and richness of bacteria in barnyardgrass rhizosphere soils, which the number of bacteria was significantly lower than that of RD23 at the family, genus and species levels (< 0.05). 3) Soil microbes significantly reduced absorption and utilization of N, P and K of barnyardgrass which co-cultured with wild rice and its descendants (RL169). In the presence of soil microbes, the absorption of N and P of barnyardgrass which co-cultured with wild rice(OL) and the absorption of N of barnyardgrass which co-cultured with rice genotypes (RL169) were significantly reduced. Multivariate analysis of variance revealed that soil microbes and allelopathic rice genotypes significantly affected N (< 0.01) or K (< 0.05) absorption of barnyardgrass and had no significant effect on P absorption; but nutrient utilization of barnyardgrass was only obviously affected by allelopathic rice genotypes (< 0.05). In conclusion, the allelopathic suppression of wild rice (OL) and its descendants (RL169) was significantly improved through soil microbe community structure changing of barnyardgrass rhizosphere soils and nutrient absorption and utilization (N, P, and K) reducing of barnyardgrass. Our study could increase further understanding of effect of soil microbes on rice allelopathy and provide a theoretical basis for the development and utilization of wild rice germplasm resources.
rice allelopathy; rhizospheric soil; soil microbial diversity; allelopathic weed suppression; nutrient absorption and utilization
2022-08-11;
2023-02-21;
2023-03-16.
10.3724/SP.J.1006.2023.22047
通信作者(Corresponding authors):张付斗, E-mail: fdzh@vip.sina.com; 张云, E-mail: zhangyun507@163.com
E-mail: xugaofeng1059@163.com
本研究由国家自然科学基金项目(31960544, 31860511), 云南省技术创新人才项目(202105AD160021), 国家重点研发计划项目(2021YFC2600400)和云南省重大专项项目(202102AE090003)资助。
This study was supported by the National Natural Science Foundation of China (31960544, 31860511), the Technological Innovation Talent Plan of Yunnan Province (202105AD160021), the National Key Research and Development Program of China (2021YFC2600400), and the Special Funds of Major Science and Technology Project in Yunnan Province (202102AE090003).
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20230314.1604.004.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

