生防菌株贝莱斯芽孢杆菌HNU24全基因组测序及分析
曹秀兰 叶雨婷 马天浩 胡安娜 曹宇 陈鹏泽 伍壮生 李鹏

关键词:贝莱斯芽孢杆菌;番茄青枯病;全基因组;次级代谢产物
中图分类号:S435.73 文献标识码:A
生物防治由于靶向明确且安全无污染,近年来已经成为发展可持续农业领域的研究热点。芽孢杆菌属的菌株是主要的生防细菌筛选目标之一,其中绝大部分是有益微生物,具有强大的生防潜力,同时根围或根际促生细菌也常来源于此属[1-2]。属内枯草芽孢杆菌(Bacillus subtilis)、苏云金芽孢杆菌(B. thuringiensis)、蜡样芽胞杆菌(B. cereus)、解淀粉芽孢杆菌(B. amyloliquefaciens)等已经被制成各类微生态制剂,广泛应用于农业、养殖、水产等领域,且成效显著[3-4]。
贝莱斯芽孢杆菌(B. velezensis)于1999 年被发现,2005 年被首次报道和命名,但不同来源菌株的形态和理化性质不尽相同[5]。已有研究结果表明贝莱斯芽孢杆菌分布广泛、生长迅速、能够产生大量次生代谢物质如抗菌蛋白、脂肽类抗生素等特性,是优良的生防菌株资源[6]。如贝莱斯芽孢杆菌对禾谷镰刀菌(Fusarium graminearum)、禾谷丝核菌(Rhizoctonia cerealis)、葡萄座腔菌( Botryosphaeria dothidea )、大丽轮枝菌(Verticillium dahliae)等多种植物病原菌有明显的拮抗活性[7-10],同时杨成等[11]的研究表明,贝莱斯芽孢杆菌Y6 可通过侵入钉螺体内干扰糖原利用而达到杀灭的目的。其强大的抑菌谱得益于多种次生代谢产物介导下的多种抗菌机制,例如竞争作用、溶菌作用、拮抗作用等[6]。王淋敏[12]、张荣胜等[13]的研究表明,使用贝莱斯芽孢杆菌制成的菌剂能够刺激植物生长,表明其也具有一定的促生长和诱导抗性的功能。FZB42 是贝莱斯芽孢杆菌的模式菌株之一,其全基因组测序与功能预测结果表明,约有10%的基因资源用于产生各类次级代谢产物,途径广泛、类型多样,且这些次级代谢产物均具备不同程度的抑菌和促进植物生长的能力,实验也证明该菌株对数十种植物病原细菌和病原真菌具有强大的抑制能力[14]。
菌株HNU24 是以強致病力茄雷尔氏菌(Ralstonia solanacearum)菌株EP1[15]为指示菌,从番茄砧木根系分离得到的一株生防菌,前期实验表明该菌株对EP1 具有较强的拮抗活性。为进一步解析HNU24 的功能机制和基因组特征,本研究采用BGISEQ-500 和ONT PromethION 平台相结合的测序技术,组装得到HNU24 的完整基因组序列,并进行基因预测、同源蛋白簇(clusterof orthologous groups of proteins, COG)、基因本体论(gene ontology, GO)、京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG )、碳水化合物活性酶数据库(carbohydrate-active enzymes, CAZy)、次级代谢产物分析,经基因组聚类分析表明HNU24 为贝莱斯芽孢杆菌,且温室实验表明其对番茄具有显著的促生长活性。相关结果对于进一步开展HNU24 在生物防治方面的研究和应用奠定基础。
1 材料与方法
1.1 材料
菌株HNU24 保藏于广东省微生物菌种保藏中心,菌株保藏编号为GDMCC61231。取HNU24单菌落接种于50 mL 的LB 液体培养基中,37 ℃,150 r/min 培养至OD600=1.0,然后在4 ℃,12 000 r/min离心10 min 收集菌体,用于基因DNA 提取,具体提取方法参照黄鹤等[16]的方法。
1.2 方法
1.2.1 基因组测序、组装和序列分析 基因组测序。基于BGISEQ-500 和ONT PromethION 测序技术平台分别对HNU24 菌株进行高通量全基因组测序,委托武汉希望生物科技有限公司进行。
基因组组装:使用Unicycler 0.4.8 软件对HUN24 菌株基因组测序产生的二代短读长和三代长读长测序数据进行混合组装,得到一条完整的环状基因组序列。
基因注释:使用prokka v1.14.5 软件对HNU24基因组中的蛋白质编码基因和tRNA、rRNA 等非编码基因进行注释。对蛋白编码基因,进一步使用eggNog-mapper v2.1.5 软件进行GO、KEGG、COG 和CAZy 等功能注释。
1.2.2 分类地位和遗传进化分析 使用GTDB-tkv1.6.0 软件分析HNU24 的基因组序列,以确定其分类学地位。该软件利用细菌基因组中广泛存在的120 个保守基因,对HNU24 菌株进行系统发育分析,寻找Genome Taxonomy Database 数据库(RS202)中与其关系最为接近的细菌物种。进一步分别使用pyani v0.2.11 软件和在线工具GGDC3.0(http://ggdc.dsmz.de/ggdc.php)计算HNU24 和其近缘细菌基因组间的平均核苷酸一致性(averagenucleotide identity, ANI)和数字DNA-DNA 杂交值,以进一步判断HNU24 的分类学地位。
1.2.3 CAZy 酶类数据库分析 碳水化合物活性酶(CAZy)按其功能分类主要包括糖苷水解酶( glycoside hydrolases, GHs )、糖基转移酶( glycosyl transferases, GTs )、多糖裂解酶(polysaccharide lyases, PLs)、碳水化合物酯酶(carbohydrate esterase, CEs)、辅助氧化还原酶(auxiliary activities, AAs)以及没有催化活性的碳水化合物结合模块(carbohydrate-binding modules,CBMs)。基于碳水化合物活性酶数据库,利用 hmmer[17]软件进行碳水化合物酶类基因的功能注释与分析。
1.2.4 次级代谢产物合成基因簇分析 使用antiSMASH(version 5.1.2)软件在线分析预测菌株HNU24 基因组中的拮抗物质合成相关基因簇。
1.2.5 纤维素降解酶活性检测 分别将HNU24和FZB42 在5 mL 的LB 液体培养基中28 ℃,180 r/min 条件下振荡培养24 h,制备纤维素培养基平板(羧甲基乙基纤维素1.0 g,磷酸钠3.8 g,琼脂糖8 g,溶解于1 L 蒸馏水中,其中羧甲基乙基纤维素需要在热水浴条件下溶解,121 ℃灭菌20 min),每皿25 mL,待培养基冷却凝固后打孔,每孔加入40 μL 的HNU24 或FZB42 菌液,培养24 h,加入适量1 g/L 的刚果红染液,以染液没过培养基为准,室温静置10 min,倒掉染液,往培养基里加入1 mol/L NaCl 溶液脱色,室温静置10 min,倒掉NaCl 溶液,重复脱色2 次后,白色背景下对培养基中形成的透明圈进行拍照记录和直径测量。
1.2.6 温室促生长实验 将1 mL(OD600=1.0)的HNU24 菌悬液在100 mL 的LB 液体培养基中振荡培养48 h(28 ℃, 180 r/min),6000 r/min 离心5 min,去除培养基后用无菌水100 mL 重悬制成悬液,制备相应菌体悬液500 mL。选出健康、长势良好且一致的番茄幼苗,分为叶面喷施清水组(A 组)、叶面喷施处理组(B 组,含HNU24)、灌根清水组(C 组)、灌根处理组(D 组,含HNU24)4 个组,每组3 个重复,每个重复10 株幼苗;叶面喷施清水组用清水进行叶面喷施处理,至叶面湿润不滴水为止;灌根清水组用清水进行灌根处理,每盆每次灌根100 mL 无菌水;叶面喷施处理组用去培养基的HNU24 无菌水重悬液进行叶面喷施,至叶面湿润不滴水为止;灌根处理组用去培养基的HNU24 无菌水重悬液进行灌根处理,每盆灌根100 mL 菌悬液。将上述处理的番茄幼苗在室温30 ℃的温室内培养,为保证植株不受水分胁迫,所有苗每隔3 d 用100 mL 的清水灌根,30 d 后记录植株株高。
2 结果与分析
2.1 测序与组装
通过对测序结果进行组装,获得了HNU24的完整基因组序列,长度为3 932 768 bp,GC 含量为46.48%,共编码3822 个基因,鉴定出99 个tRNA 基因,33 个rRNA 基因,1 个tmRNA 基因,2 个ncRNA 基因,58 个假基因,1 个SPR-RNA基因,3747 个蛋白质编码区,26 个生物核糖开关以及12 个转座酶基因(sequence read archive 授权号:SRR16216500)(图1)。
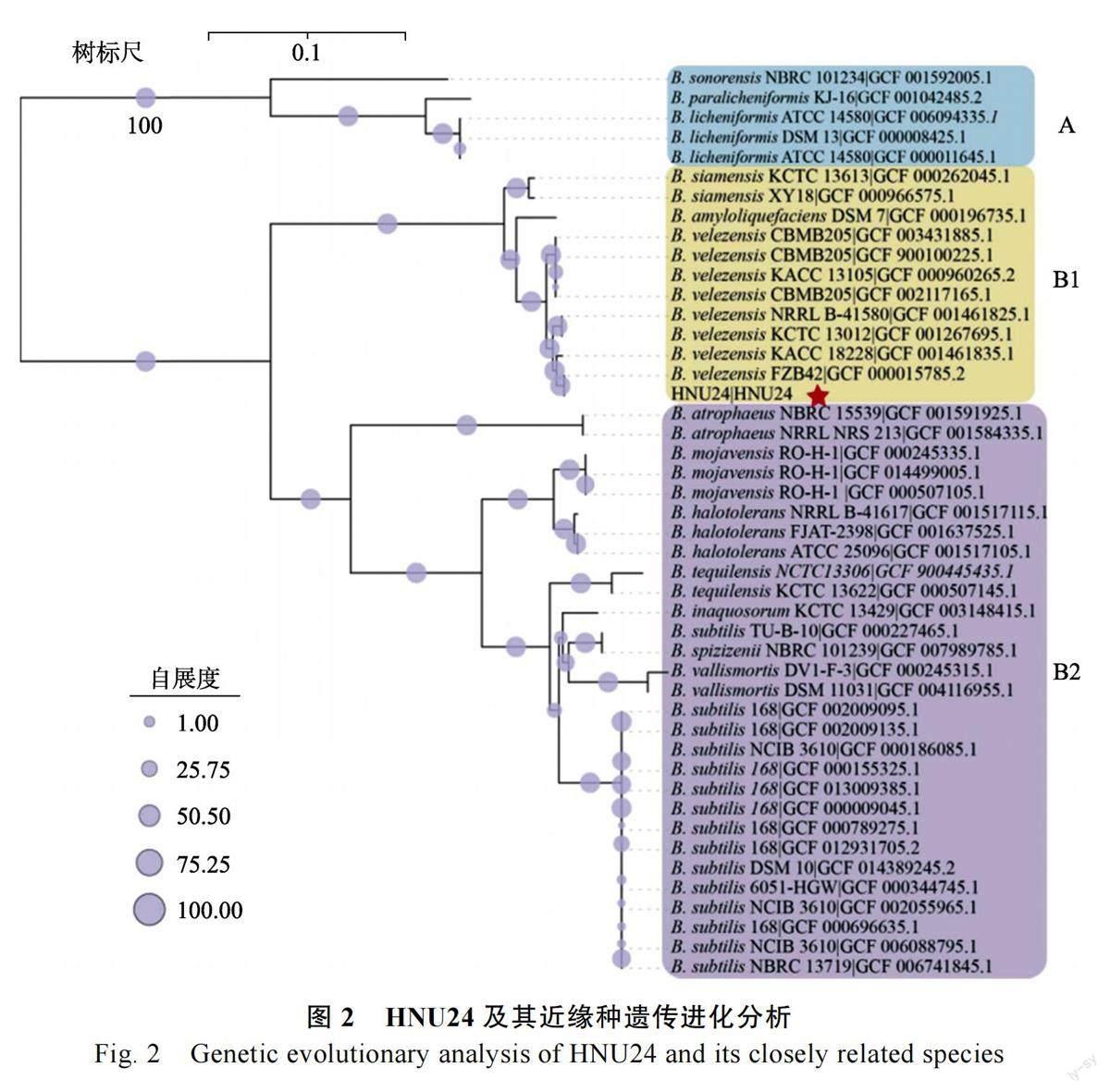
2.2 分类地位与遗传进化分析
为了进一步确定菌株HNU24 的分类地位,从数据库中下载了45 个芽孢杆菌的全基因组,通过ANI 和聚类分析,所有的菌株同HNU24 被分为A和B(B1 和B2)两个大支(图2),B. sonorensisNBRC 101234、B. paralicheniformis KJ-16、B.licheniformis ATCC 14580、B. licheniformis DSM13 等聚类为A 支,与HNU24 的ANI 值低于80%。HNU24 与B. velezensis FZB42、B. velezensis KACC18228、B. velezensis KTCT 13012 等聚类为B1 支,其ANI 值介于97%~100%之间,高于种的分类阈值95%,B. atropharus NBRC15539、B. mojavensisRO-H-1、B. halotolerans ATCC25096 等29 株菌聚类为B2 支,与HNU24 的ANI 值在93%~95%之间,低于分类阈值。综上,菌株HNU24 可归属为贝莱斯芽孢杆菌(B. velezensis),且与FZB42具有最高的基因组相似性。
2.3 基因功能注释及蛋白同源性分析
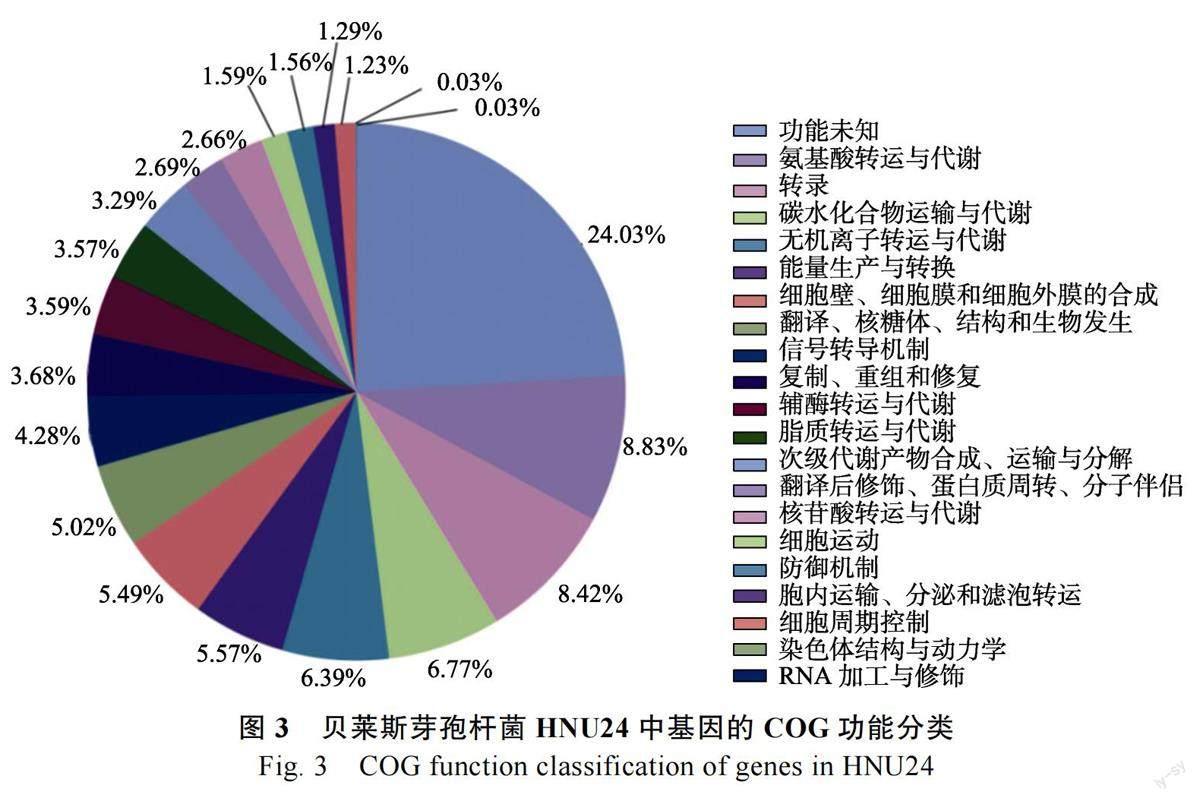
本研究对HNU24 进行了基因功能注释、COG功能分类分析。COG 功能分类统计结果表明(图3),共有3646 个蛋白具有明确的COG 分类,约占总蛋白数的95.37%。在22 个COG 类别当中,其中最多的分类是功能未知的类群(876 个),占COG 分类蛋白总数的24.03%,分类为氨基酸运转与代谢的蛋白数量次之,占8.83%,然后依次是转录相关蛋白(8.42%),碳水化合物运输和代谢相关蛋白(6.77%),无机离子转运和代谢相关蛋白(6.39%),能量生产和转换(5.57%),细胞壁/细胞膜/细胞外膜的合成(5.49%),翻译、核糖体、结构和生物发生(5.02%),信号转导机制(4.28%),复制、重组和修复(3.68%),辅酶转运与代谢(3.59%),脂质转运与代谢(3.57%),次级代谢产物合成、运输和分解(3.29%),翻译后修饰、蛋白质周转、分子伴侣(2.69%),核苷酸转运与代谢(2.66%),细胞运动(1.59%),防御机制(1.56%),胞内运输、分泌和滤泡转运(1.29%),细胞周期控制、控制细胞分裂、染色体分离(1.23%),染色体结构及动力学(0.03%),DNA 加工与修饰(0.03%)。
2.4 CAZy 酶类数据库对比分析
CAZy 酶类数据库对比结果表明菌株HNU24编码的碳水化合物活性酶基因家族43 个(表1),分别为GTs(22 个)、GHs(13 个)、CBMs(2个)、CEs(1 个)基因家族,另外还发现有复合酶类,复合类型为GH13/GH31(2 个)、AA10/CBM73(1 个)、CBM6/GH43(1 个)、GH5/GH9(1 个)。其中GT 家族最多,约占整个基因家族的50%,GH 家族次之,约占29.5%,复合酶类占整个基因家族的13.6%。其中GH9 家族对纤维素和木聚糖的降解能够发挥很大的作用,GH43、GH26、GH5、GH51 等家族也与纤维素和半纤维素的水解相关,说明HNU24 具有降解纤维素、半纤维素、木聚糖的潜力。为了进一步确定碳水化合物水解酶是否表达,利用纤维素培养基结合刚果红染色法,将其与贝莱斯芽孢杆菌模式菌株FZB42 进行纤维素降解酶活性比较,结果显示HNU24 透明圈直径和FZB42 相当,均值为18 mm(图4),说明其具备与FZB42 相当的纤维素酶降解活性。CBM50 家族的酶类够裂解几丁质和纤维素,通过连接部分糖苷水解酶,以分解几丁质和肽聚糖。推测菌株HNU24 也具备分解幾丁质和肽聚糖的潜力。
2.5 次级代谢产物预测
通过使用antiSMASH 软件预测,在HNU24基因组中预测到13 个次级代谢产物生物合成基因簇,其中8 个能够找到完全相似或者相似度极高的已鉴定基因簇( 表2 ), 分别是表面活性素(surfactin)、植物唑霉素(plantazolicin)、大环内酰亚胺(macrolactin)、杆菌烯(bacillaene)、泛革素(fengycin)、地非西丁(difficidin)、杆菌毒素(bacillibactin)、杆溶菌素(bacilysin),其相似度除表面活性素为91%外,其余均为100%,另有1种聚酮合酶类似物与丁胺菌素A/丁胺菌素B(butirosinA/butirosinB)相似度仅为7%、2 种烯萜类化合物、1 种三型酮聚合酶化合物以及1 种非核糖体多肽类化合物的基因簇尚未在数据库中比对出相似化合物基因簇,极有可能是未知的化合物。
2.6 生防菌HNU24 温室促生长实验
为了检测HNU24 的促生长活性,本研究以番茄(东绿明星番茄)为研究对象开展了温室促生长实验(图5),结果显示通过在叶面喷施处理和灌根处理HNU24 菌悬液,与对照相比,均对番茄株高有显著促进作用,且灌根的促进作用更加明显。采用灌根方式和叶面喷施方式施加HNU24时,HNU24 促进番茄株高增长率分别为26.2%和18.6%。
3 讨论
通过采用BGISEQ-500 和ONT PromethION平台相结合的测序技术得到了一株对茄雷尔氏菌具有拮抗活性的菌株HNU24 的全基因组序列。通过与枯草芽孢杆菌、解淀粉酶芽孢杆菌和贝莱斯芽孢杆菌等主要芽孢菌属的菌株基因组的ANI值和聚类分析,结果显示HNU24 归属为贝莱斯芽孢杆菌,并与菌株FZB42 的亲缘关系最近。
CAZy 数据库预测结果表明,HNU24 可以产生纤维素合酶、阿拉伯聚糖酶、阿拉伯呋喃糖苷酶、几丁质合成酶等酶类,具有降解纤维素、半纤维素、木聚糖、几丁质等物质的潜力。本研究中纤维素降解实验也进一步证明其纤维素降解活性。相对于其他芽孢杆菌,HNU24 预测出了较高比例的复合酶类,涉及糖苷水解酶家族内的复合、碳水化合物结合模块与糖苷水解酶和辅助氧化还原酶与碳水化合物结合模块的跨家族复合,表明此类酶在功能发挥方面可能存在一定的复合性,或是能够与其他酶类发生互作。另外,通过对比CAZy 数据库中贝莱斯芽孢杆菌与HNU24 的各酶类分布情况,结合已经报道的其他芽孢杆菌碳水化合物酶类数量及分布情况[18-21],发现数据库中注释的116 个贝莱斯芽孢杆菌的碳水化合物酶的平均数量约110~130 个,且各酶类数量分布比例相对稳定,但是HNU24 预测出的碳水化合物酶总量远低于其他贝莱斯芽孢杆菌平均数量,仅为平均水平的三分之一,且并未预测出多糖裂解酶(PLs),AAs 也只以复合酶类的形式存在,但其仍旧具备极大的抗菌活性和促生能力。这也使得HNU24 区别于其他菌株。可能是该菌株分离自海南热带地区,其在适应海南特殊的地理及气候条件的过程中发生进化,加大了复合酶类的比例,丢失了部分功能冗余的酶类,保留了对其自身特性贡献度最大的酶类以减小自身的代谢成本[22]。
根据次级代谢产物预测结果,HNU24 能够通过多种途径产生不同类型的13 种次级代谢产物,涵蓋了同类代表菌株FZB42 中所有已被证实的具有抗菌活性的所有活性代谢产物。在已鉴定出相似度极高的8 个次生代谢产物中,在FZB42 或芽孢杆菌属内其他菌株上均有相关功能报道,表面活性素(surfactin)已被证实与介导生物膜的形成有关[23];泛革素(fengycin)能够抑制真菌菌丝生长,如黄曲霉ABA-31[24];杆溶菌素(bacilysin)对细菌和真菌具有广谱抗菌活性,研究报道其为贝莱斯芽孢杆菌拮抗革兰氏阴性食源性致病菌的主要参与者[25];杆菌烯(bacillaene)是一类通过抑制蛋白合成来拮抗细菌和真菌的多烯类抗生素,NANNAN 等[25]和EREGA 等[26]的研究表明,其能够对空肠弯曲菌的生物膜形成起抑制作用[27];大环内酰亚胺(macrolactin)、地非西丁(difficidin)是大环内酯类抗生素,常见于各类芽孢杆菌中,有研究显示地非西丁有助于解淀粉芽孢杆菌扩宽抗菌范围[28-29],菌毒素(bacillibactin)的直接抗菌活性能够扩大B. amyloliquefaciens MBI600抗菌范围[30]。与属内其他芽孢杆菌相比,如B.subtilis 168T、B. amyloliquefaciens DSM7T 等[28-29],HNU24 产生次级代谢产物的种类更多,途径更广。值得关注的是,有1 个基因簇并未找到高相似度的同源蛋白,仅与Streptomyces sp. A2991 中saccharide 途径合成的butirosin A 或butirosin B有7%的相似度,另有4 个基因簇在数据库中并未寻找到同源蛋白,这表明在HNU24 中可能存在新的抗菌物质有待开发。
通过对贝莱斯芽孢杆菌HNU24 基因序列信息的解析,从基因组层面展示其在生物防治方面和促进植物生长方面具有巨大潜力,对今后该菌株的开发应用方向提供一定的理论基础和指导。

