热应激对奶牛GNAS启动子区DNA甲基化水平的影响
陈玉梅,张聪聪,胡丽蓉,房浩,窦金焕,郭刚,王炎,刘巧香,王雅春,徐青
热应激对奶牛启动子区DNA甲基化水平的影响

1北京交通大学生命科学与生物工程研究院,北京 100044;2中国农业大学动物科技学院,北京 100193;3北京农学院,北京 102206;4北京首农畜牧发展有限公司,北京 100029;5北京生物种业创新联合体,北京 100125
【目的】热应激严重影响奶牛的生产和健康,是制约奶业持续健康发展的重要因素。DNA甲基化作为一种重要的表观遗传调控机制参与动物的热应激反应,但其在奶牛热应激过程中的作用和分子机制研究较少。研究通过检测奶牛热应激相关的DNA甲基化变化,筛选和验证DNA甲基化相关的关键基因,为奶牛热应激的表观遗传机制研究积累数据。【方法】以北京市三元绿荷金银岛牧场的24头中国荷斯坦牛(泌乳阶段及胎次相同) 为研究对象,分别于春季 (非应激期,2017年4月份) 和夏季(热应激期,2017年7月份) 采集试验个体的血液,提取DNA,共获得48份DNA样本。首先,随机选择其中15头奶牛,并随机分为3组,每组5份DNA样本混合,通过全基因组重亚硫酸盐测序法(Whole- genome bisulfite sequencing, WGBS) 检测奶牛基因组DNA的甲基化状态,筛选差异甲基化区域 (Differential methylation region, DMR; 1000 bp windows, 500 bp overlap,<0.05) 及相关基因,利用PROMO和Methprimer软件预测基因启动子的转录因子结合位点和CpG岛区。然后,39 ℃热处理牛乳腺上皮细胞 (Bovine mammary gland epithelial cells, Mac-T)不同时间 (24 h, 48 h, 72 h),MTT法检测细胞活力。最后,利用亚硫酸盐测序法 (Bisulfite sequencing PCR, BSP)分别对春夏季的24头奶牛及39 ℃热处理不同时间的Mac-T细胞中目的基因启动子区的甲基化状态进行分析。【结果】通过WGBS共得到49 861个DMRs,其中一个DMR注释到基因GNAS复合体基因座(GNAS complex locus,)的启动子区,其整体甲基化水平在夏季热应激期极显著上调(<0.001),且该区域预测到一个352 bp的CpG岛,包含Sp1、C/EBP等重要转录因子的结合区域。24头奶牛个体中启动子区31个CG位点的整体甲基化水平在热应激期显著上调(<0.05),与WGBS结果一致,其中,21号(-113 bp,Chr13:57532733)和27号CG (-63 bp,Chr13:57532683) 位点甲基化水平显著上调(<0.05)。Mac-T 细胞热处理48和72 h后,细胞活力极显著下降(<0.01),启动子的CG 位点整体甲基化水平显著上调 (<0.05),21号和27号CG均为显著上调的差异甲基化位点,与个体水平结果一致。【结论】热应激会引起奶牛启动子甲基化水平增加,是奶牛热应激DNA甲基化调控的潜在靶基因。
热应激;奶牛;WGBS;DNA甲基化;GNAS基因
0 引言
【研究意义】全球气候的持续变暖对人类发展、动物生存和地球的生态平衡造成了威胁[1-2],日趋严重的热应激问题制约了畜牧业的持续健康发展,而气候变暖以及产奶量提高所导致的奶牛代谢热增加都加重了热应激对奶牛养殖业的影响,如导致奶牛生产性能、繁殖性能和免疫能力的下降,乳房炎、子宫内膜炎等多种疾病发病率的增加[3-7],对奶牛行业造成了巨大的经济损失。因此,研究奶牛热应激的分子机制,从表观遗传调控角度探索潜在的关键基因,对于寻找奶牛热应激抗性育种的分子标记和思路具有重要意义。【前人研究进展】近年来,奶牛热应激的相关研究取得了较大的进展。通过大量热应激相关的奶牛生理、生化和生产性能表型的收集和分析,全基因组关联分析等研究,定位了重要的热应激相关的数量性状基因座,鉴定了显著相关的遗传标记[6-8]。随着高通量检测技术的发展,组学分析已应用到奶牛热应激研究中,发现热应激会引起奶牛体内基因表达、代谢物和胃肠道微生物的显著变化,并影响多个生物学通路的生物功能[9-11]。由此可见,奶牛热应激是一个复杂过程,由多基因和非遗传因素共同作用,涉及的分子机制复杂,需要从更多角度包括表观遗传学等进行探索研究。DNA甲基化是调节基因表达的重要表观遗传修饰方式之一[12],在动植物应对环境压力过程中起重要的调节作用。研究表明DNA甲基化参与生物的热应激反应[13-15]。2021年,LIVERNOIS等报道,热应激会引起荷斯坦牛血细胞基因组DNA甲基化水平的变化[16],进一步证实了DNA甲基化在奶牛热应激反应过程中的重要作用。DNA甲基化的检测技术众多[17],而亚硫酸盐预处理DNA后检测甲基化为主流方法,其中全基因组重亚硫酸盐测序法(whole-genome bisulfite sequencing, WGBS)可获得单碱基分辨率的全基因组甲基化水平,而通过亚硫酸盐测序法(Bisulfite sequencing PCR, BSP)可快速准确地获得基因局部区域的甲基化状态。基因GNAS复合体基因座(GNAS complex locus,)(Entrez ID: 281793)位于奶牛基因组13号染色体上的57465317—57532635区域,全长67 319 bp,包括多种转录本,主要编码G蛋白和神经内分泌蛋白55等,在信号转导和甲状旁腺激素反应的生物过程中发挥重要的作用。研究表明,GNAS是一个印迹基因,表现出亲本特异性甲基化,其转录本NESP55表现为父源等位基因甲基化,仅母本表达,异常甲基化还可导致疾病的发生[18]。GNAS基因的甲基化变化与假性甲状旁腺功能减退症[19]和恶性胰腺囊肿[20]相关,但GNAS基因的甲基化状态与功能的相关在奶牛上未检索到相关报道。【本研究切入点】虽已有研究表明DNA甲基化与奶牛的热应激反应密切相关,但相关的分子机制和潜在作用仍不清楚,而GNAS基因启动子在奶牛热应激反应中的DNA甲基化的变化还未见报道。【拟解决的关键问题】本研究首先通过WGBS检测和比较春季(非热应激期)和夏季(热应激期)奶牛全基因组DNA甲基化的状态,筛选与奶牛热应激相关的差异甲基化区域和关键基因,然后采用BSP法,在个体和细胞水平上进一步验证关键基因GNAS的启动子区热应激前后甲基化水平的变化,从而探索奶牛热应激相关的表观遗传调控机制,以期为奶牛抗热应激育种积累数据。
1 材料与方法
1.1 试验动物
选择泌乳阶段和胎次相同、无亲缘关系的24头中国荷斯坦牛为试验动物,分别在春季非应激期(2017年4月,38<THI(Temperature humidity index)<68)和夏季热应激期(2017年7月,THI>72[21])采集尾静脉抗凝血,提取DNA。记录和计算牛场的温度和湿度,根据公式THI=0.8×AT+[RH×(AT-14.4)]+46.4[21]计算THI值。从24头奶牛中随机选择15头用于WGBS测序,24头牛全部应用于GNAS基因的BSP检测。所有奶牛来自北京市大兴区北京三元绿荷金银岛牧场,试验个体饲喂条件及营养水平遵从牧场管理要求。具体试验动物的分组和基因组DNA甲基化的检测、分析和验证流程如图1所示。
1.2 细胞培养、鉴定和热应激处理
在牛乳腺上皮细胞系(Mac-T,BFN607200656,青旗生物技术发展有限公司)中进行细胞实验。细胞培养液为含10 %胎牛血清和1 %青霉素与链霉素的高糖DMEM(Dulbecco’s modified eagle medium)完全培养基。培养过程中观察细胞形态并拍照。通过免疫荧光对Mac-T细胞进行鉴定:5×104个/孔接种细胞于24孔板中制作细胞爬片。待细胞长到80%—90%后,4%多聚甲醛固定15 min后用0.2% TritonX-100处理5 min,清洗后用免疫染色封闭液QuickBlockTM封闭15 min,再滴加CK8(Immunoway,YT1275)或CK18(Abcam,ab668)一抗4 ℃过夜,然后加入FITC标记的二抗(Immunoway,RS004;ABclonal,AS019)室温避光孵育30 min,洗涤后加入DAPI(4',6-diamidino- 2-phenylindole)室温避光孵育10 min,于荧光显微镜下观察并拍照,PBS孵育替换一抗作为阴性对照。细胞热应激处理过程:37 ℃正常培养24 h后,于39 ℃分别培养24、48和72 h,37℃培养相同时间的细胞作为对照组。每个时间点细胞热处理后,立即提取热处理组和对照组细胞的基因组DNA,每个试验独立重复3次。
1.3 细胞活力检测
以3×103个/孔接种Mac-T细胞于96孔板中,37 ℃正常培养24 h后每个试验组再39 ℃分别培养24 h、48 h和72 h,然后每孔加入MTT溶液继续培养4 h后去掉上清,加入DMSO低速振荡10 min,于490 nm 处检测吸光值。根据以下公式计算细胞相对活力:细胞活力=[(39 ℃试验组OD值-空白对照OD值)/(37 ℃对照组OD值-空白对照OD值)]×100%。
1.4 DNA提取和重亚硫酸盐修饰
参照Blood DNA Midi Kit试剂盒(成都福际生物技术有限公司)的使用说明提取血液和细胞样品中的基因组DNA。使用紫外分光光度计检测DNA的浓度及纯度,1%琼脂糖凝胶电泳检测DNA样品的完整性,纯度在1.8≤A260/280≤2.0之间,且无降解的DNA样品视为合格,用于后续试验。使用EZ DNA Methylation-Gold KitTM(D5005, Zymo Research)试剂盒进行重亚硫酸盐修饰。

图1 技术路线图
1.5 全基因组重亚硫酸盐测序(WGBS)
在24头中国荷斯坦牛个体中,随机选择其中15头,并随机分为3组,每组5头奶牛的DNA样本进行混合后建库,通过Illumina HiSeq4000平台-进行WGBS测序,结果获得3组非应激期和3组热应激期奶牛的全基因组甲基状态测序数据。通过FastQC(v0.11.0)和Fastp(v0.21.0)对测序原始数据进行评估和质控,Bismark(v0.20.1)进行参考基因组序列比对和统计,R包Methylkit进行DMR(1000 bp windows, 500 bp overlap,<0.05)鉴定和注释,以支持甲基化的reads数/(支持甲基化的reads数+不支持甲基化的reads数)计算甲基化水平。
1.6 重亚硫酸盐测序法(BSP)检测GNAS启动子甲基化水平
从NCBI数据库中获得基因的基因序列(NC_037340.1),利用PROMO在线软件预测转录因子结合区域(http://alggen.lsi.upc.es/cgi-bin/promo_v3/ promo/promoinit.cgi?dirDB=TF_8.3),使用Methprimer在线软件(http://www.urogene.org/cgi-bin/methprimer/ methprimer.cgi)预测GNAS基因启动子区的CpG岛,并以长度>300 bp、GC %>50.0 %,及观察值/期望值≥0.6 的标准鉴定CpG岛。以重亚硫酸盐修饰后的DNA为模板,通过巢式PCR扩增目标片段,BSP的巢式PCR引物如表1所示。PCR反应条件为:98 ℃ 30 s,98 ℃ 10 s,56.5 ℃ 30 s,72 ℃40 s,40个循环,72 ℃ 5 min。通过%琼脂糖凝胶电泳纯化PCR产物,送至生工生物(上海)工程股份有限公司进行测序。甲基化水平计算公式:CG位点上C的甲基化水平=C峰高/(C峰高+T峰值)。
1.7 统计学分析
通过卡方检验统计DMR,配对T检验计算非应激组和热应激组奶牛的组间差异的显著性,T检验计算细胞水平差异的显著性。<0.05表示差异有统计学意义,以平均值±标准差表示结果。
2 结果
2.1 GNAS基因启动子区奶牛热应激相关DMR的甲基化水平分析
根据WGBS的分析结果,如图2-A所示,夏季热应激组奶牛和春季非应激组相比,统计发现49 861个DMR(<0.05),其中有4 789个DMR位于启动子区,并注释对应到4 069个基因。其中,DMR(Chr13:57532501—57533500,命名为DMR1)位于基因的启动子和5’-UTR内,即转录起始点前879 bp 和后121 bp(图2-C)。WGBS检测结果如图2-B显示,夏季热应激组奶牛的DMR1整体甲基化水平极显著高于非应激组(<0.001)。通过Methprimer软件分析发现DMR1包含一个352 bp的CpG岛,位于GNAS基因的启动子-364—-12 bp区。本研究对包含该CpG岛在内的-454—+144 bp区域(命名为GNAS1)的甲基化水平进行了进一步分析,图2-C对DMR1、GNAS1、及CpG岛的位置等信息进行了详细的说明。PROMO预测发现,GNAS1区包含36个CG位点,具有Sp1、AP2、C/EBP等重要转录因子潜在的结合位点(图2-D)。WGBS分析结果显示,夏季热应激组奶牛GNAS1区的所有CG位点甲基化水平显著高于春季非应激组(44.20% vs 57.31%,<0.05,图2-B)。综上,热应激组和非应激组个体DMR1和GNAS1区CG位点甲基化水平的显著差异关系密切。
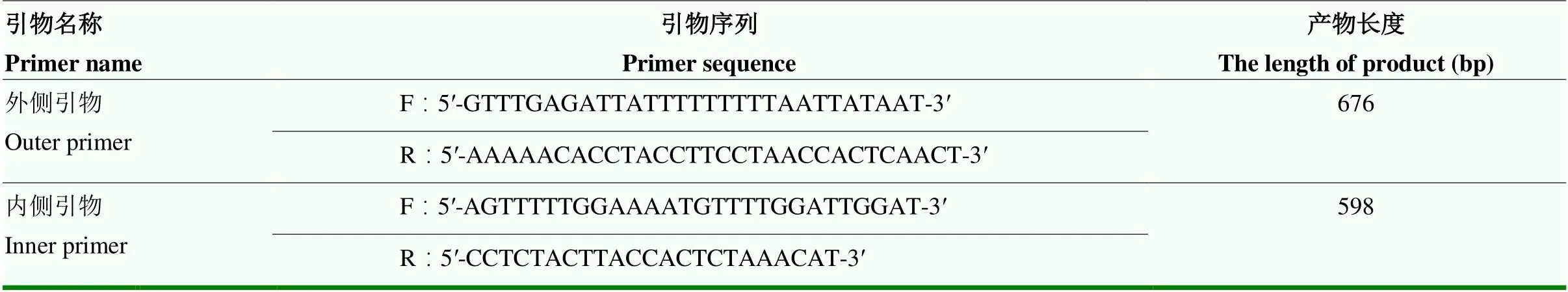
表1 GNAS基因的巢式PCR引物
2.2 奶牛热应激下GNAS1甲基化水平显著升高
通过BSP法分别对24头奶牛热应激期和非应激期的GNAS1片段中CG位点的甲基化状态进行检测。如图3-A所示,BSP方法清楚地检测到了31个CG位点的甲基化状态。在检测的48个奶牛样本中,夏季热应激条件下,GNAS1的CG位点整体甲基化水平显著升高(<0.05,图3-B),与WGBS检测结果一致。对每一个CG位点的甲基化水平进行分析,结果如图3-C所示,31个CG位点甲基化水平在夏季热应激条件下都呈上升趋势,其中21号和27号CG(分别位于-113 bp和-63 bp处,即基因组上Chr13:57532733和Chr13:57532683处)的甲基化水平分别显著上调7.37%和7.72%(<0.05)。
2.3 Mac-T细胞鉴定
对使用的Mac-T细胞进行鉴定。培养过程中,Mac-T细胞稳定传代且贴壁生长,呈现典型的上皮细胞形态(图4),低密度时呈现分散生长的多角形单个细胞(图4-A),密度增大后呈不规则的鹅卵石样单层聚集(图4-B),符合上皮细胞形态学特点。Mac-T表达上皮细胞特异性的角蛋白CK8和CK18,其免疫荧光鉴定结果如图5所示,本研究所用细胞大小清晰,经FITC染色的细胞质可与DAPI染色的细胞核完全重合,且阴性对照无绿色荧光。形态学观察和免疫荧光鉴定表明实验所用细胞为牛乳腺上皮细胞Mac-T。
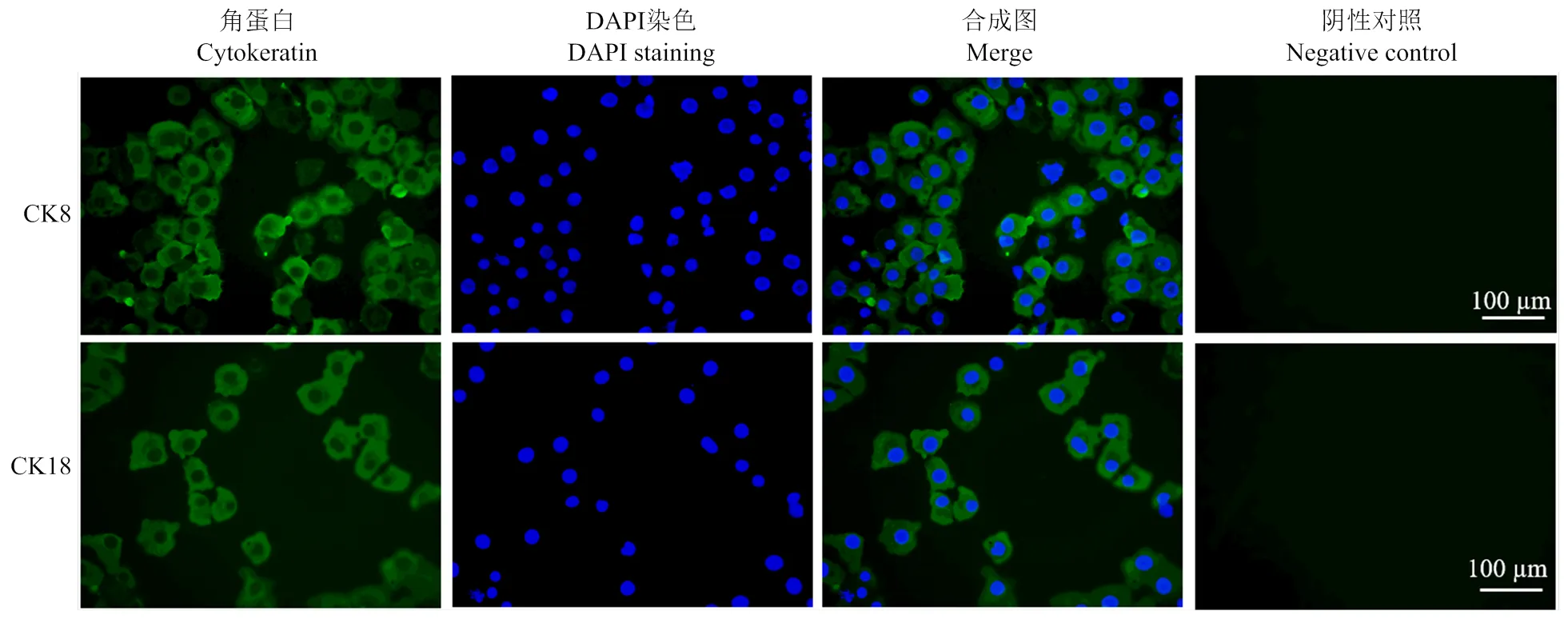
图5 免疫荧光鉴定Mac-T细胞
2.4 Mac-T细胞热处理后细胞活力降低和GNAS1甲基化水平升高
利用MTT法检测热应激对细胞活力的影响,结果表明39 ℃热处理影响Mac-T细胞活力。如图6所示,与对照组相比,48和72 h热处理组细胞活力极显著下降(<0.01),而24 h热处理组的细胞活力变化不明显。通过BSP法检测了Mac-T细胞不同热处理组的的甲基化水平,结果表明热处理后GNAS1甲基化水平显著升高。如图7-A所示,39 ℃热处理48 h后,GNAS1的CG位点甲基化的平均水平从58.93%上调至65.02%(<0.01),热处理72 h后则上调到63.84%(<0.05)。对于单个CG位点,热处理48 h和72 h后的细胞中,31个CG位点都呈现甲基化水平上调趋势,而21号和27号CG位点甲基化水平的上调较显著(图7-B)。综上,热应激会引起牛乳腺上皮细胞基因启动子甲基化水平的显著上调,21号和27号CG位点可能是热应激相关的重要甲基化位点。
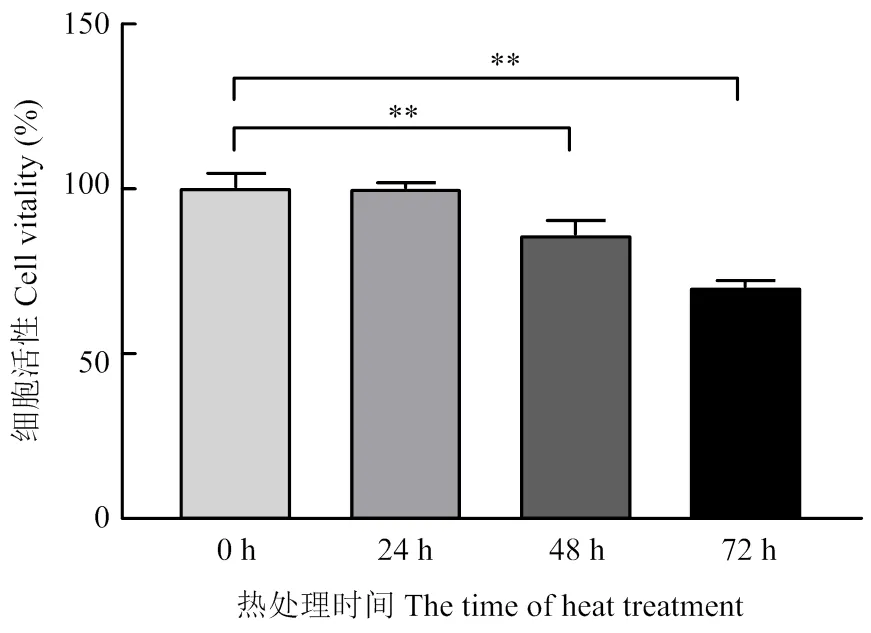
**表示P<0.01;** represents P<0.01
3 讨论
3.1 奶牛热应激研究现状
目前,热应激问题是奶牛养殖业面临的巨大挑战之一。热应激会严重降低奶牛的生产性能[22]和免疫能力[23],引发乳房炎和其他健康问题,甚至会直接导致奶牛死亡[24]。生物机体可通过热休克反应[25]、脂肪代谢[26]和氧化应激[27]等调节方式响应热应激,但DNA甲基化在奶牛热应激过程中发挥的作用鲜少报道。因此,本研究从DNA甲基化的角度出发,在个体和细胞水平上,采用不同方法组合检测和分析奶牛热应激后基因组甲基化的变化,为奶牛热应激的表观遗传机制研究积累数据和提供思路。
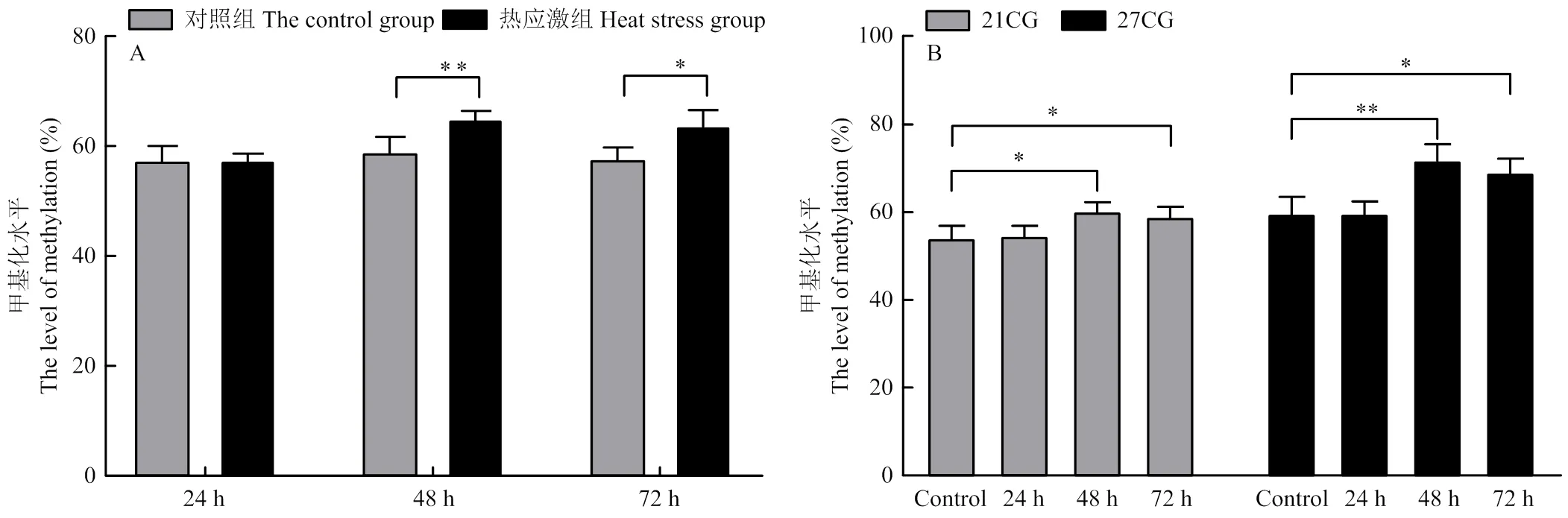
A:Mac-T细胞39 ℃热处理不同时间后GNAS1的甲基化水平;B:Mac-T细胞39 ℃热处理不同时间后21和27号CG位点的甲基化水平。*表示P<0.05;**表示P<0.01
3.2 奶牛基因组甲基化检测方法
WGBS被称为甲基化检测的金标准,虽价格昂贵且数据分析复杂,但特异性和灵敏度较高,能够获得个体全基因组范围内的甲基化状态[28],常用于小样本全基因组甲基化状态的检测。BSP法通过一代测序技术则能准确快速获得部分区域的甲基化状态[29],常用于大样本中已知基因启动子区域甲基化水平的检测。WGBS在牛的甲基化检测中已有广泛的应用,比如2020年,Zhou等采用WGBS检测分析了荷斯坦牛16个组织的全基因组甲基化水平状态和不同组织的差异[30],WANG等[31]对不同乳蛋白和乳脂率的泌乳荷斯坦牛乳腺组织中全基因组DNA甲基化进行了检测,而IBEAGHA-AWEMU等[32]对DNA 甲基化在奶牛的回肠和回肠淋巴结对鸟分枝杆菌亚种反应中的作用进行了研究。本研究首先利用WGBS检测了热应激下小样本奶牛全基因组甲基化状态的变化关系,筛选到关键基因,再通过BSP法在大样本个体和细胞模型中深入验证热应激下关键基因启动子区域的甲基化水平的变化。根据检测方法的特点,经济、快速、准确地获得了试验结果。
3.3 热应激对GNAS基因DNA甲基化的影响
DNA甲基化是指在DNA甲基转移酶(DNA methyltransferases,DNMTs)的催化下,以S-腺苷甲硫氨酸为甲基供体,将甲基转移到胞嘧啶的第五位碳原子上,使DNA分子的碱基结合甲基的过程[33]。作为一种重要的表观遗传修饰,DNA甲基化与细胞生长、胚胎发育和衰老等生命活动密切相关[34],同时参与生物对环境的适应性过程。长期的热应激可导致机体DNA甲基化的改变。KOROTKO等[35]在2021年研究了热应激不同处理时间拟南芥的基因组DNA甲基化变化,发现热应激降低了DNA甲基化水平,并发现了Hsp70等关键差异甲基化基因。VINOTH等[36]发现鸡胚胎发育过程中的,热应激可引起启动子区域甲基化水平的变化,从而提高鸡出生后的耐热能力,表明启动子区域甲基化是促进表观遗传适应的重要因素。在2021年LIVERNOIS[16]的报道中,分析了热应激前后奶牛外周血单核细胞基因组启动子区的DNA甲基化变化,结果提示启动子区域的DNA甲基化状态与奶牛对热应激反应的响应有关。已有的研究说明DNA甲基化与奶牛热应激关系密切,在热应激下基因的DNA甲基化水平可发生异常改变。在本研究中,奶牛个体和细胞热应激后,GNAS基因启动子的甲基化水平显著升高,表明GNAS基因通过DNA甲基化参与了奶牛的热应激反应,可能是调控奶牛响应热应激的一种重要方式。
2018年报道氧应激后TPM2基因的甲基化水平增加[37],Corbett等[38]总结NR3C1启动子的高甲基化与应激暴露相关,同时ZHANG等[39]报道同型半胱氨酸引起GPX4基因高甲基化,与氧化应激有关,以上与本研究结果相似,表现为应激条件下基因的甲基化水平增加。热应激引起基因甲基化水平的变化,可能与机体内的DNMTs(形成和维持DNA甲基化的关键酶)的表达量和活性变化等因素有关。已有研究表明应激会改变机体DNMTs表达,导致DNA甲基化水平的改变,牛卵母细胞热应激24 h后DNMT1基因表达水平显著增加[40],而酒精刺激导致了神经前体细胞和小鼠胚胎成纤维细胞DNMT3A基因的表达水平上调[41]。本研究中GNAS基因启动子的DNA甲基化水平在热应激后显著增加,可能与DNMTs表达上调有关。在人上,GNAS基因与假性甲状旁腺功能减退症、骨异型增生和垂体瘤等疾病的发生发展有关,LI 等[42]报道GNAS基因的甲基化状态可能是一种用于卵母细胞质量检测的新型表观遗传标记。刘明哲等[43]在GNAS基因上获得了与头孢类药物过敏高度相关的DNA甲基化差异位点,而POIRIER等[44]在泌乳早期牛卵母细胞中发现了代谢相关的差异甲基化区域。在本研究中,热应激后奶牛GNAS基因的启动子CpG岛的CG胞嘧啶整体甲基化水平显著升高,且所有CG位点的胞嘧啶甲基化水平在热应激后都呈现上升趋势。基因启动子区CpG岛的甲基化具有重要作用,可通过影响转录因子等调控元件的结合而调控基因表达[45]。在该研究中,采用PROMO预测GNAS基因启动子的转录因子结合位点,发现重要转录因子C/EBP和Sp1结合区域富含多个CG位点,且其位点的甲基化水平均上调,而Sp1的结合区域中的27号CG位点热应激后甲基化水平显著增加。Sp1是甲基化敏感转录因子,其与基因启动子区域的结合受到DNA甲基化的影响[46],DNA 甲基化可抑制Sp1与靶基因的结合,导致基因表达沉默。许多启动子区域富含CG的基因,例如FOXF2、KEAP1和NDRG2等的表达受到 Sp1和DNA甲基化的联合作用的调控[46-48]。Sp1和DNMT1通过与靶基因启动子区域竞争性结合可调控靶基因的甲基化状态和转录表达。本研究中,GNAS的Sp1结合区域的CG位点甲基化水平显著增加,可能会干扰Sp1与GNAS启动子的结合,从而影响基因的表达,因此27号CG位点可能是奶牛热应激GNAS基因的关键甲基化位点,后续可进一步分析该位点的DNA甲基化在奶牛热应激中的作用。目前,GNAS基因甲基化在热应激中的功能研究鲜少报道。本研究发现了热应激下GNAS基因启动子甲基化水平的变化趋势,为奶牛热应激研究提供了新的候选表观遗传标志,具体的甲基化调控机制和功能有待更深入的挖掘和探索。
4 结论
本研究通过WGBS和BSP检测了奶牛个体和细胞水平上的DNA甲基化状态,结果显示热应激条件下启动子的甲基化水平显著增加,且在31个CG位点中,位于基因组上Chr13:57532733 (-113 bp)和Chr13:57532683(-63 bp)处的CG位点的甲基化水平热应激后显著上调。综上,通过分析了奶牛热应激与GNAS基因启动子甲基化的关系,提示DNA甲基化在动物热应激反应中的重要调控功能。
[1] ROSSIELLO M R, SZEMA A. Health effects of climate change- induced wildfires and heatwaves. Cureus, 2019, 11(5): e4771.
[2] ALI M Z, CARLILE G, GIASUDDIN M. Impact of global climate change on livestock health: Bangladesh perspective. Open Veterinary Journal, 2020, 10(2): 178-188.
[3] RANJITKAR S, BU D P, WIJK M, MA Y, MA L, ZHAO L S, SHI J M, LIU C S, XU J C. Will heat stress take its toll on milk production in China? Climatic Change, 2020, 161(4): 637-652.
[4] BAGATH M, KRISHNAN G, DEVARAJ C, RASHAMOL V P, PRAGNA P, LEES A M, SEJIAN V. The impact of heat stress on the immune system in dairy cattle: a review. Research in Veterinary Science, 2019, 126: 94-102.
[5] BERNABUCCI U, BIFFANI S, BUGGIOTTI L, VITALI A, LACETERA N, NARDONE A. The effects of heat stress in Italian Holstein dairy cattle. Journal of Dairy Science, 2014, 97(1): 471-486.
[6] 胡丽蓉, 康玲, 王淑慧, 李玮, 鄢新义, 罗汉鹏, 董刚辉, 王新宇, 王雅春, 徐青. 冷热应激对北京地区荷斯坦牛产奶性能及血液生化指标的影响. 中国农业科学, 2018, 51(19): 3791-3799. doi:10. 3864/j.issn.0578-1752.2018.19.015.
HU L R, KANG L, WANG S H, LI W, YAN X Y, LUO H P, DONG G H, WANG X Y, WANG Y C, XU Q. Effects of cold and heat stress on milk production traits and blood biochemical parameters of Holstein cows in Beijing area. Scientia Agricultura Sinica, 2018, 51(19): 3791-3799. doi:10.3864/j.issn.0578-1752.2018.19.015. (in Chinese)
[7] ABBAS Z, SAMMAD A, HU L R, FANG H, XU Q, WANG Y C. Glucose metabolism and dynamics of facilitative glucose transporters (GLUTs) under the influence of heat stress in dairy cattle. Metabolites, 2020, 10(8): 312.
[8] KHAN A, KHAN M Z, UMER S, KHAN I M, XU H T, ZHU H B, WANG Y C. Cellular and molecular adaptation of bovine granulosa cells and oocytes under heat stress. Animals: An Open Access Journal from MDPI, 2020, 10(1): 110.
[9] LIU S H, YUE T T, AHMAD M J, HU X W, ZHANG X X, DENG T X, HU Y, HE C J, ZHOU Y, YANG L G. Transcriptome analysis reveals potential regulatory genes related to heat tolerance in Holstein dairy cattle. Genes, 2020, 11(1): 68.
[10] ZHAO S G, MIN L, ZHENG N, WANG J Q. Effect of heat stress on bacterial composition and metabolism in the rumen of lactating dairy cows. Animals: An Open Access Journal from MDPI, 2019, 9(11): 925.
[11] KOCH F, ALBRECHT D, GÖRS S, KUHLA B. Jejunal mucosa proteomics unravel metabolic adaptive processes to mild chronic heat stress in dairy cows. Scientific Reports, 2021, 11: 12484.
[12] GAO F, LUO Y L, LI S T, LI J, LIN L, NIELSEN A L, SØRENSEN C B, VAJTA G, WANG J, ZHANG X Q, DU Y T, YANG H M, BOLUND L. Comparison of gene expression and genome-wide DNA methylation profiling between phenotypically normal cloned pigs and conventionally bred controls. PLoS ONE, 2011, 6(10): e25901.
[13] ZHAO J G, LU Z G, WANG L, JIN B. Plant responses to heat stress: physiology, transcription, noncoding RNAs, and epigenetics. International Journal of Molecular Sciences, 2020, 22(1): 117.
[14] OUELLET V, LAPORTA J, DAHL G E. Late gestation heat stress in dairy cows: Effects on dam and daughter. Theriogenology, 2020, 150: 471-479.
[15] WU J, ZHANG W W, LI C H. Recent advances in genetic and epigenetic modulation of animal exposure to high temperature. Frontiers in Genetics, 2020, 11: 653.
[16] LIVERNOIS A M, MALLARD B A, CARTWRIGHT S L, CÁNOVAS A. Heat stress and immune response phenotype affect DNA methylation in blood mononuclear cells from Holstein dairy cows. Scientific Reports, 2021, 11(1): 11371.
[17] KHODADADI E, FAHMIDEH L, KHODADADI E, DAO S, YOUSEFI M, TAGHIZADEH S, ASGHARZADEH M, YOUSEFI B, KAFIL H S. Current advances in DNA methylation analysis methods. BioMed Research International, 2021, 2021: 8827516.
[18] JÜPPNER H. Molecular definition of pseudohypoparathyroidism variants. The Journal of Clinical Endocrinology & Metabolism, 2021, 106(6): 1541-1552.
[19] MANTOVANI G, DE SANCTIS L, BARBIERI A M, ELLI F M, BOLLATI V, VAIRA V, LABARILE P, BONDIONI S, PEVERELLI E, LANIA A G, BECK-PECCOZ P, SPADA A. Pseudohypoparathyroidism and GNAS epigenetic defects: Clinical evaluation of Albright hereditary osteodystrophy and molecular analysis in 40 patients. The Journal of Clinical Endocrinology & Metabolism, 2010, 95(2): 651-658.
[20] FAIAS S, DUARTE M, PEREIRA L, CHAVES P, CRAVO M, DIAS PEREIRA A, ALBUQUERQUE C. Methylation changes at the GNAS imprinted locus in pancreatic cystic neoplasms are important for the diagnosis of malignant cysts. World Journal of Gastrointestinal Oncology, 2020, 12(9): 1056-1064. doi:10.4251/wjgo.v12.i9.1056.
[21] MCDOWELL R E, HOOVEN N W, CAMOENS J K. Effect of climate on performance of holsteins in first lactation. Journal of Dairy Science, 1976, 59(5): 965-971.
[22] HAMMAMI H, BORMANN J, M’HAMDI N, MONTALDO H H, GENGLER N. Evaluation of heat stress effects on production traits and somatic cell score of Holsteins in a temperate environment. Journal of Dairy Science, 2013, 96(3): 1844-1855.
[23] KOCH F, THOM U, ALBRECHT E, WEIKARD R, NOLTE W, KUHLA B, KUEHN C. Heat stress directly impairs gut integrity and recruits distinct immune cell populations into the bovine intestine. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(21): 10333-10338.
[24] TOKUHISA K, TSURUTA S, DE VRIES A, BERTRAND J K, MISZTAL I. Estimation of regional genetic parameters for mortality and 305-d milk yield of US Holsteins in the first 3 parities. Journal of Dairy Science, 2014, 97(7): 4497-4502.
[25] KIM W S, NEJAD J G, PENG D Q, JUNG U S, KIM M J, JO Y H, JO J H, LEE J S, LEE H G. Identification of heat shock protein gene expression in hair follicles as a novel indicator of heat stress in beef calves. Animal, 2020, 14(7): 1502-1509.
[26] HENG J H, TIAN M, ZHANG W F, CHEN F, GUAN W T, ZHANG S H. Maternal heat stress regulates the early fat deposition partly through modification of m 6 A RNA methylation in neonatal piglets. Cell Stress & Chaperones, 2019, 24(3): 635-645.
[27] WANG Y R, YANG C X, ELSHEIKH N A H, LI C M, YANG F X, WANG G L, LI L. HO-1 reduces heat stress-induced apoptosis in bovine granulosa cells by suppressing oxidative stress. Aging, 2019, 11(15): 5535-5547.
[28] LI Q, HERMANSON P J, SPRINGER N M. Detection of DNA methylation by whole-genome bisulfite sequencing. Methods in Molecular Biology (Clifton, N J), 2018, 1676: 185-196.
[29] AKIKA R, AWADA Z, MOGHARBIL N, ZGHEIB N K. Region of interest methylation analysis: A comparison of MSP with MS-HRM and direct BSP. Molecular Biology Reports, 2017, 44(3): 295-305.
[30] ZHOU Y, LIU S L, HU Y, FANG L Z, GAO Y H, XIA H, SCHROEDER S G, ROSEN B D, CONNOR E E, LI C J, BALDWIN R L, COLE J B, VAN TASSELL C P, YANG L G, MA L, LIU G E. Comparative whole genome DNA methylation profiling across cattle tissues reveals global and tissue-specific methylation patterns. BMC Biology, 2020, 18(1): 85.
[31] WANG M Q, BISSONNETTE N, DUDEMAINE P L, ZHAO X, IBEAGHA-AWEMU E M. Whole genome DNA methylation variations in mammary gland tissues from Holstein cattle producing milk with various fat and protein contents. Genes, 2021, 12(11): 1727.
[32] IBEAGHA-AWEMU E M, BISSONNETTE N, BHATTARAI S, WANG M Q, DUDEMAINE P L, MCKAY S, ZHAO X. Whole genome methylation analysis reveals role of DNA methylation in cow's ileal and ileal lymph node responses tosubsp. paratuberculosis infection. Frontiers in Genetics, 2021, 12: 797490.
[33] WEBER M, SCHÜBELER D. Genomic patterns of DNA methylation: targets and function of an epigenetic mark. Current Opinion in Cell Biology, 2007, 19(3): 273-280.
[34] TATE P H, BIRD A P. Effects of DNA methylation on DNA-binding proteins and gene expression. Current Opinion in Genetics & Development, 1993, 3(2): 226-231.
[35] KOROTKO U, CHWIAŁKOWSKA K, SAŃKO-SAWCZENKO I, KWASNIEWSKI M. DNA demethylation in response to heat stress in. International Journal of Molecular Sciences, 2021, 22(4): 1555.
[36] VINOTH A, THIRUNALASUNDARI T, SHANMUGAM M, UTHRAKUMAR A, SUJI S, RAJKUMAR U. Evaluation of DNA methylation and mRNA expression of heat shock proteins in thermal manipulated chicken. Cell Stress & Chaperones, 2018, 23(2): 235-252.
[37] ZHANG J, ZHANG J, XU S, ZHANG X, WANG P, WU H, XIA B, ZHANG G, LEI B, WAN L, ZHANG D, PANG D. Hypoxia-induced TPM2 methylation is associated with chemoresistance and poor prognosis in breast cancer. Cellular Physiology and Biochemistry, 2018, 45(2): 692-705.
[38] CORBETT R J, LUTTMAN A M, WURTZ K E, SIEGFORD J M, RANEY N E, FORD L M, ERNST C W. Weaning induces stress-dependent DNA methylation and transcriptional changes in piglet PBMCs. Frontiers in Genetics, 2021, 12: 633564.
[39] ZHANG X Y, HUANG Z B, XIE Z A, CHEN Y L, ZHENG Z Y, WEI X A, HUANG B, SHAN Z, LIU J H, FAN S W, CHEN J, ZHAO F D. Homocysteine induces oxidative stress and ferroptosis of nucleus pulposus via enhancing methylation of GPX4. Free Radical Biology and Medicine, 2020, 160: 552-565.
[40] PAVANI K C, ROCHA A, BARON E, LOURENÇO J, FAHEEM M, DA SILVA F M. The effect of kinetic heat shock on bovine oocyte maturation and subsequent gene expression of targeted genes. Zygote (Cambridge, England), 2017, 25(3): 383-389.
[41] MIOZZO F, ARNOULD H, DE THONEL A, SCHANG A L, SABÉRAN-DJONEIDI D, BAUDRY A, SCHNEIDER B, MEZGER V. Alcohol exposure promotes DNA methyltransferase DNMT3A upregulation through reactive oxygen species-dependent mechanisms. Cell Stress & Chaperones, 2018, 23(1): 115-126.
[42] LI Q N, LI A, SUN S M, LIU W B, MENG T G, GUO X P, SCHATTEN H, SUN Q Y, OU X H. The methylation status in GNAS clusters May Be an epigenetic marker for oocyte quality. Biochemical and Biophysical Research Communications, 2020, 533(3): 586-591.
[43] 刘明哲, 郭华林, 冯彦, 李建国, 李鹏飞, 高彩荣, 郭相杰. 过敏者外周血DNA甲基化差异. 法医学杂志, 2021, 37(2): 211-214.
LIU M Z, GUO H L, FENG Y, LI J G, LI P F, GAO C R, GUO X J. DNA methylation differences in peripheral blood of patients with anaphylaxis. Chinese Journal of Rehabilitation Medicine, 2021, 37(2): 211-214. (in Chinese)
[44] POIRIER M, TESFAYE D, HAILAY T, SALILEW-WONDIM D, GEBREMEDHN S, RINGS F, NEUHOFF C, SCHELLANDER K, HOELKER M. Metabolism-associated genome-wide epigenetic changes in bovine oocytes during early lactation. Scientific Reports, 2020, 10: 2345.
[45] ZEMACH A, MCDANIEL I E, SILVA P, ZILBERMAN D. Genome- wide evolutionary analysis of eukaryotic DNA methylation. Science, 2010, 328(5980): 916-919.
[46] TIAN H P, LUN S M, HUANG H J, HE R, KONG P Z, WANG Q S, LI X Q, FENG Y M. DNA methylation affects the SP1-regulated transcription of FOXF2in breast cancer cells. Journal of Biological Chemistry, 2015, 290(31): 19173-19183.
[47] GUO D, WU B, YAN J H, LI X S, SUN H M, ZHOU D S. A possible gene silencing mechanism: Hypermethylation of the Keap1 promoter abrogates binding of the transcription factor Sp1 in lung cancer cells. Biochemical and Biophysical Research Communications, 2012, 428(1): 80-85.
[48] SHEN L, QU X, MA Y, ZHENG J, CHU D, LIU B, LI X, WANG M, XU C, LIU N, YAO L, ZHANG J. Tumor suppressor NDRG2 tips the balance of oncogenic TGF-β via EMT inhibition in colorectal cancer. Oncogenesis, 2014, 3(2): e86.
Effect of Heat Stress on DNA Methylation ofPromoter Region in Dairy Cows

1Institute of Life Science and Bioengineering, Beijing Jiaotong University, Beijing 100044;2College of Animal Science and Technology, China Agricultural University, Beijing 100193;3Beijing University of Agriculture, Beijing 102206;4Beijing Sunlon Livestock Development Company Limited, Beijing 100029;5Beijing Innovation Consortium of Bio-breeding, Beijing 100125
【Objective】Heat stress has seriously impaired the production and health of dairy cows, causing the subsequent limitation in sustainable development of dairy industry. DNA methylation is an important epigenetic regulatory mechanism involved in an animal’s heat stress response, but the potential functions and molecular mechanisms of which are not clear. The current study was conducted to detect the DNA methylation related to heat stress in dairy cows and to identify target genes related to DNA methylation, so as to provide a better insight into the epigenetics mechanism of heat stress in dairy cows.【Method】In the study, 24 Chinese Holstein lactation cows (same lactation stage and same parity) in Sanyuan dairy farm were used for the blood samples collection in heat stress period (July in the summer of 2017) and non-heat stress period (April in spring 2017), respectively, followed by DNA extraction. To explore the DNA methylation differences in dairy cows from different heat stress period, 15 of 24 animals were randomly assigned to 3 groups (N=5 animals/group), 5 DNA samples in one group were mixed together to get a single pooled DNA sample, thus 6 pooled DNA samples including 3 from spring and 3 from summer were used for the DNA methylation detection by the whole-genome bisulfite sequencing (WGBS), then the differential methylation region (DMR; 1000 bp windows, 500 bp overlap,<0.05) and key gene were identified. PROMO and Methprimer software were used to predict transcription factor binding sites and CpG islands, respectively. Then, the bovine mammary gland epithelial cells (Mac-T) were treated at 39 ℃ for 24 h, 48 h, and 72 h, and the cell viability were detected by MTT method. Finally, using the bisulfite sequencing PCR (BSP), the methylation levels of target gene promoter in 24 dairy cows in spring and summer and Mac-T cells treated in 39℃ were examined, respectively. 【Result】Based on the DNA methylation analysis of WGBS data, 49 861 differential methylation regions (DMRs) associated with heat stress were identified. One of DMRs was attributed to the promoter area of GNAS complex locus (), whose methylation level significantly increased in heat-stressed animals (<0.001). Also, there was a 352 bp CpG island in the promoter ofcontaining potential binding sites for Sp1, C/EBP and other important transcription factors. Further the methylation status of thegene promoter region in heat stressed dairy cows were verified by BSP, and the average methylation level in all cytosine of 31 CpG sites was higher in heat stress cows than that in control groups (<0.05), which corresponding to the above WGBS results. Moreover, the 21 (-113 bp, Chr13:57532733) and 27 (-63 bp, Chr13:57532683) CpG sites showed significant differences between the spring and summer groups (<0.05). In Mac-T cells, after 48 h and 72 h heat treatment, the cell viability decreased significantly (<0.01), but the overall CG methylation level of 31 CpG sites in thegene promoter region increased significantly (<0.05), and also the similar significant methylation changes appeared in the site 21 and 27 CpG in cell. 【Conclusion】Heat stress increased the methylation levels of the promoter region of thein dairy cows as well as in cells, which indicated thatwas a potential target gene regulated DNA methylation in heat stress response of dairy cows.
heat stress; dairy cows; WGBS; DNA methylation;

10.3864/j.issn.0578-1752.2023.12.013
2022-02-09;
2022-07-28
国家自然科学基金外国青年学者研究基金(3175011045)、财政部和农业农村部:国家现代农业产业技术体系(CARS-36)
陈玉梅,Tel:18813021971;E-mail:19121601@bjtu.edu.cn。通信作者徐青,Tel:13520490069;E-mail:qingxu@bjtu.edu.cn
(责任编辑 林鉴非)

