食品能力验证样品中沙门氏菌的分离与多相分类鉴定
何肖云 饶秋华 吕新 刘兰英 傅建炜


摘 要:为沙门氏菌的快速检测与鉴定提供参考,从传统标准检测方法、特异性PCR、分子系统发育3个层面,其中传统标准检测方法基于食品安全国家标准GB 4789.4-2016《食品微生物學检验 沙门氏菌检验》、特异性PCR基于invA基因、分子系统发育基于16S rDNA,对能力验证(CFAPA-805)样品进行沙门氏菌的分离鉴定。结果表明:3种检测方法结果一致,样品341中检出沙门氏菌,其中生化鉴定、血清学鉴定与分子系统发育鉴定结果显示,此次阳性样品中的沙门氏菌为沙门氏菌生化群(Ⅰ),肠炎沙门氏菌肠炎亚种,血清分型为O:9,12:H:g, m。本次实验室的能力验证结果为“满意”,肯定实验室沙门氏菌检测能力。通过比较,特异性PCR鉴定可以确定沙门氏菌是否检出,生化鉴定、血清学鉴定和分子系统发育鉴定可以确认沙门氏菌的生化群、血清分型与分子分型。
关键词:沙门氏菌;多相鉴定;血清鉴定;特异性PCR;分子系统发育
中图分类号:TS 207.4 文献标志码:A 文章编号:0253-2301(2023)01-0051-07
DOI: 10.13651/j.cnki.fjnykj.2023.01.008
Isolation and Polyphasic Taxonomy of Salmonella in Food Proficiency Testing Samples
HE Xiao-yun, RAO Qiu-hua, LV Xin, LIU Lan-ying, FU Jian-wei*
(Institute of Agricultural Quality Standards and Testing Technology, Fujian Academy of Agricultural Sciences/
Fujian Key Laboratory of Agro-products Quality & Safety, Fuzhou, Fujian 350003, China)
Abstract: In order to provide reference for the rapid detection and identification of Salmonella, Salmonella was isolated and identified from the proficiency testing (CFAPA-805) samples from three aspects including the traditional standard detection methods, specific PCR and molecular phylogeny. The traditional standard detection method was based on the national food safety standard (GB 4789.4-2016) ″Salmonella Examination of Food Microbiological Examination″, the specific PCR was based on the invA gene, and the molecular phylogeny was based on the 16S rDNA. The results showed that the results of the three detection methods were consistent. Salmonella was detected in 341 samples, and the results of physiological and biochemical identification, serological identification and molecular phylogenetic identification showed that: the Salmonella in the positive samples was Salmonella biochemical group (Ⅰ), Salmonella enteritidis subspecies, of which the serotyping was O:9, 12:H:g, m. The ability verification result of this laboratory was ″satisfactory″, affirming the laboratory′s ability to detect Salmonella. By the comparison, the specific PCR identification could determine whether Salmonella was detected. The physiological and biochemical identification, serological identification and molecular phylogenetic identification could confirm the biochemical group, serotyping and molecular typing of Salmonella.
Key words: Salmonella; Polyphasic taxonomy; Serological identification; Specific PCR; Molecular phylogeny
沙门氏菌是全球范围内最常见的一种食源性革兰氏阴性杆菌,可导致胃肠炎、败血症等引起机体功能障碍的严重疾病[1-2]。在中国沙门氏菌同样也是主要的食源性致病菌,据报道有70%~80%的细菌性食物中毒事件均因沙门氏菌污染所致[3]。因此,作为食源性致病菌的重点监控对象,沙门氏菌的检测具有重要的卫生安全意义。目前沙门氏菌的检测技术主要包括传统标准检测方法、分子生物学方法、免疫学方法,以及生物传感器等新型检测方法[4],其中传统标准检测方法与分子生物学法应用较广。
传统标准检测方法主要基于沙门氏菌的生长生化特征进行检验[5],按照食品安全国家标准GB4789.4-2016《食品微生物学检验 沙门氏菌检验》[6],分为預增菌、选择性增菌、选择性分离、生化鉴定和血清学鉴定5个步骤,虽然存在过程繁琐、耗时费力、难以批量化等缺点[4,7],但是目前市场配套和技术成熟度高,且环境要求和技术门槛较低,具有较高的准确性,检测结果得到市场认同,是国际和国内沙门氏菌检测的黄金标准[8]。分子生物学检测方法是通过保守或特异的核酸序列比对实现细菌种属鉴定,具有灵敏、快速、特异等优势[9],目前基于16S rDNA保守序列的系统发育分析已经广泛应用于细菌的分类鉴定[10-11],而基于物种特异性基因的PCR技术在定性检测中也十分常见[12-13]。因此,本研究采用传统标准检测方法、特异性PCR和分子系统发育3种方法,对能力验证(CFAPA-805)样品进行沙门氏菌的分离鉴定,在验证实验室检测能力的同时,为沙门氏菌的快速检测与鉴定提供参考。
1 材料与方法
1.1 试验材料与仪器
1.1.1 供试样品
CFAPA-805食品中沙门氏菌的测定能力验证计划样品,样品编号为和405和341,以样品405和样品341表示,两份样品均为冻干粉末状,以西林瓶真空密封包装,由大连中食国实检测技术有限公司提供。
1.1.2 标准菌株
肠炎沙门氏菌CMCC(B)50335、枯草芽孢杆菌ATCC6633采购自广东环凯微生物科技有限公司。
1.1.3 培养基与试剂
磷酸盐缓冲液(PBS)、缓冲蛋白胨水(BPW)、四硫磺酸钠煌绿(TTB)增菌液、亚硒酸盐胱氨酸(SC)增菌液、亚硫酸铋(BS)琼脂、HE琼脂、木糖赖氨酸脱氧胆盐(XLD)琼脂、科玛嘉沙门氏菌属显色培养基(CAS)、DBI-05沙门氏菌干制生化鉴定试剂盒(北京陆桥技术股份有限公司);A~F沙门氏菌属诊断血清(宁波天润生物药业有限公司);细菌基因组提取试剂盒、PCR Mix、琼脂糖凝胶、PCR引物合成、PCR产物测序[生工生物工程(上海)股份有限公司]。
1.1.4 仪器设备
高温高压灭菌锅:SQL810C,重庆雅马拓有限公司;生物安全柜:BSC-1360ⅡA2,北京东联哈尔有限公司;恒温培养箱:IC612C,日本雅马拓公司; PCR仪:BIO-RADTM S1000,美国伯乐公司;电泳仪:DYY-6C ,北京六一仪器厂;凝胶成像仪:BIO-RADTM Gel Doc XR+,美国伯乐公司。
1.2 试验方法
1.2.1 样品处理
根据CFAPA-805《食品中沙门氏菌的测定能力验证计划 参试指导书》中的要求,在无菌条件下开启真空密封西林瓶,立即加入4 mL 无菌PBS水化溶解,吸出至无菌三角瓶内,反复用无菌PBS清洗西林瓶内壁,并将清洗液转移至上述无菌三角瓶内,最终清洗液合计40 mL,即为待测原液。
1.2.2 选择性平板分离
按照食品安全国家标准GB4789.4-2016《食品微生物学检验 沙门氏菌检验》,取25 mL待测原液转至225 mL BPW中预增菌,取1 mL预增菌18 h培养物分别转种至TTB和SC内,于42℃和36℃下分别培养24 h,BPW预增菌液、TTB增菌液及SC增军液分别划线于BS平板、XLD平板和CAS平板,挑取36℃培养24 h。
1.2.3 生化与血清鉴定
根据GB 4789.4-2016《食品微生物学检验 沙门氏菌检验》,以肠炎沙门氏菌CMCC(B)50335作为阳性对照,挑取样品405与样品341选择性平板上的可疑菌落接种至三糖铁斜面琼脂,先在斜面划线,再于底层穿刺,同时接种营养琼脂平板,于36℃培养24 h。根据三糖铁斜面琼脂的反应结果,选取对应营养琼脂平板上的可疑菌落制备菌悬液,接种至沙门氏菌干制生化鉴定试剂盒中,36℃培养18~48 h,观察并记录相应的生化结果。
根据生化结果,对判定为沙门氏菌属的菌落,用A~F沙门氏菌属诊断血清进行血清学鉴定。首先,利用生理盐水与目标培养物混合,排除自凝集反应。对无自凝现象的培养物,以生理盐水为空白对照,以肠炎沙门氏菌CMCC(B)50335为阳性对照,用A~F多价O血清和H因子血清进行进一步的O抗原鉴定和H抗原鉴定,观察血清与目标培养物的凝集现象。最后,根据凝集试验的结果,按照GB 4789.4-2016《食品微生物学检验 沙门氏菌检验》附录B 沙门氏菌属抗原表判定沙门氏菌菌型。
1.2.4 特异性PCR反应鉴定
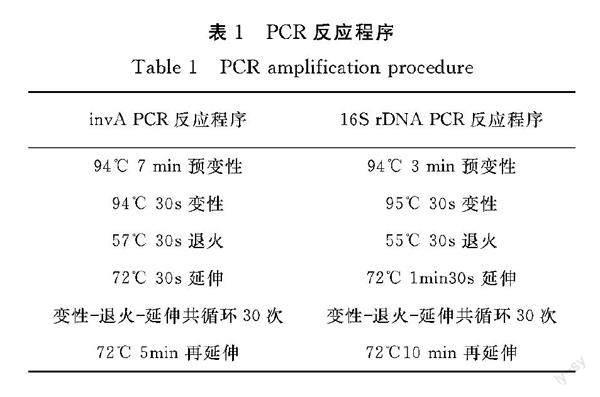
按照细菌基因组提取试剂盒的操作规程对SC增菌液进行DNA提取,得到目的基因后,测定DNA浓度并进行PCR。PCR反应体系(25 μL)为:2X SanTaq PCR Master Mix 12.5 μL,引物F 1 μL,引物R 1 μL,DNA模板 1 μL,ddH2O 9.5 μL。PCR反应程序见表1,其中特异性PCR以沙门氏菌侵袭蛋白A invA基因(F:5′-GTGAAATTATCGCCACGTTCGGGCAA-3′,R:5′-TCATCGCACCGTCAAAGGAACC-3′)作为目的基因[13], PCR产物用于电泳分析,普通PCR以16S rDNA(27F: 5′-AGAGTTTGATCCTGGCTCAG-3′, 1492R: 5′-GGTTACCTTGTTACGACTT-3′)为目的基因,以枯草芽孢杆菌ATCC6633为阴性对照, 肠炎沙门氏菌CMCC(B)50335为阳性对照,对阴性对照、阳性对照、样品405、样品341进行invA基因的特异性扩增。PCR产物送至生工生物工程(上海)股份有限公司进行测序,并将序列上传至NCBI,样品405、样品341和肠炎沙门氏菌CMCC(B)50335的GenBank Accession Number 分别标记为OM666544、OM666543、OM666545。
1.2.5 分子系统发育
将测序后的阳性对照菌株肠炎沙门氏菌CMCC(B)50335、样品405和样品341的16S序列通过BLAST进行初步判断,按照沙门氏菌的分子分型[14],选择亚利桑那亚种Salmonella enterica subsp.arizonae、双亚利桑那亚种Salmonella enterica subsp.diarizonae、肠炎亚种Salmonella enterica subsp.enterica、豪顿亚种Salmonella enterica subsp.houtenae、印度亚种Salmonella enterica subsp.indica、萨拉姆亚种Salmonella enterica subsp.salamae这6个肠炎沙门氏菌亚种和邦戈沙门氏菌Salmonella bongori作为参考序列,选择弗氏柠檬酸杆菌Citrobacter freundii作为外群,进行系统发育分析。利用MEGA v7对目标序列进行比对,构建最大似然树(ML Tree)。
2 结果与分析
2.1 选择性平板分离
沙门氏菌属因不同选择性培养基中成分及浓度的不同,呈现不同的菌落特征[15-16],在BS平板上表现为黑色有金属光泽、棕褐色或灰色菌落,周围培养基黑色或棕色,或为灰绿色菌落,周围培养基不变;在XLD平板上表现为粉红色带或不带黑色中心菌落,或为带大光泽黑色中心或全部黑色菌落,或为黄色带或不带黑色中心菌落[6];在CAS平板上表现为淡紫色菌落,或培养24~48 h后才出现紫色菌落。根据已知沙门氏菌属的菌落特征,挑选样品405和样品341中的可疑菌落,具体见表2。 对比预增菌液(BPW)和增菌液(TTB和SC)的分离结果,发现在不同选择性平板上,两种增菌程度的可疑菌落特征未表现出明显差异。对比同种增菌液在不同选择性平板上的分离结果,发现样品在不同选择性平板上表现出不一样的菌落特征,其中样品405与阳性对照差异较大,但仍可BS与XLD的沙门氏菌菌落描述中找到对应的特征,却与CAS的沙门氏菌菌落特征描述不符,样品341则表现出与阳性对照肠炎沙门氏菌CMCC(B)50335相似的菌落特征。
2.2 生化与血清鉴定
由表3可知,样品405的可疑菌落在三糖铁琼脂斜面和底层均产酸,且赖氨酸脱羧酶试验结果为阴性,可初步判断为非沙门氏菌属,结合靛基质、尿素、氰化钾的生化结果,可判定为非沙门氏菌属。样品341的可疑菌落与阳性对照菌株生化结果一致,在三糖铁琼脂斜面产碱、底层产酸、硫化氢阳性、产气阳性、赖氨酸脱羧酶试验阳性可初步判断样品341中存在可疑沙门氏菌属,靛基质、尿素、氰化钾、β-半乳糖苷酶、丙二酸盐的生化结果表明,基于GB 4789.4-2016《食品微生物学检验 沙门氏菌检验》样品341可判定为沙门氏菌生化群Ⅰ[6],基于第 2 版《伯杰系统细菌学手册》 (2005)样品341可判定为S.enterica subsp.enterica(Ⅰ)[17]。
基于生化试验的结果,对样品341和阳性对照菌株进行进一步的血清学鉴定。由表4可知,样品341和阳性对照菌株在生理盐水下表现为不凝集,表明样品341菌株与阳性对照菌株肠炎沙门氏菌CMCC(B)50335同为非粗糙型菌株。肠炎沙门氏菌CMCC(B)50335 的血清学鉴定结果O抗原为O9,12,H抗原第1相为g,m。样品341在O抗原鉴定试验中,被A~F多价O血清凝集,后续依次用O2、O4、O5等多种因子血清做凝集试验,发现样品341被O9和O4,12因子血清凝集,且不被O4因子血清凝集,查GB 4789.4-2016附录B可知,样品341的O抗原可判定为D群。在H抗原的鉴定试验中,利用单因子血清进行逐一验证,结果表明样品341被Hg和Hm因子血清凝集。综上可知,样品341的血清分型为O:9,12:H:g,m,血清鉴定结果与肠炎沙门氏菌CMCC(B)50335一致。因此样品341中检出沙门氏菌,且血清型为O:9,12:H:g,m,是肠炎沙门氏菌S.enterica serovar enteritidis。
2.3 特异性PCR反应鉴定
invA基因是沙门氏菌编码吸附和侵染上皮细胞表面蛋白的毒力基因,是沙门氏菌的侵染基础[7]。因其在血清型分类中的广谱性和沙门氏菌特异性,已被设计成沙门氏菌的特异性引物广泛应用于沙门氏菌的快检中[18]。基于invA基因的特异性PCR电泳结果见图1,阳性对照菌株肠炎沙门氏菌CMCC(B)50335与样品341表现出invA基因特异性,约280 bp处出现明显的目的条带,与报道中invA基因的大小相符[13],样品405扩增出3条模糊的条带,大小分别约为2 000、400、250 bp,明显不具备invA基因特异性,阴性对照菌株枯草芽孢杆菌ATCC6633未出现条带。由此可得,样品341与阳性对照菌株肠炎沙门氏菌CMCC(B)50335的特异性PCR结果一致,表明样品305检出沙门氏菌。
2.4 分子系统发育
由图2可知,样品405區别于沙门氏菌的7个亚种,与弗氏柠檬酸杆菌聚类在同一分支,因此,样品405应为沙门氏菌阴性。样品341和阳性对照菌株肠炎沙门氏菌CMCC(B)50335,与沙门氏菌的7个亚种聚类在同一大分支中,且对应的分支嵌在沙门氏菌7个亚种中拓扑结构中,由此可知样品341检出沙门氏菌阳性。此外,样品341聚类在Salmonella enterica subsp.enterica这一小分支中,且其分支嵌于Salmonella enterica subsp.enterica的拓扑结构中,因此样品341中沙门氏菌的分子分型为肠炎沙门氏菌肠炎亚种。
3 讨论与结论
常规分离培养作为易普及、易操作的传统基础方法仍在微生物检测领域,尤其是食品沙门氏菌常规检测中发挥重要作用[19]。在本研究中,选择性平板分离阶段的不同增菌程度菌液表现出一致的可疑菌落特征,但在日常检测中,BPW预增菌液直接划线于选择性平板中,存在杂菌蔓延速度过快,阳性菌落因不具空间竞争与营养竞争优势,导致阳性菌落瘦小不典型等情况,容易出现漏检[20]。另外,对比所用的BS、XLD与CAS 3种选择性培养基,发现CAS在分离鉴别时,受干扰菌影响较小,能更直观区分杂菌,这与研究报道中的描述相符[19-21]。CAS培养基作用原理主要是依于沙门氏菌特有的辛酯酶反应,是在生化反应的基础上开发的新技术,具有较高的灵敏性和特异性,目前在沙门氏菌的快速检测中已得到广泛应用[21-23],但是CAS同样无法做到鉴别所有沙门氏菌,因此建议选用CAS与多种选择性培养基的组合模式[19,21],并且对选择性培养基中的可疑菌落进行后续生化与血清鉴定,可提高检出率,以防漏检[24]。
生化鉴定技术主要是根据不同种属微生物酶系统、代谢途径和代谢产物的差异性,利用代谢产物的不同生化特征进而鉴定微生物种类。沙门氏菌属最早就是通过生化反应实现分类鉴定[1],第 2 版《伯杰系统细菌学手册》 (2005) 采用了目前国际认可的沙门氏菌分类方案,将沙门氏菌属分为了邦戈尔沙门菌、肠炎沙门菌(包括6个亚种)两个种。GB 4789.4-2016《食品微生物学检验 沙门氏菌检验》中依据沙门氏菌的生化反应分类方案,将沙门氏菌属划分成6个生化群,虽与主流分类方案相似,但无法完全等同。而血清分型对沙门氏菌属的分类更为细化,已多达2 600多个,其中绝大部分属于肠炎沙门氏菌[25]。本研究中,分离得到的沙门氏菌正是肠炎沙门氏菌,根据国标归属于沙门氏菌生化群I,根据《伯杰系统细菌学手册》归属于肠炎沙门氏菌肠炎亚种(I),血清型为O:9, 12:H:g,m,是较为常见的沙门氏菌株。在日常检测中,生化鉴定是必做项目,而血清学鉴定属于选做项目[6],但是若为了进一步研究沙门氏菌致病性及传染源的追踪、溯源等,血清型鉴定是不可或缺的一项[26]。
目前,对于沙门氏菌鉴定的分子技术已多有研究报道,沙门氏菌的特异性PCR是通过成功扩增沙门氏菌基因组中的特异性基因片段,实现沙门氏菌的检测,其中inva基因是沙门氏菌中编码入侵蛋白的基因,在沙门氏菌鉴定中得到广泛应用[7,12-13],PCR体系及程序已相对成熟,在本研究中也得到成功验证,在阳性对照和阳性样本中成功检出沙门氏菌,在阴性对照和阴性样本中未检出沙门氏菌。16S rDNA是细菌鉴定的通用引物,本研究中将其应用于沙门氏菌的检测中,构建的系统发育树不仅成功确定样本沙门氏菌的是否检出,而且明确参试菌种的分类,表明本研究中的沙门氏菌为肠炎沙门氏菌肠炎亚种,作为阴性样品的干扰菌为弗氏柠檬酸杆菌。与常规生化血清鉴定相比,分子方法具有简单、快速等优点,在快检技术的开发应用具有巨大的优势[7],但GB 4789.4-2016《食品微生物学检验 沙门氏菌检验》中并未涉及,因此分子方法可作为传统标准检测方法的补充。
采用传统标准检测方法、特异性PCR与分子系统发育3种不同方法对能力验证(CFAPA-805)样品进行检测,检测结果一致,样品341中检出沙门氏菌,能力验证结果为“满意”,验证了实验室沙门氏菌的检测能力。比较发现,选择性培养可以确定是否存在可疑沙门氏菌,特异性PCR可以确定沙门氏菌是否阳性检出,可应用于沙门氏菌快速检测,是传统标准检测方法的补充;生化鉴定、血清学鉴定和分子系统发育鉴定可以确认沙门氏菌的生化群为沙门氏菌生化群(Ⅰ),血清分型为O:9,12:H:g,m,分子分型为肠炎沙门氏菌肠炎亚种,适用于沙门氏菌具体鉴定分型,可应用于沙门氏菌的致病性、流行病溯源。
参考文献:
[1]毕旺来,赵巍薇,马达,等.全基因组测序和real-time PCR法检测食源性沙门氏菌parC、gyrA基因突变特征[J].食品科学,2022,43(12):296-302.
[2]曾瑜,谌委菊,全珂,等.基于脱氧核酶的食品安全快速检测方法研究进展[J].食品与机械,2022,38(6):205-212.
[3]ZHANG Y-N,BUTLER F.Risk assessment of pork products produced in China.Biosyst[J].Food Eng Res Rev,2018(23):48-51.
[4]李傲,賈波,刘枫,等.沙门氏菌分析检测方法研究进展[J].安徽农业科学,2022,50(2):5-8,18.
[5]章志超,鄢雷娜,匡佩琳.一株沙门氏菌Ⅲb60:r:e,n,x,z15的分离、鉴定及其检测方法比较[J].食品研究与开发,2020,41(21):192-198.
[6]国家卫生和计划生育委员会.食品安全国家标准食品微生物学检验沙门氏菌检验:GB 4789.4-2016[S].北京:中国标准出版社.2016:1-20.
[7]吕新,刘兰英,陈丽华,等.可视化环介导恒温扩增法检测蔬菜产地环境灌溉水源中沙门氏菌[J].食品安全质量检测学报,2021,12(2):492-498.
[8]RIVERA W L,PAVON R.Molecular Serotyping by Phylogenetic Analyses of a 1498bp Segment of the invA Gene of Salmonella[J].ASM Science Journal,2021,14:1-14.
[9]蒋玥,衣服德,许华,等.4种血清型沙门氏菌多重PCR检测方法建立及应用[J].江苏农业科学,2021,49(13):59-63.
[10]苏远科,王志宏,郭晓燕,等.16SrRNA序列分析在沙门氏菌鉴定分型中的应用[J].食品安全质量检测学报,2017,8(11):4153-4160.
[11]孟令缘,牛沁雅,廉鲁昕,等.基于16S rDNA序列,MALDI-TOF-MS和VITEK的沙门氏菌和金黄色葡萄球菌的鉴定[J].中国食品学报,2021,21(10):197-205.
[12]颜卫,赵莎莎,丁丽军,等.江苏地区沙门氏菌流行现状调查及耐药性分析[J].江苏农业科学,2019,47(2):157-159.
[13]谭燕,肖紫鸣,李小林,等.多重PCR技术检测食品中鼠伤寒沙门氏菌3种毒力基因[J].食品研究与开发,2021,42(12):171-176.
[14]刘素可,张彪,路娟娥,等.沙门氏菌在食品中的生存策略及其防控的研究进展[J].食品科学,2022,43(13):218-226.
[15]SON K,MENOLASCINA F,STOCKER R.Speed-dependent chemotactic precision in marine bacteria[J].PNAS,2016,113(31):8624-8629.
[16]BAEZ A,KUMAR A,SHARMA A K,et al.Effect of amino acids on transcription and translation of key genes in E.coli K and B grown at a steady state in minimal medium[J].New Biotechnol,2019,49:120-128.
[17]苏章庭,曾晓琮,刘秋婷,等.不同食品标准中沙门氏菌分类方案比较[J].检验检疫学刊, 2021(031-002).
[18]HEYMANS R,VILA A,HEERWAARDEN C V,et al.Rapid detection and differentiation of Salmonella species,Salmonella Typhimurium and Salmonella Enteritidis by multiplex quantitative PCR[J].PLOS ONE,2018,13(10).
[19] 刘玥,刘夏,崔琳,等.食品沙门氏菌现行国内外标准中选择性增菌和分离的等效性评估[J].中国食品卫生杂志,2022,34(3):474-481.
[20]张亓,李苨.不同增菌和培养条件下沙门氏菌检出效果的比较[J].工业微生物,2019,49(1):50-54.
[21]陈茵茵,钟卫烨,韩志杰,等.沙门氏菌在不同品牌显色培养基上的对比分析[J].食品安全质量检测学报,2020,11(16):5455-5460.
[22]王萍,董贵军,乔勇升,等.显色培养基上沙门氏菌及干扰菌的分离鉴定[J].食品研究与开发,2017,38(12):158-161.
[23]PERRY J D.A decade of development of chromogenic culture media for clinical microbiology in an era of molecular diagnostics[J].Clin Microbio Rev,2017,30(2):449-479.
[24]程浩.食品中沙门氏菌检测方法比较[J].安徽农业科学,2018,46(30):202-204.
[25]RITTER A C,TONDO E C,SIQUEIRA F M,et al.Genome analysis reveals insights into high-resistance and virulence of Salmonella Enteritidis involved in foodborne outbreaks[J].Int J Food Microbiol,2019,306:108269.
[26]郑林,祝令偉,郭学军,等.沙门氏菌主要流行血清型耐药性的研究进展[J].江苏农业科学,2020, 48(6):8-12.
(责任编辑:柯文辉)

