图自动编码器上二阶段融合实现的环状RNA-疾病关联预测
张 奕,王真梅
(1.桂林理工大学 信息科学与工程学院,广西 桂林 541006;2.广西嵌入式技术与智能系统重点实验室(桂林理工大学),广西 桂林 541006)
0 引言
环状RNA(circular RNA,circRNA)是一种新型的单链非编码RNA 分子,与传统RNA 不同,circRNA 具有封闭的环状结构,以各种形式广泛存在于真核细胞中,在调节微小RNA(microRNA,miRNA)介导的基因表达方面扮演着十分重要的角色[1-2]。现在越来越多的研究者通过研究circRNA 在疾病发生中所起到的作用和作用机制,探讨它作为生物标志物的诊断价值和在疾病治疗中的作用[3-4]。因此,与疾病相关的circRNA 被认为是一种新的疾病诊断和治疗的生物标志物,系统地了解circRNA 与疾病的关联关系是生物信息学研究的一个重要内容,有利于疾病的诊断、治疗和预后,是未来研究的新途径[5]。
现有的预测circRNA-疾病关联关系的方法可分为基于网络传播、基于机器学习和基于深度学习这3 大类。
1)基于网络传播的方法。
通过构建基于已知circRNA-疾病关联、circRNA 相似性和疾病相似性的异质网络预测circRNA-疾病关联。Fan 等[5]在circRNA 表达谱、疾病表型相似性和已知的circRNA-疾病关联构建的异质网络上,提出一种基于KATZ 测度的人类circRNA-疾病关联预测模型(computational model of KATZ measures for Human CircRNA-Disease Association,KATZHCDA)。该模型对异质网络采用简单的度量方法就能成功地预测出circRNA-疾病关联关系;但不适用于预测没有任何已知circRNA 关联的新疾病或没有任何已知疾病关联的孤立circRNA。Xiao 等[6]开发了一种基于图形多标签学习的线上方法,利用circRNA 空间和疾病空间的不同特征保证了数据的局部几何结构,将图的正则化和混合规范约束条件纳入模型,减少了训练的时间和成本;但circRNA(疾病)相似性计算仍然严重依赖已知的circRNA-疾病关联信息,预测精度不高。Lei 等[7]利用circRNA(疾病)相似性和已知circRNA-疾病关联构成的异质网络计算路径加权方法预测circRNA-疾病的关联。该模型结合circRNA 功能相似性分数、疾病语义相似性分数和高斯相互作用谱内核相似性分数,填充疾病和circRNA 相似性网络的稀疏值,并且只使用了3 步内的路径达到了减少噪声信息的效果;但该模型对数据稀疏的矩阵预测效果仍不理想。
2)基于机器学习的方法。
利用circRNA(疾病)相似性和已知circRNA-疾病关联构建circRNA(疾病)特征,并设计分类器识别与疾病相关的circRNA。Lei 等[8]提出一种计算方法,将重启随机游走算法应用于具有全局网络拓扑信息的加权特征,并采用K最近邻(K-Nearest Neighbors,KNN)算法根据特征进行分类提高预测性能;但该方法在揭示疾病和没有任何关联的新circRNA或circRNA 和没有任何关联的新疾病之间的关联关系方法略有不足。Yan 等[9]采用基于Kronecker 积的正则化最小二乘法实现潜在的circRNA-疾病关联预测;但该模型没有考虑其他相关的生物数据信息(如circRNA-miRNA 关联和序列信息),预测精度不高。Ding 等[10]开发了一个基于随机游走和逻辑回归的计算模型,重启随机游走方法可以得到每个circRNA 的全局关系信息,比仅使用基于相似性的方法性能更好;但该模型无法预测与circRNA 没有任何关联的新疾病或与疾病没有任何关联的新circRNA。
3)基于深度学习的方法。
通过整合circRNA、疾病和miRNA 等多源生物信息数据构建复杂的circRNA-疾病关联异构网络图,提取circRNA 和疾病的非线性特征,实现circRNA-疾病关联预测。Wang等[11]提出了一种融合多源生物信息的深度卷积神经网络计算方法,利用空间关系自动提取circRNA-疾病描述的深层特征,极端学习机具有快速训练和良好的归一化性能的优势,能快速准确地预测潜在的circRNA-疾病关联关系;但该方法没有优化数据,可能含有更多的噪声信息,会造成实验结果一定的偏差。Fan 等[12]融合circRNA、miRNA 和疾病之间的多种相似性和相互作用特征,构建双层卷积神经网络,有效引入了circRNA 和疾病的相似性构建circRNA(疾病)的拓扑特征;但没有考虑更多可靠的生物信息,导致预测精度不高。Li 等[13]利用图注意网络学习circRNA(疾病)的潜在表示,图卷积网络提取非线性特征,解决了传统方法中成本高和耗时长的难题;但忽略了线性特征的重要性。Deepthi 等[14]提出了一种集合方法,整合circRNA(疾病)相似性构建特征,利用深度自动编码器提取隐藏的生物模式,用随机森林分类器进行训练;但该方法需要负样本训练模型。Chen 等[15]使用矩阵补全方法填充矩阵中缺失值以达到预测良好的效果;但未能低成本快速地寻找最优参数。Lu 等[16]提出一种基于深度矩阵分解方法的circRNA-疾病关联(Deep Matrix Factorization for CircRNA-Disease Association,DMFCDA)预测。该方法利用多层神经网络捕捉非线性特征,掌握数据的复杂结构。DMFCDA 加入注意力机制,关注重要信息,忽略或减少其他信息,可以提高预测性能。Li 等[17]提出一种基于加速归纳式矩阵补全的circRNA-疾病关联(Speedup Inductive Matrix Completion for CircRNA-Disease Associations,SIMCCDA)预测模型,将circRNA-疾病关联转化为推荐系统问题,并应用归纳式矩阵补全算法预测潜在的circRNA-疾病关联关系,不仅取得了良好的预测效果,而且节省了内存和降低了训练时间成本;但SIMCCDA 不能应用于没有任何关联的新疾病或孤立circRNA 的预测。
针对以上3 类方法的固有缺陷,本文在图自动编码器的基础上,融合归纳式矩阵补全和自注意力机制对预测精度进行二阶段提高,提出新型circRNA-疾病关联预测模型——GIS-CDA(Graph auto-encoder combining Inductive matrix complementation and Self-attention mechanism for predicting CircRNA-Disease Association)模型。GIS-CDA 模型的建立过程如图1 所示,分为以下3 个步骤:

图1 GIS-CDA模型建立过程Fig.1 Construction process of GISCDA model
1)整合多源生物信息数据,包括已知circRNA-疾病关联、疾病语义相似性、circRNA 功能相似性和circRNA(疾病)高斯相互作用谱内核相似性。
2)图自动编码器对circRNA 和疾病相似性进行编码和解码。学习circRNA(疾病)潜在特征,获得circRNA(疾病)的低维表征。将学习到的特征输入归纳式矩阵补全,生成关联矩阵,提高节点之间的相似性和依赖性。模型训练后得到circRNA(疾病)特征矩阵,将两者整合得到circRNA-疾病特征矩阵,增强预测的稳定性和准确性。
3)将第2)步得到的circRNA 特征矩阵、疾病特征矩阵和circRNA-疾病特征矩阵作为数据输入,分别引入自注意力机制,提取重要特征,减少对其他生物信息的依赖。
1 模型建立
1.1 相似性网络建立
1.1.1 疾病语义相似性
疾病语义信息因其有效性和稳定性被广泛用于疾病相似性的度量[14]。本文从MeSH(Medical Subject Headings)数据库[9]获取每种疾病的相关注释词,利用有向无环图(Directed Acyclic Graph,DAG)[9]计算疾病语义相似性。任意疾病dt对疾病di的语义贡献值用(dt)表示,计算如式(1)所示:
其中σ表示语义贡献的衰减系数,根据Wang 等[18]提出的计算疾病语义相似性计算方法,σ取最优值0.5。
在DAG 中拥有更多共同部分的疾病具有更高的语义相似性[9]。矩阵DS∈Rnd×nd表示疾病语义相似性矩阵(nd表示疾病数),矩阵元素DS(di,dj)表示疾病di与dj之间的疾病语义相似性,计算如式(2)所示:
其中:T(di)表示疾病di的DAG;D(di)表示疾病di的语义值,计算如式(3)所示:
1.1.2 circRNA功能相似性
集合D={d1,d2,…,dnd}表示疾病集,max(dt,D)表示任意疾病dt在疾病集D中语义相似性的最大值,计算如式(4)所示:
功能相似的circRNA 往往与相似的疾病相关[14]。根据此假设,结合Chen 等[15]提出的miRNA 功能相似性计算方法,获 得circRNA 功能相 似性矩 阵CS∈Rnc×nc(nc表 示circRNA 数),矩阵元素CS(ci,cj)表示circRNAci与cj之间的功能相似性,计算如式(5)所示:
其中,集合Di表示与circRNAci有关联的疾病集;集合Dj表示与circRNAcj有关联的疾病集;|Di|和|Dj|分别表示集合Di和Dj中疾病的数量。
1.1.3 高斯相互作用谱内核相似性
矩阵A∈Rnc×nd表示已知的circRNA-疾病关联。如果circRNAci与疾病dj存在经实验验证的已知关联,则定义矩阵元素A(ci,dj)=1;如果任何circRNAci与疾病dj不存在经实验验证的已知关联,则定义矩阵元素A(ci,dj)=0。由于circRNA 与疾病的已知关联数量少,导致已知关联矩阵中存在大量的缺失值,呈现出固有的稀疏性。本文引入circRNA与疾病的高斯相互作用谱内核相似性填充缺失值[14]。矩阵CK∈Rnc×nc表示circRNA 高斯相互作用谱内核相似性,矩阵元素CK(ci,cj)表示circRNAci与cj的高斯相互作用谱内核相似性,计算如式(6)所示:
其中μc表示高斯相互作用谱内核相似性的控制内核带宽,控制CK(ci,cj)的大小,计算如式(7)所示:
同理,矩阵DK∈Rnd×nd表示疾病高斯相互作用谱内核相似性,矩阵元素DK(di,dj)表示疾病di与疾病dj的高斯相互作用谱内核相似性,计算如式(8)所示:
其中μd表示高斯相互作用谱内核相似性的控制内核带宽,控制DK(di,dj)的大小,计算如式(9)所示:
1.1.4 集成相似性
如前所述,本文已经获得了疾病语义相似性、circRNA 功能相似性、circRNA(疾病)高斯相互作用谱内核相似性。考虑到疾病语义相似性和circRNA 功能相似性固有存在的稀疏性,通过整合来自多个数据源的互补信息和不同的表示方法,采用集成相似性量化每对circRNA(疾病)相似性克服固有稀疏性。circRNA 集成相似性由矩阵Xc∈Rnc×nc表示,矩阵元素Xc(ci,cj)的计算如式(10)所示:
同理,矩阵Xd∈Rnd×nd表示疾病集成相似性,矩阵元素Xd(di,dj)的计算如式(11)所示:
1.2 方法概述
1.2.1 图自动编码器
Li 等[19]利用图自动编码器获得低维表示,可以提高模型的预测精度。为此,GIS-CDA 采用图自动编码器从图的节点中提取嵌入向量,学习潜在特征,重构原始输入数据,获取低维表示以达到提高预测精度的目的。
利用circRNA 集成相似性矩阵Xc和疾病集成相似性矩阵Xd,构建circRNA 特征向量c=[c1,c2,…,cnc]和疾病特征向量d=[d1,d2,…,dnd]。使用两层图卷积神经网络将关联矩阵A分别与circRNA 和疾病的特征向量相结合,得到相应的低维表示。
将Xc、Xd作为编码器[19]的输入,使用tanh 函数增加模型对非线性数据的处理能力并进行归一化。编码器对Xc编码,得到低维表示Zc,计算如式(12)(13)所示:
同理,编码器对Xd编码,得到低维表示Zd,计算如式(14)(15)所示:
由于circRNA(疾病)特征向量涵盖了结构信息,本文采用解码器[19]识别潜在的circRNA-疾病关联关系。解码器采用sigmoid 激活函数器[19],使平均激活程度小于1。矩阵Fc∈Rnc×nd表示解码器对Zc解码得到的circRNA 特征矩阵,计算如式(16)所示:
同理,矩阵Fd∈Rnd×nc表示解码器对Zd解码得到的疾病特征矩阵,计算如式(17)所示:
1.2.2 归纳式矩阵补全
GIS-CDA 借鉴归纳式矩阵补全的方法[15],利用上一步推导得到的疾病特征矩阵Fd和circRNA 特征矩阵Fc,重建circRNA-疾病关联矩阵,填补已知关联矩阵A中的缺失值。补全后的关联矩阵Q∈Rnc×nd的计算如式(18)所示:
GIS-CDA 采用最小化损失函数训练参数,以达到优化损失函数的目的[20],在没有过拟合的情况下降低损失,提高优化效率。模型优化过程如式(19)所示:
其中:L表示损失函数;λ表示平衡因子,取值为10-8,W∈Rnc×nd表示权 重矩阵;‖ ‖·F表示矩阵的弗罗贝尼乌斯范数。
1.2.3 自注意机制
图注意力网络通过注意力机制学习图上节点的表示,为不同的邻居节点分配不同的学习权重,使节点特征之间的相关性更好地整合到模型中,取得了较好的预测性能[21]。为此,GIS-CDA 引入自注意力机制,通过矩阵的秩不等式提高预测精度[22],计算如式(20)所示:
其中:rank(*)表示矩阵的秩;α∈(0,1)为Fc和Fd之间的平衡系数,α取值为0.5。
由此,将Fc和Fd整合为最终的circRNA-疾病特征矩阵F∈Rnc×nd,计算如式(21)所示:
将推导得到的矩阵Fc、Fd和F作为数据输入,令Fc、Fd和F分别等价于自注意力机制中的查询、关键字和值。通过点乘方式计算Fc和Fd中两个向量之间的相似性,softmax 优化后,再和F中每个向量点乘得到对应的自注意力层的输出向量,用矩阵E∈Rnc×nd表示,计算如式(22)所示:
其中d表示隐藏层的向量维度,值设为256。
由此,将式(19)更改写为式(23):
在文献[22]的基础上加入重建误差,计算如式(24)所示:
综上,本文模型的总损失函数如式(25)所示:
其中:β表示平衡因子,为方便计算,将β的值设为1;λ的值设为10-8。
2 实验与结果分析
2.1 实验数据集和环境
从circR2Disease 数据库[23]中获取经实验验证的739 个circRNA-疾病已知关联关系(涉及661 个circRNA 与100 种疾病)。删除冗余数据后,只挑选与人类复杂疾病相关的650个已知关联数据(涉及585 个circRNA 与88 种疾病)作为已知关联矩阵。所有实验均在AMD 1.80 GHz CPU 和Windows 10 操作系统上完成。
2.2 评估指标
由于已知circRNA-疾病关联数量远小于未知的关联数量,从所有未经实验验证的65 361 个circRNA-疾病关联对中随机挑选650 个circRNA-疾病关联对作为负样本,circRNA-疾病关联数据中的650 个已知关联作为正样本。为减少随机样本分区的变化,采用了10 次重复的五折和十折交叉验证的平均接收者操作特征曲线下面积(Area Under Receiver Operating Characteristic curve,AUROC)、精确率-召回率曲线下面积(Area Under Precision-Recall curve,AUPR)作为评估指标评估GIS-CDA 预测性能。其中,AUROC 是以假正例率(False Positive Rate)为横坐标、真正例率(True Positive Rate)为纵坐标的接收者操作特征(Receiver Operating Characteristic,ROC)曲线下面积,AUPR 是以召回率(Recall)为横坐标、精确率(Precision)为纵坐标的精确率-召回率(Precision-Recall,PR)曲线下面积。五折和十折交叉验证后,得到GIS-CDA 的ROC 曲线和PR 曲线,如图2 所示。

图2 GIS-CDA交叉验证结果Fig.2 Cross validation results of GIS-CDA
由图2 结果可知,GIS-CDA 模型在十折交叉验证的AUROC、AUPR 值比五折交叉验证分别高出了0.75、4.47 个百分点,表明GIS-CDA 模型使用十折交叉验证利用了更多的训练数据,可以准确地评估GIS-CDA 模型在circRNA-疾病关联预测性能。
2.3 参数选择影响
对参数的分析可以定量地评估模型的稳定性[20]。本节分析Fc和Fd之间的平衡系数α、学习率l和隐藏层维度d这3 个参数值的选择对GIS-CDA 预测性能的影响。
2.3.1 隐藏层维度选择
根据文献[22]取固定学习率l为0.01,Fc和Fd之间的平衡系数α为0.5,分析隐藏层维度d对GIS-CDA 性能的影响。针对每个d∈{32,64,128,256,512}进行五折交叉验证后,得到相应的AUROC 值和AUPR 值,如表1 所示。

表1 不同隐藏层维度d下的AUROC、AUPR值Tab.1 AUROC、AUPR values with different hidden layer dimension d
隐藏层维度越高,误差越小,但是会增加模型复杂度,也可能会出现过拟合现象。由表1 可知,当维度从32 增加到256,GIS-CDA 的性能随之提升;当维度为512 时,AUROC 值和AUPR 值分别比维度256 的低1.87 和4.23 个百分点。当维度为256 时,模型的AUROC 值和AUPR 值最大。因此,本文将隐藏层维度d设定为256,既保证了模型预测性能,又节省了时间与空间成本。
2.3.2 平衡系数选择
固定l为0.01,d为256,针对每个Fc和Fd之间的平衡系数α∈{0.1,0.3,0.5,0.7,0.9}进行五折交叉验证后,得到相应的AUROC 值和AUPR 值,如表2 所示。

表2 不同平衡系数α下的AUROC、AUPR值Tab.2 AUROC、AUPR values with different balance coefficient α
Fc和Fd之间的平衡系数α不仅决定融合后circRNA-疾病特征矩阵的稀疏性,也关系到节点之间的相关性是否更好地整合到模型中。由表2 可知,当α=0.5 时,模型的AUROC 值和AUPR 值最大。
2.3.3 学习率选择
固定d为256,α为0.5,改变学习率l为常用的数值。针对每个l∈{0.001,0.005,0.01,0.05,0.1}进行五折交叉验证后,得到相应的AUROC 值和AUPR 值,如表3 所示。

表3 不同学习率l下的AUROC、AUPR值Tab.3 AUROC、AUPR values with different learning rate l
学习率l控制模型学习的速度。由表3 可知,当l为0.01时,AUROC 值和AUPR 值最大。
通过对3 种参数值的选择分析可知,当α为0.5,l为0.01 和d为256 时,GIS-CDA 可获得最佳的预测性能(即AUROC 值为0.930 3,AUPR 值为0.227 1)。
2.4 消融实验
为了验证引入的自注意力机制和重建的损失函数对模型GIS-CDA 的预测精度二次提高的效果,本文设置了3 组对比实验,具体如表4 所示。

表4 消融实验对比设置Tab.4 Comparison setting of ablation experiment
经五折交叉验证后,得到3 组对比实验的ROC 曲线和PR 曲线,如图3 所示。
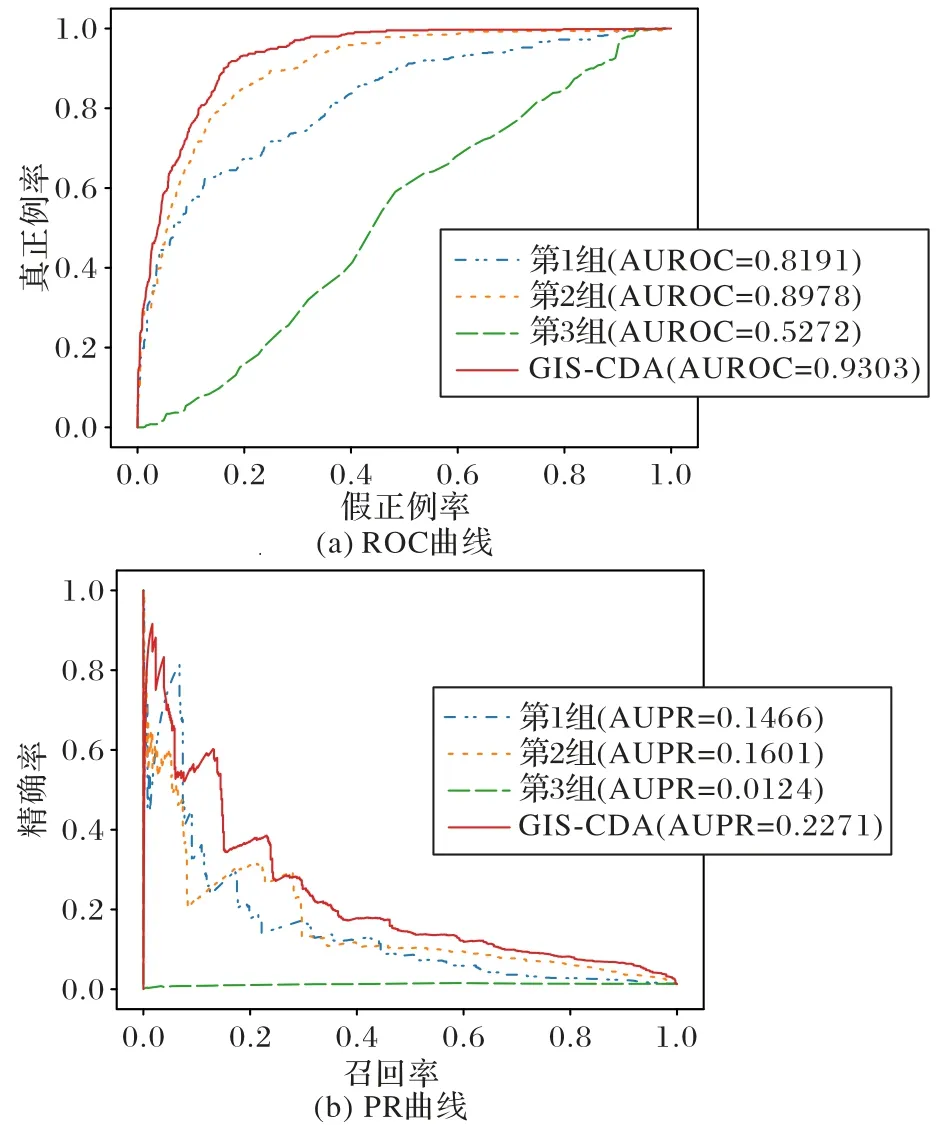
图3 消融实验结果对比Fig.3 Comparison of ablation experimental results
由图3 可知,无论是AUROC 值还是AUPR 值,GIS-CDA均优于3 组对照实验,其中:第2 组的AUROC 值比第3 组高37.06 个百分点,AUPR 值高14.77 个百分点,说明加入重建损失函数,减小了误差,提高了模型优化性能;第1 组的AUROC 值比第3 组 高29.19 个 百分点,AUPR 值 高13.42 个百分点,说明加入了自注意力机制可以减少对其他信息的依赖,对融合circRNA-疾病有较大的帮助;GIS-CDA 的AUROC值比第1、2 组分别高了11.12 和3.25 个百分点,AUPR 值分别高8.05 和6.7 个百分点,说明结合自注意力机制和重建损失函数,可以获取更多的有用信息,进而提高模型预测精度。综上表明,引入自注意力机制和重建损失函数对GIS-CDA 预测精度的提高是至关重要的。
2.5 不同数据集对比
为了进一步验证GIS-CDA 模型的预测性能,本文在circRNADisease[24]、circ2Disease[25]和circR2Disease[23]这3 个数据集上进行了拓展实验。删除冗余数据后,只挑选与人类复杂疾病相关的已知关联数据用于circRNA-疾病潜在关联预测,详细数据如表5 所示。

表5 不同数据集数据细节Tab.5 Data details of different datasets
五折交叉验证后,得到3 个数据集的ROC 曲线和PR 曲线,如图4 所示。

图4 不同数据集预测结果对比Fig.4 Comparison of prediction results on different datasets
由表5 可知,circR2Disease 的疾病数、circRNA 数和关联数均比circRNADisease 和circ2Disease 多一倍以上。由图4可知,GIS-CDA 在circ2Disease、circRNADisease、circR2Disease数据集上取得的AUROC 值分别是0.843 0、0.848 9、0.930 3,AUPR 值分别是0.159 4、0.163 6、0.227 1。实验结果表明,GIS-CDA 在不同数据集上AUROC 值和AUPR 值均取得了0.840 0 和0.150 0 以上的预测结果,说明模型具有鲁棒性,可适用于不同尺度的数据。
2.6 与现有方法对比
在相同的数据集条件下,以AUROC 和AUPR 作为评价指 标,将GIS-CDA 与KATZHCDA[5]、DMFCDA[16]、RWR(Random Walk with Restart)[26]和SIMCCDA[17]这4 个先 进模型进行性能对比,涉及到的参数均使用各自模型推荐的最优参数。五折交叉验证后的ROC 曲线和PR 曲线如图5 所示,AUROC 值、AUPR 值和运行时间如表6 所示。
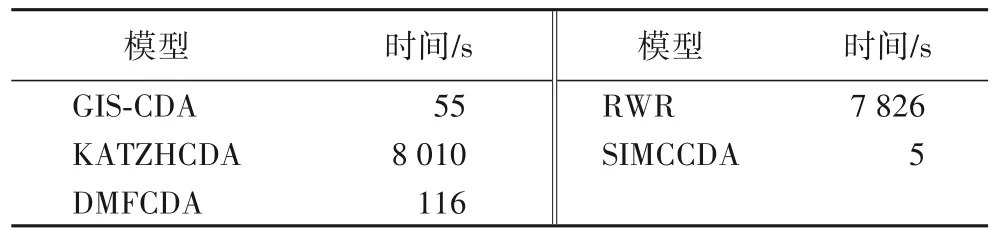
表6 所提模型与现有模型的AUROC值、AUPR值和运行时间对比Tab.6 Comparison of AUROC values,AUPR values and running time of the proposed model and existing models
运行时间方面,GIS-CDA 仅高于SIMCCDA,但GIS-CDA的AUROC 值和AUPR 值分别比SIMCCDA 的高5.01 和13.86个百分 点。此 外,GIS-CDA 的AUROC 值、AUPR 值优于KATZHCDA、DMFCDA 和RWR,其中AUROC 值分别高出了13.19、35.73 和13.28 个百分点,AUPR 值分别高出21.72、22.43 和21.96 个百分点。由此,从AUROC 值、AUPR 值和运行时间这3 方面可以得出,GIS-CDA 模型的预测性能更优。
3 案例分析
为了验证GIS-CDA 模型在真实案例中的预测效果,本文分别对神经胶质癌[27]和胃癌[28]进行了案例研究。经计算预测与此两种疾病相关的circRNA,将得到的关联预测得分按降序排列后,分别筛选排名前10 位的circRNA,如表7、8所示。

表7 前10个与神经胶质癌相关的circRNATab.7 Top 10 circRNAs associated with glioma

表8 前10个与胃癌相关的circRNATab.8 Top 10 circRNAs associated with gastric cancer
表7、8 通过在PMID 数据库中搜索相关文献和报告,得到了GIS-CDA 模型的预测结果。表中数据显示,在GIS-CDA预测的前10 位与神经胶质癌、胃癌相关的circRNA 中,各有7、8 个已被文献证实。其中:circPVT1 通过上调miR-199a-5p抑制胶质癌的生长和转移[29],circHIPK3 通过miR-524-5p/KIF2A 介导的PI3K/AKT 途径促进神经胶质癌的转移和凋亡[30];CirITCH 通过Wnt/β-catenin 途径封存miR-17 预防胃癌的发生,是胃癌的预后标志物[31];circCCDC66 通过靶向miR-618/BCL2 轴克服胃癌细胞对顺铂的耐药性[32]。案例分析进一步验证了GIS-CDA 具有识别与疾病关联circRNA 的良好性能,具有一定的应用价值。
4 结语
本文在图自动编码器基础上,引入归纳式矩阵补全与自注意力机制,通过二阶段融合实现circRNA-疾病关联预测。GIS-CDA 模型克服了现有模型训练时间长、预测准确度不高的缺陷,具有较好的预测性能。今后将整合更多的生物数据集来进一步提升模型的预测性能。

