基于胶囊网络的青少年特发性脊柱侧弯智能分型研究
段文玉 何越 杜钦红 杜钰堃 西永明 杨环
摘要:针对青少年特发性脊柱侧弯疾病的影像诊断问题,基于胶囊网络设计一种智能辅助医生诊断的脊柱侧弯分型算法,直接通过脊柱X光影像给出脊柱侧弯分型结果。该算法在胶囊网络使用共享注意力模块关注脊柱区域多尺度的特征信息,利用胶囊解码结构产生一致性的胶囊向量,使用动态路由算法共同激活脊柱畸变区域的胶囊向量完成脊柱侧弯分型的预测。实验结果表明,本算法的准确率、F1分数、G-mean等指标均优于Vgg、ResNet、EfficientNetV2等卷积神经网络分类方法。
关键词:青少年特发性脊柱侧弯;胶囊网络;注意力模块
中图分类号:TP391.41
文献标志码:A
文章编号:1006-1037(2023)02-0043-07
doi:10.3969/j.issn.1006-1037.2023.02.08
基金项目:
山东省泰山学者项目(批准号: ts20190985)资助
通信作者:
杨环,女,博士,副教授,主要研究方向为图像/视频处理与分析、视觉感知建模及质量评估、深度学习等。
青少年特发性脊柱侧弯(Adolescent Idiopathic Scoliosis,AIS)是青少年人群中最常见疾病之一,在中国的发病率为2%~5.2%[1]。AIS不仅会影响青少年的生长发育,还会造成患者运动失衡、心肺功能下降等问题。在传统诊断方式中,需要经验丰富的外科医生手动从脊柱X光影像中测量多种医学特征,进而分析判断AIS分型。由于X光影像质量参差不齐,且医学参数测量受医生主观因素的影响,造成AIS分型耗时长、结果不稳定等问题。近年来,图像分类技术得到长足发展,广泛应用在医学领域,辅助医生分析人体的解剖结构以及识别病变类型[2]。例如使用VGG-16预测新冠肺炎[3]、GoogLeNet预测乳腺癌肿块[4]、MobileNetV2预测人体皮肤病[5]等。在判断脊柱疾病方面,SVM根据脊柱曲线的中心点系数特征自动对AIS分型[6]、ResNet预测脊柱曲线弯曲程度[7]、EfficientNetV1预测脊柱侧弯曲线[8]。上述模型使用卷积神经网络在层级传递信息,使用下采样操作增大感受野,但在深层结构中丢失位置信息严重,不能有效学习图像中不同特征之间相对位置信息。有的模型在脊柱侧弯分型任务中无法很好地捕获脊柱多个弯曲区域之间的位置关系,导致AIS分型准确率降低。基于Transformer架构的ViT [9]和Swim-transformer[10]可以有效地保留不同图像块间的位置信息,但是训练这些模型需要大量的数据,并且模型的参数量巨大,对硬件设备要求过高,在医学图像小样本分类任务中容易过拟合,导致分类结果欠佳。胶囊网络可以保存特征位置信息,比Transformer架构的模型更加容易训练,在重叠手写数字集上能将错误率降低到5.2%[11],而且在小样本数据集中,相比其他深度学习方法,胶囊网络可以获得更高的准确率[12]。然而在主胶囊层中每个胶囊向量都要与对应的二维矩阵进行运算,这导致胶囊网络参数量较大,并且每个胶囊向量不能转化到同一个语义空间中,导致动态路由算法(Dynamic Routing Algorithm,DRA)[11]不能很好地捕获每个胶囊向量在特征空间中的位置关系,从而使AIS分型结果较差。在脊柱X光影像中,AIS分型决定因素主要集中在脊柱区域,背景等无关区域能够干扰AIS分型结果。为了解决上述问题,本文添加共享注意力模块融合多尺度信息,使用胶囊解码结构替代主胶囊层中的二维矩阵,使网络既可以关注脊柱区域的特征信息,又可以产生输出一致性的胶囊向量,从而使DRA有效地学习脊柱畸变区域胶囊向量的位置关系,提高AIS分型准确率。实验使用AIS分型数据集中存在类别不平衡的现象,因此使用焦点损失(Focal Loss,FL)函数减少类别不平衡和难易样本对模型的影响[13]。
1 方法
1.1 整体网络架构
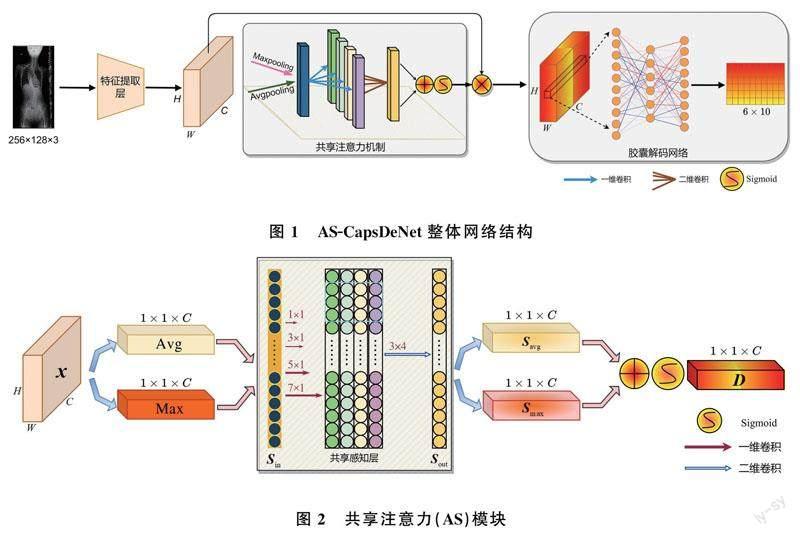
针对类别不平衡AIS分型数据设计了共享注意力胶囊解码网络(Attention Shared Capsule Decoding Network,AS-CapsDeNet),網络的整体结构由特征提取层、共享注意力(Attention Shared,AS)模块和胶囊解码网络(Capsule Decoding Network,CapsDeNet)构成,如图1所示,其中H、W、C分别代表特征图的高、宽、维度,Maxpooling和Avgpooling分别代表全局最大池化和全局平均池化。
AS-CapsDeNet的输入尺寸为256×128×3,在特征提取层中使用4个卷积层,卷积核的大小分别为3×3,3×3,5×5,7×7,卷积核的数量分别为128,64,64,64,每个卷积层后使用最大池化层提取浅层语义特征。随后将特征输入到AS模块,以激活合适的脊柱特征。将处理后的脊柱特征信息输入到CapsDeNet,在主胶囊层和数字胶囊层之间使用DRA,综合考虑X光影像脊柱区域语义特征信息,通过加权选择解码出脊柱侧弯的分类特征,最终完成分型预测。
1.2 共享注意力模块
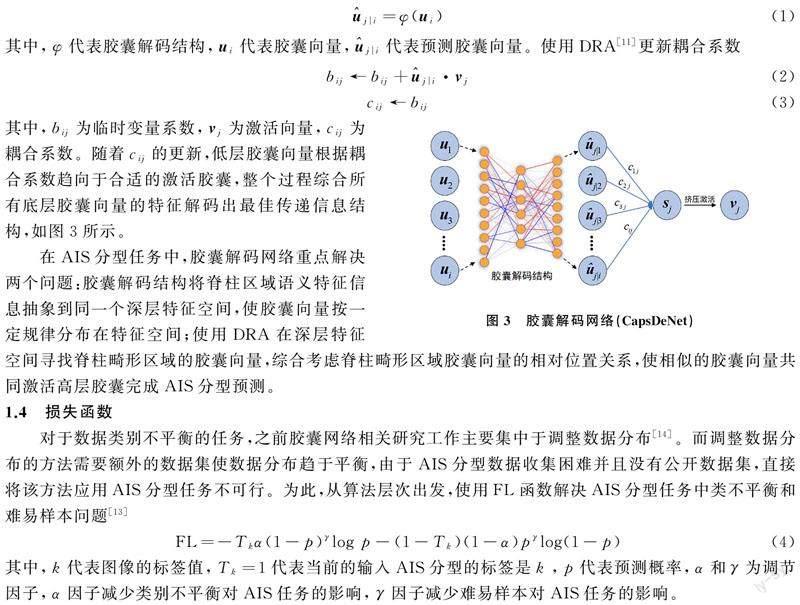
AIS分型任务中,所含脊柱畸形的特征区域只是X光影像的一小部分,大部分区域都是无关信息。提取特征信息之后,使用注意力模块让网络模型重点关注感兴趣的脊柱区域。AS模块如图2所示,对于给定的特征x(大小为H×W×C),AS模块使用全局平均池化层和全局最大池化层来简单地生成每个通道上的统计信息分别记为Avg(大小为1×1×C)和Max(大小为1×1×C),并将不同统计信息输入到共享感知网络层中。为了更加有效的捕获感兴趣脊柱区域的特征信息,本文从多尺度的角度出发,在共享感知层中引入1×1,3×1,5×1,7×1不同尺度的卷积核,分别对输入Sin提取特征,然后利用二维3×4的卷积核融合这些多尺度特征信息,得到共享感知网络层的输出Sout(大小为1×1×C)。
经过共享感知网络层编码后的Savg和Smax相加得到一组权重参数D=[d1,d2,…,dC],通过非线性激活函数(Sigmoid)激活,这组权重参数可以反映出每个维度相较于整个AIS分型重要程度。将学习到的权重参数与输入特征x在维度上进行信息融合,得到一组筛选后的语义特征信息y(大小为1×1×C)。虽然前后的特征尺寸没有发生变化,但是AS已经激活了感兴趣脊柱区域,抑制背景等不重要的信息。AS模块轻量且高效,能够融合多尺度特征提升AIS分型的准确率。
1.3 胶囊解码网络
卷积神经网络(Convolutional Neural Networks,CNN)随着网络层数不断加深,可以在深层结构融合全局上下文信息。然而随着层数的增加,CNN也会忽略特征之间的局部位置信息,在AIS分型任务中不能很好地捕获脊柱畸变区域的上下关系。虽然CNN可以使用注意力模块让网络关注感兴趣的脊柱区域细节信息,但是无法较好地表达图像中的局部依赖关系,CapsDeNet中的DRA[11]可以解决上述问题。主胶囊层对浅层特征更加敏感,如果将X光影像直接输入主胶囊层,不仅使网络参数量巨大,而且降低网络捕获特征的能力,致使整个网络AIS分型性能下降。所以对于AS-CapsDeNet底层使用CNN提取图像的特征,深层利用胶囊网络中DRA传递层级信息,既充分利用CNN的提取特征能力,又可以保留深层特征之间的局部位置关系。
胶囊是一个向量,方向代表对象属性,长度代表对象存在概率。CapsDeNet由主胶囊层、胶囊解码结构和数字胶囊层构成,数字胶囊层为输出层一共有6个不同激活胶囊向量,对应6种不同AIS分型。将主胶囊层中的每个胶囊向量输入到胶囊解码结构中,该结构由3层全连接网络组构成,可以将每个胶囊向量特征信息抽象到同一个高维特征空间中,并按一定的规律分布在该空间中。胶囊向量通过该结构可以得到一组转化后的预测胶囊向量
其中,bij为临时变量系数,vj为激活向量,cij为耦合系数。随着cij的更新,低层胶囊向量根据耦合系数趋向于合适的激活胶囊,整个过程综合所有底层胶囊向量的特征解码出最佳传递信息结构,如图3所示。
在AIS分型任务中,胶囊解码网络重点解决两个问题:胶囊解码结构将脊柱区域语义特征信息抽象到同一个深层特征空间,使胶囊向量按一定規律分布在特征空间;使用DRA在深层特征空间寻找脊柱畸形区域的胶囊向量,综合考虑脊柱畸形区域胶囊向量的相对位置关系,使相似的胶囊向量共同激活高层胶囊完成AIS分型预测。
1.4 损失函数
对于数据类别不平衡的任务,之前胶囊网络相关研究工作主要集中于调整数据分布[14]。而调整数据分布的方法需要额外的数据集使数据分布趋于平衡,由于AIS分型数据收集困难并且没有公开数据集,直接将该方法应用AIS分型任务不可行。为此,从算法层次出发,使用FL函数解决AIS分型任务中类不平衡和难易样本问题[13]
其中,k代表图像的标签值,Tk=1代表当前的输入AIS分型的标签是k,p代表预测概率,α和γ为调节因子,α因子减少类别不平衡对AIS任务的影响,γ因子减少难易样本对AIS任务的影响。
2 实验结果和分析
2.1 数据集与预处理
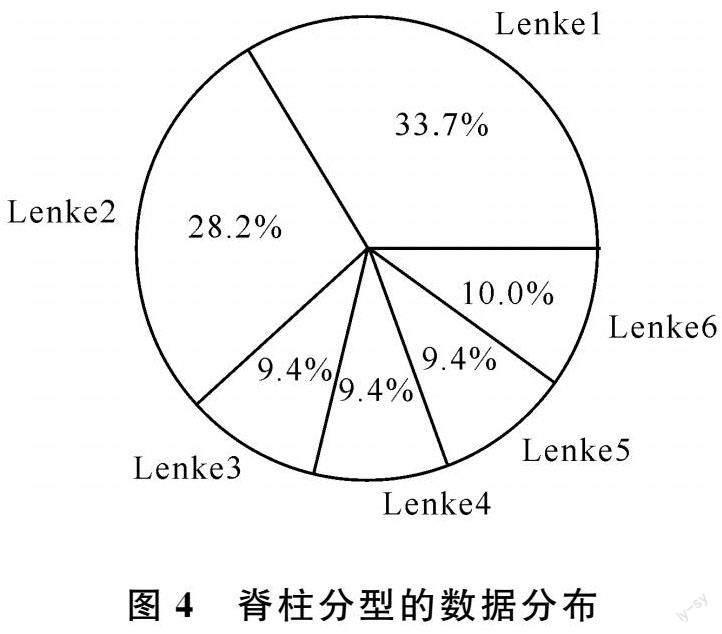
AIS分型的数据集取自青岛大学附属医院脊柱外科,共有564张X光影像数据,每张图像都由专业医生标记为Lenke1到Lenke6共6种AIS分型,分型数据分布如图4所示。X光原始影像数据不仅尺寸过于庞大,而且存在冗余的黑色区域和尺寸不一致的问题。数据预处理时,首先使用Crop函数将其裁剪,然后利用Resize函数将裁剪区域的尺寸统一缩放到256×128。因为X光影像较暗,使用Gamma校正方法调整对比度,利用归一化方法将图像的数值缩放到0~1之间。
将数据集中每个类别的数据以7∶3比例分为训练集和测试集,随机使用缩放、平移、旋转等方法增强数据,其中相对与原图像的缩放比例为[0.8,1.2],相对于原图像随机平移距离的比例为[-0.2,0.2],旋转角度为[-10°,10°],数据增强处理后的训练集数量增加一倍。
2.2 对比试验与消融实验
为了更好的训练模型,采用Adam优化器分批次训练数据,每批次包含32个图像,学习率设为0.000 1。所有实验均采用Tensorflow开源框架,使用的显卡型号为NVIDIA GeForce RTX 1080。为了评估AS-CapsDeNet模型在AIS分型任务中的有效性,使用Vgg[15],DenseNet[16],ResNet[17],InceptionV3[18],EfficientNetV1[19]等分类网络对比测试AIS分型结果,每个模型都预加载ImageNet训练权重,使用准确率(Accuracy)、F1分数(F1-score)和G-mean验证每个模型的性能,实验结果见表1。
CapsNet是基础的网络,没有添加AS模块和胶囊解码结构,使用FL函数。AS-CapsNet在CapsNet基础上添加了AS模块,AS-CapsDeNet在AS-CapsNet基础上添加胶囊解码结构。由表1可知,AS-CapsDeNet在脊柱侧弯任务中比ResNet、EfficientNetV2等方法更具优势。这是因为AIS分型通过主次弯所在位置的相对关系进行判断,AS-CapsDeNet中的胶囊解码结构可以将脊柱区域的向量转化成一致性的输出向量并规范其位置,DRA可以有效的保留向量之间的长依赖关系和相对位置信息,从而使网络的性能提升。为了证明AS模块更加有效,在CapsNet基础上添加ECA和CBAM注意力模块进行对比,可以发现AS相比ECA和CBAM在准确率、F1分数、G-mean指标上均有提升。AS-CapsDeNet在AIS分型任务中比CNN鲁棒性更强,相比CBAM和ECA,使用AS更加有效。表2为AS模块、胶囊解码结构和FL函数对AS-CapsDeNet的影响。在添加AS模块后, AS-CapsNet和AS-CapsDeNet的准确率要比CapsNet高出3%~6%。在添加胶囊解码结构后,AS-CapsDeNet的错误率相比AS-CapsNet更低,这是因为在AS-CapsNet中每一个胶囊向量都要与对应的二维矩阵进行空间上的转换,而在AS-CapsDeNet中所有的胶囊向量都要输入到同一个胶囊解码结构中,在高维特征空间中产生一致性的输出,这使后续的DRA更加高效准确地学习到脊柱畸变区域胶囊向量的位置关系,并一同激活完成AIS分型的预测。CapsNet、AS-CapsNet、AS-CapsDeNet去掉DRA(NoDRA)后,网络转变为传统的CNN,致使网络性能下降。对比边界损失(Margin Loss,ML) 和FL函数可知,FL函数可以减少数据类别不平衡和难易样本对实验的影响。
图5为 CapsNet、AS-CapsNet、AS-CapsDeNet在AIS分型任务中的ROC曲线图,可以观察到AS-CapsDeNet模型曲线下面积比其他模型大,达到99.36%,表明AS-CapsDeNet使用AS模块、胶囊解码结构和FL函数可以在不平衡的AIS分型任务中提升模型的准确率和抗干扰能力,可以为医生提供高效、精确的辅助诊断建议。
为了更好观察注意力模块对胶囊网络的影响,使用Grad-CAM[25]可视化注意力模块模块融合后的特征图。为了与胶囊网络进行对比,在EfficientNetV2中添加多种注意力模块,使用EfficientNetV2作为AS-CapsDeNet中的特征提取层,结果如图6所示。使用AS后可以使AS-CapsNet、AS-CapsDeNet、EfficientNetV2更加关注脊柱区域,减少背景等干扰信息。使用胶囊解码结构后,AS-CapsDeNet和EfficientNetV2可以观察到更远更广阔更连续的脊柱区域。这也间接表明了CapsDeNet的作用机制,使用胶囊解码结构以后,可以将底层脊柱区域的胶囊向量解码到同一个深层特征空间中,此时脊柱区域胶囊向量语义信息更加丰富并且按一定规律分布在该空间中。后续的DRA可以有效从中学习到这些语义特征信息之间的位置关系,不会丢失局部位置信息,从而使脊柱区域的注意力热图更加广阔更加连续。
3 结论
本文提出一种用于辅助诊断脊柱侧弯分型的方法,在AS-CapsDeNet使用AS模块关注脊柱区域特征信息,使用胶囊解码结构将脊柱区域的胶囊向量,转化到更加深层次的特征空间中并规范其位置,利用DRA选择脊柱畸形区域胶囊向量一同激活完成AIS分型的预测。实验表明FL函数可以使AS-CapsDeNet有更强的鲁棒性,可以减低类别不平衡对脊柱分型任务的影响,辅助医生诊断AIS分型。
参考文献
[1]傅声帆,黄孔阳,吕畅,等. 儿童、青少年特发性脊柱侧凸症相关知识调查及健康教育[J]. 浙江创伤外科. 2008(4): 358-359.
[2]黎英,宋佩华. 迁移学习在医学图像分类中的研究进展[J]. 中国图象图形学报, 2022, 27(3): 672-686.
[3]DASH A K, MOHAPATRA P. A fine-tuned deep convolutional neural network for chest radiography image classification on COVID-19 cases[J]. Multimedia Tools and Applications, 2022, 81(1): 1055-1075.
[4]HASSAN S A, SAYED M S, ABDALLA M I, et al. Breast cancer masses classification using deep convolutional neural networks and transfer learning[J]. Multimedia Tools and Applications, 2020, 79(41-42): 30735-30768.
[5]AKAY M, DU Y, SERSHEN C L, et al. Deep learning classification of systemic sclerosis skin using the MobileNetV2 model[J]. IEEE Open Journal of Engineering in Medicine and Biology, 2021, 2: 104-110.
[6]譚志强. 基于U-net的特发性脊柱侧弯Lenke分型算法研究[D]. 北京:中国科学院大学, 2019.
[7]YANG J L, ZHANG K, FAN H W, et al. Development and validation of deep learning algorithms for scoliosis screening using back images[J]. Communications biology, 2019, 2(1): 1-8.
[8]AMIN A, ABBAS M, SALAM A A. Automatic detection and classification of scoliosis from spine x-rays using transfer learning[C]// 2022 2nd International Conference on Digital Futures and Transformative Technologies (IcoDT2). Rawalpindi, 2022: 1-6.
[9]DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16×16 words: Transformers for image recognition at scale[C/OL]// International Conference on Learning Representations. 2020.[2022-05-05]. https://arxiv.org/abs/2010.11929.
[10] LIU Z, LIN Y T, CAO Y, et al. Swin transformer: Hierarchical vision transformer using shifted windows[C/OL]// IEEE/CVF International Conference on Computer Vision. Montreal, 2021. [2022-05-05]. https://openaccess.thecvf.com/content/ICCV2021/html/Liu_Swin_Transformer_Hierarchical_Vision_Transformer_Using_Shifted_Windows_ICCV_2021_paper.html.
[11] SABOUR S, FROSST N, HINTON G E. Dynamic routing between capsules[C]// 31st International Conference on Neural Information Processing Systems. Long Beach, 2017: 3859-3869.
[12] BAYDILLI Y Y, ATILA U. Classification of white blood cells using capsule networks[J]. Computerized Medical Imaging and Graphics, 2020, 80: 101699.
[13] LIN T Y, GOYAL P, GIRSHICK R, et al. Focal loss for dense object detection[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020, 42(2): 318-327.
[14] AFSHAR P, MOHAMMADI A, PLATANIOTIS K N, et al. Brain tumor type classification via capsule networks[C]// 25th IEEE International Conference on Image Processing (ICIP). Athens, 2018: 3129-3133.
[15] SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[C/OL]// International Conference on Learning Representations (ICLR). San Diego, 2015: 1-14. [2022-05-05]. https://arxiv.org/pdf/1409.1556.pdf.
[16] HUANG G, LIU Z, VAN DER MAATEN L, et al. Densely connected convolutional networks[C]// 30th IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Honolulu, 2017: 2261-2269.
[17] HE K M, ZHANG X Y, REN S Q, et al. Deep residual learning for image recognition[C]// IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Seattle, 2016: 770-778.
[18] SZEGEDY C, VANHOUCKE V, IOFFE S, et al. Rethinking the inception architecture for computer vision[C]// IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Seattle, 2016: 2818-2826.
[19] TAN M X, LE Q V. Efficientnet: Rethinking model scaling for convolutional neural networks[C]// 36th International Conference on Machine Learning. Long Beach, 2019: 6105-6114.
[20] MA N N, ZHANG X Y, ZHENG H T, et al. Shufflenet v2: Practical guidelines for efficient cnn architecture design[C]// 15th European Conference on Computer Vision (ECCV). Munich, 2018: 122-138.
[21] HOWARD A, SANDLER M, CHU G, et al. Searching for mobilenetv3[C]// IEEE/CVF International Conference on Computer Vision. Seoul, 2019: 1314-1324.
[22] TAN M X, LE Q V. Efficientnetv2: Smaller models and faster training[C]// 38th International Conference on Machine Learning(ICML). Electr Network. 2021: 7102-7110.
[23] WOO S H, PARK J, LEE J Y, et al. Cbam: Convolutional block attention module[C]// 15th European Conference on Computer Vision (ECCV). Munich, 2018: 3-19.
[24] WANG J Y, Chen Y B, CHAKRABORTY R, et al. Orthogonal convolutional neural networks[C]//2020 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Seattle, 2020: 11505-11515.
[25] SELVARAJU R R, COGSWELL M, DAS A, et al. Grad-cam: Visual explanations from deep networks via gradient-based localization[C]// IEEE International Conference on Computer Vision. Venice, 2017: 618-626.

