大豆GmMADS4基因克隆、亚细胞定位及功能分析
薛迎斌,宋 佳,李枭艺,李小豪,陈经烨,伍萍珍,朱胜男,刘 颖
(1广东海洋大学化学与环境学院,广东 湛江 524088;2国家耐盐碱水稻技术创新中心华南中心,广东 湛江 524088;3广东海洋大学滨海农业学院,广东 湛江 524088;4岭南师范学院生命科学与技术学院,广东 湛江 524048)
MADS是一类重要的转录因子,其命名来源于酿酒酵母的mini chromosome maintenance 1(MCM1)、拟南芥AGAMOUS(AG)、金鱼草DEFICIENS(DEF)和人类Serum response factor(SRF)基因,它们所编码蛋白的N端都含有由50~60个氨基酸组成的MADS-box保守结构域[1]。植物MADS家族从系统发育上可分为I型和II型(又称M IKC型)2种,二者的区别主要在于有无K-box结构域[2-3]。关于I型MADS基因家族成员的报道较少。在水稻中,I型的OsMADS78和OsMADS79在种子发育早期发挥了重要的调控作用[4];拟南芥中研究比较深入的I型MADS是AtAGL62,参与调控拟南芥胚乳发育过程中的细胞分化[5]。II型MADS基因是植物所特有的,也是目前研究最多的,其中被广泛熟知的是参与花器官的形成和发育,如经典的花器官发育模型“ABC(D)E模型”中控制花萼和花瓣形成的AP1、控制花瓣和雄蕊形成的AP3、控制雄蕊和心皮形成的AG等基因,均属于MADS家族成员[3]。除了参与调控开花,MADS基因还被报道参与植物多种生长发育过程。水稻的OsMADS25通过正调控生长素的累积参与促进主根和侧根的生长[6];豆科作物大豆Glycine max的MADS成员GmNMH7能够抑制结瘤相关基因的表达,进而在根瘤的形成和发育过程中扮演负调控角色[7]。MADS家族基因还参与植物应对非生物胁迫的适应性调控[8]。拟南芥AtAGL6通过抑制盐胁迫响应基因的表达,负调控植物耐盐[9];水稻OsMADS25响应ABA信号,正调控植株对低温的适应性[10];超量表达野生大豆MADS基因GsMAS1,增加了拟南芥对铝毒的耐受性[11]。此外,多个MADS基因被报道参与缺磷的响应,如水稻的OsMADS23、OsMADS25和OsMADS27[12],但具体分子功能还有待进一步研究。
大豆是我国重要的粮油作物,然而我国大豆生产严重不足,每年约1亿吨需要进口[13]。限制大豆生产的因素很多,其中,磷作为植物生长必需的大量营养元素,在土壤中很容易被吸附固定,导致土壤有效磷含量普遍偏低,低磷胁迫是限制我国大豆生长和产量的重要因素之一[14-15]。研究大豆磷营养调控机理,挖掘调控大豆适应低磷胁迫的重要转录因子,对于磷高效大豆品种的遗传改良和培育具有重要意义[15-16]。MADS家族转录因子在大豆全基因组共有163个成员[17],但是目前有功能报道尤其是有响应低磷胁迫功能的相对较少。本研究克隆了1个大豆II型MADS成员−GmMADS4,并通过生物信息学、亚细胞定位、表达模式分析以及超量表达该基因对根系磷稳态的影响分析,进一步挖掘该基因可能的功能,为深入解析GmMADS4在大豆生长发育以及低磷胁迫适应过程中的功能奠定基础。
1 材料与方法
1.1 供试材料
本研究所用植物材料为大豆品种‘粤春03-3’,由华南农业大学根系生物学研究中心惠赠;烟草Nicotiana benthamiana为本氏烟草。所用菌株为大肠埃希菌DH5α、根癌农杆菌GV3101、发根农杆菌K599。
1.2 试验处理及样品准备
不同组织部位表达模式及缺素处理表达模式分析参考Yao等[18]的方法。挑选大小一致的大豆种子,播种于砂土中进行萌发,5 d后选择长势一致的幼苗,转移到1/2 Hoagland正常营养液中进行培养。对于不同组织部位样品,分别在培养的第32天(大豆开花时)收获根、茎、叶、花,第50天收获种子。对于缺素处理样品,幼苗在正常营养液培养1周后,取部分幼苗分别转移到无氮、无磷、无钾的1/2Hoagland营养液(北京酷来搏公司)中处理24 h,然后收获根和新叶以备分析。试验于2021年4—6月在日光温室进行,白天平均气温30℃,夜晚平均气温25℃,日平均光照时间为13 h。收获后的样品立即用液氮冷冻,之后放入−80℃条件下保存,用于RNA提取。
用Trizol试剂(Invitrogen公司)分别提取根、茎、叶、花和种子的总RNA,具体方法参照其说明书中的步骤。测定提取的总RNA的浓度和纯度,检测合格后,取1μL的总RNA,根据逆转录试剂盒(Promega公司)说明书去除基因组DNA后进行逆转录反应,获得相应的cDNA,保存于−20℃,作为载体构建和RT-qPCR的模板。
1.3 生物信息学分析
GmMADS4基因基本信息包括开放阅读框(Open reading frame,ORF)长度、外显子/内含子数目、氨基酸长度,从Phytozome数据库(https://phytozome-next.jgi.doe.gov/)获得。GmMADS4蛋白相对分子质量及等电点通过Expasy数据库(https://web.expasy.org/compute_pi/)获得。GmMADS4蛋白保守结构域的预测通过NCBI conserved domains(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行。基因结构通过GSDS网站(http://gsds.gao-lab.org/index.php)获得。蛋白二级结构通过Prabi网站(https://npsaprabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行预测。GmMADS4基因启动子(5′UTR上游2 000 bp的DNA序列)顺式作用元件预测通过PlantCARE网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行。GmMADS4互作蛋白预测通过STRING网站(https://www.string-db.org/)根据氨基酸序列进行。系统进化树分析通过MEGA 5.05软件完成,采用邻接法(Neighbor-joining method),Bootstrap设置为1 000次。
1.4 载体构建
以大豆花的cDNA为模板,用基因特异引物(OE-GmMADS4-F/R)对GmMADS4基因ORF全长进行PCR扩增。50μL的PCR反应体系:10×Ex Taq Buffer 5μL、dNTP 4μL、正反向引物各1μL、cDNA模板2μL以及ddH2O 37μL。PCR反应程序:94℃预变性3min;94℃变性30 s、59℃退火30 s、72 ℃延伸1 min,30个循环;72 ℃保温10 min。扩增出的目标基因片段纯化回收后,先连入pMD18-T载体,经测序无误后,用Sac I和Xba I进行双酶切,再通过T4 DNA连接酶将GmMADS4基因连入目标载体pTF101s,获得GmMADS4-pTF01s重组质粒,检测无误后转入发根农杆菌K599备用。
构建GmMADS4融合绿色荧光蛋白(GFP)的表达载体,用基因特异引物GFP-GmMADS4-F/R(酶切位点为Kpn I和BamHI)扩增GmMADS4基因ORF全长,并连入目标载体pBEGFP,测序无误后转化农杆菌GV3101备用。引物设计通过PerlPrimer软件完成,引物序列见表1。
1.5 亚细胞定位分析
GmMADS4蛋白亚细胞定位通过烟草叶片表皮细胞瞬时表达[19]进行分析。将含有GmMADS4-pBEGFP载体质粒的农杆菌GV3101活化后扩大培养,菌体收集后用重悬液(包含10mmol/LMgCl2、10mmol/LMES以及0.1 mmol/L乙酰丁香酮)重悬,使D600nm为0.5左右,静置2~3 h,通过注射器导入生长3~4周的烟草叶片下表皮,转化3 d后即可进行观察。以转化空载体pBEGFP的叶片为对照。GFP荧光的观察通过激光共聚焦显微镜进行,激发光波长为488 nm,发射光波长为507 nm。
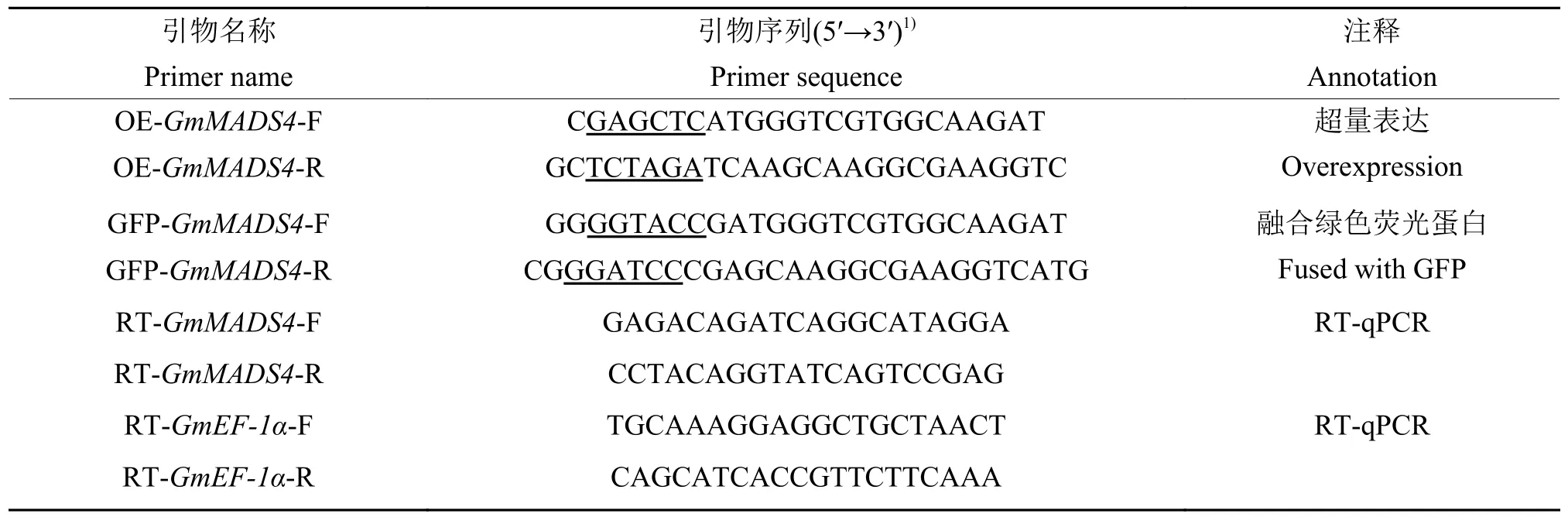
表1 本研究所用到的引物Table 1 Primers used in this study
1.6 RT-qPCR分析
将大豆待测cDNA样品稀释10倍作为模板,RT-qPCR参考Liu等[20]的方法,试剂采用Go Taq qPCR Master Mix(Promega,美国),通过StepOnePlusTM实时荧光定量PCR仪(ABI,美国)完成,程序设置为95℃30 s;95℃5 s、60 ℃15 s,40个循环;72 ℃30 s。基因定量引物RT-GmMADS4-F/R和内参基因GmEF-1α(Glyma17g23900)定量引物RT-GmEF-1α-F/R见表1。目的基因的相对表达量用2-ΔΔCt法计算。
1.7 大豆复合植株转化
大豆复合植株转化参考Zhuang等[21]的方法。种子萌发后,用注射器的针头分别蘸取适量携带pTF101s空载体和GmMADS4-pTF101s的K599发根农杆菌,然后在大豆幼苗子叶节的位置穿孔并涂抹菌体,进行侵染。约10 d左右可以看到有毛根长出,待毛根长到10 cm左右,把原来的主根剪掉,留下毛根,即为转基因复合植株。将超量表达GmMADS4和对照转基因复合植株转移到正常营养液中培养2周,收获地上部和毛根进行相关指标的测定。
1.8 生物量的测定
植株生物量用干质量表示,样品收获后立即在105℃烘箱杀青30min,然后调到75℃烘干至恒质量,称取植株总干质量;每个处理有4次独立的生物学重复。
1.9 可溶性磷含量的测定
将0.1 g毛根用液氮速冻并研磨至粉碎,加1 200μL双蒸水继续研磨以充分提取毛根中的可溶性磷;将研磨混合液转入2 m L离心管,于4℃、14 000 r/min离心30min;取上清液采用钼锑抗比色法[22]测定磷含量,测定波长为700 nm。
2 结果与分析
2.1 GmMADS4基因结构及保守结构域预测
随着基因组数据库的更新,本研究获得了1个大豆MADS成员,基因座位号为Glyma01g37470,命名为GmMADS4,该基因ORF长度为732 bp,编码244个氨基酸,预测蛋白相对分子质量为28 000,等电点为9.31。GmMADS4基因结构如图1A所示,该基因由7个外显子和6个内含子组成,两端各有1段非编码区。通过NCBI conserved domains保守结构域预测网站分析GmMADS4蛋白保守结构域,结果表明,该基因编码蛋白N端第2—77个氨基酸,为保守的MADS结构域,说明该基因属于MADS超家族,此外还含有1个K-box结构域(图1B)。
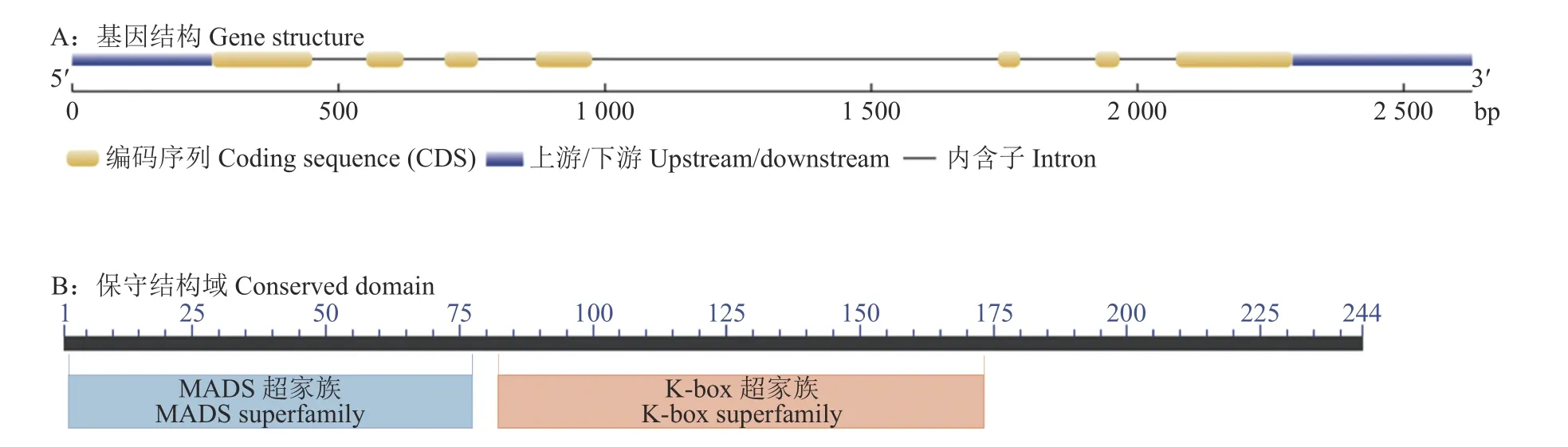
图1 GmMADS4基因结构及保守结构域预测Fig.1 Gene structure and conserved domain prediction of GmMADS4
2.2 GmMADS4蛋白二级结构预测
进一步分析了GmMADS4蛋白二级结构,结构表明GmMADS4蛋白包含多个α螺旋、β转角、延伸链和无规则卷曲(图2)。其中,α螺旋最多,占51.44%;其次为无规则卷曲,占23.05%;延伸链占16.05%;β转角最少,占9.47%(图2)。

图2 GmMADS4蛋白二级结构预测Fig.2 Secondary structure prediction of GmMADS4
2.3 MADS家族蛋白进化树分析
为了预测GmMADS4基因的功能,本研究将GmMADS4蛋白序列与已报道的模式植物拟南芥和水稻MADS家族成员,以及大豆MADS家族其他成员进行了系统进化树分析。这些MADS成员大致可以分成2个亚家族,分别为亚家族I和亚家族II(图3)。I型MADS成员包括已报道的拟南芥的AtAGL 23/62和水稻的OsMADS78/79;Gm MADS4属于II型MADS成员,该类型的MADS成员还包括拟南芥的AtAP1、AtAG、AtSOC1、AtANR1、AtP1、AtAP3、AtAGL 12/14/15/16/17/18/21,水稻的OsMADS20/23/25/26/27/62,大豆的GmNMHC5、GmNMH7、GmSEP1以及GmAGL1/11/15,其中GmMADS4与拟南芥的AtAP3序列相似性较高(图3)。
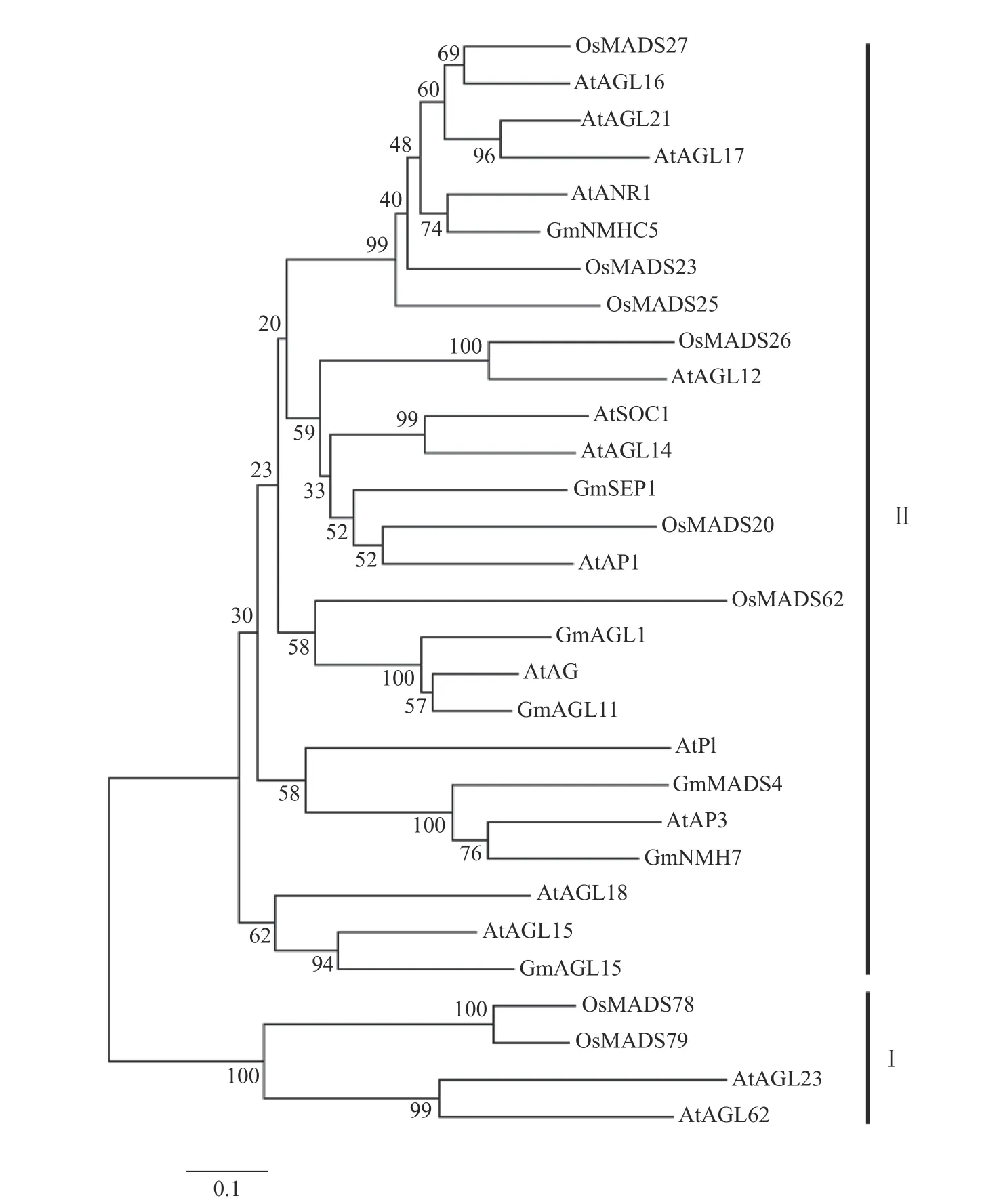
图3 MADS家族蛋白系统进化树分析Fig.3 Phylogenetic tree analysis of MADS family proteins
2.4 GmMADS4基因启动子顺式作用元件分析
取GmMADS4基因5'UTR上游2 000 bp的序列为启动子,分析其中的顺式调控元件,结果表明,在GmMADS4基因启动子中含有多种类型的顺式调控元件。其中最多的是典型的光响应元件,有22个,包括9个Box 4、5个G-Box、3个GT1-motif、2个TCCC-motif、2个AE-box和1个LAMP-element元件。其次是植物激素响应相关的元件,有9个,其中响应茉莉酸甲酯的有4个,包括2个CGTCA-motif和2个TGACG-motif;还有4个脱落酸响应元件ABBRE和1个赤霉素响应元件P-box。除了光响应和激素响应元件外,还包括胚乳表达相关元件GCN4_motif、分生组织表达相关元件CAT-box等参与生长发育调控的元件,厌氧感应元件ARE,以及MYB和MYC转录因子介导的多种逆境胁迫响应元件。
2.5 GmMADS4互作蛋白预测
通过STRING网站对GmMADS4的互作蛋白进行了预测,结果发现,有5个候选蛋白可能与GmMADS4(GLYMA 01G37470.1)存在互作关系(图4)。其中,GmMADS8(GLYMA 13G09660.1)、GmMADS9(GLYMA 14G24590.1)、GmMADS10(GLYMA04G42420.1)、GmMADS7(GLYMA06G12380.1)为MADS家族蛋白,GmLFY1(GLYMA04G37900.2)为LEAFY家族蛋白。
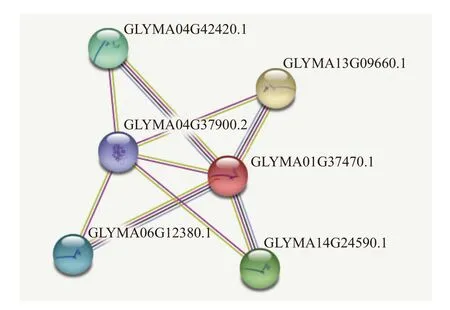
图4 GmMADS4互作蛋白预测Fig.4 Interaction protein prediction of GmMADS4
2.6 GmMADS4基因克隆及超量表达载体构建
为了分析GmMADS4基因功能,本研究用特异性引物OE-GmMADS4-F/R通过PCR克隆了GmMADS4基因ORF全长,结果如图5A所示;PCR扩增获得1条单独的条带,位置在750 bp左右,与GmMADS4基因ORF全长732 bp大小一致。经测序无碱基突变后,进一步连接到超量表达载体pTF101s上。图5B为GmMADS4-pTF101s重组质粒酶切检测,与重组质粒酶切前(泳道1)相比,经Sac I和Xba I限制性内切酶双酶切后(泳道2)在略低于800 bp的位置出现了1个条带,大小与GmMADS4基因ORF全长一致,说明重组质粒构建成功。
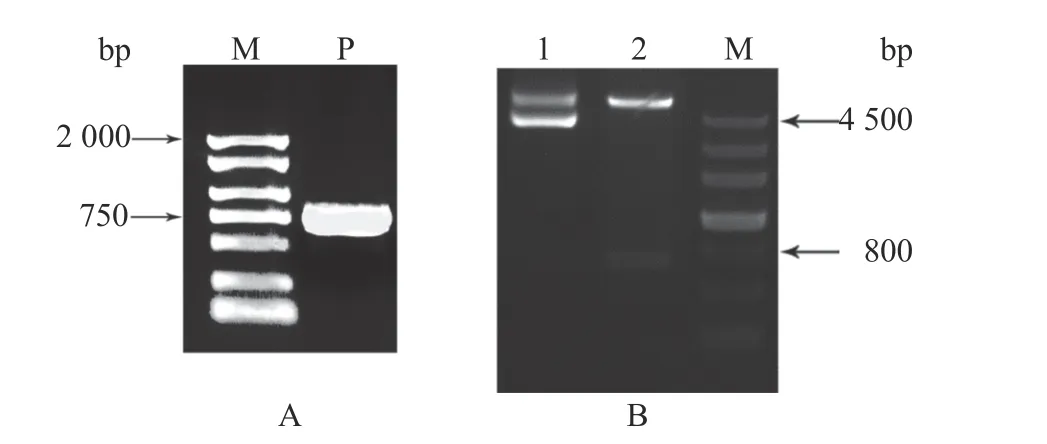
图5 GmMADS4基因克隆及酶切检测Fig.5 Cloning and enzyme digestion of GmMADS4
2.7 GmMADS4在大豆不同部位的表达模式
为了分析GmMADS4基因主要在大豆哪个部位发挥功能,本研究通过RT-qPCR分析了该基因在大豆根、茎、叶、花和种子中的表达量(图6)。在大豆根、茎、叶、花和种子中都能检测到GmMADS4基因的表达;值得注意的是,GmMADS4主要在花和种子中有较高的表达量,尤其在花中的表达量最高,而在根、茎和叶中表达量相对较低,尤其在茎中表达量最低(图6)。GmMADS4基因在花中的表达量约是种子中的1.7倍,同时约是根、茎和叶中表达量的50倍以上。
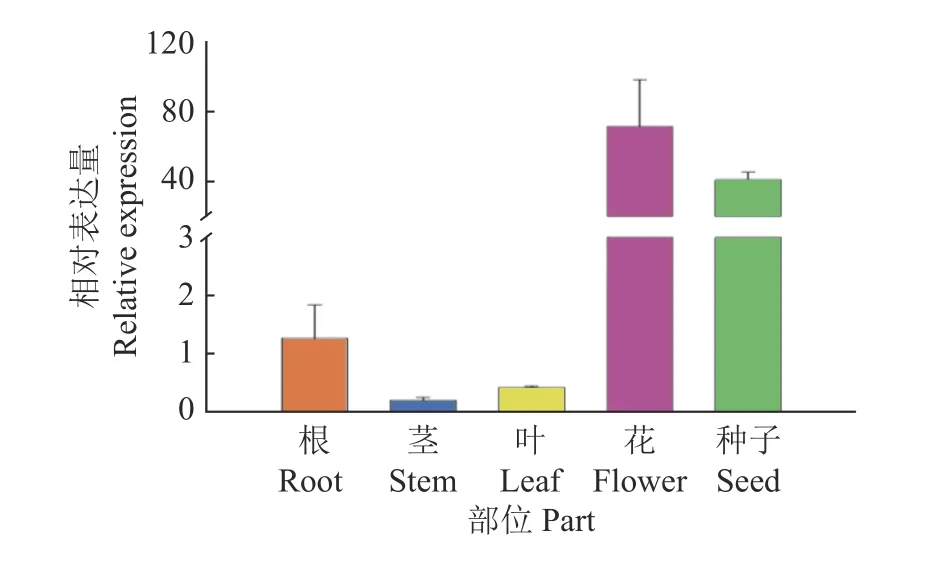
图6 GmMADS4在大豆不同部位的表达分析Fig.6 Expression pattern analysis of GmMADS4 in different soybean organs
2.8 GmMADS4在大豆根和叶中响应缺素的表达模式
进一步分析了GmMADS4基因对缺氮、缺磷、缺钾胁迫的响应,结果表明GmMADS4在大豆叶和根中响应多种非生物胁迫(图7)。GmMADS4在大豆根中主要受缺氮和缺磷处理上调表达,而对于缺钾处理没有响应;与正常处理的对照组相比,GmMADS4在缺氮和缺磷处理下的表达量分别提高了11.2和6.6倍(图7A)。在大豆叶部,缺氮、缺磷和缺钾处理均上调GmMADS4的表达;与正常处理的对照组相比,GmMADS4在缺氮、缺磷和缺钾处理叶中的表达分别提高了12.0、0.7和5.2倍(图7B)。
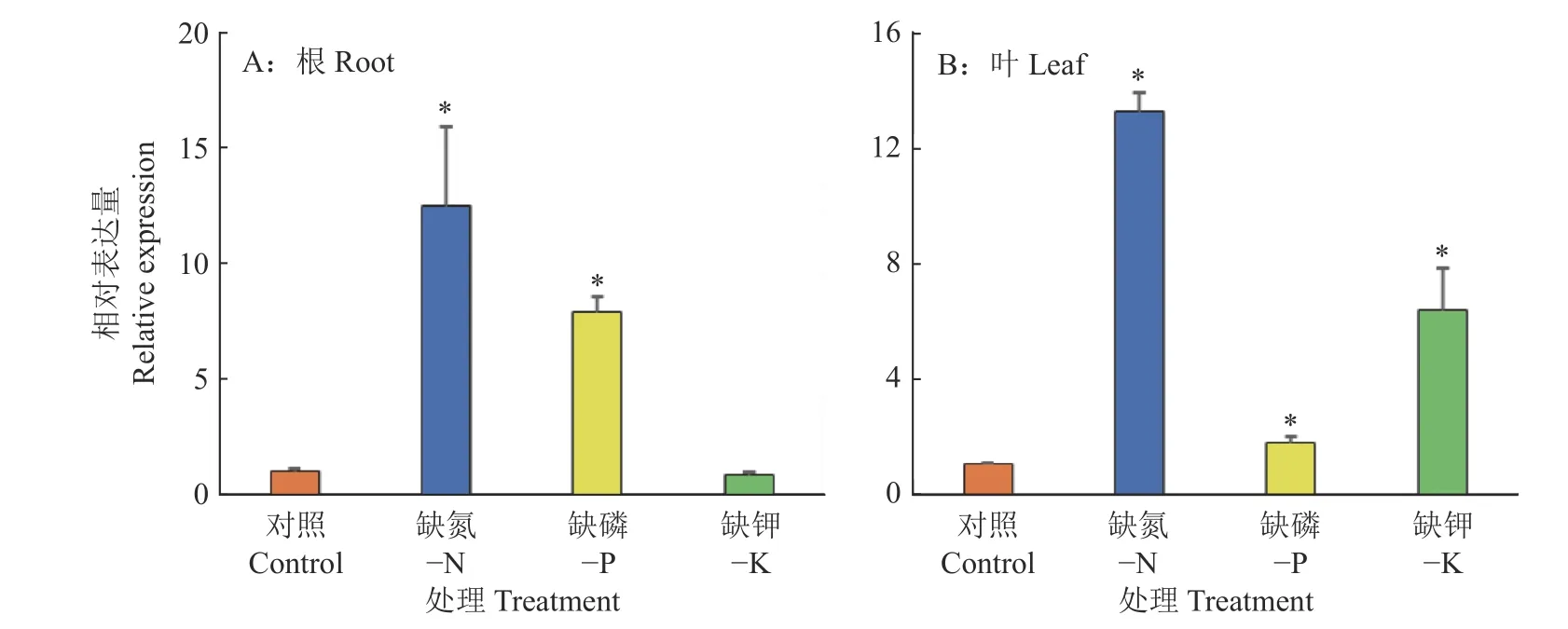
图7 GmMADS4在大豆根和叶中响应缺素的表达模式分析Fig.7 Expression pattern analysis of GmMADS4 in response to element deficiency in soybean roots and leaves
2.9 GmMADS4亚细胞定位分析
为了研究GmMADS4蛋白在亚细胞水平的具体定位,本研究通过烟草叶部瞬时转化,将携带GmMADS4融合GFP(GmMADS4-GFP)的农杆菌GV 3101转化烟草叶片,通过激光共聚焦显微镜观察荧光位置,其中以GFP空载体为对照。转化GFP空载体的叶片在细胞核、细胞质、细胞膜都有较强的绿色荧光,而转化GmMADS4的叶片主要在细胞核有较强绿色荧光,此外在细胞膜也有微弱的绿色荧光(图8)。这些结果表明GmMADS4主要定位在细胞核。

图8 GmMADS4亚细胞定位分析Fig.8 Subcellular localization analysis of GmMADS4
2.10 超量表达GmMADS4影响大豆可溶性磷浓度
为了研究GmMADS4基因的功能,本研究进一步利用发根农杆菌K599,通过下胚轴复合植株转化法,获得超量表达GmMADS4的大豆转基因复合植株,即根系为转基因毛根、地上部位正常的大豆植株。将超量表达株系与转化空载体的对照株系在大豆营养液中培养14 d,观察表型。与对照相比,超量表达GmMADS4有促进毛根生长、减缓地上部生长的趋势,但是从毛根、地上部以及整株干质量上看差异均不明显(图9)。超量表达GmMADS4虽然没有改变复合植株的生物量(图10A),但是显著增加了毛根的可溶性磷含量(图10B),与对照相比,超量表达GmMADS4转基因复合植株毛根中的可溶性磷含量显著增加51%(图10B)。

图9 超量表达GmMADS4转基因复合植株的生长表型Fig.9 growth phenotypes of overexpressing GmMADS4 transgenic composite plants
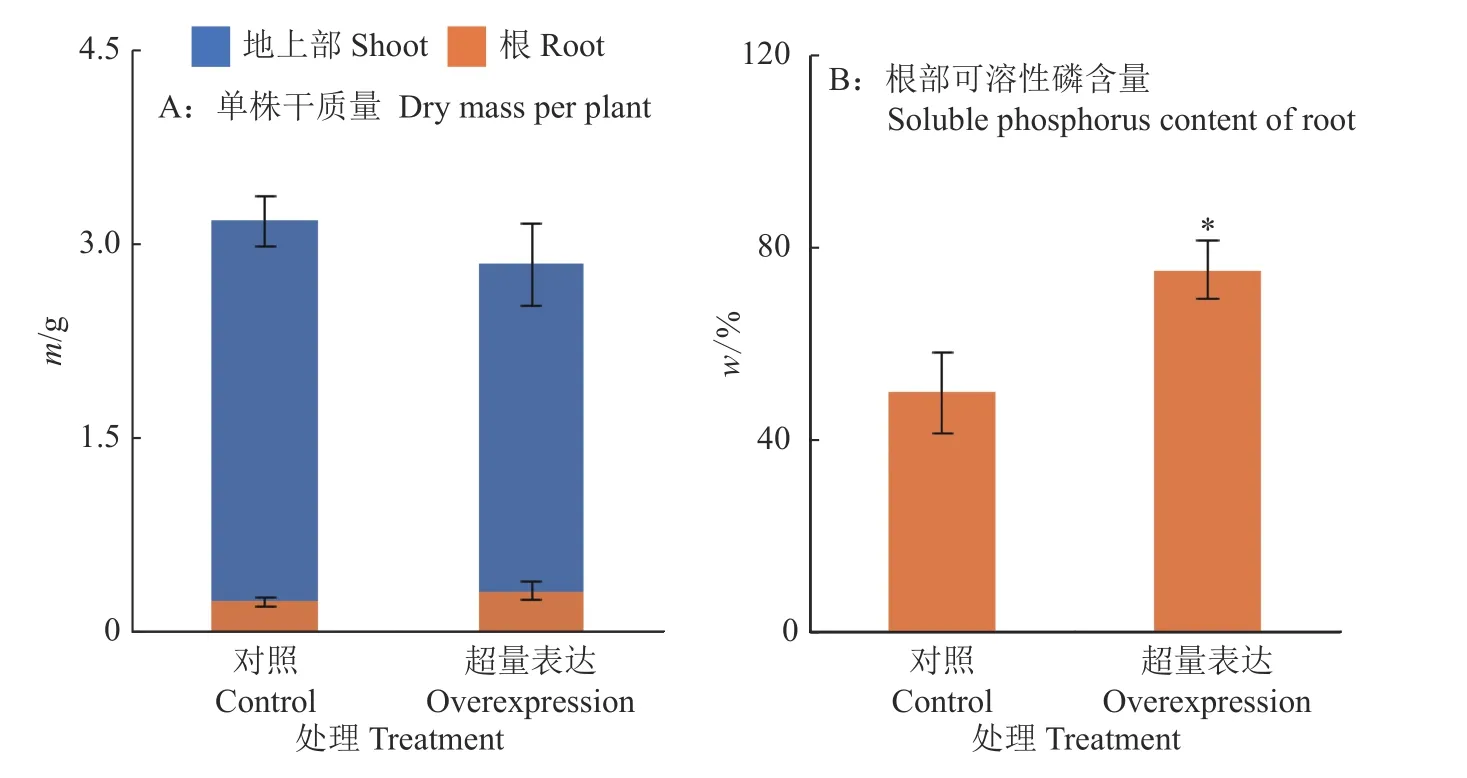
图10 超量表达GmMADS4对大豆复合植株生物量和可溶性磷含量的影响Fig.10 Effects of overexpressing GmMADS4 on biomass and soluble phosphorus content of soybean composite plants
3 讨论与结论
MADS转录因子家族在植物多个生长发育过程中发挥重要的调控作用。本研究克隆了大豆II型MADS成员−GmMADS4,从进化关系上,GmMADS4与拟南芥MADS家族成员AP3的序列相似性最高。AP3属于经典的ABC模型中的B型基因,在花器官识别中扮演重要角色[23-24],同时,不同部位表达模式分析表明GmMADS4基因在大豆花中表达量最高,暗示GmMADS4可能在花器官形成和发育中发挥重要作用。在拟南芥花分生组织发育的调控过程中,拟南芥MADS成员SEP3与AG、AP3和PI均存在明显互作,以异源二聚体的形式发挥功能[25]。本研究通过互作蛋白预测发现GmMADS4与其他4个MADS成员GmMADS7/8/9/10存在相互作用,表明GmMADS4可能以复合体的形式参与调控作用。虽然关于大豆GmMADS7/8/9/10的具体功能还未见报道,但表达模式分析表明GmMADS7/8/9/10这4个成员均主要在花中特异表达[17]。这些结果表明,GmMADS4可能与其他蛋白互作,以异源二聚体的形式调控花器官的形成和发育。除了在花中高表达,GmMADS4在大豆种子中也有较高的表达量,且GmMADS4启动子中含有胚乳表达相关元件GCN4_motif,暗示GmMADS4还参与大豆种子发育。互作蛋白预测发现GmMADS4与GmLFY 1也存在互作,GmLFY1被报道主要在发育中的豆荚和种子中表达,而在茎尖分生组织中不表达,可能参与大豆的种子发育而不是开花[26]。这些结果表明,GmMADS4可能在大豆种子发育调控中也发挥重要功能。
MADS转录因子已被报道参与植物多种非生物胁迫,并在调控网络中扮演关键角色[27]。本研究发现,GmMADS4在根和叶中响应缺氮和缺磷胁迫上调表达,同时在叶中也受缺钾胁迫上调表达,暗示GmMADS4可能参与氮、磷或钾元素缺乏胁迫的适应性调控。在拟南芥中,MADS家族成员AtAGL14/19/20/21/44均在根部受缺氮胁迫上调表达,其中,AtAGL20同时还受缺磷胁迫上调表达[28-29]。超量表达AtAGL44 (也即AtANR1)能够促进拟南芥根系发育,增加侧根数目和长度,且这种调控方式依赖于硝酸根离子的存在[30]。水稻OsMADS61在根部受缺氮胁迫上调表达[12],相反,OsMADS27/57在根部受缺氮胁迫下调表达[12,31]。其中,OsMADS57已被证明通过直接调控OsNRT2.3a的表达,进而调控水稻中硝酸根离子从根往地上部的长距离运输[32]。此外,在水稻中多个MADS基因在根部受缺磷胁迫下调表达,如OsMADS23/25/27/57[12,31]。除了拟南芥和水稻,在小麦180个MADS成员中有54个MADS基因的表达在根部响应缺磷,其中TaMADS21/93/121这3个成员受缺磷胁迫上调表达超过4倍[33]。但是,关于MADS基因参与磷信号调控功能的报道较少。本研究发现,在大豆毛根中超量表达GmMADS4能够增加毛根中可溶性磷的含量,暗示GmMADS4可能参与调控大豆根系磷稳态。
综上所述,大豆GmMADS4在花和种子中高表达,可能通过与其他转录因子互作参与调控花和种子发育。另一方面,低磷胁迫上调GmMADS4在根系中的表达,且超量表达GmMADS4增加了转基因毛根的可溶性磷含量。因此,GmMADS4可能在大豆耐低磷胁迫中发挥重要调节作用。

