栽培小麦Brock中抗白粉病相关基因TaRPP13-1B的克隆及功能分析
刘晓颖 张 驰 王雪晴 杨晨晓 王光钰 卞云迪 方 芳 王 颖 王振英,*
栽培小麦Brock中抗白粉病相关基因的克隆及功能分析
刘晓颖1张 驰1王雪晴1杨晨晓1王光钰1卞云迪1方 芳2王 颖2王振英1,*
1天津师范大学生命科学学院 / 天津市动植物抗性重点实验室, 天津 300387;2天津市宝坻区林业发展服务中心, 天津 301899
白粉病是小麦生产中的主要病害之一, 发掘抗病基因并实现抗病基因转育是提高作物抗病性的最经济有效的方法。本研究克隆了一个位于小麦染色体1B上、具有典型CC、NBS和LRR结构域的基因。接种白粉菌后,基因在抗病小麦Brock和BJ-1中表达量虽然出现上下调波动, 但平均表达水平一直高于感病小麦品种京411。采用病毒诱导的基因沉默和转基因过表达技术进行功能分析, 发现抑制目标基因表达, 抗病小麦品种Brock对白粉菌的抗性显著降低; 过表达的转基因小麦津强5号对白粉菌抗性明显提高。说明基因参与小麦抗白粉病的防御反应过程。该研究为小麦抗病品种的选育提供了有价值的备选基因。
小麦; NBS-LRR类基因;; 抗白粉病
目前小麦中已克隆的抗白粉病基因中多数是NBS-LRR (nucleotide binding site leucine rich repeat, NBS-LRR)类基因, 其中又以CNL (coiled-coil NBS-LRR, CNL)类基因居多[1], 如、、、、、、及其等位基因, 携带这些抗性基因的小麦品种田间抗性良好, CNL类基因在小麦白粉病防治上发挥了重要作用[2-6]。(recognition of, RPP)首先在拟南芥抗霜霉病反应中被鉴定到, 具有典型的CNL结构域, 它通过识别效应蛋白ATR13, 从而诱发抗病反应[7]。研究发现,基因具有多态性, 其LRR结构域的多态性影响了不同生态型拟南芥对不同病原小种的特异性识别[8-9]。Rentel等[10]、Ramachandran等[11]发现, 霜霉病效应蛋白ATR13能够抑制ATR13/RPP13复合体诱发超敏反应。目前, 小麦中已经鉴定到多个同源基因, 这些基因在基因序列上都存在较大差异, 且基因功能涉及小麦生长的多个方面。例如,同源基因在小麦抗白粉病[12-14]、小麦抗锈病[15]和小麦面粉颜色方面均发挥作用[16]。在其他植物的研究中发现,同源基因在柑橘抗白色念珠菌感染[17]、姜黄抗茎腐病[18]、番茄抗黄叶卷曲病毒[19]、葡萄抗白粉病[20]、花生抗黄曲霉病[21]和百合的杂种株高优势[22]也发挥作用。基因在抵抗真菌、细菌、病毒侵染和植物生长发育等多个方面发挥重要作用。
前期工作中, 课题组配制了京411的抗病近等基因系BJ-1, 对白粉菌侵染早期(12 hpi)感病小麦京411及其抗病近等基因系BJ-1应答基因转录组比较分析中发现, 位于1B染色体的在小麦抗病近等基因系BJ-1中表达量上调, NCBI/Blast比对分析发现该基因具有典型的NBS- LRR类基因结构, 为基因家族的同源基因[13,24], 命名为。本研究克隆了这个潜在的抗性基因, 并对其在不同抗、感小麦中的序列组成进行了比较; 分析了在白粉菌胁迫下的表达模式, 通过降低和过表达基因表达水平, 探究该基因在小麦抵抗白粉菌侵染过程中的贡献, 为小麦抗病品种的选育提供了新的备选基因。
1 材料与方法
1.1 试验材料
Brock是一个抗白粉病小麦品种, 从英国引进, 在苗期和成株期均高抗小麦白粉病。京411是我国北方种植的农艺性状优良的小麦品种, 但易感白粉病。本实验室于2012年, 以Brock为抗病基因供体, 京411为轮回亲本, 通过连续回交6次, 创制了京411抗病近等基因系BJ-1。BJ-1的产量、生长周期、株高、分蘖等特性与轮回亲本京411一致, 它的抗病等级为1级。而抗病基因供体亲本Brock为0级。BJ-1农艺性状上偏京411, 抗白粉病特性偏Brock[23]。抗白粉病小麦品种Brock, 感白粉病小麦品种京411和抗病近等基因系BJ-1用于小麦全长cDNA的克隆及白粉菌诱导表达分析。感病春麦品种津强5号用于过表达载体的转化。白粉菌菌株E09 (f. sp.E09,E09)用于苗期抗病性鉴定, 由中国农业科学院植物保护研究所提供。
1.2 TaRPP13-1B全长cDNA的克隆
根据的序列信息设计引物, 用Primer premier 5.0设计引物TaRPP13-1B-F和TaRPP13-1B-R。将感病小麦品种京411、抗病近等基因系BJ-1和抗病小麦品种Brock培养至第1片叶子完全展开后, 采用抖拂法将白粉菌(E09)接种于小麦叶片12 h后, 取叶片用RNAiso Plus (TaKaRa,中国大连)分别提取总RNA, M-MLV反转录试剂盒(宝生物, 中国大连)反转录成cDNA。分别以3个小麦品种(系)的cDNA为模板, 扩增目的基因。PCR反应体系25 μL, cDNA 1 μL、上下游引物各0.5 μL、5× GXL缓冲液5 μL、dNTP混合物2 μL、PrimeSTAR GXL DNA聚合酶0.5 μL、双蒸水10.5 μL。反应程序为: 94℃ 5 min; 98℃ 10 s, 55℃ 15 s, 68℃ 3 min, 35个循环; 68℃ 7 min。PCR产物经1%琼脂糖凝胶电泳检测回收。按照DNA A-Tailing试剂盒方法加A尾(宝生物, 中国大连), 连接pGEM-T Easy载体(Promega, 美国)并转化大肠杆菌DH5α, 挑取LB Amp+(氨苄青霉素, ampicilline)筛选培养基上的阳性克隆, 经PCR和酶切鉴定后送苏州金唯智生物科技有限公司测序。
3'和5'RACE片段的扩增按SMART RACE cDNA扩增试剂盒(Clontech, 美国)的说明书进行。根据已经掌握的部分序列, 用Primer 5.0设计特异引物RPP13-1B-GSP1F和RPP13-1B- GSP2F分别进行3'RACE反应的第1和第2轮PCR; 设计特异引物RPP13-1B-5RACE-1R和RPP13-1B- 5RACE-2R分别进行5'RACE反应的第1和第2轮PCR。将3'和5'RACE第2轮PCR扩增的片段分别克隆至pGEM-T Easy载体, 经测序验证后, 将3'RACE、5'RACE及部分序列进行拼接, 得到拼接后的全长cDNA序列。根据拼接的全长cDNA序列设计引物-ORF-F/R扩增的全长cDNA, 将扩增产物连接至pGEM-T载体并经测序验证。本研究所用引物序列见表1。

表1 本研究的引物及序列
将基因序列提交NCBI GenBank进行Blast比对;利用DNAMAN软件对序列进行同源性分析, 构建系统发育树。利用cDNA序列的ORF在小麦联盟网站(http://202.194.139.32)进行比对, 分析其染色体定位, 预测DNA序列组成。
1.3 TaRPP13-1B基因的表达分析
将小麦京411、Brock和BJ-1幼苗培养至一叶一心期时, 抖拂法接种E09 2、4、8、12、24和48 h后分别提取叶片总RNA, 反转录成cDNA。以SYBR Green I作为荧光染料, 利用荧光定量PCR法(quantitative real time PCR, qRT-PCR) (ABI 7500 FAST, 美国)分析白粉菌侵染后基因在苗期京411、Brock和BJ-1中的表达水平。用2–DDCT法[25]计算白粉菌侵染过程中目标基因的相对表达量变化。具体步骤按照FastStart Universal SYBR Green Master (Roche, 美国)试剂说明书进行, PCR体系20 μL, 2× Master Mix (SYBR Green I) 10 μL、上下游引物各1 μL、cDNA 1 μL。将上述反应体系混合均匀, 进行荧光定量PCR扩增。扩增程序: 50℃ 2 min; 95℃ 10 min; 95℃ 15 s, 58℃ 30 s, 40个循环。以作为内参, 每个反应重复5次, 实验重复3次。
1.4 利用病毒诱导的基因沉默(virus-induced gene silencing, VIGS)技术分析TaRPP13-1B的功能
根据的测序结果, 在基因的非保守区设计引物RPP13-1B-V-F/R, 扩增带有I接头的基因片段。BSMVγ:载体经I酶切、去磷酸化后, 与沉默基因片段连接, PCR验证后获得基因沉默载体BSMVγ:。BSMVα和BSMVγ经I酶切, BSMVβ经I酶切后, 体外转录成BSMV病毒, 将病毒摩擦接种于抗病小麦Brock叶片表面沉默目标基因。以GKP buffer组、BSMV:组和BSMV:组为对照组。待BSMV:对照组小麦第三叶白化, 选取少量小麦叶片提取总RNA, 利用qRT-PCR法分析目标基因的沉默效率, 具体步骤同1.3。在已接种病毒的Brock叶片表面接种白粉菌, 接种白粉菌48 h、72 h和7 d后, 利用考马斯亮蓝染色, 在显微镜下观察白粉菌孢子萌发状况[26], 照相记录接菌7 d后叶片表面的白粉菌生长状态。明确基因功能。每组统计5片叶子, 每片叶子统计100个白粉菌孢子, 实验重复3次。
1.5 利用农杆菌介导的遗传转化法进行TaRPP13- 1B功能鉴定
以连接有序列的T载体为模板, 扩增获得带有E II和II酶切位点和接头序列的的全长, 利用无缝克隆试剂(全式金, 中国北京)将连接至pCAMBIA1301的相应酶切位点。将连接产物转至大肠杆菌DH5α, PCR验证后, 最终获得包含全长, 启动子为35S的过表达载体35S:。利用本实验室改良的农杆菌介导的小麦成熟胚遗传转化法[27-28], 将含有35S:的农杆菌C58C1转入感病春麦津强5号成熟胚, 收获T0代种子。利用潮霉素筛选、PCR和RT-PCR鉴定转基因小麦株系。以转化pCAMBIA1301质粒的津强5号为对照组。利用潮霉素筛选35S:株系至T1代。将T1代转基因小麦培养至一叶一心期, 在小麦叶片接种白粉菌E09, 逐日观察叶片表型变化直至第7天, 拍照记录。逐株调查材料的侵染类型, 按照病叶分级标准分级记录, 标准如下。1级: 病斑占叶面积的10%以下; 2级: 病斑占叶面积的10%~25%; 3级: 病斑占叶面积的26%~50%; 4级: 病斑占叶面积的51%~ 80%; 5级: 病斑占叶面积的80%以上[29-30]。
2 结果与分析
2.1 TaRPP13-1B基因的克隆
根据序列设计一对特异引物TaRPP13-1B-F/R, 在京411、Brock和BJ-1中扩增得到基因的cDNA序列。为确认该序列是否为全长, 进一步利用RACE技术扩增了该基因的5'和3'非编码区。最后克隆得到基因的cDNA全长3232 bp, 包含2934 bp的ORF以及104 bp和194 bp的5'和3'非编码区。感病小麦品种京411和抗病近等基因系BJ-1中的基因序列与Brock相同。在小麦联盟网站分别进行Blast分析, 推测该基因位于1B染色体; DNA序列长3453 bp, 由2个外显子(1~933 bp和1453~3453 bp)和1个内含子组成(934~1452 bp) (图1-A)。进一步的氨基酸比对分析发现, 该基因编码蛋白具有CC、NB-ARC和LRR结构域, 也是一个典型的CNL类型蛋白(图1-B)。蛋白质的同源比对及进化树分析发现, 其氨基酸序列与二穗短柄草(XP_010237386.1), 乌拉尔图小麦(EMS68464.1)和玉米(XP_00864940.1)中RPP13的同源性较高, 分别为93%、79%和66% (图1-C)。根据染色体定位信息, 将其命名为。
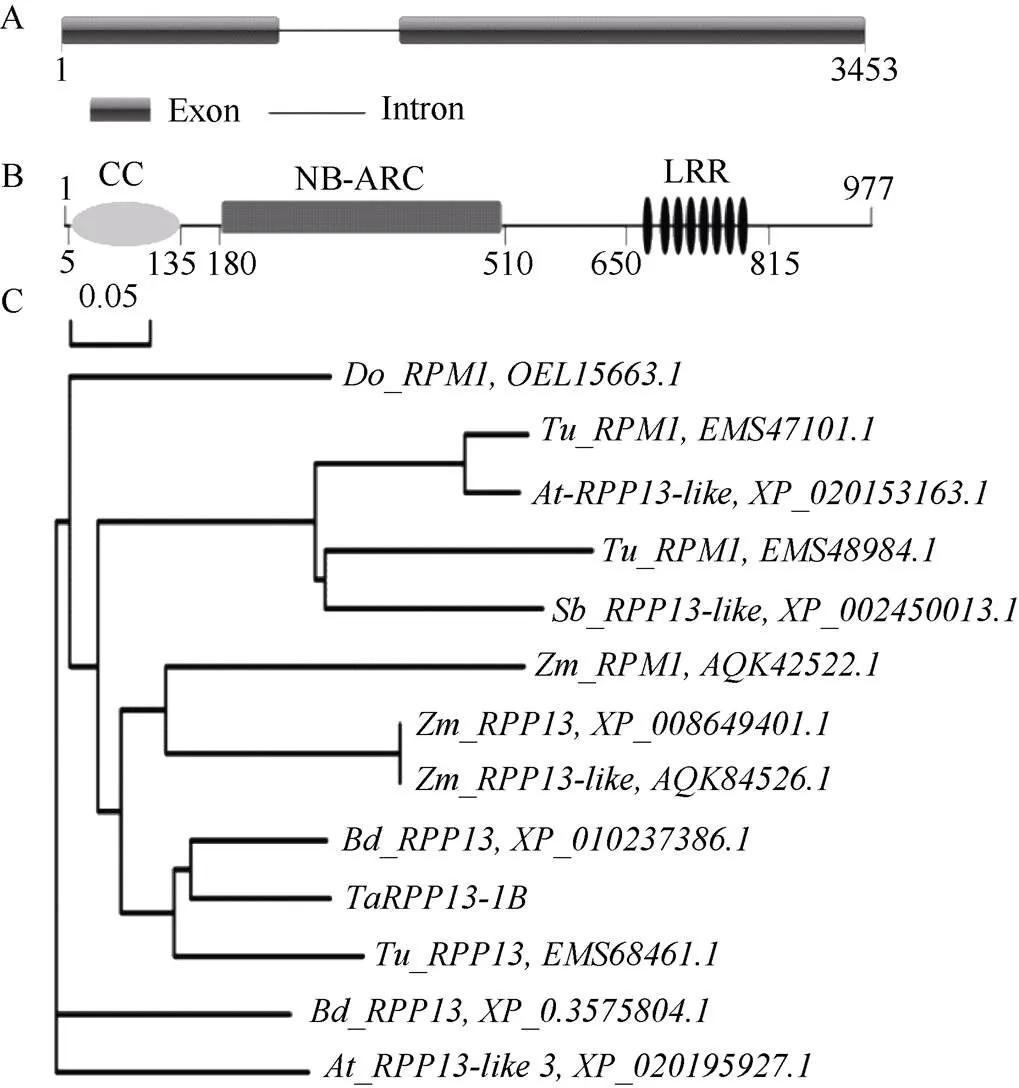
图1 TaRPP13-1B基因的结构域分析
A:的基因结构; B: TaRPP13-1B蛋白的结构域; C: 不同植物中RPP13蛋白的进化树。
A: predictive schematic forsequence; B: predictive domain schematic for coding TaRPP13-1B protein; C: the phylogenetic tree of diverse RPP13-like proteins family.
将与的基因序列在IWGSC和小麦联盟网站进行Blast比对发现,和基因分别定位于染色体1B和7D, 这与最初的参考基因序列预期一致。这两个基因均与基因家族具有较高同源性, 具有典型CNL结构域, 但二者的氨基酸序列一致性仅有23.2% (图2), 由此我们认为这两个基因在小麦抵抗白粉菌侵染过程中的作用有待进一步深入研究。
2.2 TaRPP13-1B基因的表达分析
在抗病小麦品种Brock中, 除白粉菌侵染4 hpi,基因表达短暂下调,基因表达水平一直高于抗病近等基因系BJ-1和感病小麦品种京411; BJ-1中的本底表达量与京411持平, 2 hpi时表达量逐渐上调, 4 hpi达到高峰, 之后逐渐下降; 京411中基因表达模式与BJ-1一致, 但整体表达量低于抗病近等基因系BJ-1。虽然表达量远低于BJ-1, 但表达高峰同样出现在4 hpi; 之后, 表达量开始下降。总之, 抗病材料中虽然部分接菌时间段内(4 hpi)基因的表达出现下调, 但基因的表达水平一直高于感病品种(图3)。故推测基因参与小麦对白粉菌的抗性反应。

图2 TaRPP13-1B和TaRPP13-3的同源性分析
2.3 瞬时沉默TaRPP13-1B基因能够显著降低小麦对白粉菌的抗性
2.3.1基因沉默小麦的表型分析 由于基因在抗病小麦品种Brock中表达水平较高, 在VIGS实验中以Brock为沉默对象。待Brock的第二片叶展开, 接种重组病毒。继续培养至第三片叶展开, 进行表型观察, 结果如图4-A所示。BSMV:对照组中小麦Brock叶片白化, 说明沉默体系有效; 利用荧光定量qRT-PCR检测各组材料中的基因的表达量, 从图4-C可以看出, BSMV:实验组中目标基因表达量低于另外两个对照组, 说明瞬时沉默基因表达成功。接种白粉菌7 d后, 观察BSMV:实验组、BSMV:对照组以及GKP-buffer对照组的叶片表面白粉菌生长状态(图4-B)。接种白粉菌7 d后, BSMV:实验组植株的叶片上已经出现大量白色菌斑, 2个对照组叶片上也出现菌斑, 但数量少于实验组。这些结果说明降低抗病小麦Brock中基因表达, 能够增加白粉菌成功侵染效率, 小麦叶片的抗病力下降。
2.3.2基因沉默小麦叶片孢子生长情况分析 进一步观察接种白粉菌孢子不同时间点的孢子萌发状况。接种白粉菌孢子48 h后, 对照组叶片上的白粉菌孢子多数无法侵入, 为畸形附着胞(图5-A, B), 而BSMV:实验组叶片上则开始出现附着胞(图5-C); 接种白粉菌72 h后, 对照组中可以发现少量附着胞, 但仍以纤细型或分瓣型的畸形附着胞为主(图5-D, E), BSMV:实验组叶片上开始出现少量次级菌丝(图5-F), 实验组叶片上白粉菌孢子生长速度明显快于对照组, 在接菌早期就侵染成功; 接种白粉菌7 d后, 对照组和实验组叶片上均发育出分生孢子, 但实验组小麦叶片表面的孢子数量更多(图5-G~I)。这些结果说明降低抗病小麦Brock中基因表达后, 小麦叶片上白粉菌孢子萌发速度加快, 分生孢子数量增多, 小麦的抗病力下降。
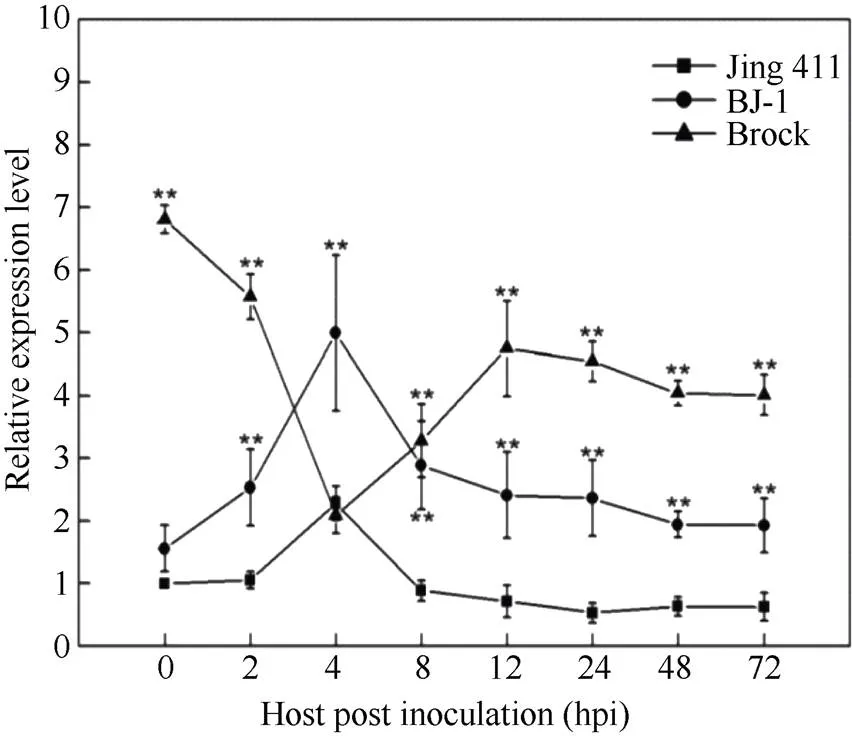
图3 TaRPP13-1B基因的白粉菌诱导表达分析
Jing 411: 感病小麦品种京411; BJ-1: 京411抗病近等基因系; Brock: 抗病小麦品种; Student’s测验,*:< 0.05;**:< 0.01。
Jing 411: wheat susceptible variety; BJ-1: near isogenic line; Brock: wheat resistant variety; Student’s-test,*:< 0.05;**:< 0.01.
2.4 农杆菌转化法鉴定TaRPP13-1B基因的功能
2.4.1 感病小麦津强5号的遗传转化及转基因植株的分子鉴定 利用农杆菌介导的遗传转化法将重组载体转入津强5号小麦成熟胚, 并对该基因的T0代植株进行转基因鉴定, 获得11株T0代植株(图6)。培养转基因苗至T1代, 利用qRT-PCR检测目标基因表达, 发现部分转基因小麦中的目标基因表达量显著高于对照组, 平均为对照组的3.74倍, 最高的达到对照组的5倍(T1-1~T1-4号转基因植株), 最低约为对照组的2.6倍(T1-2和T1-3号转基因植株) (图7)。说明成功转入津强5号小麦并稳定遗传,实现目标基因在受体中表达。
2.4.2T1代转基因植株的抗病性鉴定 为了进一步验证该基因的抗白粉病特性, 分别选取了4个过表达的T1代转基因株系(T1-4、T1-6、T1-8和T1-11)培养至一叶一心期, 利用E09侵染转基因小麦及其对照组, 观察叶片表型。接种白粉菌48 h和72 h时, 野生型和转基因小麦叶片表面的白粉菌孢子的生长情况相近, 无显著差别。白粉菌侵染7 d后, 野生型和转基因小麦叶片表面都出现白色菌落, 但转基因小麦叶片表面菌落少于野生型(图8-A)。为了进一步确定转基因小麦对白粉病的抗病程度, 利用ImageJ对转基因小麦及其对照组叶片病斑面积进行统计分析, 结果如图8-B所示, 对照组叶片病斑面积为64.6%, 发病等级为4级(感病)。4个转基因小麦叶片病斑面积较小, 在10%~25%之间, 发病等级均为2级。以上结果说明, 过表达提高了感病小麦津强5号的抗病性,基因是抗白粉病相关基因。
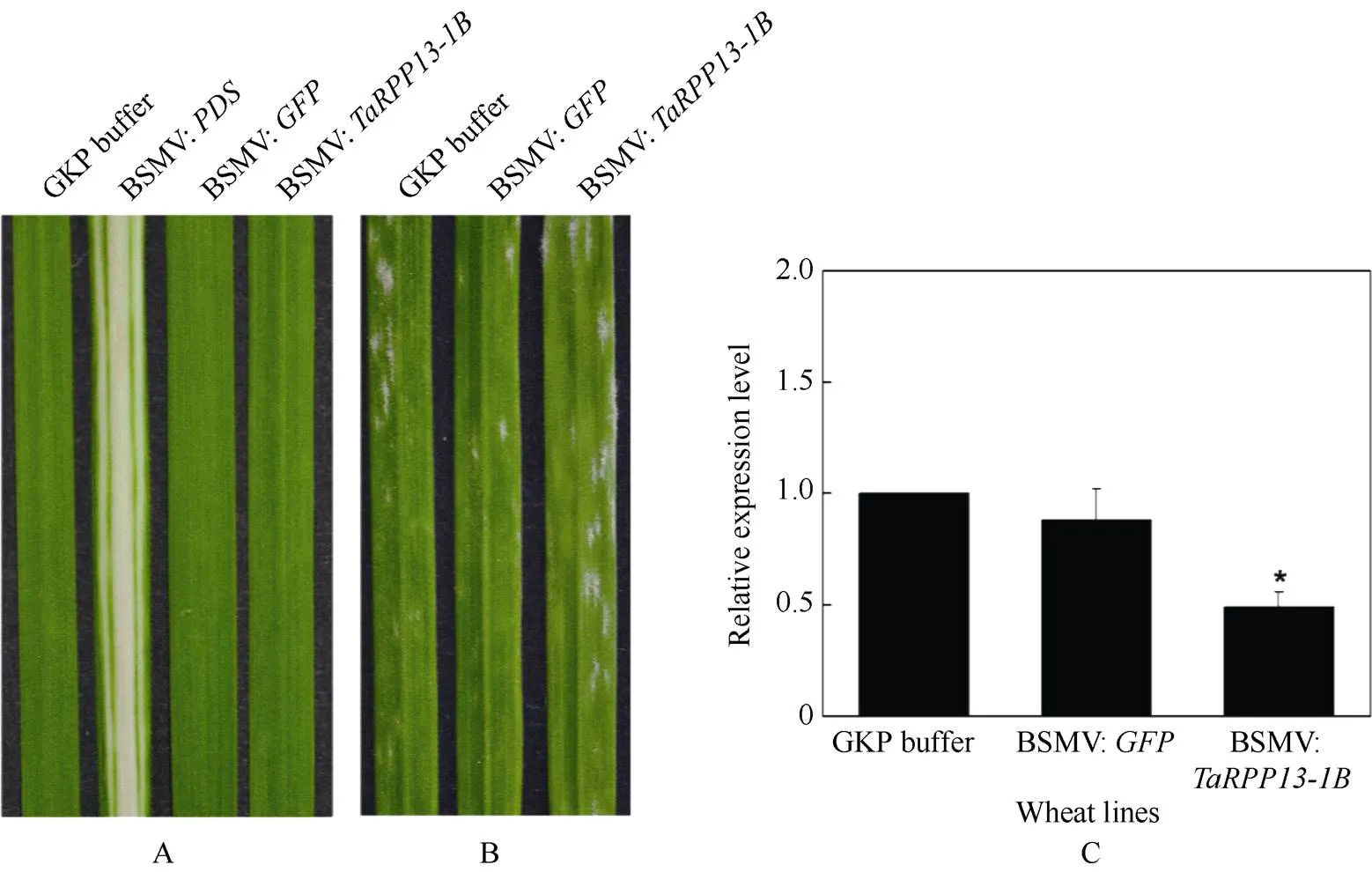
图4 沉默TaRPP13-1B后小麦抗病性变化
A: Brock接种BSMV病毒后表型; B: 沉默叶片接种白粉菌E09的表型观察; C: 各实验组中基因的相对表达量(*:< 0.05)。
A: phenotype of Brock leaves inoculated with BSMV; B:-knockdown plants inoculated withE09; C: the relative expression of(*:< 0.05).
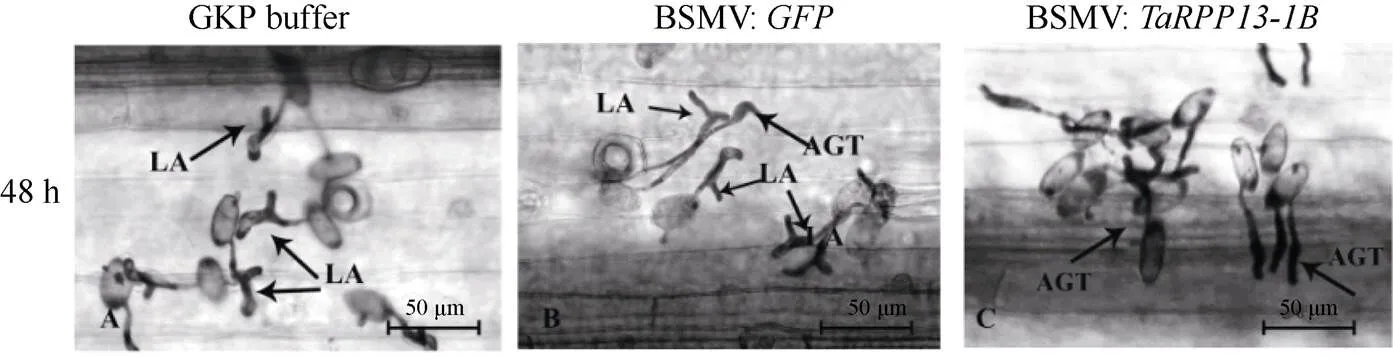
(图5)
LA: 分瓣型畸形附着胞; SA: 纤细型附着胞; AGT: 喙型附着胞; SH: 次生菌丝; BC: 念珠状分生孢子。
LA: lobed appressoria; SA: slender appressoria; AGT: appressorium germ tube; SH: the secondary hypha; BC: beaded conidia
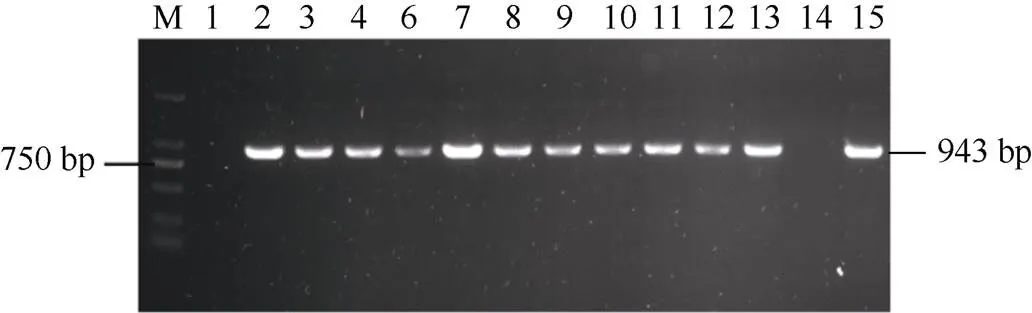
图6 TaRPP13-1B T0代转基因小麦的PCR检测
M: DL2000 marker; 1: 1301对照组; 2: 质粒阳性对照; 3~14: T0代转基因植株。
M: DL2000 marker; 1: control; 2: positive control; 3–14: T0transgenic wheat.
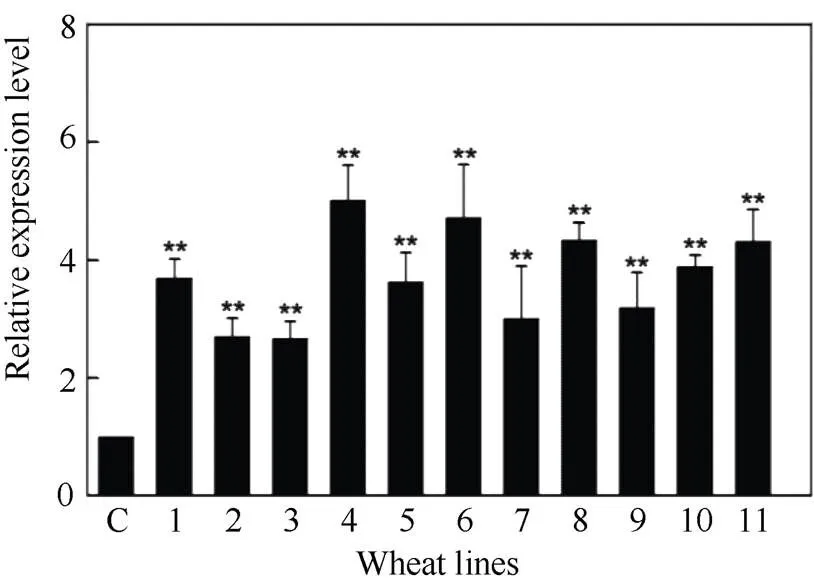
图7 TaRPP13-1B在T1代转基因小麦中的表达量分析
C: 对照组; 1~11: T1-1~T1-11 转基因单株(**:< 0.01)。
C: control; 1–11: T1-1–T1-11: transgenic wheat (**:< 0.01).
3 讨论
植物中克隆了大量的CNL类抗病基因。是Yahiaoui等[31]利用图位克隆法克隆得到第1个抗白粉病基因, 包含典型的CNL结构域。Brunner等[32-33]将及其等位基因转化感病六倍体小麦, 所有转基因小麦均表现出田间抗白粉菌特性。也是一个典型的CNL抗病基因。目前, 通过国家和省级品种审定携带基因小麦新品种有30多个,基因是中国小麦白粉病抗性育种利用的重要抗源[3,34-35]。[36-39]、[40]和(及其等位基因)[41-43]也都是CNL抗白粉病基因, 携带这些抗性基因的小麦品种均表现出良好的田间抗性。这些已有研究结果说明CNL类基因在小麦白粉病防治上发挥了重要作用。
前期研究中, 我们对感病小麦京411及其抗病近等基因系BJ-1的染菌前后的转录组分析中发现了位于染色体1B的同源基因,该基因在白粉菌侵染后表达量上调, 且在抗病材料上调明显[24]。本研究以为基础, 结合RACE技术, 克隆了Brock、BJ-1和京411中基因cDNA序列, 3个小麦品种中的cDNA序列相同, 它与二穗短柄草(XP_010237386.1)有较高同源性(93%), 与乌拉尔图小麦(EMS68464.1)有较高同源性(79%)。虽然不同抗性小麦品种中的基因cDNA序列相同, 但目标基因在抗/感小麦品种中的表达量差异显著, 抗病小麦中表达水平高于感病小麦。所以我们推测可能是不同小麦品种中的启动子区或上游调控基因存在差异, 但仍需进一步研究证明。与基因一样[13], 它们都属于基因家族。在IWGSC进行序列比对分析后发现, 二者位于不同的染色体, 序列一致性仅有23.2%。根据二者基因序列很难预测基因功能之间的联系。
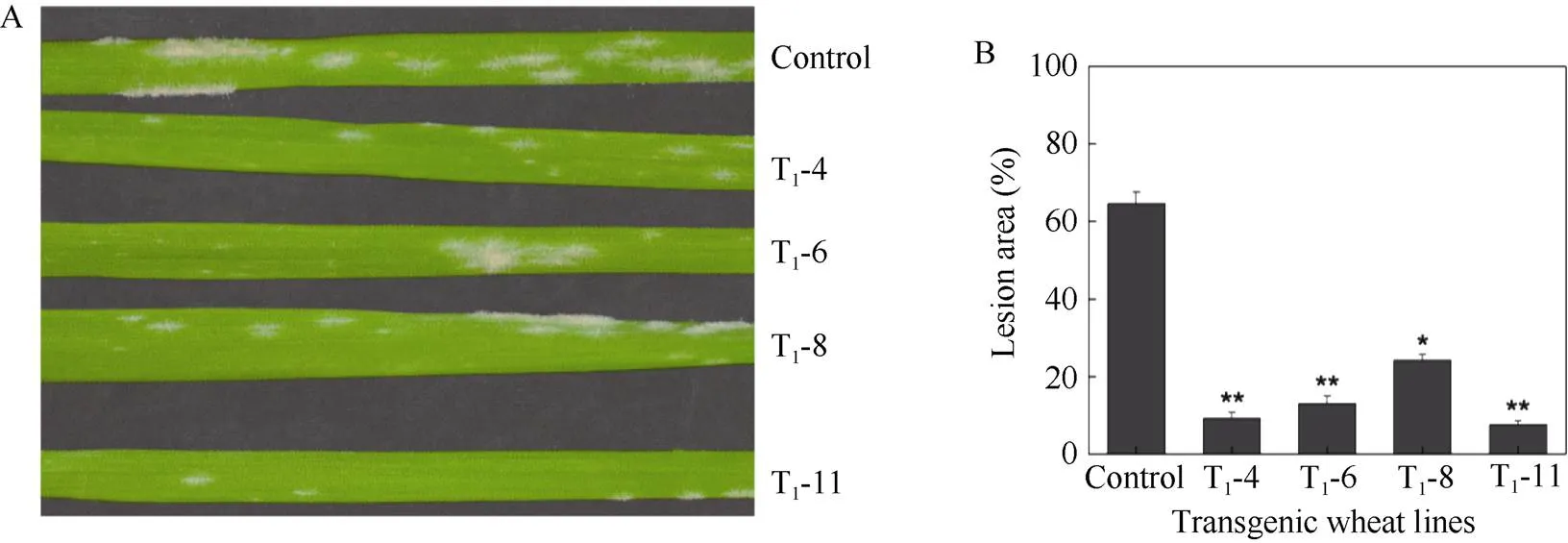
图8 过表达TaRPP13-1B基因提高小麦对白粉菌E09的抗性
A: 转基因小麦在白粉菌E09侵染7 d后的叶片表型; B: 白粉菌E09侵染下的小麦叶片病斑面积(**:< 0.01)。
A: phenotype of transgenic wheat leaves inoculated withE09 for 7 days; B: the lesion area in transgenic wheat leaves (**:< 0.01).
利用VIGS技术沉默基因, 白粉菌侵染实验表明, 接菌48 h和72 h后, 实验组叶片上白粉菌生长速度明显快于对照组, 说明目标基因表达量降低有利于病原菌侵染, 目标基因在病原菌侵染早期发挥作用。接菌7 d后的实验组和对照组叶片上都出现肉眼可见的白粉菌孢子, 但对照组叶片上孢子堆较少, 这表明基因表达被抑制后在一定程度上降低了抗病小麦植株的抗性。进一步构建过表达基因的转基因小麦, 接种白粉菌7 d, 发现转基因小麦叶片表面白粉菌生长数量和状态明显慢于野生型津强5号。这些研究结果进一步确认,基因参与小麦-白粉菌互作过程, 发挥正向调节作用。
4 结论
本研究利用RACE技术克隆了小麦。沉默/过表达目标基因后的功能研究表明, 该基因参与小麦对白粉菌侵染的早期抗病防御反应, 在小麦-白粉菌互作过程中发挥正向作用, 研究结果为小麦白粉病抗性品种的选育提供备选基因。
[1] Kourelis J, Van Der Hoorn R A L. Defended to the nines: 25 years of resistance gene cloning identifies nine mechanisms for R protein function., 2018, 30: 285–299.
[2] Zou S H, Wang H, Li Y W, Kong Z S, Tang D Z. The NB-LRR geneconfers powdery mildew resistance in wheat., 2018, 218: 298–309.
[3] Xing L P, Hu P, Liu J Q, Witek K, Zhou S, Xu J F, Li W H, Gao L, Huang Z P, Zhang R Q, Wang X E, Chen P D, Wang H Y, Jones D G, Karafiátová M, Vrána J, Bartoš J, Doleže l J, Tian Y C, Wu Y F, Cao A Z.fromencodes a CC-NBS-LRR protein conferring powdery mildew resistance in wheat., 2018, 11: 874–878.
[4] Li M M, Dong L L, Li B B, Wang Z Z, Xie J Z, Qiu D, Li Y H, Shi W Q, Yang L J, Wu Q H, Chen Y X, Lu P, Guo G H, Zhang H Z, Zhang P P, Zhu K Y, Li Y W, Zhang Y, Wang R G, Yuan C G, Liu W, Yu D Z, Luo M C, Fahima T, Nevo E, Li H J, Liu Z Y. A CNL protein in wild emmer wheat confers powdery mildew resistance., 2020, 228: 1027–1037.
[5] Xie J Z, Guo G H, Wang Y, Hu T Z, Wang L L, Li J T, Qiu D, Li Y H, Wu Q H, Lu P, Chen Y X, Dong L L, Li M M, Zhang H Z, Zhang P P, Zhu K Y, Li B B, Deal K R, Huo N X, Zhang Y, Luo M H, Liu S Z, Gu Y Q, Li H J, Liu Z Y. A rare single nucleotide variant inconfers powdery mildew resistance in common wheat., 2020, 228: 1011–1026.
[6] Koller T, Brunner S, Herren G, Hurni S, Keller B. Pyramiding of transgenicalleles in wheat results in improved powdery mildew resistance in the field., 2018, 131: 861–871.
[7] Hall S A, Allen R L, Baumber R E, Baxter L A, Fisher K, Bittner-Eddy P D, Rose L E, Holub E B, Beynon J L. Maintenance of genetic variation in plants and pathogens involves complex networks of gene-for-gene interactions., 2009, 10: 449–457.
[8] Allen R L, Meitz J C, Baumber R E, Hall S A, Lee S C, Rose L E, Beynon J L. Natural variation reveals key amino acids in a downy mildew effector that alters recognition specificity by anresistance gene., 2008, 9: 511–523.
[9] Rose L E, Bittner-Eddy P D, Langley C H, Holub E B, Beynon J L. The maintenance of extreme amino acid diversity at the disease resistance gene,, in., 2004, 166: 1517–1527.
[10] Rentel M C, Leonelli L, Dahlbeck D, Zhao B, Staskawicz B J. Recognition of theeffector ATR13 triggers resistance against oomycete, bacterial, and viral pathogens., 2008, 105: 1091–1096.
[11] Ramachandran S R, Yin C, Kud J, Tanaka K, Mahoney A K, Xiao F, Hulbert S H. Effectors from wheat rust fungi suppress multiple plant defense responses., 2017, 107: 75–83.
[12] Hu W G, Wang Q H, Wang S W, Wang M M, Wang C Y, Tian Z R, Liu X L, Ji W Q, Zhang H. Gene co-expression network analysis provides a novel insight into the dynamic response of wheat to powdery mildew stress., 2020, 99: 44–45.
[13] Liu X Y, Zhang C Q, Zhang L L, Huang J G, Dang C, Xie C J, Wang Z Y., a CC-NBS-LRR like gene located on chr 7D, promotes disease resistance to wheat powdery mildew in Brock., 2020, 168: 688–699.
[14] 王艳红, 肖莹, 郑舒扬, 刘晓颖, 王振英. 白粉菌诱导的小麦品种Brock的差异表达基因解析. 天津师范大学学报(自然科学版), 2015, 35(2): 71–76.
Wang Y H, Xiao Y, Zheng S Y, Liu X Y, Wang Z Y. Analysis on differentially expressed genes of wheat Brock inoculated byf. sp..(Nat Sci Edn), 2015, 35(2): 71–76 (in Chinese with English abstract).
[15] Ramachandran S R, Yin C, Kud J, Tanaka K, Mahoney A K, Xiao F, Hulbert S H. Effectors from wheat rust fungi suppress multiple plant defense responses., 2016, 107: 75–83.
[16] Chen J H, Zhang F Y, Zhao C J, Lyu G G, Sun C W, Pan Y B, Guo X Y, Chen F. Genome-wide association study of six quality traits reveals the association of thegene with flour colour in Chinese bread wheat., 2019, 17: 2106–2122.
[17] 刘登全, 王园秀, 崔朝宇, 秦双林, 欧阳慧, 蒋军喜. 晚熟温州蜜柑 NBS-LRR类抗病基因同源序列的克隆及分析. 江西农业大学学报, 2016, 38: 83–89.
Liu D Q, Wang Y X, Cui C Y, Qin S L, Ou-Yang H, Jiang J X. Cloning and analysis of NBS-LRR disease-resistant gene analogs inMarc., 2016, 38: 83–89 (in Chinese with English abstract).
[18] Prabhukarthikeyan S R, Manikandan R, Durgadevi D, Keerthana U, Harish S, Karthikeyan G, Raguchander T. Bio-suppression of turmeric rhizome rot disease and understanding the molecular basis of tripartite interaction among,and., 2017, 111: 23–31.
[19] Chen T, Lv Y, Zhao T M, Li N, Yang Y w, Yu W, He X, Liu T, Zhang B L. Comparative transcriptome profiling of a resistant vs. susceptible tomato () cultivar in response to infection by tomato yellow leaf curl virus., 2013, 8: e80816.
[20] Han L J, Weng K, Ma H, Xiang G Q, Li Z Q, Wang Y J, Liu G T, Xu Y. Identification and characterization of erysiphe necator-responsive MicroRNAs in Chinese wild vitis pseudoreticulata by high-throughput sequencing., 2016, 7: 621.
[21] Khan S A, Zhang C, Ali N, Gandeka M. Highdensity SNP map facilitates fine mapping of QTLs and candidate genes discovery forresistance in peanut ()., 2020, 133: 2239–2257.
[22] Howlader J, Robin A, Natarajan S, Biswas M K, Nou I S. Transcriptome analysis by RNA-Seq reveals genes related to plant height in two sets of parent-hybrid combinations in easter lily ()., 2020, 10: 9082–9097.
[23] 黄建国. 小麦抗白粉病基因 SSR 分子标记筛选及AP2/ERF转录因子家族分析. 天津师范大学硕士学位论文, 天津, 2021.
Huang J G. Screening for SSR Markers Linked to Powdery Mildew Resistance Gene and Bioinformatics Analysis of AP2/ERF Transcription Factor Family in Wheat. MS Thesis of Tianjin Normal University, Tianjin, China, 2021 (in Chinese with English abstract).
[24] 刘晓颖. 小麦品种Brock响应白粉菌侵染早期抗病相关基因表达和功能分析. 中国农业大学博士学位论文, 北京, 2021.
Liu X Y. Expression and Functional Analysis of Genes Associated with Early Powdery Mildew Resistance in Wheat Variety Brock. PhD Dissertation of China Agricultural University, Beijing, China, 2021 (in Chinese with English abstract).
[25] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2‒ΔΔCTmethod., 2001, 25: 402–408.
[26] Liu X Y, Wang J Y, Fan B L, Shang Y T, Sun Y F, Dang C, Xie C J, Wang Z Y, Peng Y K. Agene in wheat contributes to the early defence response against wheat powdery mildew., 2018, 166: 116–122.
[27] 栗聪, 雒景吾, 张磊, 田增荣, 刘新伦, 吉万全. 小麦成熟胚再生体系优化及优良受体基因型筛选. 麦类作物学报, 2014, 34: 583–590.
Li C, Luo J W, Zhang L, Tian Z R, Liu X L, Ji W Q. Optimizing the regeneration system from mature embryo and screening of elite wheat genotypes., 2014, 34: 583–590 (in Chinese with English abstract).
[28] 王艳丽, 叶兴国, 刘艳鹏, 杜丽璞, 徐惠君. 农杆菌敏感小麦基因型的筛选研究. 麦类作物学报, 2005, 25(6): 6–10.
Wang Y L, Ye X G, Liu Y P, Du L P, Xu H J. Screening of wheat genotype sensitive toinfection., 2005, 25(6): 6–10 (in Chinese with English abstract).
[29] 王怡, 彭永康. 抗、感白粉病春小麦感染白粉菌后抗病特性的细胞学观察. 天津农业科学, 2014, 20(4): 1–4.
Wang Y, Peng Y K. Cytological analysis of spring wheat with different resistance to powdery mildew induced by Bgt., 2014, 20(4): 1–4 (in Chinese with English abstract).
[30] 河南省植保站. 河南省主要农作物病虫害测报办法. 郑州: 河南科学技术出版社, 1995. pp 11–18.
Plant Protection and Quarantine Station of Henan Province. Forecasting Methods of the Main Crop Diseases and Insect Pests in Henan Province. Zhengzhou: Henan Science and Technology Press, 1995. pp 11–18 (in Chinese).
[31] Yahiaoui N, Srichumpa P, Dudler R, Keller B. Genome analysis at different ploidy levels allows cloning of the powdery mildew resistance genefrom hexaploid wheat., 2004, 37: 528–538.
[32] Brunner S, Hurni S, Herren G, Kalinina O, Burg S V, Zeller S L, Schmid B, Winzeler M, Keller B. Transgenicwheat lines show resistance to powdery mildew in the field., 2011, 9: 897–910.
[33] Brunner S, Stirnweis D, Quijano C D, Buesing G, Herren G, Parlange F, Barret P, Tassy C, Sautter C, Winzeler M. Transgenicmultilines of wheat show increased powdery mildew resistance in the field., 2012, 10: 398–409.
[34] He H G, Zhu S Y, Zhao R H, Jiang Z N, Ji Y Y, Ji J, Qiu D, Li H J, Bie T D., encoding a typical CC-NBS-LRR protein, confers broad-spectrum resistance to wheat powdery mildew disease., 2018, 11: 879–882.
[35] 王海燕, 肖进, 袁春霞, 徐涛, 于春艳, 孙昊杰, 陈佩度, 王秀娥. 携带抗白粉病基因的小麦–簇毛麦小片段易位染色体在不同小麦背景中的传递率及遗传稳定性. 作物学报, 2016, 42: 361–367.
Wang H Y, Xiao J, Yuan C X, Xu T, Yu C Y, Sun H J, Chen P D, Wang X E. Transmission and genetic stability of no-homoeologous small fragment wheat–translocation chromosomes within various cultivar backgrounds of common wheat., 2016, 42: 361–367 (in Chinese with English abstract).
[36] Sánchez-Martín J, Steuernagel B, Ghosh S, Herren G, Hurni S, Adamski N, Vrána J, Kubaláková M, Krattinger S G, Wicker T, Doležel J, Keller B, Wulff B B H. Rapid gene isolation in barley and wheat by mutant chromosome sequencing., 2016, 17: 221.
[37] 陈芳, 乔麟轶, 李锐, 刘成, 李欣, 郭慧娟, 张树伟, 常利芳, 李东方, 阎晓涛, 任永康, 张晓军, 畅志坚. 小麦新种质CH1357抗白粉病遗传分析及染色体定位. 作物学报, 2019, 45: 1503–1510.
Chen F, Qiao L Y, Li R, Li C, Li X, Guo H J, Zhang S W, Chang L F, Li D F, Yan X T, Ren Y K, Zhang X J, Chang Z J. Genetic analysis and chromosomal localization of powdery mildew resistance gene in wheat germplasm CH1357., 2019, 45: 1503–1510 (in Chinese with English abstract).
[38] 陈芳. 小偃麦衍生品系抗白粉病基因的遗传定位及图位克隆. 山西大学博士论文, 山西太原, 2020.
Chen F. Genetic Mapping and Positional Cloning of Powdery Mildew Resistance Genes in the Derived Lines from a Wheat-Hybrid. PhD Dissertation of Shanxi University, Taiyuan, Shanxi, China, 2020 (in Chinese with English abstract).
[39] 李根桥, 房体麟, 朱婕, 高亮亮, 李闪, 解超杰, 杨作民, 孙其信, 刘志勇. 普通小麦品种Brock抗白粉病基因分子标记定位.作物学报, 2009, 35: 1613–1619.
Li G Q, Fang T L, Zhu J, Gao L L, Li S, Xie C J, Yang Z M, Sun Q X, Liu ZY. Molecular identification of a powdery mildew resistance gene from common wheat cultivar Brock., 2009, 35: 1613–1619 (in Chinese with English abstract).
[40] Zou S H, Wang H, Li Y W, Kong Z S, Tang D Z. The NB-LRR geneconfers powdery mildew resistance in wheat., 2017, 218: 298–309.
[41] Zhou F S, Kurth J, Wei F S, Elliott C, Valè G, Yahiaoui N, Keller B, Somerville S, Wise R, Schulze-Lefert P. Cell-autonomous expression of Barleyconfers race-specific resistance to the powdery mildew fungus via aindependent signaling pathway., 2001, 13: 337–350.
[42] Halterman D A, Wei F, Wise R P. Powdery mildew-inducedmRNAs are alternatively spliced and contain multiple upstream open reading frames., 2003, 131: 558–567.
[43] Chang C, Yu D S, Jiao J, Jing S J, Schulze-Lefert P, Shen Q H. Barley MLA immune receptors directly interfere with antagonistically acting transcription factors to initiate disease resistance signaling., 2013, 25: 1158–1173.
Cloning and functional analysis ofgene related to powdery mildew resistance in wheat cultivar Brock
LIU Xiao-Ying1, ZHANG Chi1, WANG Xue-Qing1, YANG Chen-Xiao1, WANG Guang-Yu1, BIAN Yun-Di1, FANG Fang2, WANG Ying2, and WANG Zhen-Ying1,*
1College of Life Sciences, Tianjin Normal University, Tianjin Key Laboratory of Animal and Plant Resistance, Tianjin 300387, China;2Forestry Development Service Center of Baodi District, Tianjin 301899, China
Powdery mildew caused byf. sp.() is a severe wheat disease in China. Cloning and pyramiding of different resistance genes to improve crop disease resistance is one of the most cost-effective methods. In this study,on chromosome 1B, which encodes the CC, NB-ARC, and LRR domains, was isolated from common wheat. The relative expression level ofin Brock and BJ-1 fluctuated afterinoculation, but the average expression levels were always higher than the susceptible wheat Jing 411. The function ofwas elucidated by virus-induced gene silencing (VIGS) and overexpression transgenic technique. Silencing ofresulted in decreased disease resistance in Brock. Overexpression ofimproved disease resistance in the transgenic wheat seedlings of Jinqiang 5 cultivar. The above results demonstrated thatwas involved in the defense response of wheat to powdery mildew, which provided valuable genetic resources for breeding of resistant varieties.
wheat;gene;; powdery mildew resistance
10.3724/SP.J.1006.2023.21003
本研究由国家自然科学基金项目(31071671)和天津市科技支撑项目(18YFZCNC01100, 17JCZDJC34100)资助。
This study was supported by the National Natural Science Foundation of China (31071671) and the Tianjin Science and Technology Program (18YFZCNC01100, 17JCZDJC34100).
王振英, E-mail: skywangzy@tjnu.edu.cn
E-mail: skylxy@tjnu.edu.cn
2022-01-16;
2022-05-05;
2022-05-25.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220525.1330.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

