基于3D UNet结合Transformer的肝脏及肝肿瘤自动分割
戴振晖,简婉薇,朱琳,张白霖,靳怀志,杨耕,谭翔,王学涛
广州中医药大学第二附属医院 放射治疗区,广东 广州 510006
引言
肝癌是临床常见的恶性肿瘤,其发病率和死亡率呈逐年增加的趋势[1]。放射治疗是肝肿瘤的有效治疗手段之一,而肝脏和肝肿瘤的勾画是放射治疗的关键步骤,需要医生在患者CT图像上手动逐层勾画,耗时耗力,并且放疗医生之间的勾画存在主观差异,因此在临床上需要探寻自动分割肝脏和肝肿瘤的工具[2-4]。然而在CT图像中肝脏与周围危及器官的强度对比度低,难以精确分割肝脏,同时肝肿瘤对于不同患者在大小、形状、位置和数量各异,肝肿瘤在肝脏内的边界并不清晰,这给肝脏和肝肿瘤自动分割带来了挑战[5]。
为解决以上问题,已有研究者提出了不同的分割模型,如基于强度阈值、区域增长和机器学习等方法的模型。Zheng等[6]基于统一水平集方法融合了区域信息和边缘信息来加强轮廓识别;Wong等[7]提出基于知识约束的2D区域生长方法肿瘤分割模型;Kuo等[8]提出学习带有纹理特征向量的支持向量机分类器来进行肝肿瘤分割。然而这些方法依赖于手工勾画的特征,并且特征表达能力有限。最近基于卷积神经网络(Convolutional Neural Network,CNN)的深度学习方法在肝脏和肝肿瘤分割问题上取得了巨大成功[9-10],其中UNet已成为医学图像分割任务中最流行的卷积网络[11-12]。但传统的UNet仍然存在一定的缺陷,例如,为提高学习效率,跳跃连接层传输特征图中低分辨率信息,导致图像特征模糊[13]。Han等[14]提出了结合UNet的长程连接和ResNet的短程残差连接的2.5D深度CNN,并认为2.5D模型具有更深、更宽的网络架构,同时能容纳3D信息。Li等[15]提出了一种新颖混合密集连接H-Dense UNet,可以有效探测并优化切片内特征和3D上下层信息,以实现准确的肝脏和肿瘤分割,肝脏和肝肿瘤分割结果的Dice相似性系数(Dice Similarity Coefficient,DSC)分别是0.961和0.722。Liu等[16]提出了一个空间特征融合卷积网络从CT图像中分割肝脏和肝肿瘤,肝脏和肝肿瘤分割结果的DSC分别是0.937和0.592。Bi等[17]通过具有多尺度融合的新型级联ResNet架构能够更精确地区分肝脏与肝脏病变的边界。Kaluva等[18]利用密集连接全CNN(DenseNet)对CT图像中的肝脏及其肿瘤进行全自动两阶段级联分割,肝脏和肝肿瘤分割结果的DSC分别是0.912和0.492。本文拟在3D UNet中加入了Res残差模块和Swim Transformer模块,提出了一个新型的卷积和Transformer结合的Res-Swim-UNet模型,此外,本文采用两阶段的学习策略,该策略首先在腹部CT图像中定位肝脏,然后在肝脏中定位肝肿瘤,使用两阶段策略可以解决肝肿瘤小目标分割的困难并降低分割假阳性,以期有效地提取局部和全局图像特征,实现肝脏和肝肿瘤的精确分割。
1 资料与方法
1.1 数据集
本文使用MICCAI 2017 LiTS挑战赛数据集训练自动分割模型,训练完成的模型分别在LiTS数据集和本地数据集上测试。LiTS数据集包含131例腹部CT图像和放疗医师手动勾画肝脏及肝肿瘤轮廓,图像像素矩阵为512×512,层厚范围为0.45~5.00 mm,本地数据集包含16例肝肿瘤患者CT图像和放疗医师手动勾画的肝脏及肝肿瘤轮廓,本地数据在西门子Sensation Open(Siemens Healthcare,Forchheim,德国)CT扫描仪获取,像素矩阵为512×512,层厚为3 mm。100例LiTS数据用于训练分割模型,10例LiTS数据用于验证,分别用21例LiTS数据和16例本地数据测试分割模型性能。放疗医师手动勾画的轮廓作为 Ground Truth(GT),自动分割轮廓与GT进行对比,以评估自动分割模型的性能。
1.2 算法流程
本文提出了一个两阶段的肝脏和肝肿瘤分割算法,算法流程如图1所示,主要包括4个部分:预处理、肝脏分割(第一阶段)、肿瘤分割(第二阶段)和后处理。① 预处理阶段:使用阈值法和形态学方法从原始CT图像生成躯体掩膜,基于躯体掩膜,本文从原始CT图像中裁剪出有效的躯体部分并对其进行归一化以进行深度神经网络的训练。本文将所有图像的像素分辨率归一化为0.7 mm×0.7 mm×1.0 mm,截取CT值范围为-70~150 HU并归一化为0~1。肝脏分割和肿瘤分割阶段依次执行,均使用本文提出的Res-Swim-UNet深度神经网络模型。肝脏和肿瘤分割模型只需要分别关注自己的分割目标,这可以简化它们单个模型的任务难度,使它们可以更加精准地分割单一目标。② 肝脏分割阶段:模型需要从腹部CT图像中定位肝脏并将其精确分割;基于分割出的肝脏区域,肿瘤分割模型仅需要从肝脏器官中分割肿瘤,这剔除了肝脏以外的组织的干扰,明显降低了肿瘤定位的难度。③ 后处理阶段:本文通过取最大连通域方法去除肝脏分割结果中的假阳性,然后合并肝脏和肿瘤的分割结果,最后将其放回原始图像中的对应位置并将图像插值回原始尺寸。
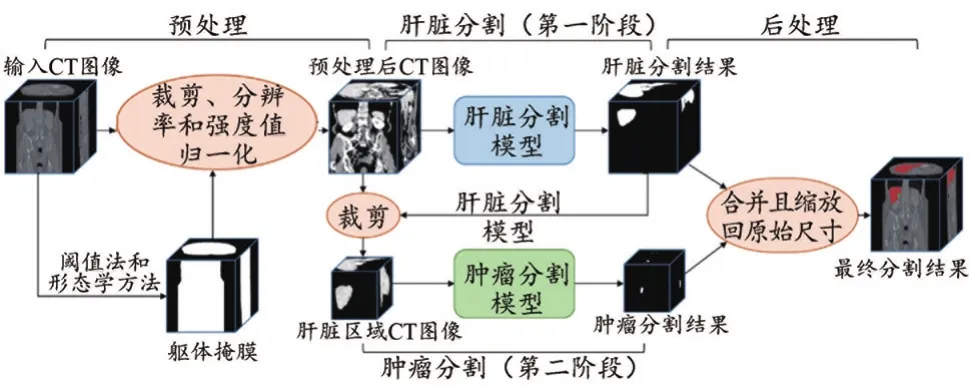
图1 肝脏和肝肿瘤分割流程图
1.3 Res-Swim-UNet网络结构
鉴于Res残差模块和具有窗口移动的Swim transformer模块在图像特征提取的高效性能[19-20],本文分别在3D UNet中加入了Res残差模块和Swim Transformer模块,提出了一个新型的卷积和Transformer结合的Res-Swim-UNet模型。与常规3D UNet网络一样,本文的网络框架包括编码器、解码器、瓶颈层和跳跃连接4个部分(图2)。编码器部分由6个卷积层、2个残差模块和4个最大池化层构成。编码器通过卷积层和残差模块提取图像特征,使用最大池化层对特征进行下采样,最终可获得5个分辨率水平的特征图。跳跃连接部分通过特征拼接的方式将编码器的多尺度特征图传输给解码器。解码器与编码器结构对称,同样包括6个卷积层和2个残差模块,而解码器中的最大池化层变为4次插值将图放大回原始分辨率,此外最后一层增加了一层1×1×1的卷积和sigmoid激活函数以生成预测概率图。瓶颈层在U型结构的最底部,这里的特征分辨率最低。由于Swim Transformer模块的计算量随分辨率大小呈线性关系,本文仅在瓶颈层加入了2个Swim Transformer模块,这既降低了模型计算成本,又可以有效提高模型性能。
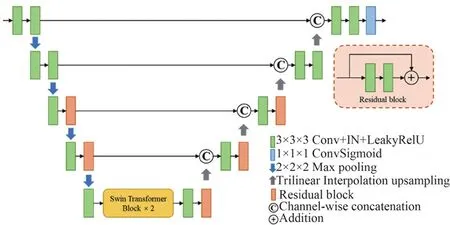
图2 Res-Swim-UNet网络结构
传统的Transformer使用多头自注意力模块建立全局信息依赖,使其可以更好地提取和分析全局信息。而Swim Transformer是基于移动窗口构建的(图3),Swim Transformer模块由连续的Swim transformer子模块构成。每个Swim transformer子模块包括归一化层、多头自注意力模块,残差连接和一个两层的多层感知机(Multilayer Perceptron, MLP)。基于窗口的多头自注意力模块(Window Multi-heads Self-Attention,W-MSA)和基于滑动窗口的多头自注意力模块(Shifted Window Multi-heads Self-Attention,SW-MSA)分别用于2个连续的Swim transformer子模块。Swim Transformer模块的可用公式(1)~(4)描述。

图3 Swim Transformer模块
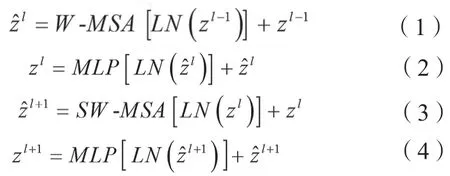
式中,和zl分别表示W-MSA模块和MLP模块的第lth模块的输出,LN表示层归一化(Layer Normalization),其中自注意力模块可表示为公式(5)。
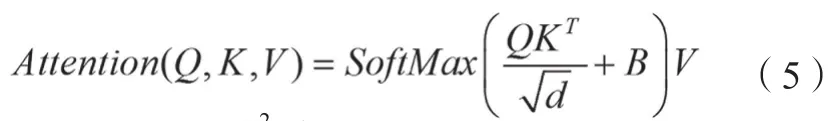
式中,Q,K,VRM2×d为3个矩阵,由输入特征图分别经过3个卷积层得到。KT为K的转置矩阵,d为比例缩放因子本文中设置为64,B为可学习的偏置参数。
1.4 损失函数和训练细节
本文分别训练肝脏分割模型和肿瘤分割模型,损失函数均为交叉熵损失与Dice损失之和。交叉熵损失的定义如公式(6)所示。

式中,a和b分别表示标签图像和模型预测图像,N表示输入图像的像素总个数,ai表示图像a中第i个像素的值。Dice损失如公式(7)所示。
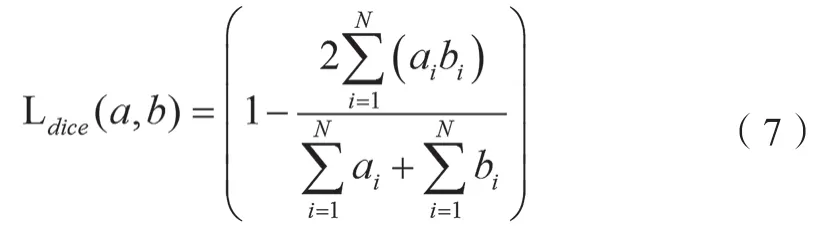
在训练过程中,本文使用超参数a来平衡2个损失函数的影响,具体见公式(8)。

肝脏和肿瘤分割模型均基于块训练,在训练阶段,随机从图像中截取像素矩阵为128×128×128大小的图像块作为模型输入。使用在线的数据扩增技术以提高模型泛化能力,扩增操作包括随机缩放、随机旋转、随机翻转、伽马变换、随机噪声和弹性形变。模型训练使用Adam优化器和ploy的学习率下降策略,基础学习率为0.0002[21]。损失函数中的超参数a设置为0.5,在12 GB内存的NVIDIA Titan Xp GPU进行模型训练,训练时长约21 h,在测试阶段,每例患者数据的处理时间约为40 s。
1.5 实验设置
本文进行了对比实验,以分析所提出的Res-Swim-UNet模型的有效性,基于100例LiTS数据集分别训练原始UNet模型和Res-Swim-UNet模型,对比2个分割模型的性能。为验证本文提出的模型在临床应用中的泛化能力,本文在本地数据集上测试了所提出模型的性能。
1.6 评估指标
本文选择了多个度量指标从多个角度评估所提出模型的肝脏和肝肿瘤分割性能,分别为DSC、体积重叠误差(Volumetric Overlap Error,VOE),其中DSC值越大表示分割结果越好,VOE越小表示分割结果越好。以DSC=0.6作为肝肿瘤分割优劣的阈值,DSC≥0.6评为优,DSC<0.6评为劣,分别计算入选优组和劣组患者的肿瘤长轴平均长度。其中DSC计算方式如公式(9)所示,VOE计算方式如公式(10)所示。
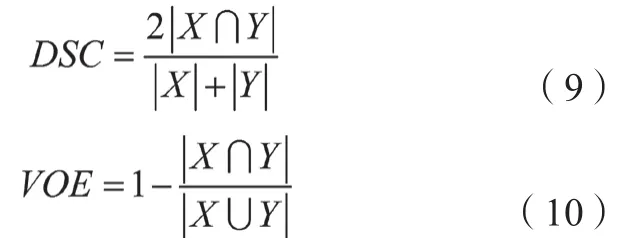
式中,X表示放疗医生手动勾画轮廓,Y表示自动分割的轮廓。
2 结果
2.1 Res-Swim-UNet模型与UNet对比
如表1~2所示,在肝脏分割结果上Res-Swim-UNet模型相对于UNet模型DSC提高了1.6%,VOE降低了1.3%;在肝肿瘤分割结果上Res-Swim-UNet模型相对于UNet模型DSC提高了13.5%,VOE降低了5.9%,证实本文提出的分割模型与原始UNet模型相比有一定的优越性。同时与现有文献[16-17]在LiTS数据集上的分割指标相比,本文提出的模型同样在DSC指标上优势明显。如表3所示,对于肝肿瘤分割,Res-Swim-UNet模型分割结果未达到优的肿瘤长轴平均长度为18.61 mm,分割结果达到优的肿瘤长轴平均长度为49.38 mm;UNet模型分割结果未达到优的肿瘤长轴平均长度为23.10 mm,分割结果达到优的肿瘤长轴平均长度为44.93 mm。Res-Swim-UNet模型自动分割轮廓和手动勾画轮廓对比结果如图4所示,其中红色线表示手动勾画的肝脏轮廓,绿色线表示自动分割的肝脏轮廓,蓝色线表示手动勾画的肝肿瘤轮廓,黄色线表示自动分割的肝肿瘤轮廓,模型自动分割轮廓和医生手动勾画轮廓基本吻合,模型在本地数据上自动分割轮廓与手动勾画轮廓的一致性比公共数据集上低。

表1 肝脏分割结果

表2 肝肿瘤分割结果

表3 不同大小肿瘤分割效果评估

图4 Res-Swim-UNet模型分割结果
2.2 Res-Swim-UNet模型在公共数据集和本地数据集对比
如表1~2所示,本文提出的模型在LiTS公共数据集上肝脏分割结果的DSC、VOE分别是0.957、0.522,肝肿瘤分割结果的DSC、VOE分别是0.672、0.617;在本地数据集上肝脏分割结果的DSC、VOE分别是0.895、0.552,肝肿瘤分割结果的DSC、VOE分别是0.589、0.706。虽然受数据源的影响,Res-Swim-UNet模型在本地数据集上分割结果比公共数据的分割结果略有下降,但仍能在本地数据维持较高的分割精度,证明了本文提出的模型在不同数据集的有效性和鲁棒性。
3 讨论
肝脏和肝肿瘤分割是肝癌放疗计划设计中重要的步骤,自动分割模型可以提供肝脏以及肝肿瘤的精确轮廓,有助于提高放疗医生的工作效率。虽然已有研究提出用于自动分割靶区和危及器官的各种算法,但由于CT图像存在噪声,而且肝脏周围器官的组织对比度低,肝脏和肝肿瘤的自动分割仍然难以获得较高的分割精度。本文在3D UNet中加入了残差模块和Swim Transformer模块,提出了一个新型的卷积与Transformer结合的Res-Swim-UNet模型,残差模块有助于消除梯度消失、加速训练和提高模型特征提取能力的作用;Swim Transformer模块可以建立特征像素之间的全局依赖,可以提高模型对小目标分割的性能。实验结果表明,本文提出的Res-Swim-Unet模型比原始UNet模型在肝脏和肝肿瘤分割上有更高的DSC和更低的VOE。同时本文所提出的模型与先前研究[16-18]进行了比较,发现在肝脏分割方面,本文方法与Bi等[17]一致(DSC:0.957vs.0.959),优于Liu等[16]和Kaluva等[18]的分割方法,在肝肿瘤分割方面,本文方法明显优于先前研究[16-18]的方法。
2021年Luan等[5]提出了一种融合空间特征和注意力机制的CNN,从CT图像中对肝脏肿瘤进行端到端分割,DSC达到了0.613,本文提出的方法对肝肿瘤分割的DSC评分相比Luan等[5]的方法提高了9.6%。2019年李渊强等[22]提出一种基于级联CNN的全自动CT图像肝脏肿瘤分割方法,DSC为0.66。本文比其方法增加了Swim Transformer模块,发挥Transformer的自注意力机制,进一步提高了肝肿瘤分割DSC至0.672。虽然肝肿瘤分割DSC低于0.7,但本文提出的方法仍提高了肝肿瘤分割精度,由于肝肿瘤体积较小,微小的分割差异就会导致DSC指标明显下降,这也是当前肝肿瘤分割精度普遍不高的原因。本文提出的肝肿瘤自动分割方法可以实现肿瘤区域初步定位,辅助医生勾画肿瘤靶区,提高工作效率。2021年秦伟等[23]利用连心医疗的智能放疗云平台自动勾画肝脏,DSC为0.92。连心医疗的智能云勾画系统是市场上发展成熟度较高的放疗自动勾画产品,本文方法分割肝脏的DSC为0.957,肝脏分割轮廓在医生微调后就可以直接用于放疗计划设计。本文的分割模型充分发挥了残差模块和Transformer模块的优势,有效提取了CT图像的局部和全局信息,肝脏和肝肿瘤的分割性能得到明显提高。常规的自动分割模型能够为大尺寸肿瘤提供合理的分割结果,如果肿瘤尺寸减小,网络则无法准确地定位肿瘤。对于肝肿瘤分割,UNet模型分割结果未达到优的肿瘤长轴平均长度为23.10 mm,Res-Swim-UNet模型分割结果未达到优的肿瘤长轴平均长度为18.61 mm,由此可见,本文提出的Res-Swim-UNet模型在小肿瘤分割上表现更好。
本文的模型在本地数据集上肝脏和肝肿瘤分割均达到了接近公共数据集上的分割水平,模型在本地数据上评价指标略有下降,这是因为来自不同医疗中心的CT图像在肿瘤分期和像素分辨率方面存在差异,导致分割结果也会存在差异。本文提出的模型从LiTS数据集迁移到本地数据数上仍具有良好的性能,肝肿瘤分割DSC达到0.589,优于Bi等[17]的0.500和Kaluva等[18]的0.492,这体现了本文模型的有效性和鲁棒性,值得进一步推广到临床使用。
4 结论
本文提出了一种用于从CT图像中进行肝脏和肝肿瘤分割的端到端深度神经网络Res-Swim-UNet模型,其可以同时提取图像中局部和全局信息以实现肝脏和肝肿瘤的精确分割,在LiTS公开数据集和本地数据集上进行的对比实验证明了所提出的分割模型具有明显优越性。

