C-C基序趋化因子配体23是乳腺癌的新型预后标志物且与免疫浸润相关①
张江涛 吴向华 王昌利 张瀚文 阮晖朝 符小兵 杨荣乾
(广西医科大学第一附属医院胃肠腺体外科,南宁 530021)
乳腺癌是一种女性高发实体恶性肿瘤。最新数据显示,目前乳腺癌已超越肺癌,成为全球发病率最高的恶性肿瘤,同时也是导致女性死亡的首要原因[1]。部分早期乳腺癌患者受益于手术、化疗和放疗技术成熟,但晚期乳腺癌具有高转移性、恶病质等特点,目前治疗手段无法显著降低患者病死率。随着对乳腺癌发生分子机制的深入研究,多种针对抗癌位点的特异性靶向药物已用于乳腺癌临床治疗,且明显提高了乳腺癌治疗效果[2]。因此,寻找乳腺癌早期和预后标志物刻不容缓。
趋化因子是一种小细胞因子或信号蛋白,可定向趋化免疫细胞募集至感染部位,根据N端半胱氨酸的排列方式可分为4个亚族:CC趋化因子亚族、CXC趋化因子亚族、XC趋化因子亚族和CX3C趋化因子亚族。作为CC趋化因子亚族成员之一,CCL23(C-C motif chemokine ligand 23)位于染色体17q12区,目前相关研究主要集中于脑部病变和炎症性疾病等[3-5]。在肿瘤领域,CCL23/CCR1/AKT/ESR1轴可抑制肝癌细胞增殖[6];国内学者也发现CCL23在肝癌组织显著下调且影响患者预后[7-8]。414例胃癌患者血清免疫应答蛋白研究发现,CCL23和MMP1与癌症风险降低相关[9]。一项队列研究也表明,高表达CCL23的食管鳞状细胞癌患者预后良好[10]。另一项研究证明CCL23可作为预测乳腺癌患者阿霉素用药后心脏毒性的生物标志物[11]。提示CCL23在癌症发展中扮演重要角色,但CCL23如何参与乳腺癌发生发展的报道较少。
本研究基于多种在线分析工具,包括GEPIA2.0、TIMER、PrognoScan和Oncomine验证CCL23 mRNA在乳腺癌及正常组织的表达差异和预后价值,LinkedOmics和GeneMANIA数据库用于可视化CCL23在乳腺癌组织中共表达基因富集的生物学行为和蛋白互作网络,TIMER数据库阐明CCL23在乳腺癌组织中与免疫微环境的相关性。
1 材料与方法
1.1 4个在线工具分析人乳腺癌组织CCL23转录水平差异GEPIA2.0数据库(http://gepia2.cancerpku.cn/#index)是北京大学创立的一个交互式网站,包含TCGA和GTEx上千例肿瘤和正常样品RNAseq数据[12]。选择“Box Plot”构建箱线图。TIMER数据库(https://cistrome.shinyapps.io/timer/)是一个基于TCGA资源开发的免疫数据库[13]。“DiffExp”模块研究CCL23 mRNA在肿瘤和邻近正常组织的差异表达,以箱线图显示。Oncomine数据库(https://soft‐ware.oncomine.com/resource/login.html)是一个免费且包含大量癌症微阵列的数据库[14]。筛选条件为:P<0.01,Change=2。UALCAN(http://ualcan.path.uab.edu)是一个基于TCGA开发的在线分析网站[15],输入“CCL23”,选择“BRCA”板块,选择相应乳腺癌亚分类构建箱线图。
1.2 2个在线工具分析CCL23转录水平与乳腺癌患者预后的关系TCGA和GEO是目前公认的肿瘤资源库。选择基于TCGA开发的GEPIA2.0数据库生存模块,选择条件为P<0.05,“Group Cutoff:Median”。采用“Survival Map”模块将CCL23前50个共表达基因进行生存分析,以热图形式展现。PrognoScan是一个基于GEO资源开发的经典在线网站[16]。阈值设定为:CoxP-value<0.05。
1.3 2个在线工具探讨CCL23涉及的蛋白互作网络和生物通路LinkedOmics数据库(http://linkedo‐mics.org/)采用TCGA-BRCA数据Pearson检验分析CCL23共表达基因,以热图显示。利用Linkinter‐preter模块进行KEGG富集分析,设置条件为FDR<0.05,1 000次计算[17]。GeneMANIA(http://genema‐nia.org/)数据库是一个可视化分析、包括智人在内9种生物目标基因蛋白互作网络的工具[18]。输入“CCL23”和选择“homo sapiens”获取与CCL23可能存在相互作用的蛋白网络,数据来源主要为GEO、BioGRID、IRefIndex、I2D[18]。
1.4 TIMER数据库了解CCL23转录水平与免疫微环境的关系 采用Gene模块查询CCL23 mRNA与6种免疫细胞数量和肿瘤纯度的关系。另外通过相关性模块了解CCL23 mRNA与多种免疫细胞活化标志物的关系。
1.5 统计学方法 采用t检验或单因素方差分析检测肿瘤组织与正常组织表达差异;Kaplan-Meier生存分析判断CCL23 mRNA表达与乳腺癌患者预后的关系,高、低表达组以中位数区分,采用log-rank检验分析高、低CCL23表达组的生存差异,得到P值;Pearson相关性检验分析CCL23的共表达基因。采用Spearman相关性检验通过调节肿瘤纯度分析CCL23与免疫细胞标志基因的相关性。其余分析采用相关网站提供的统计学分析方式。P<0.05为差异有统计学意义。
2 结果
2.1 CCL23 mRNA在人类乳腺癌组织中呈低表达 基于Oncomine、GEPIA2.0和TIMER数据库发现,相较于乳腺正常组织,乳腺癌组织CCL23 mRNA水平显著下调。Oncomine数据库设定条件为P<0.000 1,倍数变化为2,分析显示CCL23 mRNA在乳腺癌组织中呈低表达(图1)。采用UALCAN数据库对TCGA-BRCA样品多种临床病理特征进行亚组分析(根据性别、淋巴转移状态和肿瘤等级),乳腺癌患者CCL23表达明显低于正常对照组(图2)。提示CCL23可能是乳腺癌的潜在诊断指标。
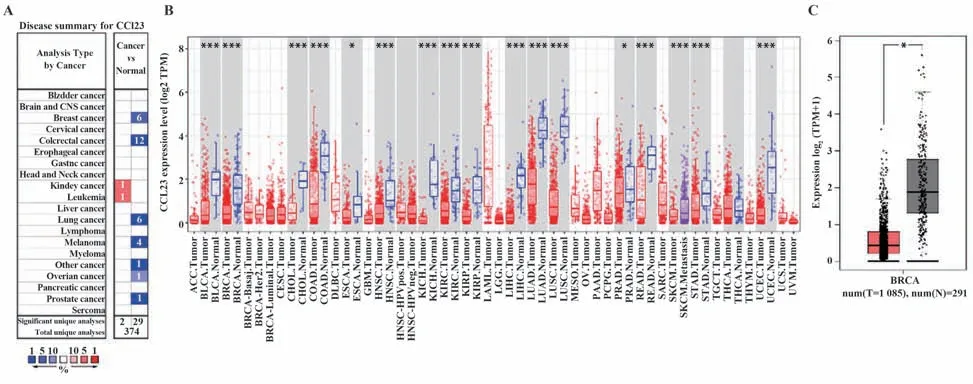
图1 CCL23 mRNA在人类乳腺癌组织中呈低表达Fig.1 CCL23 mRNA expression is decreased in human breast cancer tissues
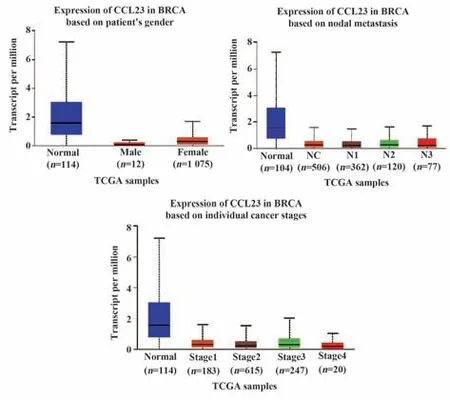
图2 UALCAN数据库分析正常乳腺组织与乳腺癌组织(不同性别、淋巴转移状态和肿瘤分期)CCL23表达差异Fig.2 Differences of CCL23 expression between normal breast tissue and cancer tissues(different gender,lymphatic metastasis status and tumor stage)in UALCAN database
2.2 CCL23 mRNA是乳腺癌患者预后的潜在生物标志物 通过在线分析工具GEPIA2.0对TCGA样本进行计算,结果表明:高表达CCL23 mRNA的乳腺癌患者预后更好[OS(HR=0.63,P=0.004 8),DFS(HR=0.63,P=0.017 0),图3]。为克服单一数据库导致的不确定性,采用PrognoScan数据库分析GEO数据集GSE6532-GPL570和GSE2034,结果表明,与GEPIA2.0分析结果趋势类似(表1)。表明CCL23具有作为乳腺癌预后标志物的潜力。
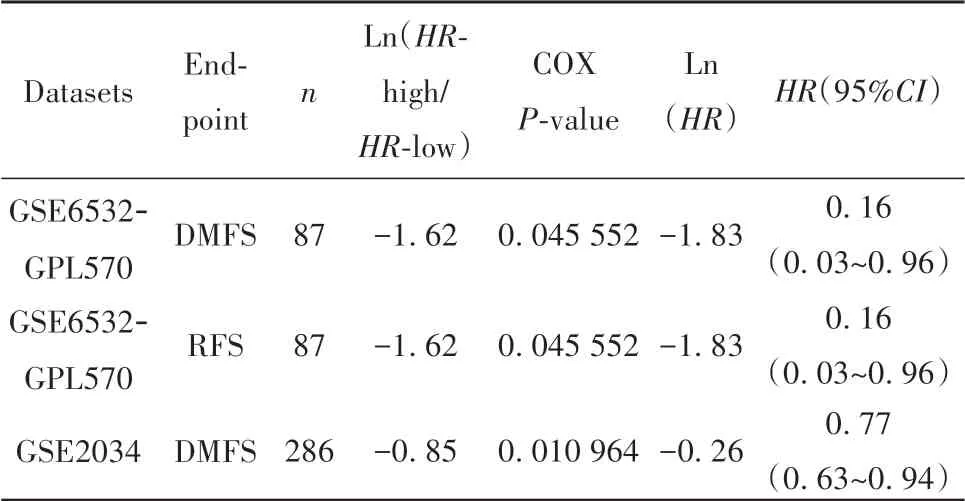
表1 GEO数据集中CCL23与乳腺癌预后的相关性Tab.1 Correlation between CCL23 and breast cancer prognosis in GEO dataset
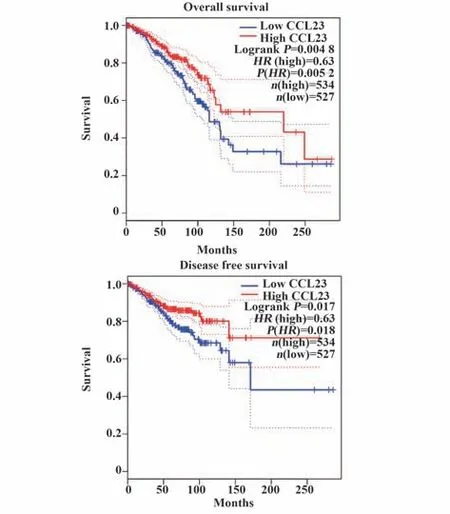
图3 GEPIA2.0数据库预后模块分析CCL23表达与乳腺癌OS和DFS的关系Fig.3 Relationship between expression of CCL23 and OS and DFS of breast cancer analyzed by prognosis module of GEPIA2.0 database
2.3 CCL23涉及的蛋白互作网络和KEGG通路LinkedOmics数据库分析得到CCL23在乳腺癌中的共表达基因,并对前50个相关性最为显著的基因生成热图。利用Linkinterpreter模块进行KEGG富集分析,结果显示:CCL23及其共表达基因主要参与免疫相关生物学行为;采用GEPIA2.0对前50个正相关和负相关基因生成生存热图,提示正相关基因中13/50个基因与患者OS相关(P<0.05),负相关基因中5/50个基因与患者OS相关(P<0.05)。Genemania数据库可视化分析发现,CCL23具有复杂的蛋白互作网络,其中CCL21和CCR1蛋白联系最为密切(图4)。
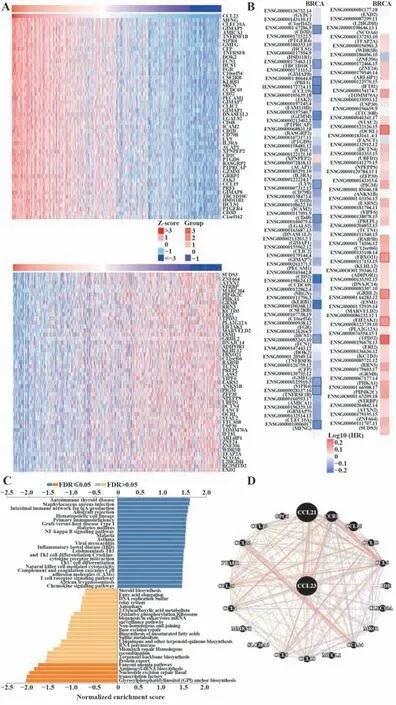
图4 CCL23涉及的蛋白互作网络和KEGG通路Fig.4 Protein interaction network and KEGG pathway involved in CCL23
2.4 CCL23 mRNA与乳腺癌组织免疫微环境的关系 选择Gene模块的BRCA队列进行数据分析,提示CCL23与肿瘤纯度呈显著负相关,与B细胞、CD4+T细胞、CD8+T细胞、巨噬细胞、树突状细胞和中性粒细胞数呈正相关(图5)。采用相关性模块通过调节肿瘤纯度发现,CCL23与多个活化CD4+T和CD8+T细胞标志物呈显著正相关(表2)。
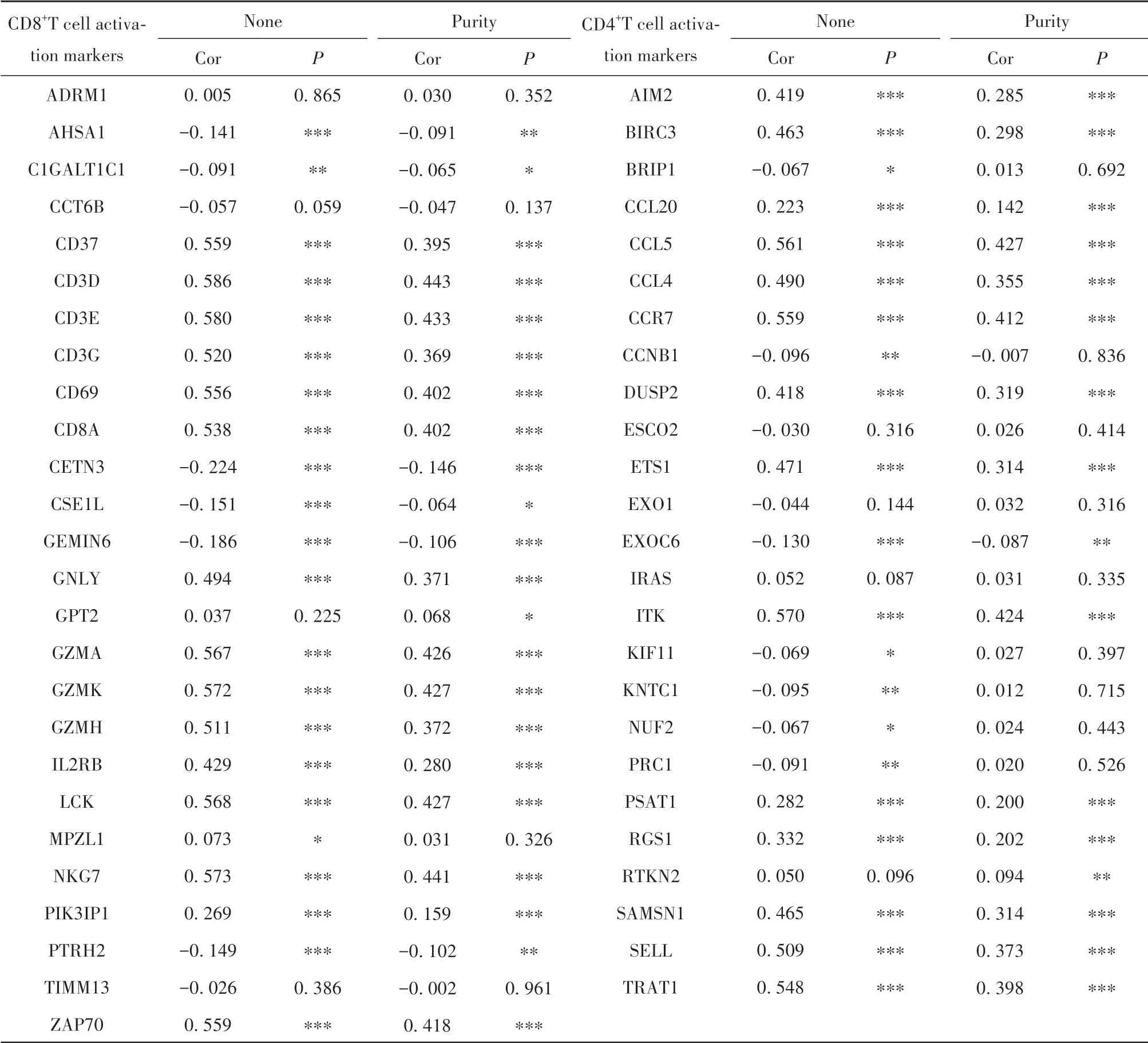
表2 乳腺癌组织中CCL23与CD8+T和CD4+T细胞活化标志物的相关性Tab.2 Correlation between CCL23 and CD8+T,CD4+T cell activation markers in breast cancer tissue
3 讨论
采用生物信息学技术对大量测序和芯片数据进行可视化分析不仅有利于了解疾病发生的分子机制,而且能够确定肿瘤关键分子靶点,为临床治疗提供新方向。免疫治疗作为肿瘤治疗的新方向,与传统治疗方式相比,具有特异性强和副作用小的优势[19]。目前PD-1和CTLA4抑制剂已在临床肿瘤治疗方面取得了显著疗效[20]。因此,探索活化肿瘤免疫细胞功能的相关分子有利于促进抗瘤免疫治疗发展。
本研究通过GEPIA2.0、Oncomine和TIMER数据库均发现相较于乳腺正常组织,肿瘤组织CCL23呈显著低表达。UALCAN数据库分析表明,女性患者CCL23 mRNA表达高于男性。恶性肿瘤转移是肿瘤治疗失败的主要原因。与正常组织相比,转移型与高分期的乳腺癌患者CCL23 mRNA表达更低。CCL23具有作为早期乳腺癌关键分子标志物的潜能。GEPIA2.0发现低表达CCL23的乳腺癌患者往往OS和DFS较短。PrognoScan分析发现,GEO多个数据集中也具有相似趋势。表明CCL23有较大潜力成为乳腺癌的预后标志物。
免疫微环境在肿瘤发生发展中扮演重要角色,越来越多的研究表明,趋化因子家族通过调节机体免疫行为参与肿瘤进展,且在恶性肿瘤免疫治疗中具有重要意义[21-22]。CCL20已被证明作用于NF-κB信号通路参与调节三阴性乳腺癌细胞株MDA-MB-231紫杉醇敏感性[23]。CCL11/CCR3轴可促进乳腺癌远处转移[24]。但CCL23如何参与乳腺癌的具体机制尚未明确。LinkedOmics数据库分析CCL23在乳腺癌中共表达的基因发现,CCL23多个共表达基因与预后相关。KEGG富集分析发现,CCL23及其共表达基因主要参与机体免疫调节。在GeneMANIA数据库中选择智人模块,分析提示CCL23蛋白主要作用于CCL21和CCR1蛋白。免疫细胞向肿瘤部位募集是由趋化因子及其特定配体介导的,与癌症进展和转移相关,CCL23/CCR1轴已被证明参与上皮性卵巢癌进展和化疗敏感性调控[25]。本研究还发现CCL21是CCL23相关性较强的趋化因子。CCL21可能是调控癌症发生发展的双向调节因子。一方面有研究发现CCL21/CCR7轴参与肿瘤细胞侵袭转移[26];另一方面有研究发现CCL21可活化T细胞功能,并在小鼠模型中得以验证,且CCL21联合PD-1抑制剂可增强抗肿瘤免疫疗效[27]。但尚未有报道表明CCL2和CCL21与肿瘤生物学行为相关,有待进一步研究。肿瘤浸润淋巴细胞可作为评估癌症患者预后和免疫治疗效果的独立指标[28]。本研究结果提示CCL23与乳腺肿瘤微环境的免疫细胞成分密切相关。TIMER数据库分析表明,CCL23与肿瘤纯度呈显著负相关,与B细胞、CD4+T细胞、CD8+T细胞、巨噬细胞、树突状细胞和中性粒细胞数量呈正相关。CCL23与CD4+T细胞、CD8+T、树突状细胞、中性粒细胞相关性较强。CD4+T细胞和CD8+T细胞在特异性杀伤肿瘤细胞有着不可或缺的作用[29];树突状细胞是机体功能最强的专职抗原递呈细胞,能够有效激活T细胞,是机体免疫应答的中心环节[30]。研究表明,N1型中性粒细胞的抗肿瘤作用主要通过分泌Ⅰ型干扰素活化自然杀伤细胞IL-18表达[31]。通过调节肿瘤纯度探讨CCL23与免疫细胞活化相关标志物的联系,结果显示:CCL23与多个活化CD4+T细胞与CD8+T细胞标志物呈显著正相关。有报道称CCL23通过结合CCR1可活化T淋巴细胞、树突状细胞和单核细胞功能,增强抗肿瘤免疫力,为本研究提供了科学依据[32]。
综上,本研究结果提示CCL23可能通过结合CCR1活化包括T淋巴细胞在内的多种免疫细胞,增强抗肿瘤免疫力,使乳腺癌患者预后良好。但以上研究仅是基于TCGA等公共数据资源分析的初步理论结果,尚需一系列体内外实验和乳腺癌病理标本进行验证。另外,课题组缺乏CCL23调节乳腺癌免疫微环境的直接证据,仍需免疫学实验进行验证。本研究结果初步阐明了CCL23作为乳腺癌预后标志物的潜在价值,并提供了一种机制。CCL23将是肿瘤免疫疗法的新方向。

