基于miRNA-mRNA 探讨西黄丸干预乳腺癌骨转移的分子机制及预后分析
邓显光,刘丽芳,袁 博*
(湖南中医药大学第一附属医院乳腺科,湖南 长沙 410007)
乳腺癌是全球第一大恶性肿瘤,发病率11.7%,死亡率6.9%,严重危害女性生命健康[1]。晚期乳腺癌患者中65%~75%发生骨转移,常伴有骨痛、骨相关事件、骨损伤等,治疗上以控制肿瘤进展、缓解疼痛、改善生活质量为原则,在化学药物治疗、内分泌治疗、靶向治疗、免疫治疗等多学科综合治疗控制乳腺癌原发灶的基础上,加用双磷酸盐、地舒单抗等骨改良药物,治疗骨相关事件[2]。 在整体观念、辨证论治理论指导下,中医药治疗乳腺癌骨转移发挥着重要作用,可有效提高乳腺癌患者生存率及生活质量,中西医结合方案具有协同增益的效用[3]。西黄丸作为广谱抗肿瘤中成药,联合唑来膦酸能显著提高乳腺癌骨转移患者的临床疗效及生存质量[4]。 研究表明,miRNA 对乳腺癌的生长以及转移有着重要作用[5],且miRNA 可作为乳腺癌治疗以及生存预后的生物标志物[6]。 miRNA-556-5p 能与靶基因3'UTR相结合,调控乳腺癌细胞的迁移、侵袭和上皮-间质转化,在乳腺癌骨转移中发挥着调控作用[7]。 为进一步明确乳腺癌骨转移分子机制,本研究以西黄丸干预乳腺癌骨转移为切入点,以miRNA-mRNA 互作为落脚点,综合多个数据库信息,采用网络药理学联合生物信息学的方法,明晰西黄丸治疗乳腺癌骨转移的分子机制及药物干预靶点,为乳腺癌骨转移的药物干预靶点及生物标志物的确定提供理论依据。
1 资料与方法
1.1 西黄丸干预乳腺癌骨转移miRNA-mRNA 互作关系构建
通过GEO 数据库(https://www.ncbi.nlm.nih.gov/geo/),分别获取GPL21572 探针的乳腺癌miRNA(GSE143564)与GPL570 探针的基因(GSE42568)表达微阵列数据。 GSE143564 表达矩阵中,将GSM 4262013~GSM4262015 样本分为正常组(Normal 组),GSM4262016~GSM4262018样本分为乳腺癌组(Tumor组);GSE42568 表达矩阵中,将GSM1045191~GSM 1045207样本分为正常组(Normal 组),GSM1045208~GSM1045311 分为乳腺癌组(Tumor 组)。 采用R 语言“limma”包,以log2FC>1 或log2FC<-1,且P<0.05为 筛 选条件[8],差异分析GSE143564、GSE42568 表达矩阵,分别筛选出差异miRNA(differentially expressed microRNAs, DEMs)与差异基因(differentially expressed genes, DEGs),绘制DEMs 环形聚类热图、DEGs 火山图与前20 位DEGs 热图(基于log2FC 绝对值)。
通过TargetScan 数据库(http://www.targetscan.org/vert_80/),逐一检索DEMs 靶基因(differentially expressed microRNA target genes, DEMTGs),构建DEMs-DEMTGs 映射关系。
通过GeneCards 数据库(https://www.genecards.org/)、OMIM 数据库(https://omim.org/),以“bone metastasis”为检索词,获取骨转移基因(bone metastasis genes, BMGs)。
通过TCMSP(https://tcmsp-e.com/)、TCMID(http://47.100.169.139/tcmid/search/)数据库,获取西黄丸中牛黄、麝香、乳香、没药的有效组分。 通过PubChem(https://pubchem.ncbi.nlm.nih.gov/)数据库,获取西黄丸各有效组分SMILES 号或结构式。 通过SwissTargetPrediction(http://www.swisstargetprediction.ch/)数据库,获取西黄丸潜在药物靶点(Xi Huang Wan Genes, XHWGs)。
通过在线韦恩图工具(http://bioinformatics.psb.ugent.be/webtools/Venn/),取DEGs、DEMTGs、BMGs、XHWGs 4 个分子靶点交集,获取西黄丸干预乳腺癌骨转移的分子靶点。
通过DEMs-DEMTGs 映射关系,映射西黄丸干预乳腺癌骨转移的分子靶点的上游DEMs,采用Cytoscape 3.8.2 构建miRNA-mRNA 互作关系。
1.2 西黄丸干预乳腺癌骨转移Hub 基因筛选及富集分析
通过STRING(https://cn.string-db.org/)数据库[9],获取西黄丸干预乳腺癌骨转移分子靶点的蛋白质-蛋白质相互作用(protein-protein interaction, PPI)网络。采用Cytoscape 3.8.2 对PPI 网络进行可视化,使用CytoHubba 中MCC 算法对PPI 网络进行拓扑分析,获取西黄丸干预乳腺癌骨转移Hub 基因。 采用R 语言“clusterProfiler”包对西黄丸干预乳腺癌骨转移分子靶点进行基因本体论(gene ontology, GO)和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes, KEGG)富集分析。根据DEMs-DEMTGs映射关系,采用Cytoscape 3.8.2 构建西黄丸干预乳腺癌骨转移Hub 基因miRNA-mRNA 核心网络。
1.3 西黄丸干预乳腺癌骨转移Hub 基因表达差异分析及数据库验证
采用R 语言,以TCGA(https://portal.gdc.cancer.gov/)数据库中乳腺浸润癌患者RNAseq 数据及临床信息作为验证集, 取1109 例乳腺浸润癌患者RNAseq数据与113 例癌旁样本RNAseq 数据进行非配对样本t 检验,将113 例癌旁组织作为正常组,113 例癌旁组织对应的乳腺癌组织作为肿瘤组,进行配对样本t 检验,以验证西黄丸干预乳腺癌骨转移Hub 基因的表达。
通过HPA(https://www.proteinatlas.org/)数据库,获取西黄丸干预乳腺癌骨转移Hub 基因的相对表达免疫组化结果。
1.4 西黄丸干预乳腺癌骨转移分子靶点预后分析
通过TCGA 数据库,采用R 语言“glmnet”“survival”包对西黄丸干预乳腺癌骨转移分子靶点进行Lasso 回归分析,十折交叉验证法筛选Lasso 的值,构建系数筛选图与变量轨迹图。
通过TCGA 数据库,采用R 语言“survminer”“survival”包,Kaplan-Meier 曲线参数法构建分子靶点与患者总体生存期(overall survival, OS)的生存曲线,评判分子靶点高、低表达在乳腺癌中的生存预后价值;采用R 语言“pROC”包构建受试者工作特征曲线(receiver operating characteristic curve, ROC),分析Lasso 回归筛选后的西黄丸干预乳腺癌骨转移分子靶点的表达的诊断价值。
通过TCGA 数据库,采用R 语言构建西黄丸干预乳腺癌骨转移分子靶点的表达与Hub 基因之间的相关性,将分子靶点表达量的中位数作为截断值,分为高、 低表达组, 将Hub 基因的表达量进行Zscore 转换,Zscore=(表达量-均值)/标准差,并进行热图可视化。
通过TCGA 数据库,以临床TNM 分期、年龄、病理阶段、分子靶点表达为自变量,以生存预后为因变量,采用R 语言单因素Cox 回归分析得到风险比(hazard ratio, HR),构建临床信息与分子靶点预后模型,HR>1 为危险因素,HR<1 为保护因素。
1.5 统计学分析
通过TCGA 数据库,采用非配对样本t 检验对ADRB1 相关的miRNA 进行差异表达分析,符合正态性和方差齐性时采用独立样本t 检验;仅符合正态性而不满足方差齐性时采用Weltch t 检验;均不满足正态性时采用Wilcoxon 秩和检验。采用R 语言“pROC”包构建ROC 曲线,分析ADRB1 相关差异表达的miRNA 的诊断价值。 采用R 语言“rms”包对ADRB1 相关差异表达的miRNA 构建列线图,设置标尺评分来表征各个miRNA 的表达情况,计算总评分预测OS 生存预后的概率。
2 结果
2.1 西黄丸干预乳腺癌骨转移miRNA-mRNA 互作关系
以GPL570 为探针的GSE42568 芯片差异分析出3364 个DEGs,其中1689 个在乳腺癌中高表达,1675 个在乳腺癌中低表达(图1A)。 根据差异倍数log2FC 的值,分别将高表达、低表达前20 位DEGs可视化并进行欧式聚类,KRT19、COL11A1、EPCAM等在乳腺中高表达,LEP、RBP4、ADH1B 等在乳腺癌中低表达(图1B)。以GPL21572 为探针的GSE143564芯片差异分析出159 个DEMs,其中140 个(miR-182、miR-494-3p、miR-3651 等)在乳腺癌中高表达,19个(miR-6722、miR-6732-5p、miR-8057 等)在乳腺癌中低表达,并将其欧式聚类及环形可视化(图1C)。
通过TargetScan 数据库,共构建了353 583 组DEMs-DEMTGs 映射关系;通过GeneCards、OMIM数据库,共获取8690 个BMGs;通过TCMSP、TCMID数据库,获取牛黄有效组分(脱氧胆酸、甲基脱氧胆酸等)共5 个、麝香有效组分(17-β-雌二醇、麝香醇、尿囊素等)共59 个、乳香有效组分(α-乳香酸、3-羟基甘遂酸、因香酚等)共8 个、没药有效组分(逆没食子酸、天竺葵素、植物类固醇Ⅳ等)共45 个,进一步通过SwissTargetPrediction 数据库共获取644个XHWGs;取DEGs、DEMTGs、BMGs、XHWGs交 集,共获取135 个西黄丸干预乳腺癌骨转移的分子靶点(图1D)。
采用Cytoscape 3.8.2 对135 个西黄丸干预乳腺癌骨转移的分子靶点构建miRNA-mRNA 互作关系,135 个分子靶点与196 个miRNA 构建出2964组miRNA-mRNA 互作关系。 详见图2。

图2 西黄丸干预乳腺癌骨转移miRNA-mRNA 互作关系
2.2 西黄丸干预乳腺癌骨转移Hub 基因筛选及GO、KEGG 富集分析
通过STRING 数据库构建的西黄丸干预乳腺癌骨转移分子靶点PPI 网络中,共有135 个节点蛋白,构成666 组PPI 关系(P<0.01)。PPI 网络初步分析结果发现EGFR、IL-6、PPARG 蛋白Degree 值最大,相互作用最强(图3A)。 MCC 算法拓扑分析筛选出前10 位作为Hub 基因,分别为CCL2、SIRT1、EGFR、PPARG、IL-6、CASP1、PTGS2、MMP-9、BCL2L1、ESR1,并进行可视化(图3B)。 GO、KEGG 共富集出1589个生物进程(biological process, BP)、26 个细胞组分(cell component, CC)、133 个 分 子 功 能(molecular function, MF)、112 个KEGG 通路,其中西黄丸治疗乳腺癌骨转移的机制可能与其影响膜上受体蛋白的蛋白酪氨酸激酶活性、激活MAPK 信号通路、参与乳腺癌骨转移的脂质代谢等生命进程有关(图3C)。 采用Cytoscape 3.8.2 对10 个Hub 基因构建miRNAmRNA 核心网络,10 个Hub 基因与95 个miRNA 构建出175 组miRNA-mRNA 核心互作关系(图3D)。
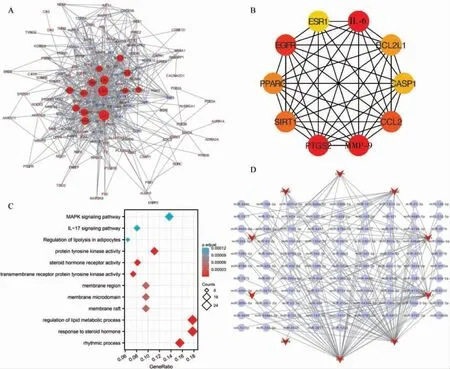
图3 西黄丸干预乳腺癌骨转移Hub 基因及GO、KEGG 富集分析结果
2.3 西黄丸干预乳腺癌骨转移Hub 基因表达差异及数据库验证结果
以TCGA 数据库为验证集,非配对与配对样本t 检验发现(图4A、B):西黄丸干预乳腺癌骨转移Hub基因中,CCL2、SIRT1、EGFR、PPARG、IL-6、CASP1、PTGS2 在乳腺癌中低表达;MMP-9、BCL2L1、ESR1在乳腺癌样本中高表达。进一步通过HPA 数据库比较乳腺癌患者中10 个Hub 基因免疫组化的相对高、低表达的结果(图4C),此结果与GSE42568 差异分析结果相互佐证。
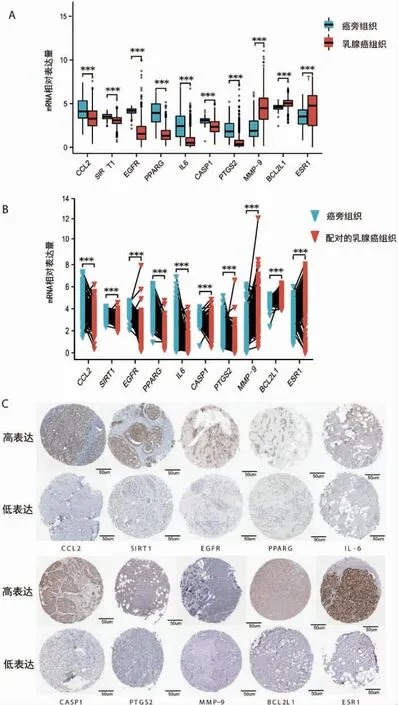
图4 西黄丸干预乳腺癌骨转移Hub 基因表达及HPA 数据库验证结果
2.4 西黄丸干预乳腺癌骨转移分子靶点预后分析结果
将135 个西黄丸干预乳腺癌骨转移的分子靶点纳入Lasso 回归分析中,构建系数筛选图与变量轨迹图(图5A、B)对其进行降维处理,结果发现西黄丸干预乳腺癌骨转移的分子靶点中,ADRB1 可作为乳腺癌独立预后因子。 非配对与配对样本t 检验发现(图5C、D),ADRB1 在乳腺癌患者组织中低表达;Kaplan-Meier 曲线(图5E)显示,ADRB1 高表达组具有较好的生存预后(P<0.05);ROC 曲线(图5F)显示,ADRB1 在患者诊断中具有较高的诊断性(AUC=0.854,95%CI=0.826~0.882)。 进 一 步 分 析ADRB1 与10 个Hub 基因表达的相关性并绘制相关性表达热图(图5G),发现ADRB1 与CCL2、EGFR、PPARG、IL-6、CASP1、PTGS2 表达呈正相关(P<0.01);ADRB1与BCL2L1 表达呈负相关(P<0.01)。
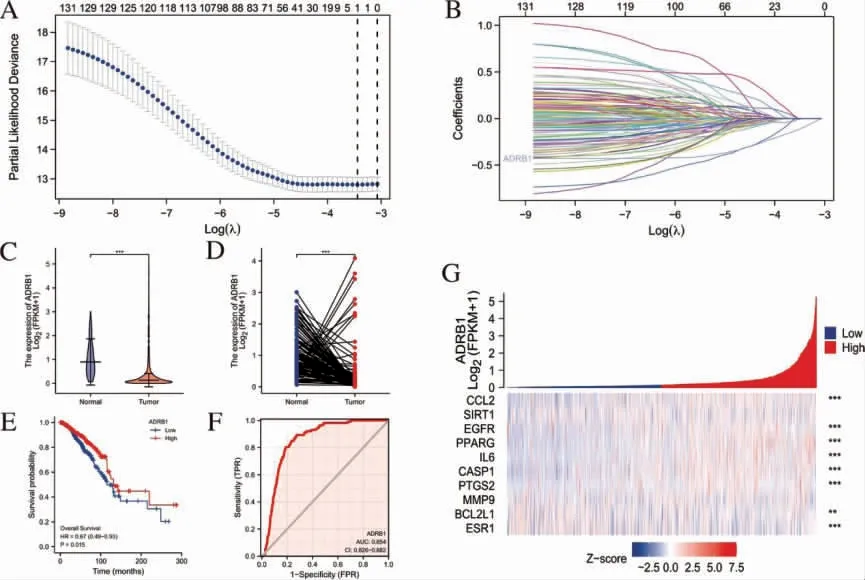
图5 西黄丸干预乳腺癌骨转移分子靶点预后分析
通过单因素Cox 分析评估ADRB1 表达及患者多种临床特征(图6),发现在TCGA 乳腺癌患者中,T4分期、N 分期(N1、N2、N3)、M 分期、年龄、病理阶段(Stage Ⅲ、Stage Ⅳ)、ADRB1 高表达与患者OS 显著相关(P<0.05),进一步多因素Cox 分析发现M 分期为危险因素(P<0.05)、年龄为危险因素(P<0.001)、ADRB1 高表达为保护因素(P<0.05),与患者OS 显著相关。
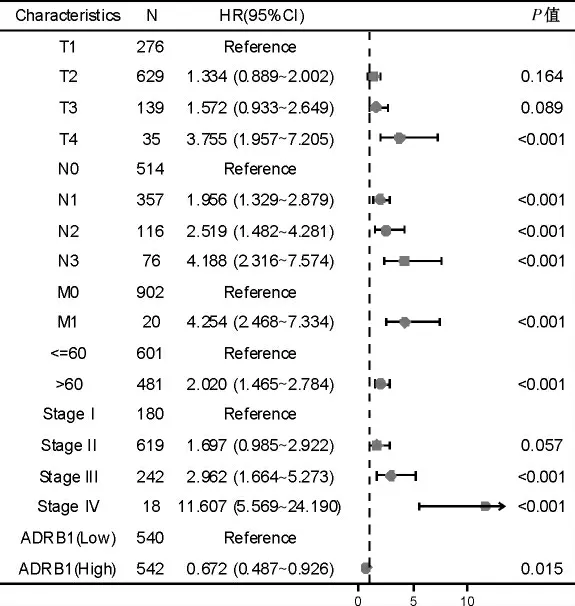
图6 ADRB1 表达与多个临床特征的单因素Cox 回归分析森林图
2.5 ADRB1 相关miRNA 差异表达、诊断及预后列线图结果
通过“1.1”构建的DEMs-DEMTGs 映射关系,映射出与ADRB1 相关的miRNA 共12 个,分别为hsamiR-500a-5p、hsa-miR-629-3p、hsa-miR-1246、hsa-miR-4665-5p、hsa-miR-3615、hsa-miR-4436b-5p、hsa-let-7d-5p、hsa-miR-30a-5p、hsa-miR-93-3p、hsa-miR-141-3p、hsa-miR-183-3p、hsa-miR-320e。 再通过非配对样本t 检验发现 (图7A),hsamiR-500a-5p、hsa-miR-629-3p、hsa-miR-4665-5p、hsa-miR-3615、hsa-let-7d-5p、hsa-miR-93-3p、hsa-miR-141-3p、hsa-miR-183-3p 在乳腺癌组织中过表达(P<0.001),而hsa-miR-1246、hsa-miR-4436b-5p、hsa-miR-30a-5p、hsa-miR-320e 在乳腺癌与正常组织中表达差异无统计学意义(P>0.05)。
对上述8 个差异表达的miRNA 构建诊断ROC曲线(图7B),发现均具有诊断价值,可作为乳腺癌骨转移的生物标志物(P<0.05),其中AUC 由高到低排序依次为:hsa-miR-141-3p>hsa-miR-183-3p>hsa-miR-629-3p>hsa-let-7d-5p>hsa-miR-93-3p>hsa-miR-3615>hsa-miR-4665-5p>hsa-miR-500a-5p;0.5≤AUC<0.7 为较低准确性,0.7≤AUC<0.9 为中度准确性,0.9≤AUC 为高度准确性。
对上述8 个差异表达的miRNA 构建列线图(图7C),差异表达miRNA 可用来预测乳腺癌骨转移患者1、2、3 年生存预后,此预测模型的一致性指数Cindex 为0.588(0.556~0.619)。
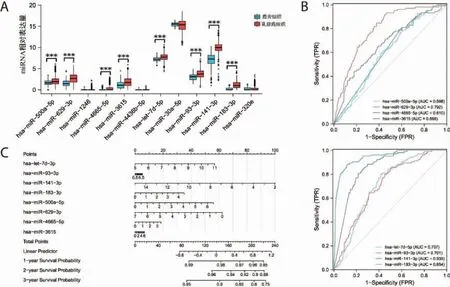
图7 ADRB1 相关miRNA 差异表达、诊断及预后列线图结果
3 讨论
乳腺癌骨转移属于中医学“骨瘤”“顽痹”“骨蚀”等范畴[10]。正气不足,髓不充骨,癌邪乘虚侵袭入骨,气、血、痰、毒、湿邪蕴积搏结于局部,且久郁化火,阻滞气血津液运行,搏结伤骨发而为瘤[11]。且乳腺癌骨转移多伴有骨痛,其病性证素多为气滞、血瘀[12]。 西黄丸由牛黄、麝香、乳香、没药组成,方中牛黄清热解毒、消肿化痰,麝香活血散瘀、消肿止痛,二者配伍,一寒一温,共奏透邪行气化瘀之功;佐以乳香、没药祛邪扶正、消肿止痛、调气活血,四药合用,共奏软坚散结、清热解毒、散瘀止痛之效[13]。研究发现,西黄丸主要活性成分有五环三萜类(如乳香酸类)、挥发油类、甾体类(如猪去氧胆酸),均具有抗肿瘤的作用[14],且西黄丸还有改变乳腺癌的肿瘤炎症微环境的作用[15]。研究发现,西黄丸联合唑来膦酸治疗乳腺癌骨转移总有效率为82.5%,能有效提高机体免疫及骨质修复能力,改善生活质量[4]。 SINGH 等[16]发现miRNAs直接参与了骨组织中乳腺肿瘤的转移;CAI 等[17]发现,miR-124 被抑制可促进体内乳腺癌细胞的骨转移,且在乳腺癌转移性骨组织中miR-124 显著减少;FENG 等[18]发现miR-124 过表达可抑制破骨细胞生成,从而抑制乳腺癌骨转移。在西黄丸干预乳腺癌骨转移明显有效的背景下,以miRNA-mRNA 相互作用为模式,进一步寻找乳腺癌骨转移预后相关的生物标志物、驱动基因及药物干预靶点具有重要意义。
本研究采用生物信息学及网络药理学方法,发现西黄丸中脱氧胆酸、麝香醇、α-乳香酸、逆没食子酸等多个有效组分,共同调控EGFR、IL-6、PPARG等135 个乳腺癌骨转移的分子靶点。 西黄丸干预乳腺癌骨转移的机制可能与干预膜上受体蛋白的蛋白酪氨酸激酶活性、激活MAPK 信号通路以及参与乳腺癌骨转移的脂质代谢等生命进程有关。同时,本研究构建了135 个乳腺癌骨转移分子靶点与196 个乳腺癌差异miRNA 交互作用关系网络,构建出2964组miRNA-mRNA 互作关系网络,锚定了10 个Hub 基因与95 个miRNA,构建出175 组miRNA-mRNA 核心互作关系,为乳腺癌骨转移生物标志的筛选提供了依据。KIM 等[19]研究发现15d-PGJ2 通过PPARG 非依赖性途径,在胞内抑制RANKL 表达及活性,抑制乳腺癌细胞增殖、迁移、侵袭和乳腺癌小鼠骨小梁的破坏。
在TCGA 中验证Hub 基因差异表达及临床相关性,研究发现西黄丸干预乳腺癌骨转移Hub 基因中,CCL2、SIRT1、EGFR、PPARG、IL-6、CASP1、PTGS2在乳腺癌样本中低表达;MMP-9、BCL2L1、ESR1 在乳腺癌样本中高表达,与差异分析结果和免疫组化结果相互佐证。 Lasso 回归分析聚焦出ADRB1 是乳腺癌患者的独立预后因子,具有良好的诊断价值及生存预后预测价值,且与CCL2、EGFR、PPARG、IL-6、CASP1、PTGS2 表达呈正相关;ADRB1 与BCL2L1表达呈负相关,进一步Cox 回归分析发现乳腺癌远处转移的M 分期、年龄是主要危险因素,而ADRB1高表达为保护因素,三者与患者OS 显著相关。 WANG等[20]通过生物信息学方法,对乳腺癌肿瘤突变负荷和免疫浸润进行分析,鉴定出ADRB1 是乳腺癌的潜在生物标志物,但是ADRB1 在乳腺癌骨转移中尚无报道,且未得到实验验证的结果。 基于此,认为ADRB1 可能是乳腺癌骨转移的有效药物干预靶点,为后续实验研究提供了理论依据。
基于miRNA-mRNA 互作模式,聚焦ADRB1 的相关miRNA,进一步寻找乳腺癌骨转移的miRNA生物标志物,并构建临床预测模型列线图,发现hsamiR-500a-5p、hsa-miR-629-3p、hsa-miR-4665-5p、hsa-miR-3615、hsa-let-7d-5p、hsa-miR-93-3p、hsa-miR-141-3p、hsa-miR-183-3p 在乳腺癌组织中高表达,且AUC 均大于0.5,具有良好的诊断价值,并构建了8 个miRNA 的列线图(C-index=0.588),可认为此8 个miRNA 可作为乳腺癌骨转移的生物标志物。KAWAGUCHI 等[21]综合分析TCGA、GEO 数据库,开发了乳腺癌转移和不良预后的miRNA 预测模型,其中包括has-miR-19a、has-miR-93 和has-miR-106a,与本研究构建的预测模型相互佐证。 基于此,本研究认为基于8 个miRNA 开发的预后模型,可预测乳腺癌骨转移的不良预后。
综上所述,本研究基于miRNA-mRNA 互作模式,采用生物信息学联合网络药理学的方法,综合分析多个数据库资源,阐述了西黄丸干预乳腺癌骨转移的分子机制与其多组分调控ADRB1 等多靶点、介导MAPK 等多通路、参与多生物进程有关,同时聚焦出8 个与乳腺癌骨转移预后不良有关的miRNA,建立了预测模型,为开发治疗乳腺癌骨转移药物以及乳腺癌骨转移生物标志物的确定提供了潜在价值,对中医药的现代化推广具有重要意义。

