基于Ghost Module的轻量级脑肿瘤3D MRI分割研究
刘丽伟, 赵 强
(长春工业大学 计算机科学与工程学院, 吉林 长春 130102)
0 引 言
胶质瘤是原发性脑肿瘤中最常见的一种,根据肿瘤细胞的恶性程度可划分为高级别胶质瘤和低级别胶质瘤两种类型[1],其中低级别胶质瘤的治愈率较高。脑肿瘤的磁共振成像MRI是当下辅助医生诊断的重要工具[2]。脑肿瘤的形状多种多样、位置分布也不尽相同。目前,脑肿瘤的诊断大多依靠有丰富临床手术经验的专家进行人工标注。分割水平多由专家的诊断水平和经验决定,带有相当大的主观性,而且分割效率低,影响患者后续治疗[3]。
随着计算机运算能力的提升,卷积神经网络被运用于医学图像语义分割领域。Long J等[4]提出FCN(Fully Convolutional Networks),将原始卷积神经网络全连接层换成卷积层,使得图像级别的分类扩展到像素级别。Ronneberger O等[5]以FCN为基础设计了2D U-Net网络,该网络由对称的编码部分和解码部分组成,且在同一深度的编码层和解码层之间添加跳跃连接进行特征融合。鉴于大多数临床成像数据是体积性的,如三维MR图像,这些模型忽略了关键的三维空间背景,Çiçek Ö等[6]利用3D卷积将其拓展为3D U-Net网络,开始用于三维MRI的分割。近年来,U-Net中的编解码结构已成为所有领先的医学图像分割方法的骨干。Milletari F等[7]提出V-Net,在编解码结构基础上,每一层加入残差机制,将上阶段采样后的特征图与该阶段的输出特征图进行融合。近几年来,Zhao T等[8]提出U-Net++,在U-Net的基础上增加了类似于Dense结构的卷积层,并融合了下一阶段卷积的特征。
尽管医学图像模型层出不穷,但缩减模型的参数量才能使得神经网络训练更加普及推广。已经有一些尝试通过使用轻量级的网络架构来实现这一要求。例如,Nuechterlein N等[9]将基于二维语义分割的快速高效的网络ESPNet扩展到三维医学图像数据,提出3D-ESPNet。Chen W等[10]利用可分离三维卷积的优势,将每个三维卷积分为三个平行分支,以减少可学习的网络参数数量,提出SD-UNet。然而,这些高效模型的性能无法与最先进的模型相比。Chen C等[11]提出DMFNet,该方法与文中初衷相近,既要保证训练精度,又要降低训练参数。它建立在多纤维单元的基础上,采用高效的群卷积,并引入了加权的三维扩张卷积操作,以获得多尺度的图像表示,用于分割。
与此同时,轻量级结构也在不断推陈出新,将此种结构引入脑肿瘤分割领域,降低网络参数量,提升训练速度。文中以Ghost Module[12]作为基础单元进行改进,并以此为基础结构进行网络搭建,为最终形成文中方法进行脑肿瘤分割研究。
1 网络结构设计
1.1 Shuffle-Ghost Module
Ghost Module是文献[12]提出的一种轻量级结构,可以用来替换任何经典CNN网络中的卷积操作,突出优势是轻量高效,相比于传统的卷积,Ghost Module第一步采用正常的卷积计算,卷积核设定为初始卷积核的1/2,同时进行channel缩减,生成部分真实feature map,然后利用cheap operation进行线性变换得到Ghost feature map,将不同的特征图进行拼接,组合完整特征层。
Shuffle-Ghost Module如图1所示。
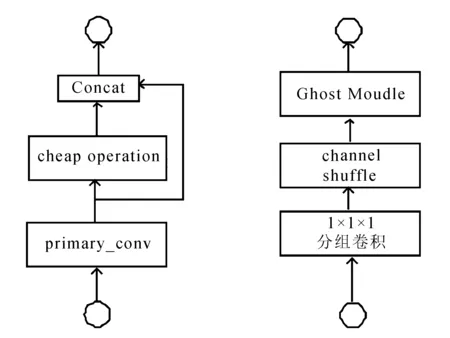
(a) Ghost Module (b) Shuffle-Ghost Module
分组卷积是一种有效的模型加速解决方案,已经被探索用于高效的网络设计。为了使得Ghost Module中的正常卷积输入特征通道等于输出特征通道,同时使得正常卷积不再承担改变channel的作用,可使用分组卷积实现相应通道改变。然而分组是一种通道稀疏连接方式(channel sparse connection),虽然分组策略可以减少参数数量,但简单地用分组卷积代替常规卷积可能会影响通道之间的信息交换,并损害学习能力。必须在分组卷积后对特征图进行channel shuffle。如果没有channel shuffle,最终输出的特征仅由一部分输入通道的特征计算得出,这种操作会阻碍信息流通,进而降低特征的表达能力。
基于以上理论,文中在进入Ghost Module前进行1×1×1分组卷积改变通道数,使得cheap operation输入通道数等于输出通道数。文献[13]同时提到过多的组,会导致MAC增加,因此,设定分组卷积为能设定的最小值,设为4。并在分组卷积后对特征图进行channel shuffle,最终形成了如图1(b)Shuffle-Ghost Module。值得注意的是,虽然cheap operation中的深度卷积本质上也是分组卷积,Ghost feature map中也会有信息损失,但考虑到结构中cheap operation只是为了增加特征图,因此不再放置channel shuffle。文中将以此结构为基础进行下文的网络搭建。
1.2 压缩路径及扩展路径改进
随着神经网络的深度加深,带来的其中一个问题就是梯度消失和梯度爆炸,它会使得训练难以收敛,通过归一化或者Normalization方法可以得到有效地缓解。然而,收敛问题得到解决之后,又存在另一个问题,随着深度加深,网络的准确度趋向于饱和,当继续加深网络,网络的准确度却会降低,并且出现这种问题并不是因为过拟合导致的[7]。文献[14]提到使用一个残差网络结构代替原来的网络结构,可以解决以上问题。
文献[15]提出一种基于全局的注意力机制GAM。以往的注意力机制忽略了保留通道和空间两个方面信息交互作用的重要性。全局注意力机制GAM能够通过减少信息约简、放大全局交互表示来加大深度神经网络的性能。全局注意力机制如图2所示。
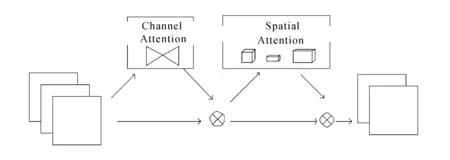
图2 全局注意力机制
对特征在通道和空间上进行重要性重新标定,将注意力更多地放在肿瘤区域上。
依托以上理论,提出两种残差结构替换编、解码路径中单纯的卷积操作。编码路径的S-GG block结构如图3所示。
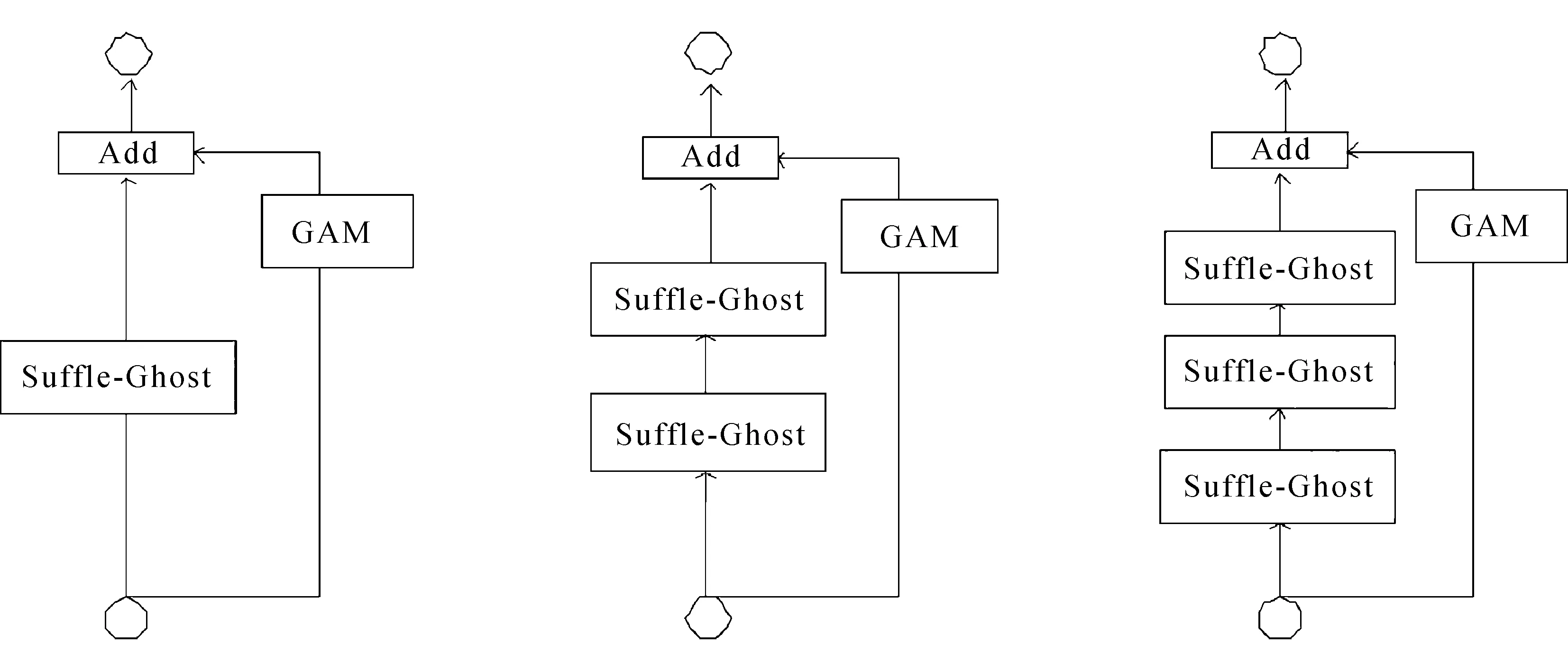
(a) Depth=1 (b) Depth=2 (c) Depth=3
结合Shuffle-Ghost Module以及GAM。不同深度的编码层输入分别经过不同个数的Shuffle-Ghost Module和GAM后与原始特征进行Add。使用GAM对特征图中肿瘤区域进行重要性标定。对于不同深度的信息,堆叠不同个数的Shuffle-Ghost Module,增加深层次的特征提取。不同深度的具体个数见图1中标注。
解码路径考虑到GAM本身带来的参数量,不再使用GAM模块,形成的S-G block如图4所示。
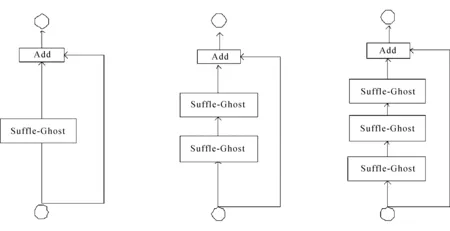
(a) Depth=1 (b) Depth=2 (c) Depth=3
与S-GG相似的是,不同深度解码层的输入经过不同个数的Shuffle-Ghost Module后与输入特征图进行Add后输出。
S-GG block和S-G block中,初始卷积核尺寸设定为5。GAM空间子模块卷积尺寸为7,padding为3。通道子模块以及空间子模块都进行了通道缩减及通道恢复,通道缩减率为4。
1.3 整体网络结构构建
文中的网络模型整体与U-Net相似。S-GG Net 网络结构如图5所示。
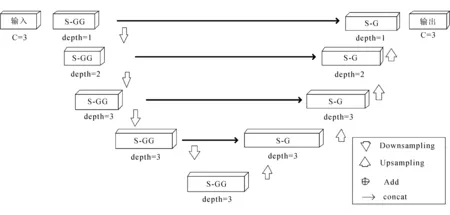
图5 S-GG Net 网络结构
主体网络由编码部分、解码部分和跳跃连接组成。使用S-GG block替换压缩路径的卷积操作,S-G block替换扩展路径的卷积操作。
文献[7]提到下采样模块不再使用池化,而使用卷积用来降低特征图的尺寸,用卷积操作代替池化,在训练期间可以有较小的显存占用。文献[15]提出使用卷积层的网络结构并不会对物体检测性能产生影响。设置下采样卷积核尺寸为2×2×2,stride=2。
上采样模块使用转置卷积,卷积核为2×2×2,stride=2,用来恢复特征图的尺寸和通道数。输入层和输出层使用普通三维卷积核为5×5×5,padding=2。输出通道设置为3,得输出通道数变为3,使输出与处理后的医生标注图片一致。最后,跳跃连接横跨在编解码的两端。
2 训 练
2.1 数据集
实验采用MICCAI提供的脑胶质瘤的公共数据集BraTs2018、BraTs2019[16-19]作为数据集。BraTs2018以4∶1的比例分别作为训练集和验证集,BraTs2019新增的50个病例作为测试集。数据集中的每名患者均包含配准后的FLAIR、T1、T2、T1C四种模态图像和真实分割标签图像GT(Ground Truth)。肿瘤分割的标签包括背景(标签0)、坏死和非增强的肿瘤(标签1)、肿瘤周围水肿(标签2)和增强肿瘤(标签4)。ET、WT和TC分别表示增强肿瘤(标签1)、整个肿瘤(标签1、2和4)以及肿瘤核心(标签1和4)的区域。
2.2 数据集预处理
先将MRI数据分块,然后对图像进行Z-score标准化,再对其进行裁剪,去除无效区域,最后将四个模态合并成四个通道。相对应的GT在分块后,同样进行标准化、裁剪,将三个标签合并成三个嵌套的子区域,最后再合并为三个通道。
2.3 损失函数
训练采用混合损失函数BCE Dice Loss[20],它是由BCE(Binary Cross Entropy) Loss和医学影像损失Dice Loss[7]组合而成。
2.4 评价指标
文中采用Dice系数、PPV,根据分割结果图和医生标注分割结果,对WT、TC、ET分割效果进行评价。评价指标为

(1)
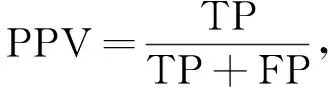
(2)
式中:TP----正确分割的血管像素数目,即真阳性;
TN----正确分割的背景像素数目,即真阴性;
FP----错误分割血管像素的背景像素,即假阳性;
FN----错误标记的背景像素,即假阴性。
除性能指标外,同样引入参数量对轻量化程度做出定量评价。
2.5 实验环境与超参数设定
实验使用的硬件设备是Intel(R) Core(TM) i9-10920X CPU @ 3.50 GHz 3.50 GHz CPU,NVIDIA RTX 3090显卡(24 GB显存),软件环境是Windows10专业工作站版、Python3.7、CUDA11.3、pytorch1.1深度学习框架。文中实验使用了余弦退火的训练策略,设置初始学习率为6e-2。实验中Epochs设置为200;Early stop设置为30,Batch size设置为2;Weight decay设置为1e-4。
3 实验与分析
3.1 对比实验设计
将文中方法与先进的医学图像分割网络以及MICCAI脑肿瘤分割挑战赛录用方法对比。在文中对比实验设定中,方法1设定为3D U-Net;方法2为U-Net改进方法3D U-Net++;方法3采用文献[20]中的UNet+Residual网络结构进行训练;方法4设定MICCAI脑肿瘤分割挑战赛录用方法DMFNet,该结构与文中有着同样的轻量化初衷;方法5添加一组使用未改进的Ghost Module构建网络,参照文中结构对编、解码层进行构建,替换Shuffle-Ghost Module在结构中的位置移除GAM,作为消融实验。方法6为文中方法S-GG Net。模型参数量及计算量见表1。

表1 模型参数量及计算量
从表1可以看出,文中的S-GG Net模型复杂度明显低于前三种医学图像分割网络,但是对于设计初衷相同的DMFNet,模型复杂度确有提升。对比方法5,参数量稍有上升,模型计算量却下降50%,可见改进的有效性。
文中方法分割评价指标结果见表2。
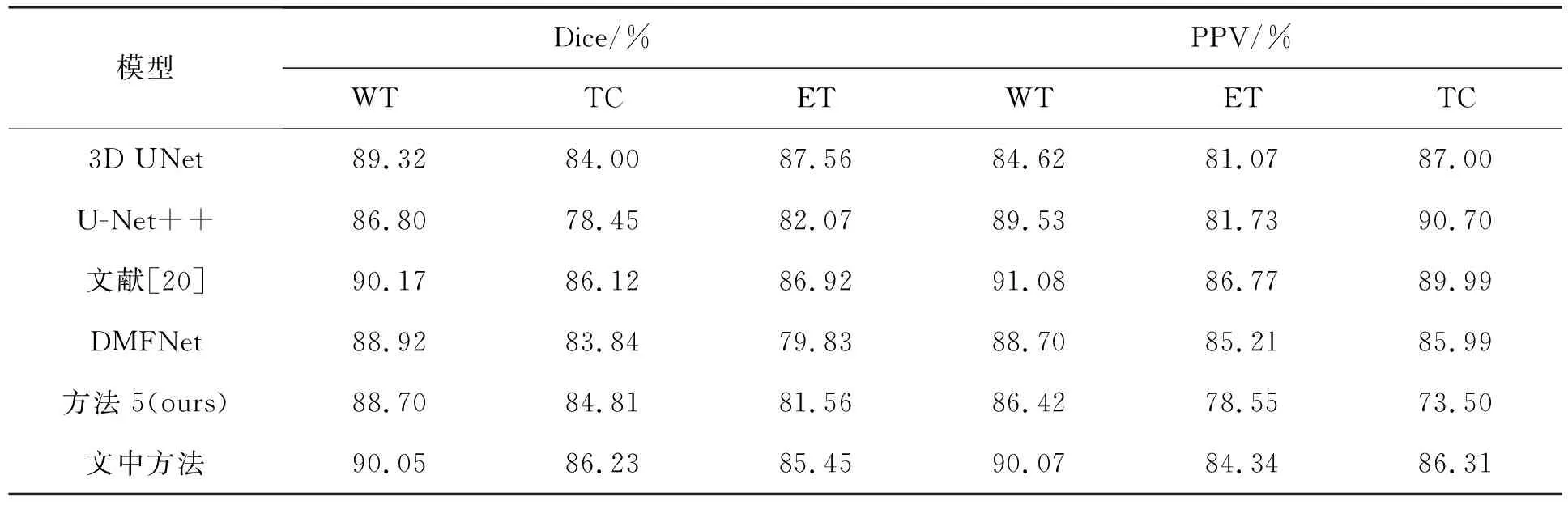
表2 分割评价指标结果
同时放置具有代表性的方法1、方法3、方法4以及文中方法的实际分割效果如图6所示。
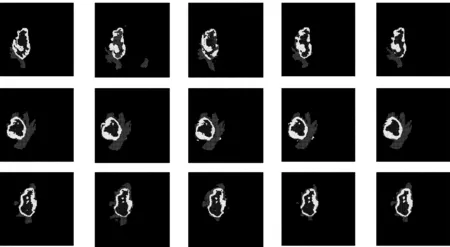
(a) GT (b) 方法1 (c) 方法3 (d) 方法4 (e)文中方法
3.2 实验结果分析
方法1作为经典的医学图像分割网络有良好的分割基准,但分割时会把健康区域识别成肿瘤区域。方法2有较为庞大的参数量及运算量,结果却不尽如人意,猜测是网络收敛性差,训练的轮次太少,不能达到网络的最优水平。方法3在占据着仅低于方法2的参数量及计算量的情况下,达到了所有方法中的最好效果,可见在网络中加入残差结构确实能增加网络的收敛速度。方法4与文中的初衷相同,但过少的参数量和计算量确实影响到分割效果,随着计算机硬件水平的提升,并不能一味地缩减参数量。相比之下,文中方法参数量计算量适中,且分割指标最为接近方法3,分割效果主观上看也贴近GT。
4 结 语
针对现存网络在3D脑肿瘤图像分割中参数量过大、训练速度过慢的问题,提出一种轻量级脑肿瘤MRI图像的分割算法S-GG Net。有6.8 M的参数和大约38 G的FLOPs,可以实现MRI中三维脑肿瘤分割的实时推理。从主观和客观两个角度评价脑肿瘤MRI图像分割的实验结果。文中网络模型极大地缩短了训练时间,并且结果贴近大型网络,更贴合医生实际诊断的需求。

