大麦种质资源抗叶斑病评价
孙逸凡,黄志磊,李葆春,姚立蓉,汪军成,司二静,杨轲,孟亚雄,马小乐,王化俊*
(1.甘肃农业大学省部共建干旱生境作物学国家重点实验室,甘肃省作物遗传改良与种质创新重点实验室,兰州 730070;2.甘肃农业大学农学院,兰州 730070;3.甘肃农业大学生命科学技术学院,兰州 730070)
大麦是一种重要的粮食作物,广泛用于酿酒工业和饲料生产之中。在大麦生长发育过程中,常受各种病菌侵害,从而造成产量损失或品质降低,影响农民收入[1-2]。大麦叶斑病是由禾旋孢腔菌(有性世代Cochilobolus sativus,无性世代Bipolaris sorokiniana)引起的叶部病害,在全世界90多个国家和地区都曾经有流行或暴发,如加拿大、美国、南非等[3]。大麦叶斑病在中国江苏、内蒙古等大麦主产区发病严重,发病田块减产幅度一般为15%~30%[4]。2012—2013年间,内蒙古呼伦贝尔市爆发叶斑病,导致大麦大幅减产,叶斑病已成为制约该地区大麦生产的主要病害之一[5]。该病是土传和种传病害,致病孢子以厚垣孢子的形式寄存于土壤和大麦、小麦的残株中,其生存年限可达8~10年,新生孢子会存留在受侵染的植株残片上。植株被病原菌侵染后,发生根冠褐变、叶片茎秆萎蔫、坏死、黑胚等症状,目前该病害尚无有效防治方法[6-7],培育抗病品种是有效的途径之一。种质资源是大麦新品种选育的物质基础,核心种质代表了大多数种质的变异。因此,对大麦核心种质进行叶斑病抗性鉴定可以为选育抗性品种奠定基础,也可为挖掘抗性基因及探究作物、菌株互作机制提供参考。
国内外在大麦抗叶斑病鉴定及抗病遗传资源利用等方面研究较深入并取得重要进展。Gyawali等[8]利用来自于摩洛哥的19种叶斑病菌对336个基因型的大麦苗期和成熟期进行了抗性鉴定,并检测出了只在苗期出现的9个QTL位点。Zhan等[9]通过QTL分析发现,大麦抗叶斑病基因分布不均,多集中在1H、3H、6H、7H染色体短臂和3H染色体长臂上。Bovill等[10]对4个抗叶斑病大麦品种成熟期和苗期的抗性位点进行了比对,发现同一品种在幼苗期和成熟期抗性位点差异明显,不同品种的抗性基因位点也并不一致。吕二锁等[11]对96份大麦材料进行了抗性鉴定,姚全杰等[12]对国内223份大麦材料的抗性鉴定,阐释了品种Azare是大麦叶斑病抗性重要种质资源。目前国内对于大麦叶斑病的研究仍然主要集中在病害防治和菌株毒性的鉴别方面,而关于大麦叶斑病抗病性遗传及抗性基因发掘的研究鲜有报道。
本研究运用SNP标记技术对200份大麦种质进行遗传多样性分析,并通过分子标记与抗大麦叶斑病的全基因组关联分析(genome-wide association study,GWAS),以期为大麦抗叶斑病基因挖掘和抗病品种选育提供理论依据。
1 材料与方法
1.1 供试材料
根据本课题组前期研究基础[12],选取具有代表性的200份不同来源的大麦品种(系)材料,由甘肃农业大学省部共建干旱生境作物学重点实验室麦类种质创新课题组提供。
1.2 SNP分子标记检测
基于Illumina SNP Genotyping技术测试平台,采用微阵列芯片(BeadArrey)技术对Illumina 9K SNP标记(包含9 000个标记)检测,由北京博奥晶典生物技术有限公司完成。试验步骤如下:用NaOH对基因组DNA分子进行变性;将基因组DNA在28℃恒温静置12 h,全部进行PCR扩增;利用随机内切酶切断扩增DNA分子,使其片段化;采用异丙醇使DNA片段沉淀,再次悬浮;芯片的微珠与50 mer长度的捕获探针相结合,而gDNA酶切后的片段不与探针DNA互补序列相连,静置12 h左右;芯片清洗,清除杂交失败或杂交错误的DNA片段;单个碱基变化以及染色,染色后,A和T、C和G将分开标记不同的荧光染料类型;芯片扫描,利用软件基于2种不同的荧光识别分析数据并计算出其分型结果。通过软件Ntsys-pc 2.02计算大麦参试材料间的遗传学相似系数。
1.3 大麦叶斑病抗性鉴定
大麦叶斑菌选用Z14484强侵染力菌株(采自甘南)。试验于2017年9月—2018年11月在甘肃农业大学人工气候室内进行,将土、沙、蛭石按体积比3∶1∶1混合均匀,经高压灭菌(121℃、30 min)后装填于直径为18 cm的花盆中。在无菌条件下用70%乙醇处理大麦种子30 s,5%次氯酸钠处理5 min,然后用无菌水冲洗3次,再用无菌滤纸将种子表面水分吸干,晾干后在培养皿中发芽,待发芽后移栽到花盆中。每份材料设置3个重复,每个重复种10株幼苗,在22℃的温室中培养。待幼苗长到三叶期时,用蒸馏水清洗叶片表面的蜡质层,最后将配置好的孢子悬浮液(0.25%Tween,99.45μL水,1×104cell·mL-1孢子含量)喷洒到叶片上,每天喷洒2次,接种3 d,在接种期间温室处于黑暗状态,接种温度22℃,湿度100%。接种9 d后调查各供试品种的发病病级[13],并计算病情指数(disease index,DI),病害和抗病等级划分如表1和表2所示。

表1 病害等级划分[13]Table 1 Level division of disease[13]

表2 抗病类型划分Table 2 Classificatrion of resistant types

式中,DI为病情指数;si为发病级数;ni为相应病级级别的株数;i为病情分级的各个级别,N为调查总株数。
1.4 数据处理
本研究通过Excel和SPSS 19.0进行数据统计和分析,运用Powermarker计算碱基对比例、基因多样性和多态性信息量(polymorphism information content,PIC)。利用软件Structure 2.3.1进 行群体遗传结构分析,预估最佳群体数K值。1~10为K值取值范围,将参数iterations调整为10 000,burnin period调整为100 000,每个K值叠加 运行10次,根据最大似然值原则确定最标准的K值为群体数目,计算Q值用作协变量,并选用PLINK1.09软件进行全基因组关联分析(GWAS),设 置maf≤0.05,显 著 性 水 平 阈 值P<10-4,使 用R3.5.2制作曼哈顿图。

式中,K为亚群数;L(K)为K的最大似然数,|L″(K)|为连续值之间差值的绝对值。
2 结果与分析
2.1大麦叶斑病的抗性鉴定
对200份大麦种质材料人工接种大麦叶斑病病原菌Z14484,接种9 d后进行抗性评价,结果(图1和表3)表明:参试材料的抗病性差异较大,病情指数分布在5.45%~95.16%之间。其中,蒙啤麦3号等17份材料属于抗性,占所有参试材料的8.5%;ZYM00695等34份材料属于抗-中抗,占所有参试材料的17%;中抗品种和中抗-中感材料各有40和43份,分别占20%和21.5%;中感材料37份,占18.5%;中感-高感材料28份,占14%;高感材料仅有1份(蒙啤麦1号),本研究中未筛选出免疫和高抗材料。

表3 大麦叶斑病抗性Table 3 Resistance to Bipolaris teres spot blotch of barley varieties 续表Continued
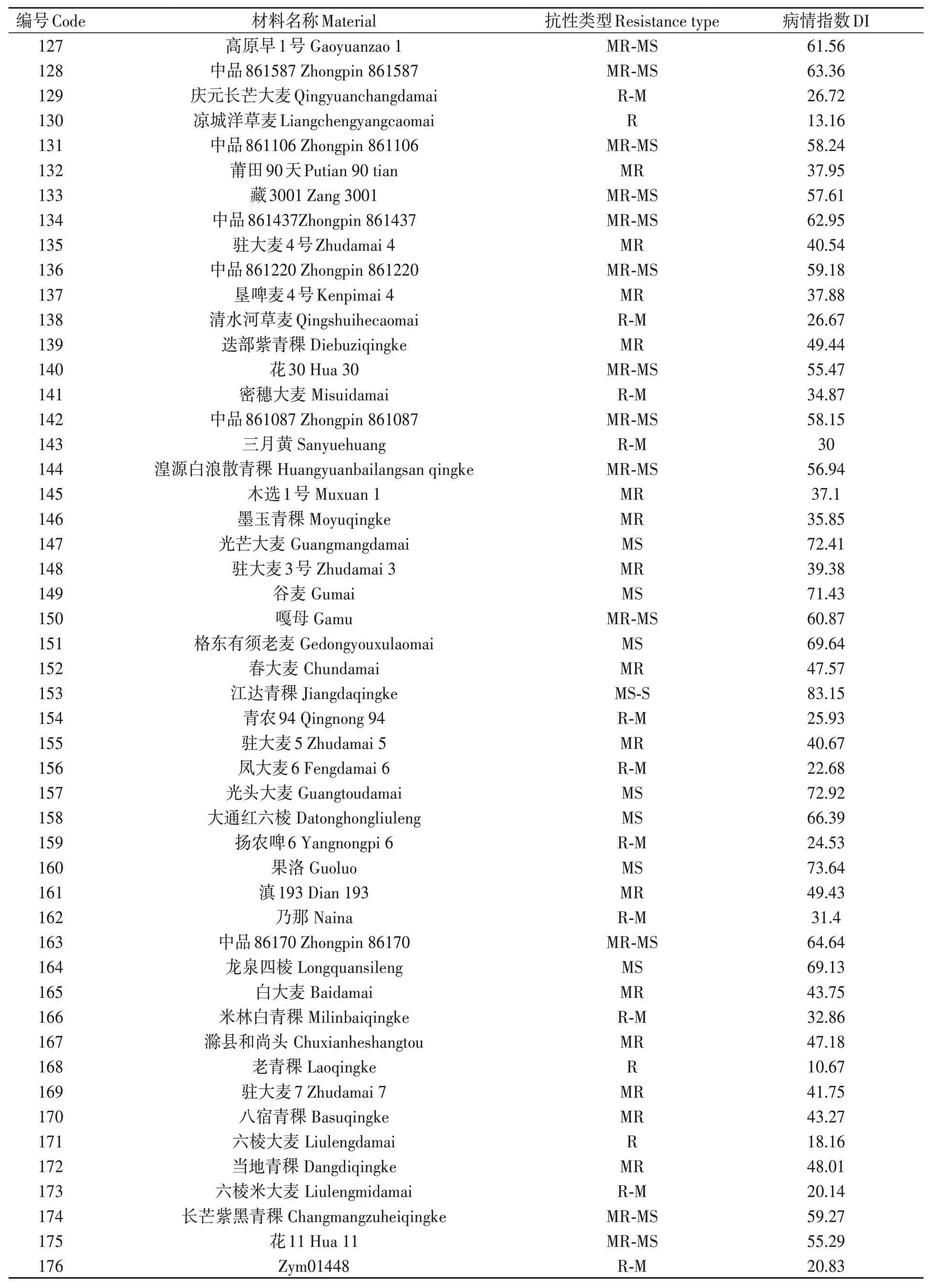
表3 大麦叶斑病抗性Table 3 Resistance to Bipolaris teres spot blotch of barley varieties 续表Continued
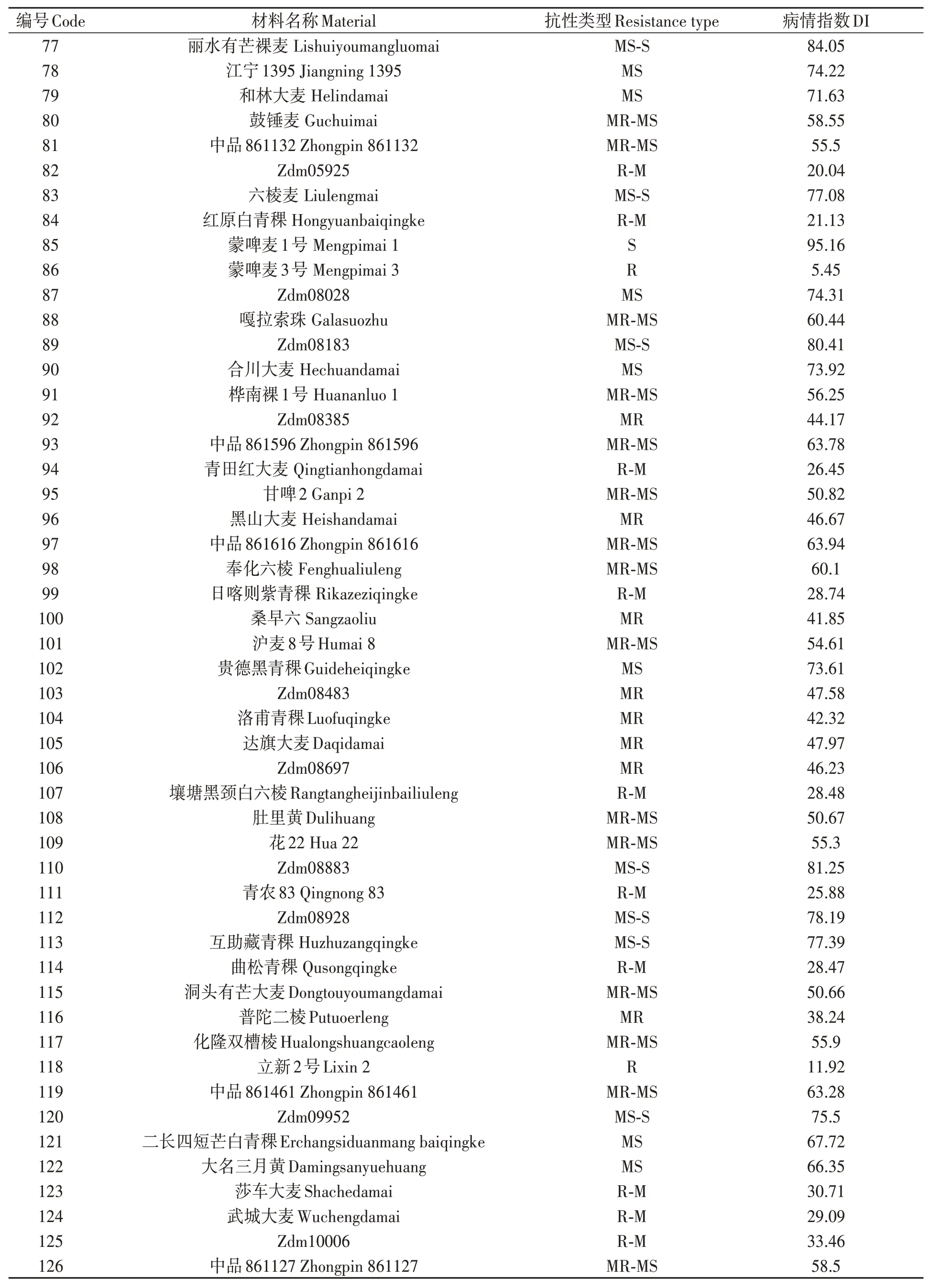
表3 大麦叶斑病抗性Table 3 Resistance to Bipolaris teres spot blotch of barley varieties 续表Continued
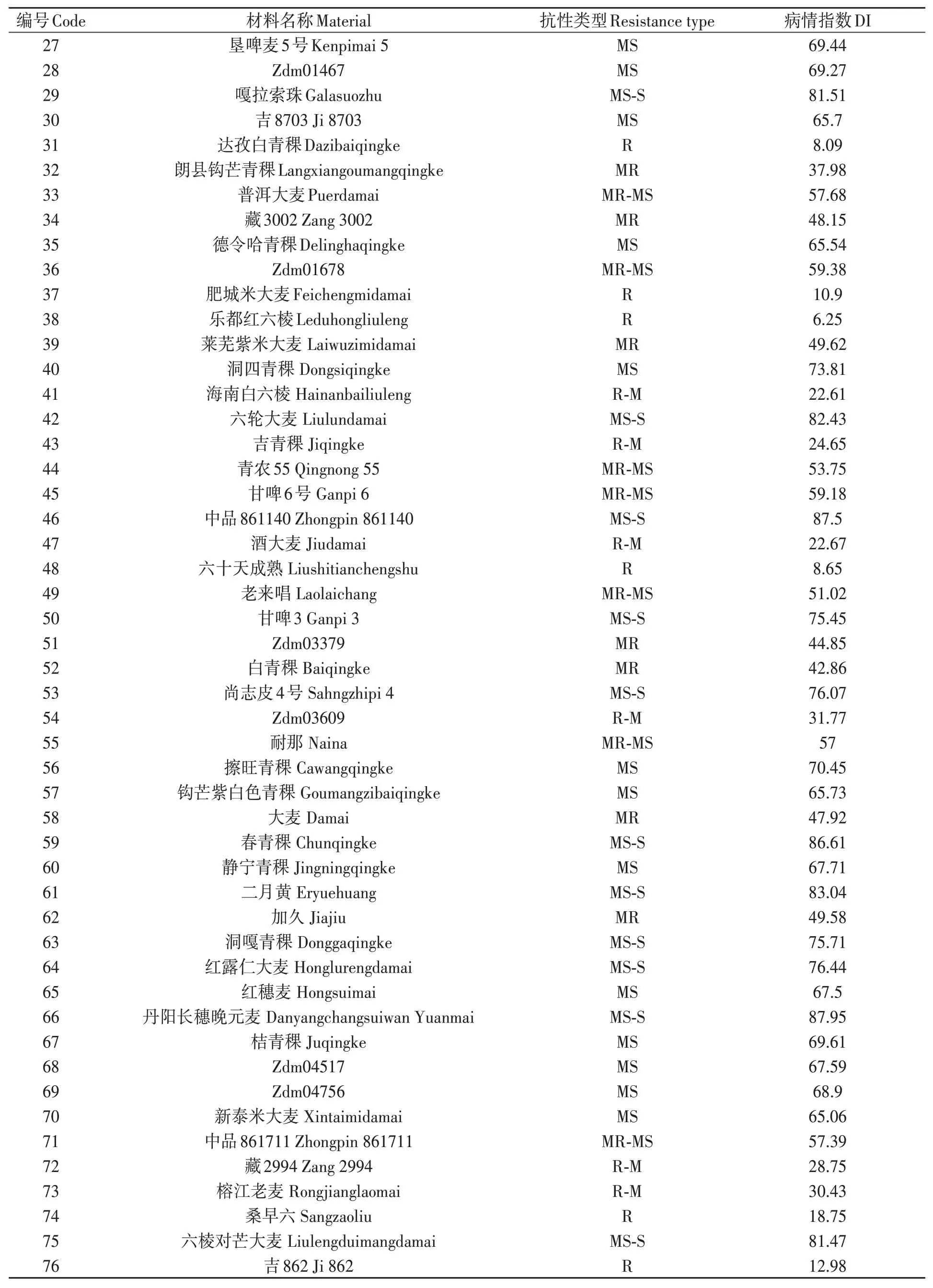
表3 大麦叶斑病抗性Table 3 Resistance to Bipolaris teres spot blotch of barley varieties 续表Continued
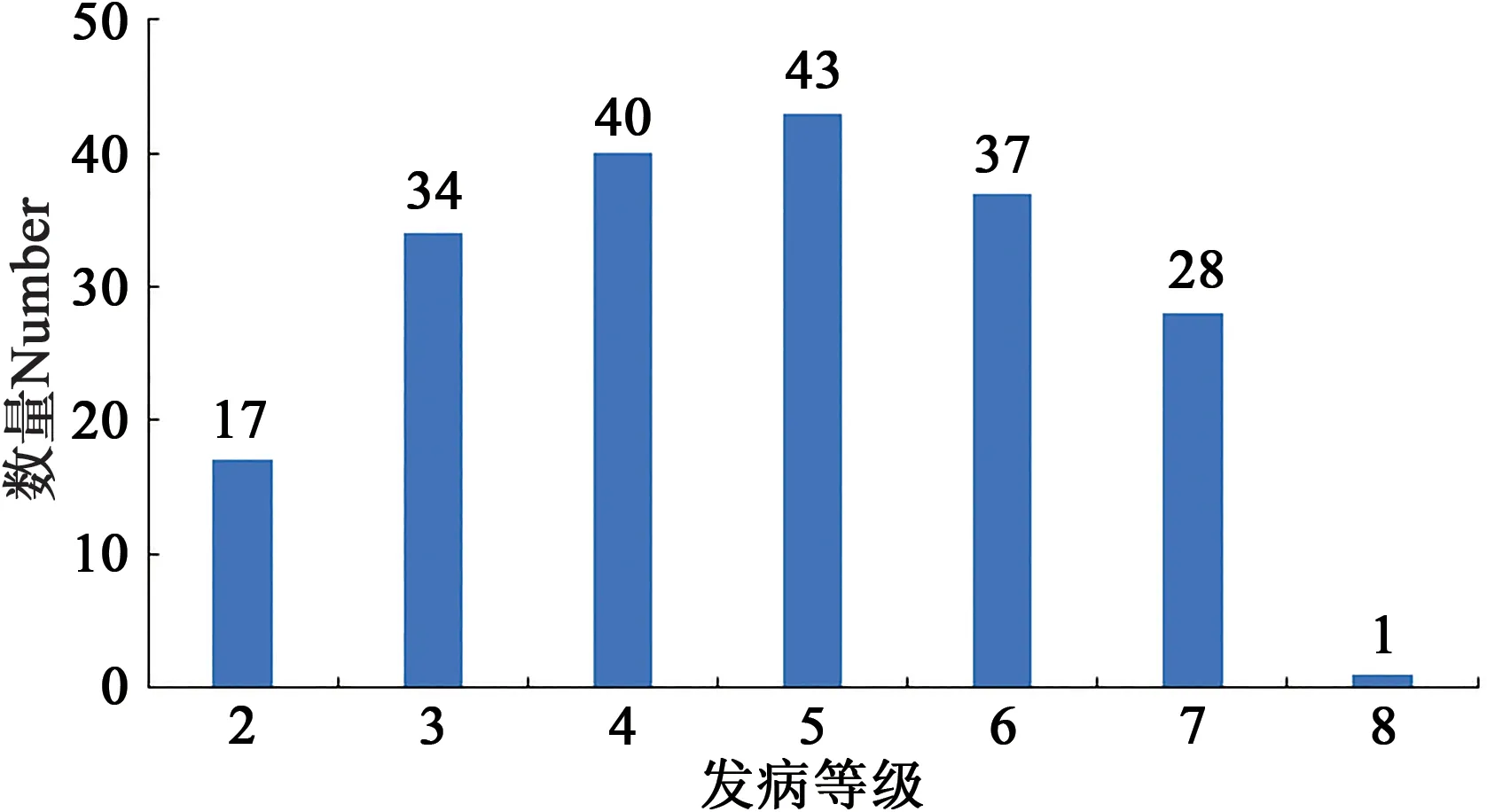
图1 群体发病级别评价Fig.1 Evaluation of incidence level of strains

表3 大麦叶斑病抗性Table 3 Resistance to Bipolaris teres spot blotch of barley varieties
2.2 大麦SNP基因型分析
本研究使用基于Illumina SNP Genotyping技术的9K基因芯片,筛选出6 454个具有特异性的SNP标记,分布在7条染色体上,平均每条染色体922个。其中,4H染色体最多,达1 288个;1H染色体最少,为642个。位点之间的距离平均为158 kb,1 H上的SNP标记最密,平均距离为207 kb;2H上的SNP标记最稀少,平均距离为131 kb。6 454个SNP标记包括8种类型的碱基替换,其中A/G类型有2 386个,在所有碱基替换类型中最多;T/A类型的最少,有104个(表4)。

表4 碱基变异比较Table 4 Comparison of base variation
2.3 群体遗传结构分析
通过SNP位点,利用Structure 2.3.1分析200份大麦材料的群体遗传结构,结果(图2和3)表明:不同亚群内大麦参试种质的相似程度最为接近,当K=3时ΔK最大,因此可以判定200份大麦被分为3个亚群,分别有100、67和33份材料。其中第I亚群主要为青海、甘肃、新疆、西藏和四川部分材料,如青海共和的海南白六棱,西藏昌都等八宿青稞、吉塘青稞,四川壤塘的壤塘黑颈白六棱等;第Ⅱ亚群主要为江苏、浙江、福建等地材料;第Ⅲ亚群主要来自于四川、山西、上海等地,如四川和林大麦等。

图2 ΔK值随K值的变化Fig.2 Change ofΔK value with K value
2.4 大麦抗叶斑病全基因组关联分析
结合SNP位点分析与抗性评价结果对全基因组进行关联分析,以每个信号区域内-logP值最高的SNP物理位置代表位点区段。检测到的GWAS信号位点如图4所示,共检测到12个位点,分别位于3号和6号染色体上。
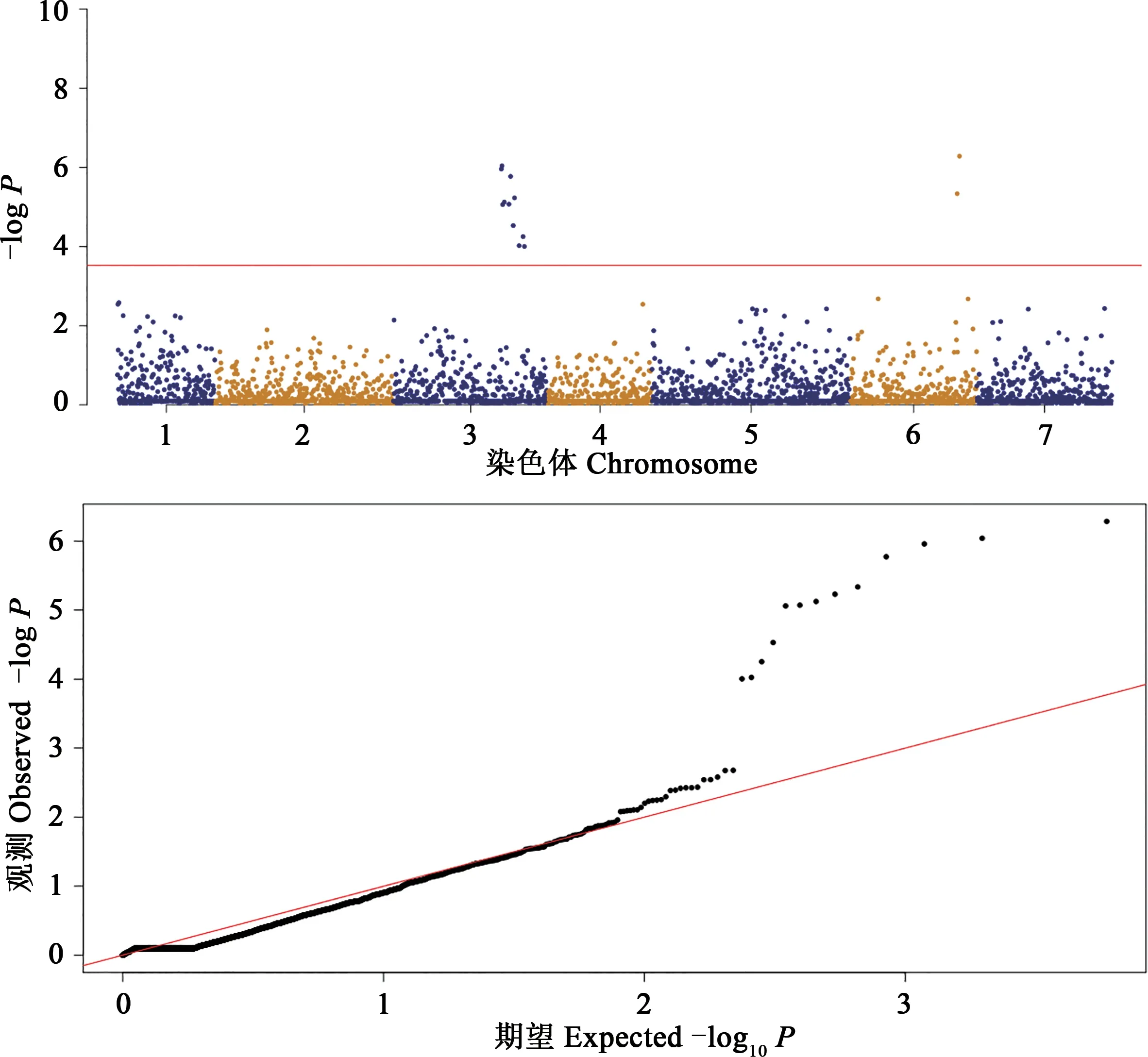
图4 大麦材料抗叶斑病曼哈顿图与Q-Q图Fig.4 Manhattan and Q-Q map of core germplasm resistance to spot blotch
2.5 大麦抗叶斑病全基因组关联位点候选基因分析
在全基因组上有12个与大麦叶斑病抗性关联的SNP位点,分别位于3H和6H染色体上,其中4个显著关联(表5)。在NCBI(https://www.ncbi.nlm.nih.gov/)上 对 这4个SNP位 点 进 行 查询,位点SCRI_RS_168529为核小体组装蛋白1∶2基 因(nucleosome assembly protein 1∶2,HORVU1Hr1G-021800.1);位点BOABC_7462109为FYVE锌指修饰的RCCI基因(RCC1 with FYVE zinc finger,HORVU3Hr1G107820.4);位点SCRI_RS_168096为铁-3超氧化歧化酶基因(Fe superoxide dismutase 3,HORVU6Hr3G020590.16);位点SCRI_RS_171415为麦类高尔基蛋白基因5(A.tauschiigolgin candidate 5,LOC109755746)。同时,位于3H上的BOPA1_11_11436和SCRI_RS_201075的POS值(与本染色体初始位点相对距离)分别为145.69和145.65,距离为42 323 kb。

表5 GWAS检测到的信号位点与SNP标记信息Table 5 Information of signal sites and corresponding SNP markers by GWAS

图3 200份大麦种质材料的群体遗传结构Fig.3 Population structure of 200 barley accessions
3 讨论
大麦叶斑病抗性材料的筛选方法主要包括田间病圃自然感病和室内人工接种鉴定。在自然环境中,由于控制发病所需条件较为困难,而室内人工接种在控制发病温度、湿度等条件下,能够显著提高接种的成功率和试验的可靠度。Gyawali等[8]通过使用来自于摩洛哥的19种叶斑病原体对336个基因型的大麦在苗期和成熟期进行了抗性鉴定,发现抗病材料仅有43份;Singh等[14]对来自于印度奥瓦拉纳西的124个大麦品种进行为期3年的抗性评估,并对发现的高抗品种BCU422、BCU1204和BCU5092进行了相关性分析;Ghazvini等[15]使用了112个不同的大麦株系对127种病原菌进行抗性鉴定,发现致病性类型多达10余种,并且不同种质对于不同菌株抗性并不相同;姚全杰等[16]通过多种叶斑病菌株对不同生长时期大麦材料进行抗性鉴定,发现233种大麦材料中只有10份材料在不同时期对不同菌株均表现出抗性;吕二锁等[17]通过2次鉴定,仅发现15种抗性材料。
本研究中不同大麦品种对大麦叶斑病抗性有明显差异,参试材料的病情指数在5.45~95.16之间。蒙啤麦1号病情指数为95.16,对Z14484表现高感;蒙啤麦3号病情指数为5.45,对菌株Z14484表现为抗性,这与郭焕强等[18]和姚全杰等[16]的研究结果基本一致。但是垦啤麦4号对于Z14484的病情指数达37.88,与姚全杰等[16]发现的垦啤麦4号抗性结果中感-高感存在较为明显差异,说明大麦对于叶斑病抗性是相对的,相同大麦材料受接种菌株侵染力和毒性强弱等条件的影响,致使大麦对于叶斑病抗性也不相同。在自然环境下大麦叶斑病致病菌株类型较多,不同菌株对于大麦叶斑病致病特性又有所区别,故在选育大麦叶斑病抗性品种时,为筛选出综合抗性较强的品种,应使用多个菌株进行筛选。
全基因组关联分析(GWAS)是对基因组中数量巨大的SNP作为遗传标记进行分型,结合表型之间的联系进行全基因组水平的关联分析,通过比较SNP与性状之间的关系,可以快速筛选出具有抗性基因的区域甚至位点。目前全基因组关联分析广泛应用到各种植物的基因挖掘之中[19-20]。魏爽等[21]在黄瓜耐热性的研究中进行了GWAS分析,检测到6个SNP位点,并筛选出相应的候选基因;任义英等[22]在甘蓝型油菜籽粒着生密度等性状的研究中进行了GWAS分析,检测到10个相关SNP位点并作出分析;王育才等[12]结合SNP位点对384种大麦材料和株高、穗长进行了分析[12,21-22]。本研究对于大麦叶斑病抗性基因进行了挖掘,发现了12个位点,其中位于3号染色体上的11_11436和SCRI_RS_201075较为接近,是较为可靠区域,另外对于SCRI_RS_201075附近的200 kb区段候选基因进行了分析,发现其中4个基因参与了大麦叶斑病抗性过程,有2个基因与大麦叶斑病抗性直接相关,即过氧化物酶-1(peroxidase-1)和铁-2超氧化歧化酶(Fe superoxide-2)基因。大麦叶斑病抗性是较为复杂的数量性状,其中牵涉到2个物种之间的互作关系,相较前人的研究结果[23-24],本研究在全基因组范围内检测到12个苗期抗叶斑病相关的SNP位点,且位于3H染色体的位点较为可信,可在今后的工作中结合QTL定位结果,继续挖掘候选基因。与此同时,本研究的试验结果也证明了GWAS是挖掘病害关联基因位点的有效手段,后续工作也应该继续就挖掘出来的位点进行更深入的研究。
本研究使用大麦叶斑病病原菌Z14484,对来源不同的200份大麦种质材料进行鉴定,结果供试材料抗性差异较大,病情指数分布在5.45%~95.16%之间。通过对供试材料进行GWAS分析,发现12个标记与大麦抗叶斑病相关联,并且筛选出4个可能存在相关性的基因。本研究为大麦叶斑病抗病育种提供了一定的理论依据。

