基于叶绿体基因组SNP的天台鹅耳枥谱系结构与分化分析
陈模舜, 杨仲毅
( 台州学院 生命科学学院, 浙江省植物进化生态学与保护重点实验室, 浙江 台州 318000 )
天台鹅耳枥(Carpinustientaiensis)属于桦木科(Betulaceae)鹅耳枥属(Carpinus),为中国特有和濒危的第三纪孑遗植物,是国家二级重点保护野生植物(章绍尧等,1993)。我国是鹅耳枥属植物的分布中心,约有33种8变种(傅立国,2003),其中东南沿海是鹅耳枥属植物一个重要的分布区,天台鹅耳枥(C.tientaiensis)和普陀鹅耳枥(C.putoensis)等为浙江地区特有(陈之端,1994)。中国植物志曾记载天台鹅耳枥产于浙江东部(中国科学院中国植物志编辑委员会,1979),浙江植物志统计天台鹅耳枥仅存5株(章绍尧等,1993),但近年来在磐安县和景宁县等地有所发现,仅间断分布于浙江省境内的天台县、磐安县、青田县和景宁畲族自治县,野外成年植株不足50株,幼年个体缺少,低于野外种群稳定存活界限,已处于极危状态。生境片断化使得野生居群变小及居群间的隔离程度增加,这将会导致遗传变异丧失和近交衰退,最终增加物种灭绝的风险(Aguilar et al.,2008; Wei & Jiang,2012)。根据IUCN物种红色名录,天台鹅耳枥属于极危(critically endangered,CR)等级。天台鹅耳枥染色体为14倍体(2n=14x=112),是桦木科倍性最高的多倍体植物(陈模舜等,2020)。天台鹅耳枥在研究桦木科分类、古植物区系、濒危机制等研究中具有很高的科研价值(章绍尧和丁炳扬,1993;王昌腾和叶春林, 2007)。
目前,仅对天台鹅耳枥组织解剖结构、光合特性对生长光强的响应、群落特征等方面进行了研究(陈模舜和柯世省,2013;陈模舜等,2020)。但天台鹅耳枥种群的进化机制与种内系统发育尚未明确,必须收集群体样本进行基因组学的分析,在基因组水平上加深对这一濒危物种居群动态的了解。叶绿体基因组(chloroplast genome,cpDNA)由于其高基因含量和保守的基因组结构,可用于研究开花植物的母系遗传,特别是多倍体植物(Birky, 1995;Soltis & Soltis, 2000)。cpDNA通常包含单亲遗传的 DNA,由于其自我复制机制和相对独立的进化,来自叶绿体的遗传信息经常被用来探索近缘种间和种内的亲缘关系(于涛等,2019)。目前,叶绿体全基因组高通量测序技术为系统进化分析提供了大量的信息位点,比较基因组结构、基因的变异和重复序列排列,有利于构建居群遗传结构、居群历史动态和谱系间分化(孙逸,2012;王佳慧,2015)。本研究通过对天台鹅耳枥叶绿体基因组高通量测序,通过对单核苷酸多态性(single nucleotide polymorphism,SNP)研究,分析多态位点和核苷酸变异,评估居群的遗传多样性水平,推断天台鹅耳枥的谱系结构和分化,为天台鹅耳枥种质资源的保护与恢复制定策略。
1 材料与方法
1.1 天台鹅耳枥样地调查及测试材料
天台鹅耳枥自然居群最高海拔相近,均在890 m以上,同属亚热带山地湿润气候,土壤pH值为酸性,森林植被覆盖率较高,植被类型为亚热带常绿阔叶林(陈模舜等,2020)。天台鹅耳枥分布范围狭窄,仅浙江省的天台县华顶山,磐安县的大盘山、高姥山和青明尖,青田县仰天湖和景宁畲族自治县上山头存在野生群落。其中天台县华顶山共存19株,青田县仰天湖仅存1株,景宁县大际乡上山头胸径20 cm以上18株,磐安县境内的大盘山共存3株,高姥山和青明尖各为1株。磐安县每个居群之间相隔20 km之内,天台县居群与景宁县居群直线距离约250 km(表1)。

表 1 天台鹅耳枥分布地的基本概况Table 1 Basic conditions of distribution regions of Carpinus tientaiensis
天台鹅耳枥材料采集自6个自然居群26个植株(包含所有居群的母株)(表1)。其中景宁县上山头样地50 m × 15 m,每隔5~6 m采集1株,共9株,平均胸径25.2 cm;孤树1株,分枝最大胸径11.1 cm。磐安县大盘山、高姥山、青明尖共采集5株,平均胸径20.41 cm。青田县仰天湖采集1株,胸径28.66 cm。天台县华顶山西茅棚采集7株,平均胸径43.08 cm,公路附近3株平均胸径20.07 cm。野外采集新鲜叶子后将其冷冻在干冰中,清洁叶片,并保存在-80 ℃的冰箱中以备后续实验研究。
1.2 cpDNA测序、组装与注释
cpDNA提取使用TIANGEN的DNAsecure新型植物基因组DNA提取试剂盒(DP320),然后在Illumina NovaSeq 6000平台,以150 bp的对端读数高通量测序,每个样本至少6 Gb的原始序列数据。在过滤原始数据并消除数据质量的影响(Phred分数Cutoff-30)之后,我们获得了高质量的数据。对获得26个天台鹅耳枥叶绿体全基因组序列,利用已发表的cpDNA序列(登录号:KY174338)作为参考序列(Yang et al., 2017),采用DOGMA软件注释完整的cpDNA(Wyman et al., 2004),使用在线程序OGDRAW(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)制作cpDNA图谱,同时对每条序列的cpDNA基本信息进行统计,包括基因组大小、基因特征和GC含量。
1.3 重复序列、IR区域与边界的扩张收缩和密码子偏好性分析
重复序列中散在重复使用Reputer软件分析,用cpLTR表示;简单重复序列(sequence of simple repeat,SSR)鉴定使用MISA(http://pgrc.ipk-gatersleben.de/misa/)工具(参数:1-10 2-5 3-4 4-3 5-3 6-3)分析。比较不同序列大单拷贝区(large single copy,LSC)、小单拷贝区(small single copy,SSC)和反向重复区(inrerted repeats,IR)长度及边界,使用在线工具IRscope(https://irscope.shinyapps.io/irapp/)完成IR区域与边界的扩张收缩分析。通过R软件分析密码子偏好并作图。
1.4 系统发育树分析
为了检查物种不同区域的系统发育,根据完整的cpDNA序列进行系统发育分析,使用RAxML 8.0软件进行极大似然树(maximum likelihood tree,ML tree)构建,结合MrBayes 3.3软件进行贝叶斯推断法构建 Bayes tree。基于cpDNA开发单倍型,运用PopART软件构建单倍型网络(Kimura, 1980;Leigh & Bryant, 2015)。使用DnaSP v6(http://www.ub.edu/dnasp/)软件分析核苷酸多样性参数,通过AMOVA分子方差分析,评估谱系间的分子变异程度和遗传分化固定指数Fst,分析得到谱系间的分化程度。
2 结果与分析
2.1 天台鹅耳枥叶绿体的基本特征
Illumina NovaSeq 6000测序平台,以150 bp的对端读数,每个样本平均产生3 000万对配对读取, 从26个天台鹅耳枥生成的数据中确定每个完整的cpDNA。天台鹅耳枥完整cpDNA由四部分组成,与鹅耳枥属(Carpinus)其他植物相似(杨霄月,2019;赵儒楠等,2021)。长度为159 281~159 841 bp,平均为159 616.2 bp,每个cpDNA由1个大单拷贝区(LSC)(88 360~88 711 bp,平均为88 522.35 bp)和1个小单拷贝区(SSC)(18 420~18 794 bp,平均为18 634.92 bp)组成,并由1对反向重复序列(IR)隔开(每个IR 26 067~26 451 bp,平均为26 229.46 bp)(表2,图1)。

表 2 天台鹅耳枥cpDNA特征Table 2 Characteristics of cpDNA of Carpinus tientaiensis
经过数据过滤后,Q20质量评分(96.46%~97.21%)平均96.98%,Q30质量评分(90.81%~92.46%)平均91.94%。基于叶绿体的统计,总体GC含量平均为36.41%,LSC、SSC、IR对应值分别为34.20%、30.09%、42.37%,IR区域的GC含量高于LSC、SSC区域。cpDNA的131个基因中,包含86个蛋白编码基因、37个tRNA基因和8个rRNA基因。其中:LSC区域包含60个蛋白质编码和22个tRNA基因; SSC区域包含12个蛋白质编码和1个tRNA基因;IR区域有7个蛋白质编码,7个tRNA和所有4个rRNA基因重复(表2,图1)。
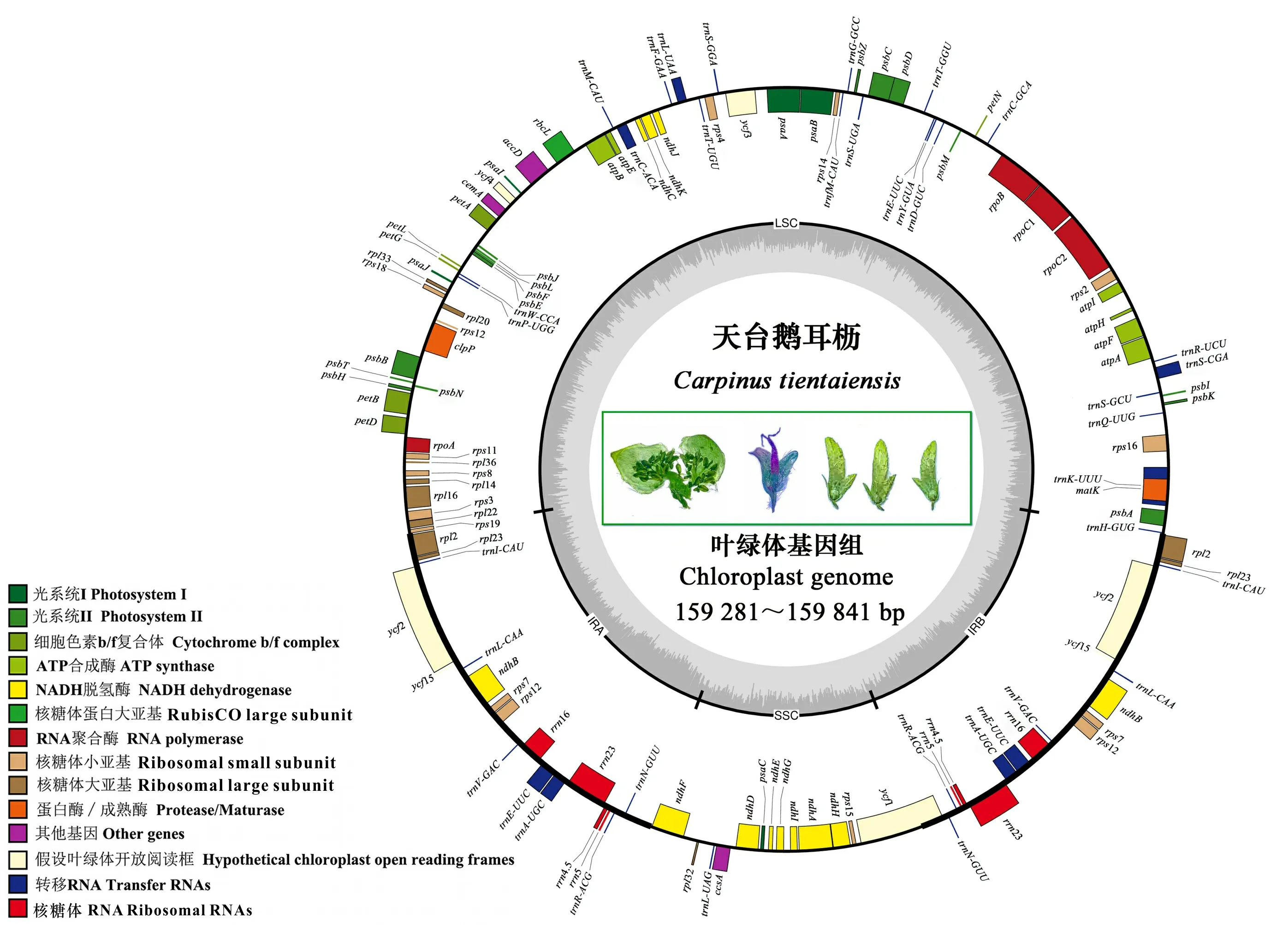
不同功能群的基因用颜色编码。外圆的基因是顺时针转录的,而内圆的基因则是逆时针转录的。内圆的虚线区域表示cpDNA的GC含量。Genes belonging to different functional groups are color-coded. Genes show the outer circle are transcribed clockwise and those inner circle are transcribed counterclockwise. Dashed area in the inner circle indicates the GC content of the cpDNA. 图 1 天台鹅耳枥cpDNA图谱Fig. 1 Gene map of Carpinus tientaiensis cpDNA
2.2 cpDNA的重复序列分析
滑动链的不匹配和重复序列的不适当重组可能导致序列变异和DNA重排(Wicke et al., 2011)。重复序列是重要的遗传标记,和物种的起源进化息息相关。重复序列一般可分为散在重复和简单重复。
散在重复使用Reputer软件分析,这里我们用cpLTR长末端重复序列表示。在天台鹅耳枥cpDNA中检测到正向重复平均32个、回文重复25个、反转重复22个(图2),这些重复中的大多数表现出10到38 bp之间的长度。简单重复序列(SSR)广泛分布于cpDNA中,包含长度为1~6 bp重复序列的短串联重复,cpDNA中的这种短串联重复是从单亲遗传过来的,SSR在基因组重组和重排中发挥重要作用,在群体遗传和进化研究中,常被用作有效的分子标记(Zhou et al., 2018)。基于SSR位于叶绿体区段类型的数量统计,天台鹅耳枥蛋白编码基因平均为48个基因、tRNA为3个基因、非编码区为41个基因。其中,蛋白编码基因中6个基因(matK、atpA、rpoB、atpB、cemA、rpl2)具有1个串联重复,1个rpoC2基因具有4个串联重复,1个ycf1基因具有2个串联重复。其中LSC 和SSC区域共有25个rps12基因串联重复,IR有10个rps12串联重复基因。
在cpDNA中有87个不同的SSR类型重复了10次以上,其中单核苷酸的数量最多,平均有50个,占总数的57.47%;其次是四核苷酸,为14个,二核苷酸为12个,三核苷酸、复合核苷酸各为4个,五核苷酸发现的比较少,为3个,而六核苷酸没有发现(图3)。在cpDNA中发现的SSR通常由A/T重复组成,很少包含G/C串联重复序列,其中单核苷酸由A/T碱基组成,占92%,这些SSR丰富了cpDNA的AT。cpSSR碱基重复序列中10条占31.20%、11条占16.95%、12条占34.44%、13条占5.65%、14条以上占11.76%(图4)。cpSSR分区IRA、IRB各占6.58%,LSC占68.59%,SSC占17.96%,其中LSC重复序列最多(图5)。在天台鹅耳枥cpDNA中,观察到SSR具有丰富的碱基重复和重复条数,可以作为居群进化研究有用的遗传信息。
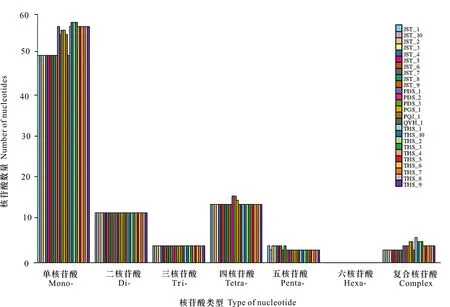
Mono-, Di-, Tri-, Tetra-, Penta-, Hexa-表示1~6核苷酸SSR,Complex表示复合核苷酸SSR。Mono-, Di-, Tri-, Tetra-, Penta-, Hexa- indicate 1 to 6 nucleotide SSR, and Complex indicates complex nucleotide SSR.图 3 天台鹅耳枥cpSSR的类型数量Fig. 3 Number of cpSSR types detected in Carpinus tientaiensis

图 4 天台鹅耳枥cpSSR碱基序列条数分布Fig. 4 Frequencies distribution of cpSSR base sequences identified in Carpinus tientaiensis

图 5 天台鹅耳枥SSR在LSC、IR和SSC区域的频率Fig. 5 Frequencies of identified SSR in LSC, IR and SSC regions of Carpinus tientaiensis
2.3 密码子偏好性分析
密码子在遗传信息的传递中起着重要作用,是核酸和蛋白质之间的联系。通过对所有蛋白质编码的cpDNA和氨基酸序列进行统计分析,天台鹅耳枥cpDNA序列的65.65%是蛋白编码基因,结果显示蛋白质密码子的相似性,其中AUG、UUA、AGA、GCU、UCU的频率较高,而CUG、GUG、AGC、CUC、CUG的频率较低(图6)。在这些密码子中,蛋白质编码基因中最常见的氨基酸是异亮氨酸(Ile),其在cpDNA中出现1 146次。相对同义密码子使用(relative synonymous codon usage,RSCU)值分析表明,色氨酸(Trp)蛋白质编码基因RSCU=1,表示该密码子没有偏好性。其中:47.62%密码子的RSCU>1,大多数(28/30,93.33%)以A或T(U)结尾;50.79%密码子的RSCU<1,大多数(30/32,93.75%)以C或G结尾(图6)。
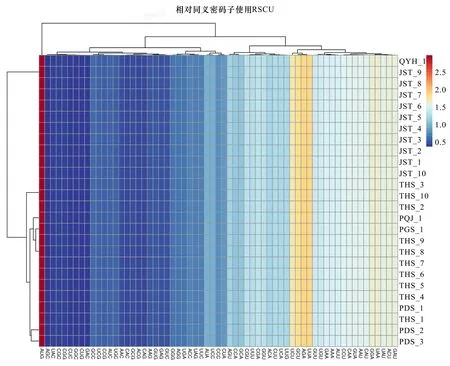
图 6 天台鹅耳枥相对密码子使用(RSCU)值聚类热图Fig. 6 Heat map of the relative synonymous codon usage (RSCU) value of Carpinus tientaiensis
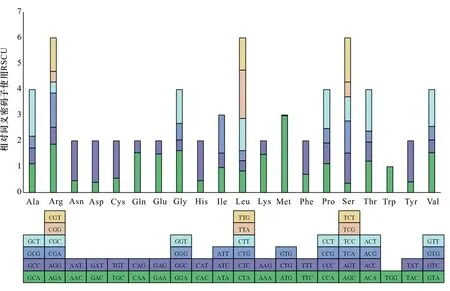
Ala. 丙氨酸; Arg. 精氨酸; Asn. 天冬酰胺; Asp. 天冬氨酸; Cys. 半胱氨酸; Gln. 谷氨酰胺; Glu. 谷氨酸; Gly. 甘氨酸; His. 组氨酸; Ile. 异亮氨酸; Leu. 亮氨酸; Lys. 赖氨酸; Met. 甲硫氨酸; Phe. 苯丙氨酸; Pro. 脯氨酸; Ser. 丝氨酸; Thr. 苏氨酸; Trp. 色氨酸; Tyr. 酪氨酸; Val. 缬氨酸。Ala. Alanine; Arg. Argnine; Asn. Asparagine; Asp. Aspartate; Cys. Cysteine; Gln. Glutamine; Glu. Glutamate; Gly. Glycine; His. Histidine; Ile. Isoleucine; Leu. Leucine; Lys. Lysine; Met. Methionine; Phe. Phenylalanine; Pro. Proline; Ser. Serine; Thr. Threonine; Trp. Tryptophan; Tyr. Tyrosine; Val. Valine.图 7 天台鹅耳枥蛋白质编码基因中20个氨基酸密码子含量Fig. 7 Codon contents of 20 amino acids in all protein-coding genes of Carpinus tientaiensis
在天台鹅耳枥物种的蛋白质编码cpDNA中,20个氨基酸由63个密码子编码,其中除天冬氨酸(Asp)外,大多数氨基酸都具有密码子偏好性。总共确定了40个密码子偏好,其中涉及19个氨基酸。在优选的密码子中,63.49%表现出较高的偏好(图7)。该结果进一步揭示了天台鹅耳枥cpDNA的相对保守性,因为高密码子偏好也是高等植物中的常见现象(喻凤和韩明,2021)。
2.4 cpDNA变异检测
单核苷酸多态性(SNP)和基因组结构变异在进化过程中至关重要(Britten et al., 2003)。cpDNA结构性变异有插入/缺失、转换、颠换和基因组结构重排。在26个天台鹅耳枥cpDNA中鉴定了314条SNPs,JST居群SNPs平均为132条、QYH为126条、PDS为18条、PGS为18条、PQJ为8条、THS为12条。以居群植株与THS _1 cpDNA比对,结果表明JST居群碱基颠换数(transversion,Tv)平均为95个、QYH为90个、PDS为16个、PGS为15个、PQJ为6个、THS为12个(表3)。研究的所有成对序列比较表明,颠换次数多于转换次数,这在其他分类群中也被发现(Stoltzfus & Norris, 2016)。
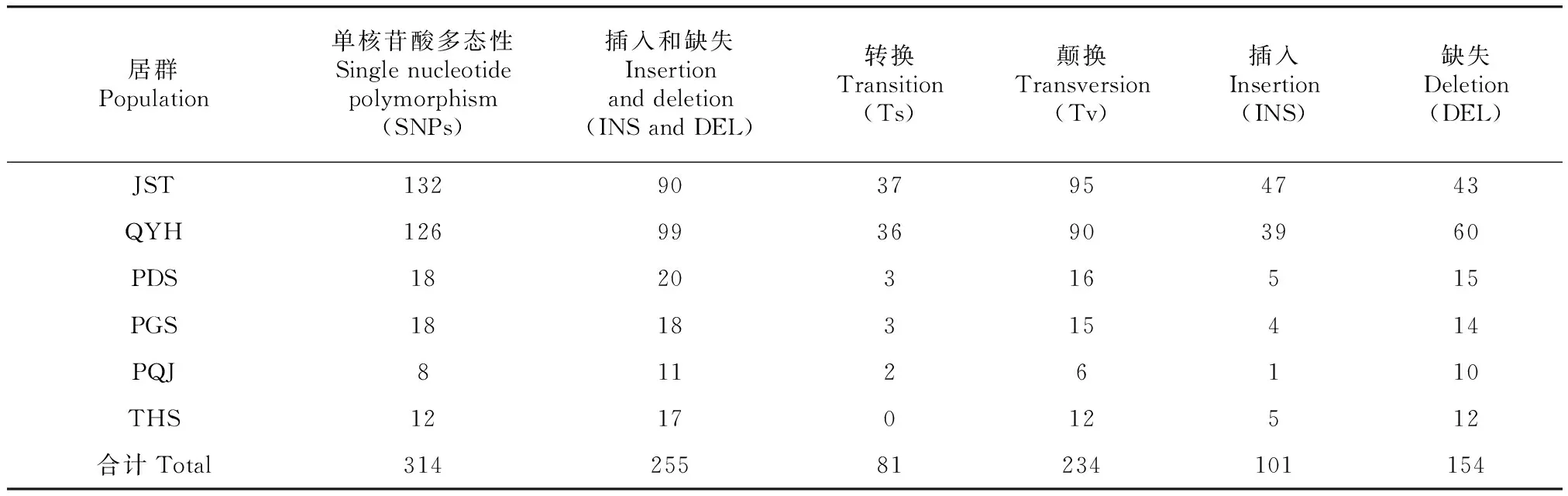
表 3 天台鹅耳枥叶绿体比较基因组统计表Table 3 Chloroplast comparative genome statistical table of Carpinus tientaiensis
2.5 IR区域与边界的扩张收缩
被子植物的cpDNA是高度保守的,IR 区域和单拷贝(single copy,SC)边界区域的扩展和收缩是造成高等植物cpDNA长度变化的主要机制(Saina et al., 2018)。比较26个完整的天台鹅耳枥cpDNA的IR/SC边界区域,发现在连接位置上有明显差异。其中,rpl2基因的长度都是1 513 bp,其端点较保守,离JLA、JLB基因间隔区都是71 bp。trnN-GUU的长度是72 bp,离JSB、JSA基因间隔区变化为1 516~1 864 bp,不超过以下基因间隔区的终点。rps19基因在JSB/IRb边界处由JSB向IRb延伸3 bp。ycf1基因跨越JSA/IRa区,4 211~4 553 bp位于SSC区域内,向IRb延伸1 192~1 540 bp(图8)。这4个cpDNA中IR/SC边界处的变异导致了整个cpDNA序列长度的差异,并在其他植物中也已发现(Yin et al., 2018)。
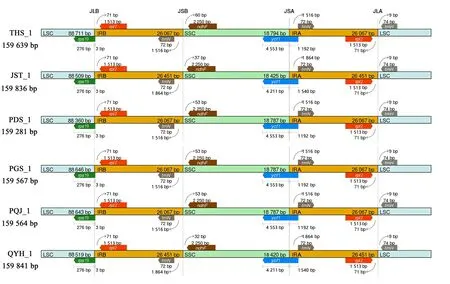
基因用彩色框表示。基因特征上方的数字表示基因末端与边界位点之间的距离。Genes are denoted by colored boxes. The numbers above the gene features indicate the distance between the ends of genes and the border sites.图 8 天台鹅耳枥cpDNA的IR-SC区的边界比较Fig. 8 Comparison of IR-SC border positions across cpDNA of Carpinus tientaiensis
2.6 基于完整的cpDNA序列的群体分析
2.6.1 单倍型网络构建 通过天台鹅耳枥cpDNA分析,运用一段遗传连锁的核酸序列的变异来区分,构建单倍型网络(haplotype network)。在系统发育分析时,核苷酸碱基一个插入或缺失作为一次进化事件进行编码分析。天台鹅耳枥单倍型中的大多数具有居群单倍型,几乎没有观察到居群之间的单倍型共享。在地理上接近的天台县(THS)(H1~H3)、景宁县(JST)(H4~H6)、磐安县大盘山(PDS)(H7~H9),各居群内具有相同单倍型;而磐安县青明尖(PQJ)H10、磐安县高姥山(PGS)H11和青田县(PQJ)H12是在单个标本中发现的私有单倍型。根据统计单倍型网络图(图9),揭示了两组单倍型,在此研究中称为浙东组THS居群和浙西组JST居群,由95个突变步骤隔开。根据多态位点分析JST居群和THS居群可变位点和插入,在rps16-trnQ序列有3个核苷酸取代,分别为T→C(位置275)、A→G(位置299)和T→G(位置1254)区分了浙东组THS居群(T、A和T)和浙西组JST居群(C、G和G)。从网络图推测可能的种群历史,除来自THS居群和JST居群单倍型之间碱基突变为95个较远外,居群组单倍型之间演化关系呈现星状中心辐射。THS居群的单倍型演化出PDS亚群、PQJ亚群和PGS亚群,居群组单倍型之间差异仅2~6个碱基突变,THS居群与PDS亚群单倍型之间的替代链接提示可能存在同源性;JST居群的单倍型演化出QYH亚型,2个单倍型之间少量差异(1~2个碱基突变),显示出天台鹅耳枥居群在历史上遇到瓶颈后曾发生局部扩张。
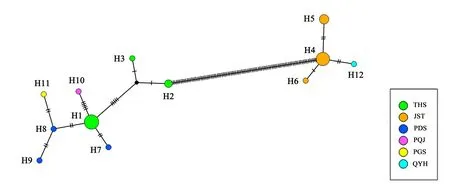
网络图中每个圆圈代表一个单倍型,每个圆圈的大小与观察到的频率成比例,圆圈颜色代表不同的居群,分支上的短线表示单倍型间的碱基替换数。Each circle in the network represents a haplotype, the size of the circle represents the frequency of haplotypes, and different colors in the circle represent different populations, and short lines on branches represent base substitutions between haplotypes.图 9 基于cpDNA的天台鹅耳枥单倍型网络Fig. 9 Network of haplotypes of Carpinus tientaiensis based on cpDNA
2.6.2 系统进化树构建 cpDNA包含丰富的系统发育信息,已被广泛用于近缘种间和种内水平的系统发育重建。使用cpDNA数据,解决了与各种系统发育困难群体相关的长期争议。为了评估天台鹅耳枥系统发生关系,使用26个天台鹅耳枥植株全叶绿体序列进行系统发育分析。通过极大似然树(RAxML)和贝叶斯推断法(MrBayes)生成系统树,系统发育分析每个节点数字为极大似然法(ML)支持值(Bootstrap support,BS)(%)和贝叶斯(BA)后验概率(posterior probability,PP)(%)(Stamatakis, 2014;Xie et al., 2018)。
在完整cpDNA序列中,通过系统分析,6个自然居群分为THS居群和JST居群(100BS/100PP)。THS居群包含第一分支THS_10和THS_2~3(65BS/83PP);第二分支PQJ_1(61BS/60PP),PDS_1(59BS/73PP),PGS_1和PDS_2~3(70BS/100PP),THS_1和THS_4~9(70BS/100PP)。JST居群包含第一分支JST_7和JST_9~10(89BS/100PP);第二分支JST_1~6、JST_8和QYH_1(85BS/100PP)(图10)。LSC、SSC数据集的拓扑结构与物种cpDNA系统树具有一致性,在种内进化枝中仅发生了细微的拓扑差异,支持形成单系的群体。

A. cpDNA ML系统发育树; B. cpDNA Bayes系统发育树。分支数表示 ML Bootstrap支持值/Bayes后验概率。A. cpDNA ML system development tree; B. cpDNA Bayes system development tree. Number of the branches indicate ML Bootstrap support value/Bayesian posterior probability.图 10 基于完整cpDNA序列的最大似然和贝叶斯推理方法重建6个分类群的系统发育树Fig. 10 Phylogenetic tree reconstruction of six taxa using maximum likelihood and Bayesian inference methods based on the complete cpDNA sequences
2.6.3 谱系结构分析 利用DnaSP计算遗传多样性参数,通过AMOVA分子方差分析,评估谱系间的分化变异程度,遗传分化固定指数Fst分析得到谱系间的分化程度。核苷酸取代和插入/缺失变异揭示了6个天台鹅耳枥分布区中的12个cpDNA单倍型(Nh),这些单倍型中的25%是在单个植株中发现的,几乎没有观察到居群之间的共享单倍型。谱系间的分化可以通过对单倍型多样性指数(Hd)与核苷酸多样性指数(Pi)进行分析,其数值越大,说明其遗传多样性越高(周文漪,2014;Nikulin et al., 2020)。其中,PGS亚群、PQJ亚群和YHS亚群由单一植株组成,仅有私有单倍型。JST居群Pi为0.000 01,THS居群Pi为0.000 02,PDS居群Pi为0.000 03,所有居群核苷酸多样性的变异均较低(Pi<0.005)。从单倍型多样性指数看,JST居群Hd为0.6,THS居群Hd为0.511, JST居群和THS居群单倍型多样性相对较低,其中PDS亚群Hd为1,由于PDS居群仅有3株个体,单倍型多样性偏高(表4)。

表 4 谱系多样性参数统计表Table 4 Statistical table of genealogical diversity parameters
天台鹅耳枥6个居群的分子变异分析(AMOVA)结果表明,居群间的遗传分化固定指数Fst为0.970 9,谱系间分化较大,表明在整个遗传变异中居群间遗传变异占97.09%,居群内遗传变异占2.91%,居群间的遗传变异大于居群内(表5)。这种分布格局的主要原因是生境的片段化,地理隔离阻碍了居群间的基因交流(孙逸,2012;郑鑫,2015;Nikulin et al., 2020)。

表 5 谱系分子方差检验统计表Table 5 Statistical table of genealogical molecular variance test
3 讨论与结论
完整的cpDNA序列可提供丰富的系统发育信息来源,天台鹅耳枥cpDNA的长度平均为159 616.2 bp,GC含量相似,为36.41%,说明该物种cpDNA的高度保守性质。重复分析显示在天台鹅耳枥cpDNA中发现LTR重复序列包括正向重复平均32个、回文重复25个、反转重复22个,SSR重复序列不同类型87个。这些重复中的大多数位于蛋白编码区、非编码区和tRNA中。在藻类和被子植物基因组中LTR重复序列很常见,是促进cpDNA重排的主要因素之一,并且许多重排终点都与此类重复序列相关(Pombert et al., 2005;Zhang et al., 2020)。在所有个体中,SSR通常由A/T重复组成,其中单核苷酸由A/T碱基组成,占92%,大多数蛋白质编码基因都具有高度的密码子偏好性,在优选的密码子中,63.49%表现出较高的偏好,叶绿体密码子的第三个A/T偏好较高。相关研究表明基因组AT含量与重复序列的动力学以及叶绿体蛋白编码基因的密码子偏向性有关(Yu et al., 2019;Wu et al., 2020)。
cpDNA SNP的数量及碱基颠换为物种之间的系统发育解析提供了有益的标记(郑鑫,2015;Nikulin et al., 2020)。根据溯祖理论,通过单倍型网络图,结合地理信息我们可以推断种群的起源、扩散历史(Huang et al., 2014)。通过对SNP多态位点和核苷酸变异分析,单核苷酸取代显示天台鹅耳枥分为天台县居群(THS)和景宁县居群(JST),亚群间距离相近的优先聚为一支。除来自THS居群和JST居群单倍型关系较远外,居群组大部分单倍型之间仅有2~6个碱基突变,某几个同一或者相近地理居群的单倍型具有聚类现象,这可能由于该物种对亚热带湿润气候或暖温带气候的环境要求较高造成(陈之端,1994)。天台鹅耳枥具有居群组单倍型,没有居群之间的单倍型共享,这种单系进化枝可能是由于地理隔离导致短时期内居群之间基因交流较少造成的(Nikulin et al., 2020)。所有居群核苷酸多样性的变异均较低(Pi<0.005),JST居群和THS居群单倍型多样性较低(Hd为0.5~0.6), 表明最近发生过居群瓶颈效应(孙逸,2012;周文漪,2014)。由于晚第三纪及第四纪的气候动荡及冰期更替,天台鹅耳枥退缩到狭小区域的避难所,度过冰期;或群体局部扩张到湿润地区的森林中(陈之端,1994;Qi et al., 2012)。
通过对天台鹅耳枥谱系结构与分化的研究,对遗传多样性比较高的居群制定资源保存和引种驯化策略。在6个自然居群中都检测到了独特的单倍型,尤其THS居群、JST居群和PDS亚群具有较高的单倍型多样性, 栖息地面积较大,内部环境较稳定,需要加强栖息地保护,以维持天台鹅耳枥高的遗传多样性。天台鹅耳枥居群规模较小、隔离程度较高,居群间呈现较大的遗传分化,致使种源不断减少,是急需保护的濒危植物。对繁殖衰退的居群应开展遗传拯救,引入以花粉为主导基因流实验,移入新个体或基因型而减缓遗传侵蚀进而提高天台鹅耳枥种群生存力。

