融合密集连接与多尺度卷积的脑肿瘤MRI图像分割算法
杨述斌,王 锋,董春林
(武汉工程大学 电气信息学院,湖北 武汉 430205)
0 引 言
脑肿瘤是在人脑中不正常生长的细胞,有较高的发病率和病死率,严重危害着人类的健康。胶质瘤作是最常见的脑部肿瘤之一,可分为高级别胶质瘤(High-Grade Glioma,HGG)与低级别胶质瘤(Low-Grade Glioma,LGG)两种。HGG的生存率低,术后效果差,但如果在LGG阶段进行及时的治疗,可取得较好的术后效果[1]。核磁共振成像(Magnetic Resonance Imaging,MRI)是目前广泛应用于脑肿瘤检测的非侵入式成像技术,主要产生四个模态的图像[2],医生结合多个模态的图像来确定患者肿瘤的位置和区域范围,从而制定相应的手术方案。然而,一次MRI会产生大量的图片,对于图片的分割,传统的分割方法主要依赖专家的手工标注,这不仅费时费力,而且易受主观的影响,分割精度的偏差势必会影响后续的治疗,给患者带来额外的痛苦。因此,提出一个精确的脑肿瘤分割算法来给医生提供参考,具有十分重要的意义。
1998年,LECUN等[3]首次将卷积神经网络应用到图像分类,与传统方法相比,基于深度学习的方法具有自主适应特定任务的特点,可以高效地完成任务。近年来,卷积神经网络在图像分割领域具有突出的表现。LONG等[4]将全卷积网络用于语义分割,主要是将卷积神经网络末尾的全连接层替换为卷积层,实现了较为准确的分割。其后,DONG等[5]提出一种基于U-Net的2D脑肿瘤分割网络,提高了脑肿瘤分割的精度。ZHANG等人[6]为了防止层数过深导致梯度消失、梯度爆炸的情况,将残差模块融入到U-Net模型中,主要将传统的卷积块替换为残差结构,取得了较好的分割效果。MCKINLEY等[7]通过加入扩张卷积的方法来扩大感受野,从而获得大量的全局信息,提高定位精度来优化分割性能。牟海维等[8]人使用密集连接结构与U-Net进行融合,将密集连接应用于跳跃连接上,可以更好地弥补下采样的损失。
基于上述分析,本文提出了一种改进的U-Net算法来对肿瘤整体(Whole Tumor,WT)、肿瘤核心(Tumor Core,TC)和增强肿瘤(Enhancing Tumor,ET)区域进行分割。在原始框架上引入密集连接机制、空洞卷积、多尺度卷积来优化算法对于病灶的定位能力。
1 U型算法的构建
在脑部MRI图像中,病灶不规则且大小不一,这给分割带来一定的影响。为了提高算法对于脑肿瘤的分割精度,本文基于传统的U-Net框架[9]进行改进。首先,针对传统编码中上下文联系不密切的问题,利用密集连接机制将各通道的特征进行融合卷积,增强上下文间的联系;其次,由于传统卷积在编码和解码连接处定位不准确,利用空洞卷积可以在不增加参数量的情况下提高算法定位精度;最后,为防止在解码环节中特征提取不够全面,将传统的卷积块替换为多尺度融合卷积,防止过大或者过小病灶的丢失。
1.1 算法结构
本文提出的算法结构如图1所示。该算法保留了U-Net传统的编码、跳跃连接和解码结构。在编码环节,首先将尺寸为4×160×160的图片进行两次卷积以及一次最大池化,变成64×80×80,经过四次相同操作后变为512×10×10。本文在原始结构中引入了密集连接机制,将每层的输入由原始输入变为之前所有层的结合,提高特征的重复利用率,减少参数的流失。在编码和解码相连处,对编码环节得到的512×10×10的特征先将通道扩大为1 024后引入空洞卷积,最终变为1 024×10×10。空洞卷积可以增大卷积的感受野,提高定位精度。在解码环节,首先把1 024×10×10的特征进行上采样后与对应的编码层的特征进行拼接,后经过两次多尺度卷积变为512×20×20,重复四次操作之后变为64×180×180。本算法将传统的3×3卷积替换为多尺度卷积,充分提取跳跃连接传入的特征,避免参数丢失造成算法的退化,最后将解码传入的特征进行通道数为3的卷积操作,进行三个病灶区域的划分。
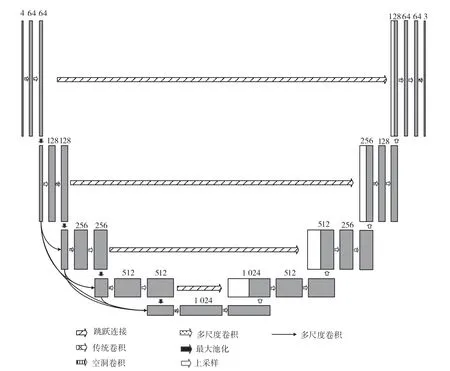
图1 网络整体结构图
1.2 密集编码部分
传统的U-Net架构只是简单地将输入的特征进行两次3×3卷积后利用最大池化操作传递到下一层,过程比较单一。随着层数的加深,网络会逐渐丢失浅层的细节信息。为加强上下层间的联系,保留更多的信息,本文在原始U-Net算法的基础上,在编码环节采用密集连接机制的思想[10]融合不同层数的特征,结构如图2所示。

图2 编码密集连接结构
传统的密集连接是将输出特征与之前特征进行拼接,最终的通道数会随着深度的加深而越来越大。为了降低后续的参数量,本文将原始密集连接的拼接改为特征融合,并且由于不同层数的图片尺寸和通道数不同,因此在特征融合之前使用1×1卷积以及最大池化进行参数匹配,使网络在保持参数量的情况下对特征进行复用,最大限度地减少参数的丢失,提高分割精度。
1.3 空洞连接卷积
原始算法将编码环节提取到的特征直接经过两个卷积模块,模块操作为先3×3卷积,后经过BN和ReLU后传递给解码环节。由于编码环节提取到的特征参数十分巨大,在比较小的感受野容易丢失部分位置信息,以至于削弱算法对于病灶的定位能力,因此,扩大算法的感受野是很有必要的。目前,增大感受野的方法主要有扩大卷积核以及增加层数两种方法。然而,增加层数容易导致算法参数量的增大。空洞卷积可以在不增加参数量的前提下增加卷积的感受野[11]。
由于经过编码环节后的特征大小为512×10× 10,并且过于扩大卷积的感受野易导致算法定位能力的退化,因此本文采用空洞系数为1的空洞卷积,在保证参数量的情况下提高算法对于病灶的定位能力。
1.4 多尺度解码部分
为避免因层数太深而导致特征的丢失,传统的U-Net算法提出跳跃连接的思想,将浅层特征与深层特征进行拼接,然后利用两个3×3卷积提取特征信息。但是传统的卷积容易导致部分参数的丢失,而多尺度的信息对于医学图像分割是十分重要的。为了充分提取拼接后的多尺度特征,本文将传统卷积替换为多尺度混合卷积块,利用不同大小的卷积可以提取不同尺度的特点[12],避免过大或者过小参数的流失,如图3所示。首先为了减少参数量,将输入的特征首先经过1×1的卷积来降低通道数,其后经过1×1,3×3,5×5的混合卷积来提取多尺度的特征,避免某些过大或过小特征的流失。并且3×3最大池化可以增强一些有用特征,减少无关特征,最大限度地减少参数的丢失,提高模型的分割精度。
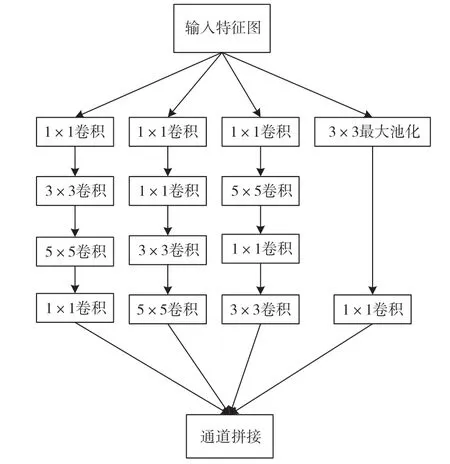
图3 多尺度卷积块
2 实验结果及分析
2.1 实验数据及预处理
实验数据为BraTs2019脑肿瘤分割竞赛数据集,其中包含335例患者的MRI的脑部影像,其中高级别脑肿瘤患者259例,低级别脑肿瘤患者76例,图像尺寸为240×240×155,其对应的金标准由多位专家联合标注。比赛要求[13]是在病灶图像中分割出浮肿区域(ED)、增强肿瘤区域(ET)以及坏疽区域(NET),分别将其标记为绿色、黄色和红色,并将这三个区域合并为三个嵌套的区域进行评估,主要包括肿瘤整体(WT=ED+ET+NET)、肿瘤核心(TC=ET+NET)以及增强肿瘤(ET=ET)。
为提高模型的分割性能,增强模型的鲁棒性,首先对数据进行预处理。原始图片中脑部周围存在大量的黑边,这不仅会给模型训练带来额外的功耗,并且对分割精度也造成一定的影响。因此将脑部周围黑边进行裁剪,删除一些脑部占比非常小的图片。由于三维图片数量少并且三维卷积参数量过大,因此将剪切后的图片进行切片后得到的四模态的数据进行合并处理。最终每一张图片的尺寸大小为4×160×160,划分成训练集18 923例,测试集3 219例。
2.2 实验配置
本文实验环境CPU为Intel i5-12400F,GPU为NVIDIA RTX 3060 12 GB,内存16 GB,操作系统 为Windows10,深度学习框架为Pytorch1.8+CUDA11.1,使用自适应估计优化方法(Adam),训练批次设置为100,批次大小为16,学习率为0.000 1。在此环境下进行训练。
2.3 实验结果与分析
为了验证本文改进算法的性能,将FCN8、U-Net、Dense-UNet与改进算法进行对比,结果如表1所示。由表1可以看出,FCN8作为一种基础算法,其提取特征的能力较差,以至于分割结果也较差;U-Net在FCN的基础上引入了跳跃连接,融合浅层和深层的特征,能够提取更多的信息,因此其分割结果略有提升;Dense-UNet将传统的跳跃连接改成了密集跳跃连接,可以更好地融合浅层特征与深层特征,无论是Dice系数还是Hausdorff距离都是比传统的算法更优。本文密集连接用于编码环节,对特征进行重用,相较于Dense-UNet,本文算法存在不同程度的优化,特别是在ET的分割上,本文的Dice系数提升了0.04,可见本文算法具有很好的分割效果。

表1 四种模型的肿瘤分割结果
为了更加清晰地表达,图4、图5为不同算法的预测结果相较于基准(Ground Truth,GT)的对比。对于图4这种简单的分割,每个算法都可以大致地分割出病灶区域,只是在细节上略有差别,主要表现为红色坏疽区域分割不完全以及绿色浮肿区域多分割,但本文算法在三个区域的分割上都更加精细。针对图5这种难度较大的图像,各种算法的分割结果的差距就十分巨大。例如FCN8算法,针对红色坏疽区域可以说几乎错分割,连在图4分割效果很好的Dense-UNet算法以及改进的算法也表现得较差。但本文算法分割结果相对于其他模型虽然分割得不够完全,但是几乎不存在错分割的情况。相比之下,本文算法的分割结果与GT图像更加 接近。
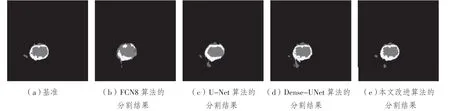
图4 不同算法对MRI图像(简单)的分割结果对比
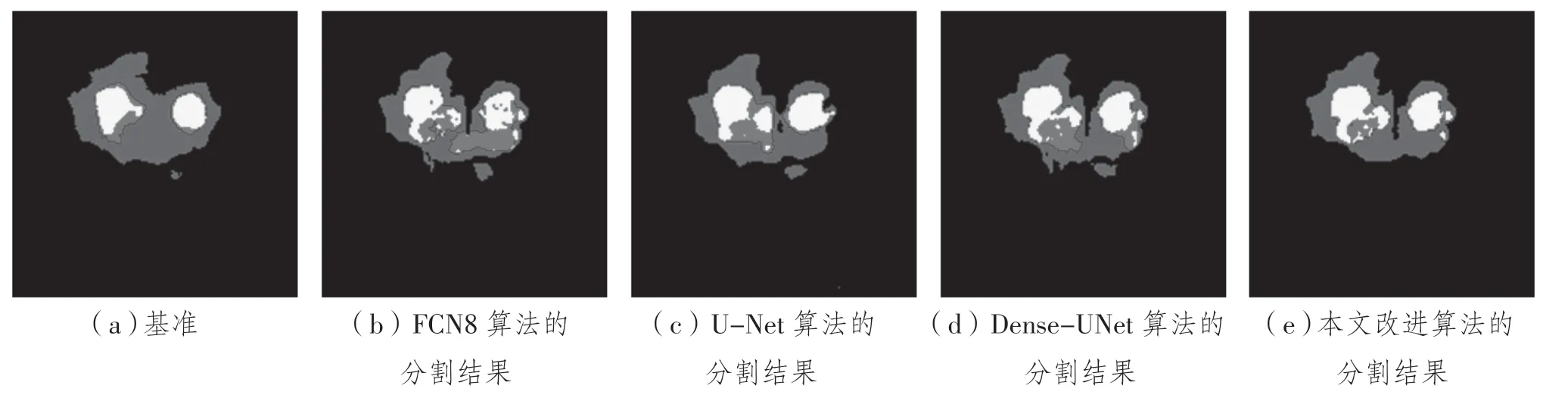
图5 不同算法对MRI图像(难度较大)的分割结果对比
3 结 语
本文针对上下层联系匮乏、特征欠提取等问题,提出了一种改进的脑肿瘤MRI图像分割算法,主要在原始算法中引入密集连接机制、多尺度卷积以及空洞卷积来增强上下文的联系并且提高算法提取特征的能力。在BraTs2019数据集上进行验证,本算法在肿瘤整体区域、核心区域、增强区域的Dice分割指数达到0.853 2,0.872 1,0.796 0,Hausdorff距离分别为2.608 9,1.585 2,2.741 6。相较于原始网络,本文模型在Dice系数上提高了0.03,0.02,0.06,在Hausdorff距离上缩小0.1,0.2,0.2。实验结果表明,本算法所使用的模型可以较好地提取特征,有效避免参数丢失,实现较高的分割精度。

