茶树R2R3-MYB转录因子CsTT2表达分析及功能初步鉴定
王玉源,刘任坚,刘少群,舒灿伟,孙彬妹,郑鹏*
茶树R2R3-MYB转录因子CsTT2表达分析及功能初步鉴定
王玉源1,刘任坚1,刘少群1,舒灿伟2,孙彬妹1,郑鹏1*
1. 华南农业大学园艺学院,广东 广州 510642;2. 华南农业大学植物保护学院/广东省微生物信号与作物病害防控重点实验室/群体微生物研究中心,广东 广州 510642
儿茶素是茶树中特色的次生代谢产物之一,是影响茶叶的品质与风味的主要组分,具有抗氧化、抗病毒、降脂减肥等药理功效。通过系统发育进化树分析、基因表达模式分析和分子生物学试验对茶树儿茶素生物合成相关调控因子CsTT2的功能进行初步鉴定。结果显示,CsTT2是R2R3-MYB转录因子,与拟南芥中调控次生代谢产物的MYB转录因子同在一个分支。在茶树品种顶芽组织中总儿茶素含量较高,和儿茶素生物合成相关基因的表达水平也较高。亚细胞定位、酵母试验和双荧光素酶报告系统试验结果表明,定位在细胞核中,其编码的蛋白是具有转录激活能力的调控因子,可以结合儿茶素生物合成关键基因的启动子激活其表达。
茶树;R2R3-MYB转录因子;;儿茶素生物合成
茶树[(L.) O. Kuntze]属于山茶属(Theaceae)灌木或小乔木,主要包括中国种(var)和阿萨姆种(var)两个栽培品种[1],是中国、日本、印度和肯尼亚等地区重要的经济作物[2]。儿茶素属于类黄酮物质,是茶树中重要的次生代谢产物,在茶树的各个器官中均有分布。儿茶素含量占茶叶干重的12%~24%,占多酚类总量的70%~80%[3]。儿茶素作为茶树中重要的次生代谢产物之一,不仅能够影响茶叶的品质风味,还具有抗癌[4]、抗衰老[5-6]、抗氧化[7]、抗病毒[8-9]、保护神经[10]、调节免疫力[11]及降脂减肥[12]等药理功能。
在植物中,许多次生代谢产物的生物合成途径是保守的。研究表明,茶树中类黄酮生物合成途径可以参照大多数模式植物,特别是拟南芥中花青素的生物合成途径[13]。与植物类黄酮生物合成途径比较,儿茶素类化合物种类较多,其生物合成途径涉及多条代谢途径,是相对复杂、需要受多种因素协调调控的过程。前人研究发现,茶树儿茶素生物合成通路主要包括莽草酸途径、苯丙烷途径、非酯型儿茶素合成和酯型儿茶素合成等4个环节[13-17]。目前,茶树儿茶素生物合成过程中主要的催化酶基因已经被克隆,包括苯丙氨酸解氨酶、肉桂酸羟化酶、4-香豆酸CoA连接酶、查尔酮合成酶、查尔酮异构酶、黄烷酮3-羟化酶、3',5'-类黄酮羟化酶、二氢黄酮醇还原酶、无色花色素还原酶、花青素合酶、花青素还原酶等[18]。茶树中的儿茶素又分为非酯型儿茶素和酯型儿茶素。非酯型儿茶素生物合成的主要途径已经清晰[19-21],而酯型儿茶素的生物合成通路尚未明确。有研究者推测,酯型儿茶素是由非酯型儿茶素没食子酯化得到,但这一猜测并未被试验证实[13]。Niemetz和Gross认为1--没食子酰--葡萄糖是单宁合成的有效酰基供体和受体[22-23]。Liu等[24]在此基础上进一步证实,表儿茶素(EC)和表没食子儿茶素(EGC)在没食子酰基-1----葡萄糖转移酶(UGGT)和1--没食子酰基---没食子酰基转移酶(ECGT)的催化下合成酯型儿茶素。与此同时,酯型儿茶素也会在水解酶的作用下水解得到非酯型儿茶素EC和EGC[25]。也有研究发现,基因家族能够参与非酯型儿茶素转化为酯型儿茶素的过程[26]。近年来,有关茶树儿茶素生物合成的调控已有相关报道。Luo等[27]研究发现,CsWRKY31和CsWRKY48能够结合甲基化儿茶素生物合成相关基因、和的启动子,抑制它们的表达。Wang等[28]研究发现,在烟草中过表达,转基因植株中儿茶素单体的含量上升,且烟草中相关合成基因和的表达上调。综上所述,茶树儿茶素的生物合成途径已经取得了许多研究进展,然而其生物合成的分子调控机制还需进一步探究。
MYB是植物中最大的转录因子家族,它在植物的生长发育、植物响应生物和非生物胁迫以及调控植物次生代谢产物生物合成方面起着重要作用[29]。它通常具有两个不同的区域,一个是位于N端的高度保守的DNA结合域,另一个是位于C端的负责调节蛋白质活性的调节区域。根据结构域所含R结构的数量,植物中的MYB转录因子可分为4类,分别是1R-MYB/MYB-related(1个R结构)、R2R3-MYB(2个R结构)、R1R2R3-MYB蛋白(3个R结构)和4R-MYB(4个R结构)[29]。在植物中,大多数MYB属于R2R3-MYB类转录因子[30],通常作为转录激活子或者抑制子直接或间接调控类黄酮生物合成中相关结构基因的表达,从而影响物质的积累[31-33]。TT2类转录因子是R2R3-MYB蛋白,通过激活相关合成酶基因的表达调控次生代谢产物的生物合成[34-37]。目前,在许多植物中,TT2类转录因子已被克隆和鉴定,包括拟南芥[38]、葡萄[39]、草莓和[33]、苹果和[40]、杨树[41]等,这些基因都参与调控植物中次生代谢产物的生物合成。
本研究通过系统发育进化树分析、基因表达模式分析和分子生物学试验对茶树儿茶素生物合成相关调控因子CsTT2的功能进行初步鉴定,为研究茶树儿茶素生物合成的分子调控机理奠定理论基础。
1 材料与方法
1.1 试验材料
试验材料为岭头单丛(LTDC)、红柄单丛(HBDC)和龙井长叶(LJCY)茶树品种的多年生灌木茶苗,种植于华南农业大学茶树种质资源圃。于2020年,分别采摘3个茶树品种一芽二叶顶芽组织,用液氮速冻后置于–80℃冰箱贮存,用于提取RNA和测定儿茶素含量;采摘LTDC品种的一芽二叶顶芽、嫩叶、成熟叶、老叶、根、茎、花和果实组织,用液氮速冻后置于–80℃冰箱贮存,用于提取RNA;以上每个样本设置3个生物学重复。此外,试验用到的本氏烟草(),种子来自本实验室。
1.2 同源比对分析及系统发育进化树构建
将拟南芥AtTT2(AT5G35550)蛋白序列比对到茶树基因组数据库(http://tpdb.shengxin.ren/index.html)中,找到茶树同源基因(),并下载其蛋白序列。将CsTT2的蛋白序列放入到NCBI数据库中进行比对,下载其他物种中同源蛋白序列,并在DNAMAN软件中进行蛋白序列比对分析。从拟南芥基因组数据库(https://www.arabidopsis.org)下载拟南芥所有MYB蛋白序列,用clustalW软件对CsTT2蛋白序列和拟南芥所有MYB蛋白序列进行完全比对分析。根据已有报道,使用Jalview软件截取R2R3-MYB蛋白的保守结构域区域[42],再使用MEGA-X软件中的邻接法进行系统发育进化树的构建。
1.3 儿茶素含量测定
儿茶素(C)、EC、没食子儿茶素(GC)、EGC、表儿茶素没食子酸酯(ECG)和表没食子儿茶素没食子酸酯(EGCG)标准样品购自上海源叶生物科技有限公司。用70%甲醇溶液将标准样品分别配制成质量浓度为0.01、0.1、0.2、0.5 mg·mL-1的标准液,用于儿茶素单体标准曲线的构建。
使用冷冻干燥机将样品冻干,用研钵将样品磨成粉末状,准确称取0.1 g粉末加入到含有5 mL 70%甲醇的10 mL离心管中,浸提5 min后(期间上下振荡),70℃水浴超声振荡30 min,在12 000 r·min-1下离心5 min,用1 mL注射器吸取上清液,通过0.45 μm微孔过滤器过滤到棕色样品瓶中,最后用Waters Alliance 2489高效液相色谱仪(Waters Technologies,美国)测定样品中儿茶素的含量。上机条件:柱温,(30±5)℃;流速,1 mg·mL-1;波长,280 nm;进样量,10 μL;上机程序如表1所示。
1.4 实时荧光定量PCR(qRT-PCR)
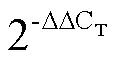

表1 梯度程序
1.5 基因表达模式分析
从茶树基因组数据库(http://tpdb.shengxin.ren/index.html)中下载茶树顶芽、嫩叶、成熟叶、老叶、根、茎、花和果实等8个不同组织部位的转录组数据,用TBtools软件进行相关基因表达量热图的构建[45],分析基因表达模式。
1.6 载体构建
根据的CDS序列以及载体酶切位点,使用CE Design V1软件设计同源重组引物,引物见表3,下划线部分为载体的酶切位点。以白叶单丛的cDNA为模板,使用PrimeSTAR酶进行PCR扩增。PCR产物经过琼脂糖凝胶电泳后回收目的条带,连接到对应的线性化载体上,转化至DH5后进行鉴定并测序,将阳性克隆提取质粒,用于转化农杆菌和酵母菌。
1.7 亚细胞定位试验
将的CDS序列(去除终止密码子)构建到pEAQ-EGFP载体上,以空白载体为对照,使用热激法转化农杆菌感受态GV3101。用侵染液(200 μmol·L-1AS,10 mmol·L-1MgCl2,10 mmol·L-1MES,PH为5.6,现配现用)将活化后的菌体重悬至OD值为0.4~0.6,活化2 h后,将空载体(35S::CsTT2::EGFP)与细胞核marker(35S::NLS::Dsred)按体积比1∶1混合,使用无针头注射器将菌液从叶片下表皮注射到本氏烟草中,黑暗保湿培养48 h后,用生物显微镜BX53(OLYMPUS,日本)进行观察和拍照。
1.8 酵母自激活试验
将全长CDS序列以及根据保守结构域短截后的序列构建到pGBKT7(BD)载体上,以空白载体为对照,将重组产物转化到酵母感受态Y2HGold。用100 μL ddH2O重悬阳性单克隆菌,分别在SD/-Trp和SD/-Trp-His-Ade培养基上点5 μL菌液,30℃倒置培养3 d后,用X--gal对菌斑进行染色,然后观察和拍照。
1.9 转录激活试验
将全长CDS序列构建到pEAQ-PBD载体上,以空白载体为阴性对照,pEAQ-PBD-VP16为阳性对照,将重组产物转化到农杆菌感受态GV3101。用侵染液(200 μmol·L-1AS,10 mmol·L-1MgCl2,10 mmol·L-1MES,PH为5.6,现配现用)将活化后的菌体重悬至OD值为0.2,活化2 h后,将阴性对照/阳性对照/pEAQ-PBD-CsTT2与报告载体(GAL-LUC)按体积比1∶1混合,使用无针头注射器将菌液从叶片下表皮注射到本氏烟草中,黑暗保湿培养24 h,再正常培养48 h后,使用Promega公司的双荧光素酶报告系统试剂盒检测萤火虫荧光素酶(Firefly luciferase,LUC)和海肾荧光素酶(Renilla luciferase,REN)的活性。

表2 定量引物序列
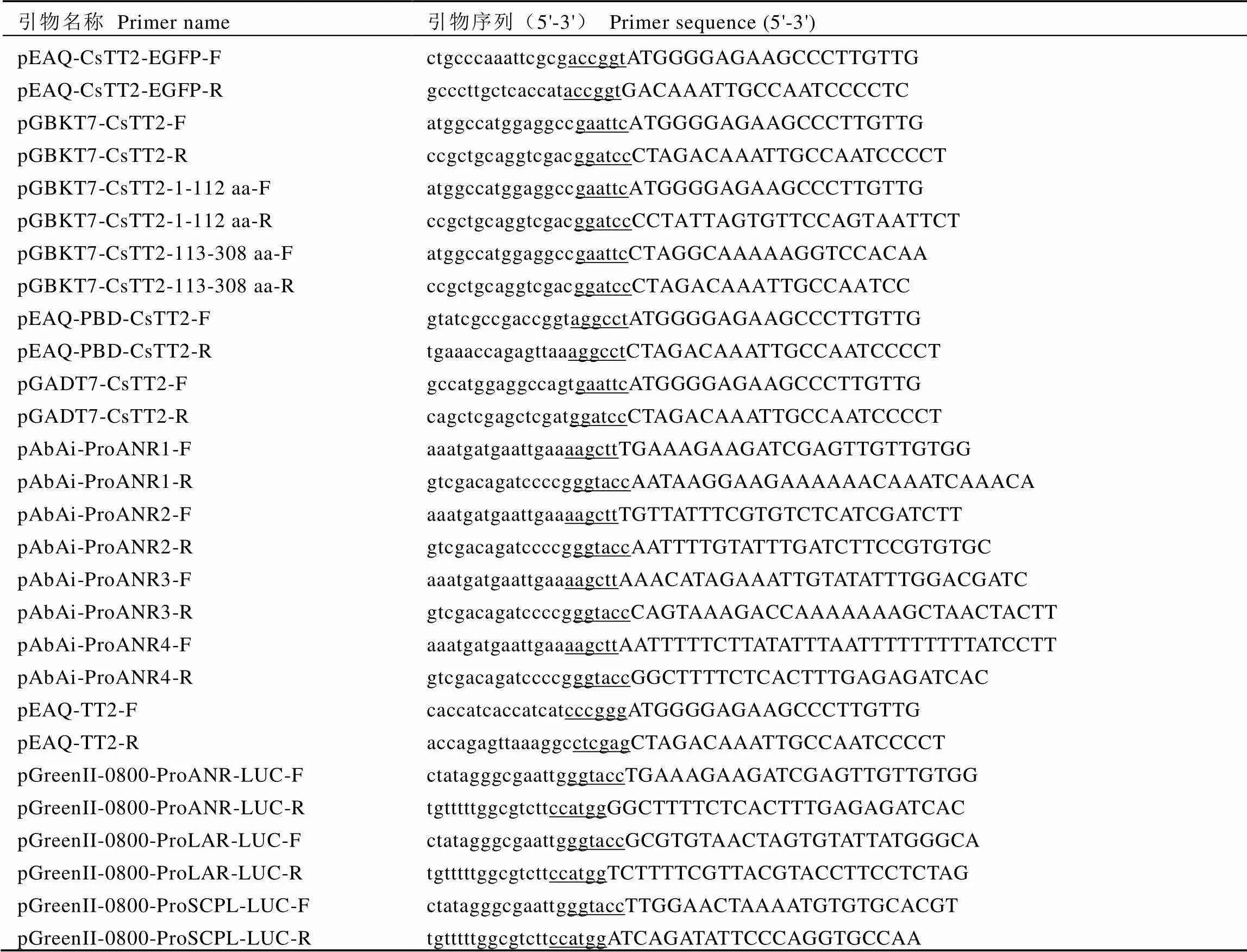
表3 载体引物序列
1.10 酵母单杂交试验
使用在线数据库JASPAR(https://jaspar.genereg.net)对儿茶素合成基因启动子中可能存在的MYB结合元件进行预测和统计。
根据预测结果,将儿茶素合成基因启动子短截为4个片段,分别构建到pAbAi载体上,并整合到Y1HGold酵母菌中,形成诱饵特异性报告菌株,筛选每个启动子的自激活抑制浓度(其中,ProLAR和ProSCPL的自激活活性在AbA质量浓度为1 000 ng·mL-1下仍无法抑制,因此没进行后续试验)。将基因的CDS全长序列构建到pGADT7(AD)载体上,再转化到诱饵特异性报告菌株中。用ddH2O重悬阳性单克隆菌,将浓度OD600调为0.2、0.02、0.002,分别在SD/-Leu和SD/-Leu+AbA培养基上点5 μL菌液,30℃倒置培养3 d后,进行观察和拍照。
1.11 双荧光素酶试验
将全长CDS序列构建到pEAQ载体上,将儿茶素合成基因启动子(、、)构建到pGreenII-0800-LUC载体上,以空白载体pEAQ作为对照,将重组产物转化到农杆菌感受态GV3101。用侵染液(200 μmol·L-1AS,10 mmol·L-1MgCl2,10 mmol·L-1MES,pH为5.6,现配现用)将活化后的菌体重悬至OD值为0.2,活化2 h后,将pEAQ/pEAQ-CsTT2+pGreenII-0800-ProANR-LUC/pGreenII-0800-ProLAR-LUC/pGreenII-0800-ProSCPL-LUC分别按体积比20∶1混合,使用无针头注射器将菌液从叶片下表皮注射到本氏烟草中,黑暗保湿培养24 h,再正常培养48 h后,使用Promega公司的双荧光素酶报告系统试剂盒检测Firefly和REN值。
1.12 数据统计与分析
数据方差分析使用IBM SPSS Statistics 22软件完成,图片处理和组合使用Adobe Illustrator 2020软件完成。
2 结果与分析
2.1 CsTT2保守结构域及系统发育进化树分析
多序列比对结果显示,CsTT2具有两个串联不完整重复序列R2和R3,表明CsTT2是R2R3-MYB转录因子(图1)。在拟南芥中,根据DNA结合域和C端氨基酸基序的保守性,对MYB转录因子家族进行了分组;其中,R2R3-MYB蛋白被分成23个亚组[46]。本研究对CsTT2和拟南芥中所有MYB蛋白进行了系统发育进化树分析,将蛋白分为6个主要亚组,Group Ⅵ又进一步分为Group Ⅵa、Group Ⅵb、Group Ⅵc、Group Ⅵd和Group Ⅵe。结果显示,CsTT2和拟南芥S2、S3、S5、S6、S7和S15中的R2R3-MYB蛋白聚集在同一亚组(Group Ⅳ)中(图2)。S7中的成员可以调控黄酮醇的合成[47],S6中的成员可以调控营养生殖过程中花青素的合成[48],S5中的成员可以调控拟南芥种皮原花青素的合成[49],而S3中的成员可以激活导管中的木质素生物合成[50-51]。因此,CsTT2可能参与调控茶树类黄酮次生代谢物生物合成的过程。
2.2 茶树顶芽儿茶素含量与儿茶素生物合成相关基因表达水平分析
儿茶素主要分布在茶树的顶芽部位。本研究使用高效液相色谱法测定LTDC、HBDC和LJCY 3个茶树品种顶芽部位中6种常见儿茶素的含量(图3-A)。如图3-B所示,在3个茶树品种中,GC的含量没有显著差异,而HBDC中EGC和C的含量显著高于其他两个品种,EC的含量显著高于LJCY。此外,LTDC和HBDC中EGCG的含量显著高于LJCY,而ECG的含量则相反。

注:葡萄:VvTT2(RVW44110.1),木豆:CcMYB6(XP_020227123.1),杨梅:MrTT2(KAB1209459.1),鸡血藤:SsTT2(TKY54429.1),大豆:GmMYB23(XP_003541301.1),拟南芥AtTT2(AT5G35550)
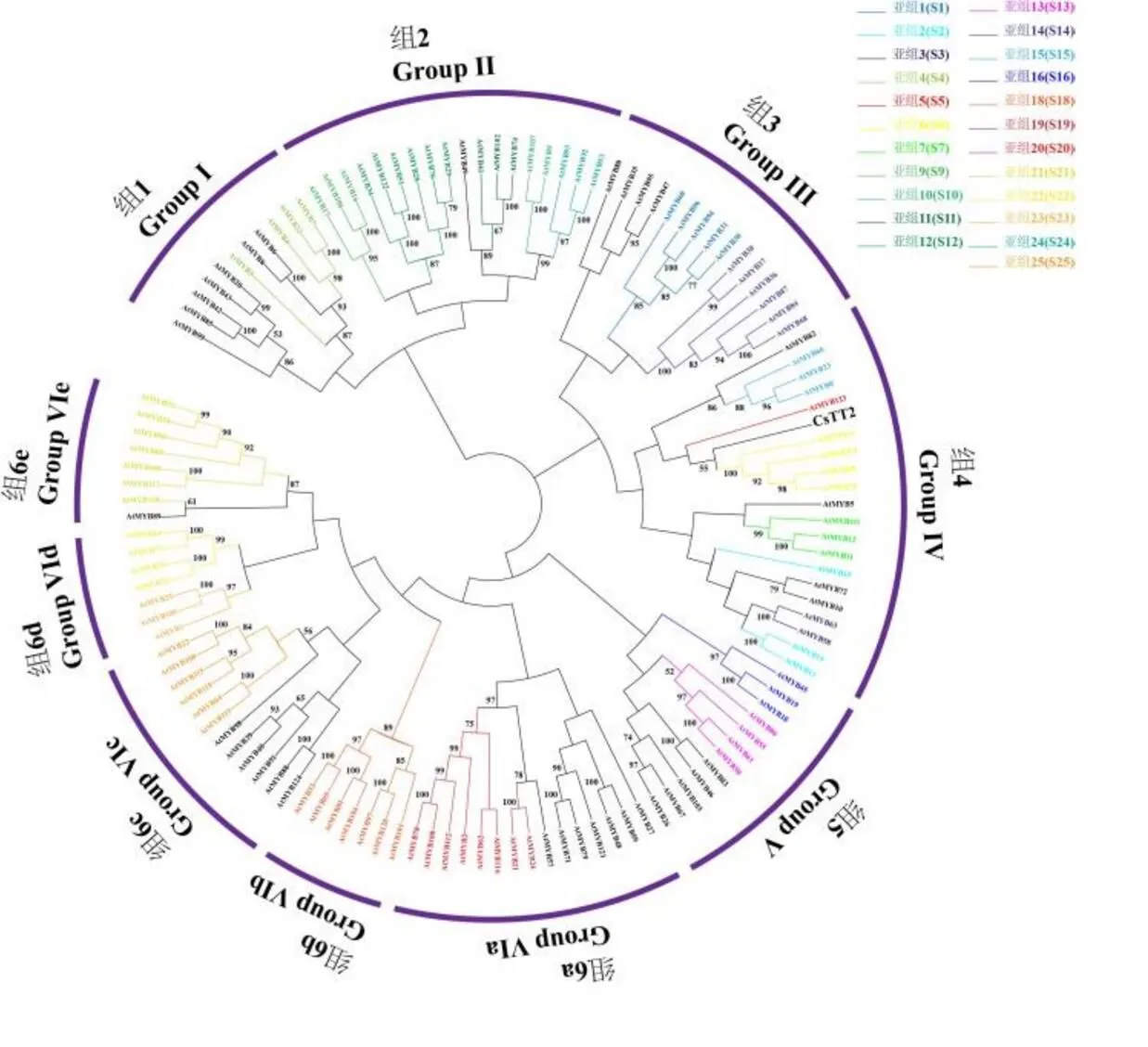
注:拟南芥中的R2R3-MYB蛋白被分为23个亚组[46],每个亚组对应分支的颜色如图右上方所示,而黑色分支为除R2R3-MYB外的其他MYB成员。本研究构建的系统发育进化树主要分为6个亚组,对应分组情况如图紫色弧线所示
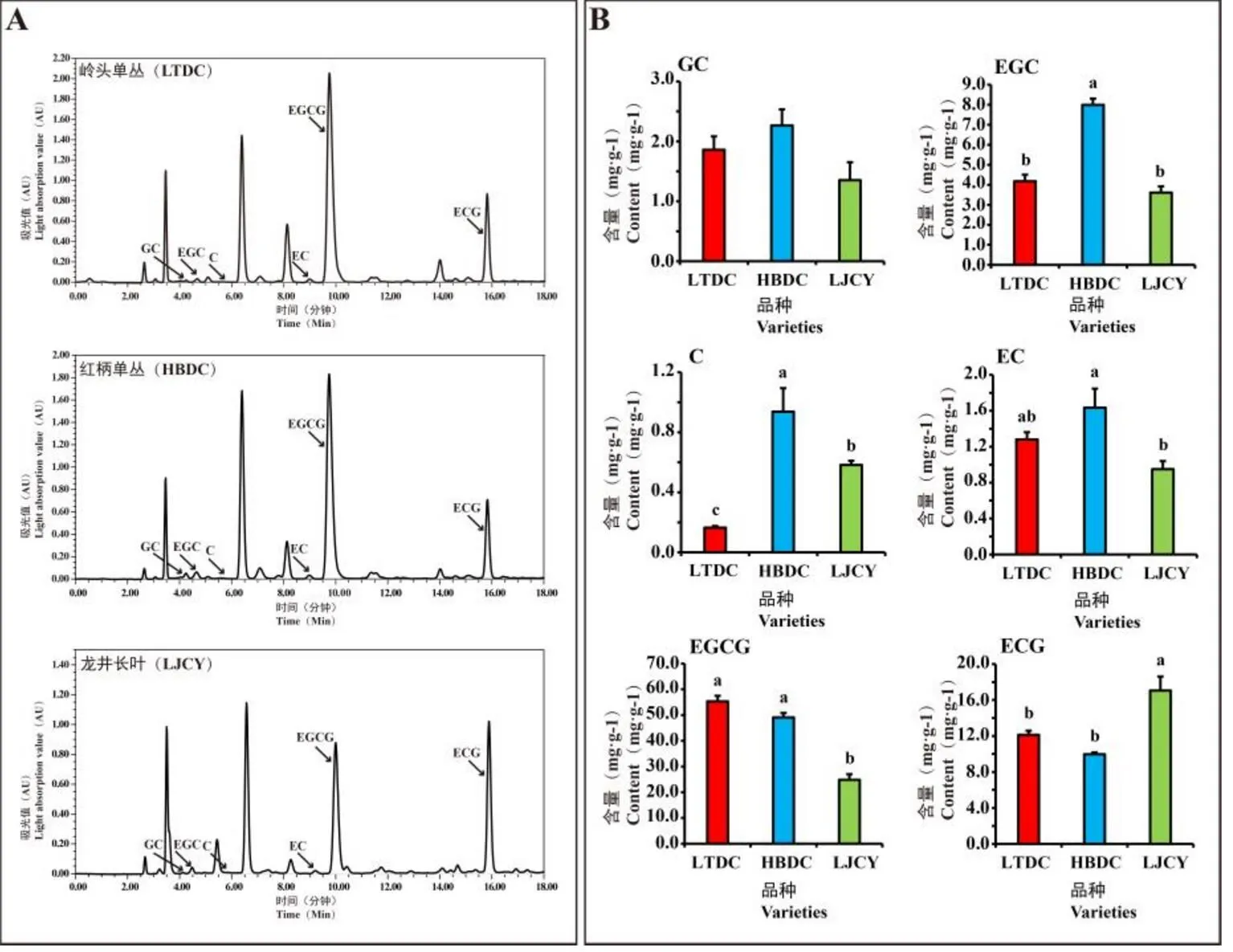
注:A. 液相色谱峰图。B. 3个茶树品种中6种儿茶素单体的含量。数据是用平均值±标准误来表示,图中不同的字母表示显著不同的值(P<0.05, Tukey’s test)
Fig.3 Contents of catechincomponents in apical bud of different tea cultivars
进一步对3个茶树品种顶芽的总儿茶素含量以及儿茶素生物合成相关基因的表达量进行分析。结果显示,LTDC和HBDC中总儿茶素含量显著高于LJCY(图4-A)。此外,在LTDC和HBDC中,除了()外,目的基因和儿茶素生物合成相关基因()、()、()、()和()的表达量均显著高于LJCY(图4-B和图4-C)。
2.3 CsTT2转录活性分析
为了进一步分析CsTT2的转录活性,本研究还进行了亚细胞定位、双荧光素酶报告系统试验和酵母自激活试验。结果显示,转入pEAQ-EGFP空载体的本氏烟草叶片中的细胞膜和细胞核均激发出绿色荧光,转入pEAQ-CsTT2-EGFP的本氏烟草叶片中仅细胞核激发出绿色荧光(图5-A),这表明定位在细胞核中。双荧光素酶报告系统结果显示,相对于pEAQ-PBD阴性对照,CsTT2可以显著提高Firefly LUC的活性(图5-B),表明CsTT2在植物中具有转录激活功能。为了进一步研究CsTT2的激活区域,根据保守结构域的位置将CsTT2序列短截后进行酵母自激活试验。结果显示,CsTT2蛋白的激活区域主要位于C端的113—308氨基酸(图5-C)。上述结果表明,定位在细胞核中,是具有转录激活能力的调控基因,且转录激活区域主要位于C端的113—308氨基酸处。
2.4 CsTT2结合调控儿茶素合成基因ANR启动子活性的分析
对和儿茶素生物合成相关基因(、、、、和)在茶树8个不同组织部位中的转录组数据进行分析。结果显示,()、()和()在茶树8个不同组织部位中的表达模式和一致,主要在茶树的幼嫩芽叶部位中表达,且表达量在嫩叶组织中最高(图6-A)。qRT-PCR结果进一步证明,、()、()和()在LTDC 8个不同组织部位中的表达模式一致,均在顶芽和嫩叶中高表达(图6-B)。
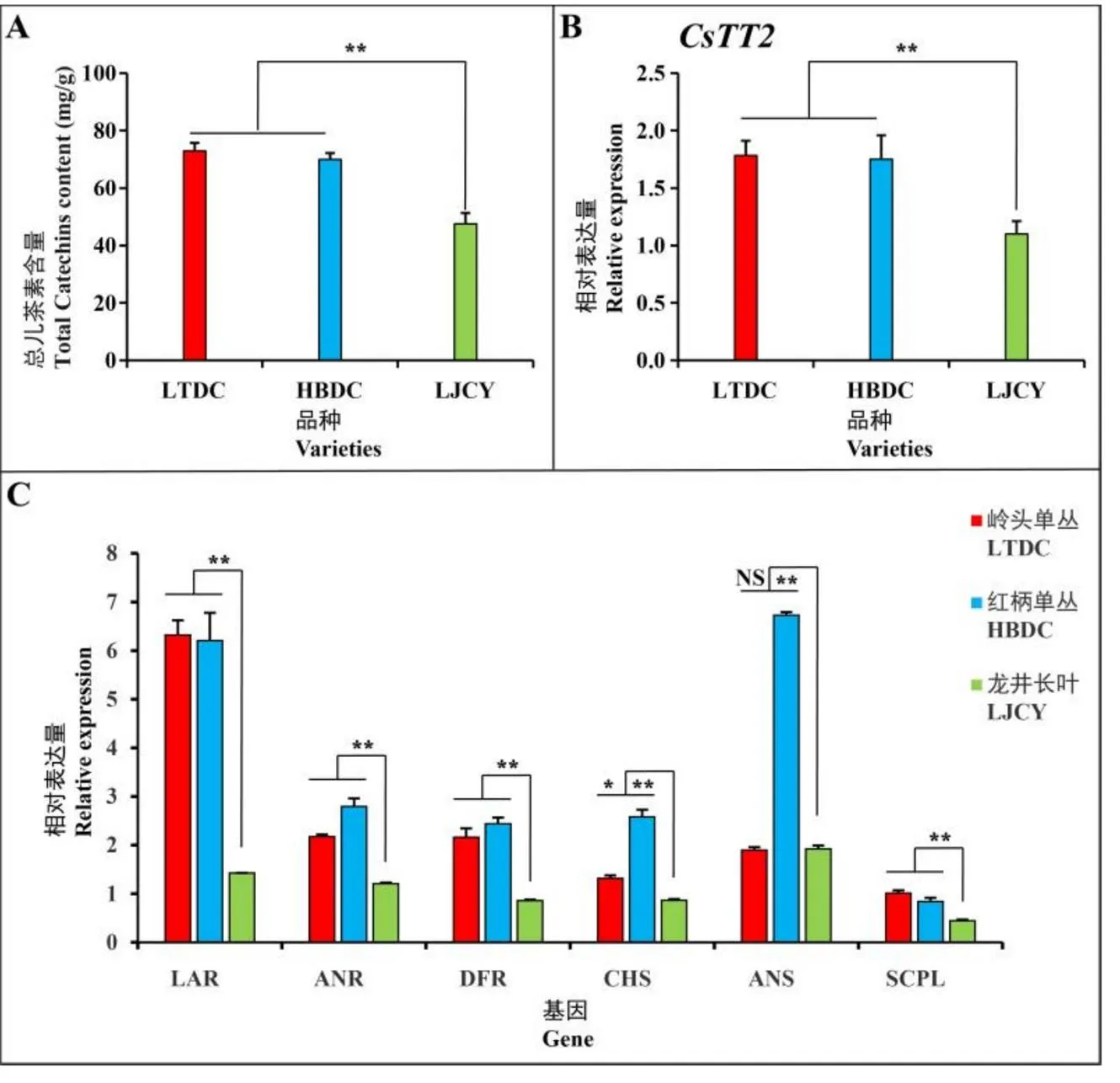
注:A. 3个茶树品种顶芽中总儿茶素的含量。B. CsTT2在3个茶树品种顶芽中的表达量。C. 儿茶素生物合成相关基因(LAR、ANR、DFR、CHS、ANS和SCPL)在3个茶树品种顶芽中的表达量。数据是用平均值±标准误来表示,采用Student’s test与对照“LJCY”进行显著性分析(**,P<0.01)
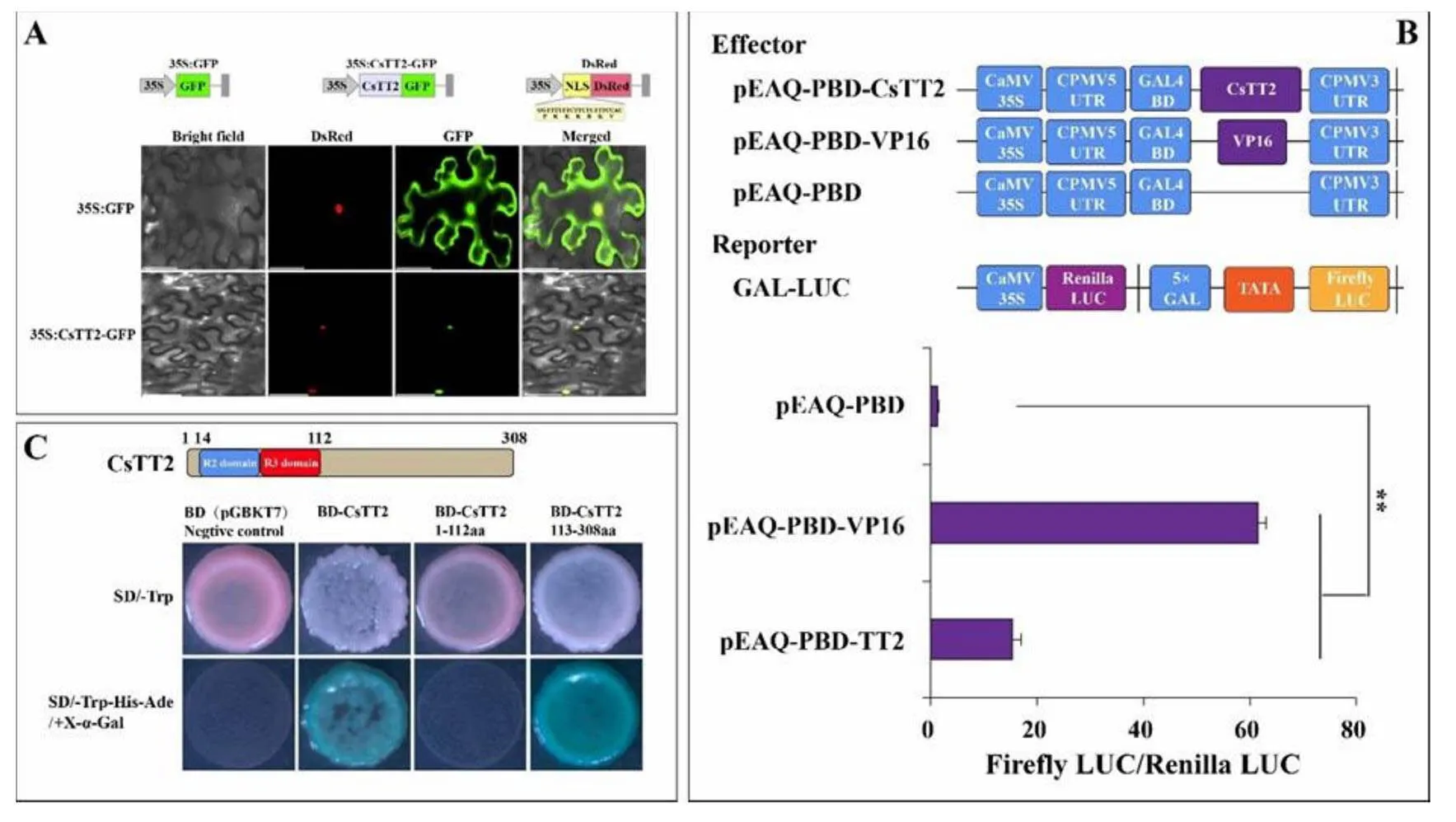
注:A. CsTT2的亚细胞定位分析。比例尺=50 μm。Bright field:明场通道;DsRed channel:红色荧光通道;GFP channel:绿色荧光通道;Merged channel:融合通道。B. CsTT2在烟草中的转录活性分析。pEAQ-PBD和pEAQ-PBD-VP16为阴性和阳性对照。数据是用平均值±标准误来表示,采用Student’s test与阴性对照进行显著性分析(**,P<0.01)。C. CsTT2在酵母中的转录活性分析。BD载体为阴性对照。图上方为CsTT2蛋白结构图。SD/-Trp代表SD培养基缺少色氨酸;SD/-Trp-His-Ade/+X-α-Gal代表SD培养基缺少色氨酸、组氨酸、腺嘌呤且添加X-α-Gal
Fig.5 Subcellular localization and transcription activity analysis of CsTT2
进一步通过酵母单杂交试验验证CsTT2与启动子的结合能力。如图7-A所示,克隆出来的启动子区域(–1 bp至–1 423 bp)可能存在16个MYB-bingding-element。根据预测结果,将其启动子短截成4段约为350 bp的短片段,进行后续试验。与AD+pAbAi-ProANR2酵母菌相比,转化AD-CsTT2+pAbAi-ProANR2的菌株能在SD/Leu和SD/Leu+AbA的平板上生长,这说明CsTT2能够与的启动子相结合(图7-B)。此外,双荧光素酶报告系统试验结果显示,在植物体内,CsTT2能够激活启动子,进而驱动下游萤火虫荧光素酶报告基因的表达,增强Firefly LUC的活性;而它不能激活和的启动子(图7-C)。上述结果表明,CsTT2蛋白能够结合儿茶素生物合成相关基因的启动子,进而激活的表达。
3 讨论
茶树儿茶素不仅能决定茶叶的品质和风味,还具有抗氧化、抗癌和降脂减肥等药理功效。目前,茶树儿茶素生物合成途径的研究已取得了重要的进展,然而其分子调控机制尚未清晰。许多研究表明,R2R3-MYB转录因子在植物次生代谢产物的生物合成过程中起着重要作用[52-54]。本研究通过系统发育进化树分析、基因表达模式和分子生物学试验对茶树儿茶素生物合成相关调控因子CsTT2的功能进行初步鉴定。
在系统发育进化树中,同一分支的基因往往具有相似的功能。本研究通过系统发育进化树分析发现,CsTT2与拟南芥中S2、S3、S5、S6、S7和S15中的R2R3-MYB蛋白聚集在同一亚组中(图2)。S3、S5、S6和S7中的成员均参与调控拟南芥类黄酮次生代谢物的生物合成[47-51]。此外,S2中的成员AtMYB15能够控制以及其他基因的表达去响应低温胁迫[55],S15中的成员参与调控拟南芥毛状体以及根毛的形成[56]。这表明CsTT2不仅可能参与调控茶树类黄酮次生代谢生物合成,还可能响应低温胁迫以及参与调控茶树毛状体和根毛的形成。基因表达模式分析有利于挖掘该基因的潜在功能。本研究通过高效液相色谱法和qRT-PCR技术测定茶树品种顶芽的儿茶素含量以及儿茶素生物合成相关基因的表达量。结果显示,在儿茶素含量高的品种中,目的基因和儿茶素生物合成相关基因(、、、和)的表达量均上调(图4)。
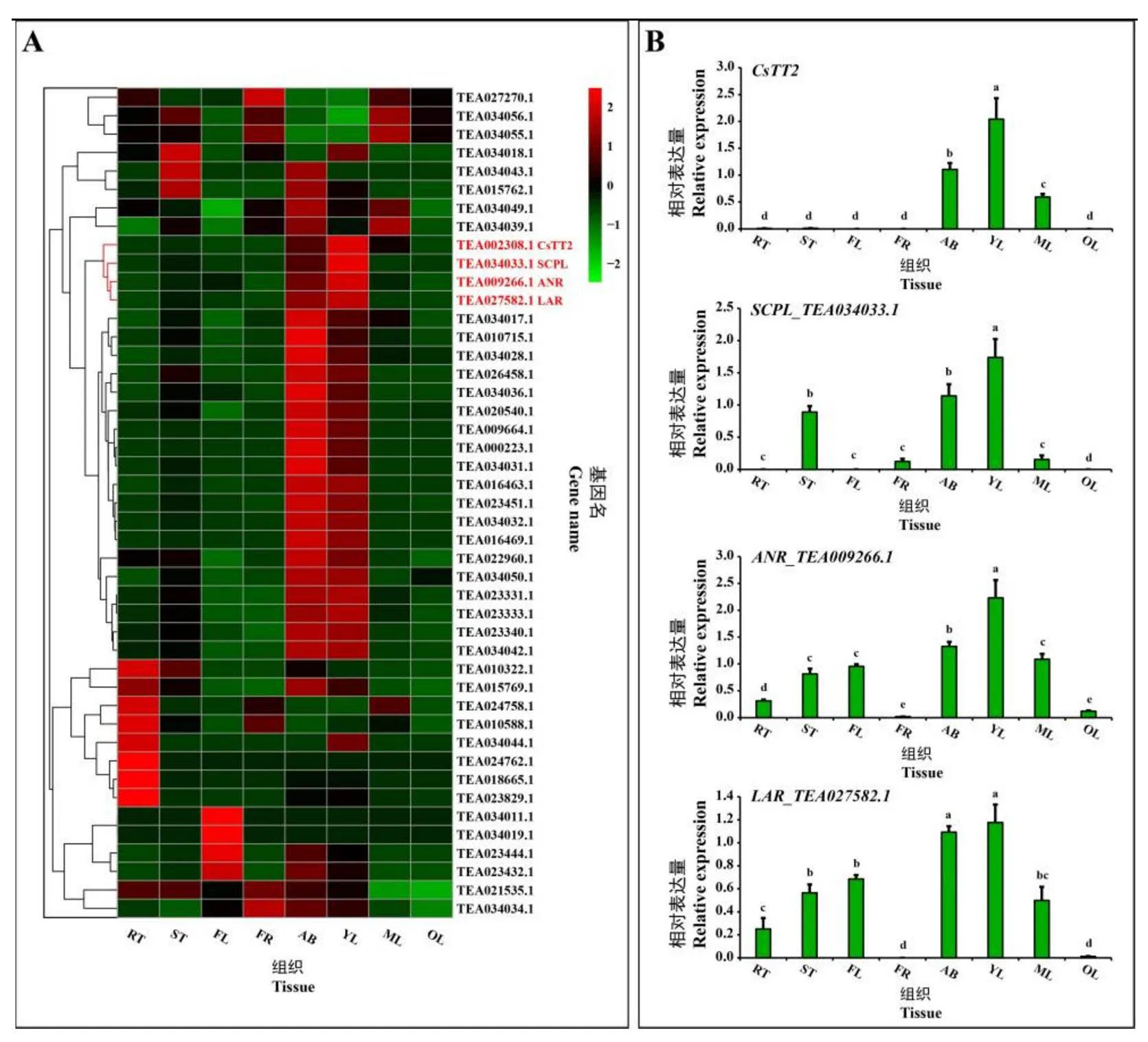
注:A. CsTT2和儿茶素生物合成相关基因在茶树8个不同组织部位的转录组数据分析。热图右侧显示每个基因的ID号。B. CsTT2和儿茶素生物合成相关基因在LTDC的8个不同组织部位的qRT-PCR分析。数据是用平均值±标准误来表示,图中不同的字母表示显著不同的值(P<0.05,Tukey’s test)。RT:根,ST:茎,FL:花,FR:果实,AB:顶芽,YL:嫩叶,ML:成熟叶,OL:老叶
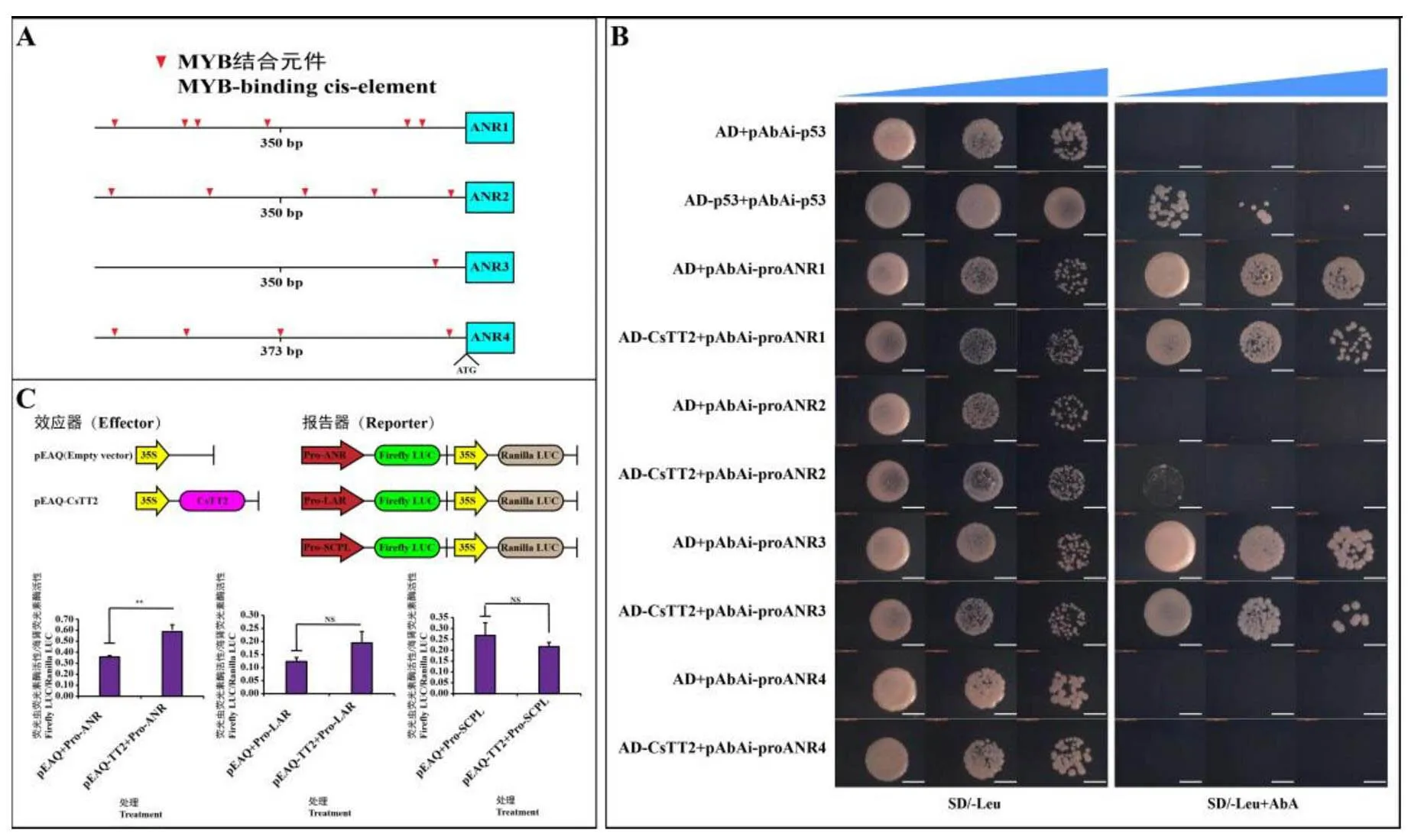
注:A. 儿茶素生物合成相关基因ANR启动子MYB转录因子结合元件的预测。红色的三角符号表示MYB转录因子特异结合元件。B. CsTT2结合儿茶素生物合成相关基因ANR启动子的鉴定。ProANR1:–1 074 bp至–1 423 bp,ProANR2:–724 bp至–1 073 bp,ProANR3:–374 bp至–723 bp,ProANR4:–1 bp至–373 bp。SD/-Leu表示SD培养基缺乏亮氨酸,SD/-Leu+AbA表示SD/-Leu添加AbA,AbA使用浓度:ProANR1和ProANR3: 1 000 ng·mL-1,ProANR2:850 ng·mL-1,ProANR4:900 ng·mL-1;蓝色三角符号代表菌液稀释的倍数。C. CsTT2调控儿茶素生物合成相关基因(ANR、LAR和SCPL)启动子活性的分析。数据是用平均值±标准误来表示,采用Student’s test与对照(pEAQ+ProANR/ProLAR/ProSCPL)进行显著性分析(**,P<0.01)
在茶树中,基因具有多个拷贝。其中,在烟草中过表达,转基因植株的花青素含量得到积累,而MYB5b能够调控儿茶素单体C和EC的合成。在烟草中过表达(),烟草中类黄酮物质生物合成相关基因(、、和)的表达量上调,但是转基因株系中的花青素含量并没有明显变化[28,57-58],它如何影响相关基因的表达以及是否参与调控茶树中儿茶素生物合成还有待进一步探究。因此,为了进一步研究CsTT2的功能,本研究进行了亚细胞定位、酵母自激活、酵母单杂交以及双荧光素酶报告系统试验。结果表明,定位在细胞核中,是具有转录激活能力的调控因子(图5)。进一步试验分析发现,CsTT2可以结合儿茶素生物合成重要基因的启动子,从而激活它的表达(图7)。CsTT2有可能是通过直接激活基因的表达,进而调控茶树儿茶素的生物合成。若后续能在茶树本体上直接进行基因功能验证,则更能进一步鉴定CsTT2的调控功能。
本研究通过基本的生物信息学分析和一系列分子生物学试验对CsTT2的功能进行初步鉴定。结果表明,CsTT2可以结合茶树儿茶素生物合成重要合成基因的启动子,并激活其表达。本研究为研究茶树儿茶素生物合成的分子调控机制奠定理论基础。
[1] Ming T, Bartholomew B. Theaceae [J]. Flora China, 2007, 12: 366-478.
[2] Zhao M, Zhang N, Gao T, et al. Sesquiterpene glucosylation mediated by glucosyltransferase UGT91Q2 is involved in the modulation of cold stress tolerance in tea plants [J]. New Phytol, 2020, 226(2): 362-372.
[3] 宛晓春. 茶叶生物化学[M]. 3版. 北京: 中国农业出版社, 2003.
Wan X C. Biochemistry of tea: the third edition [M]. 3rd ed. Beijing: China Agriculture press, 2003.
[4] Zhao M, Yu Y, Sun L, et al. GCG inhibits SARS-CoV-2 replication by disrupting the liquid phase condensation of its nucleocapsid protein [J]. Nature Communications, 2021, 12(1): 2114.doi: 10.1038/s41467-021-22297-8.
[5] Xiong L G, Chen Y J, Tong J W, et al. Epigallocatechin-3-gallate promotes healthy lifespan through mitohormesis during early-to-mid adulthood in[J]. Redox Biology, 2018, 14: 305-315.
[6] Yuan H, Li Y, Ling F, et al. The phytochemical epigallocatechin gallate prolongs the lifespan by improving lipid metabolism, reducing inflammation and oxidative stress in high-fat diet-fed obese rats [J]. Aging cell, 2020, 19(9): e13199. doi: 10.1111/acel.13199.
[7] Lwxa B, Shang C, Tsza B, et al. Green tea derivative epigallocatechin-3-gallate (EGCG) confers protection against ionizing radiation-induced intestinal epithelial cell death bothand[J]. Free Radical Biology and Medicine, 2020, 161: 175-186.
[8] Zhang Z, Zhang X, Bi K, et al. Potential protective mechanisms of green tea polyphenol EGCG against COVID-19 [J]. Trends in Food Science & Technology, 2021, 114: 11-24.
[9] Zhao J, Blayney A, Liu X, et al. EGCG binds intrinsically disordered N-terminal domain of p53 and disrupts p53-MDM2 interaction [J]. Nature Communications, 2021, 12(1): 986. doi: 10.1038/s41467-021-21258-5.
[10] Bernier L P, York E M, Kamyabi A, et al. Microglial metabolic flexibility supports immune surveillance of the brain parenchyma [J]. Nat Commun, 2020, 11(1): 1559. doi: 10.1038/s41467-020-15267-z.
[11] Liu Z S, Cai H, Xue W, et al. G3BP1 promotes DNA binding and activation of cGAS [J]. Nat Immunol, 2019, 20(1): 18-28.
[12] Yang C S, Hong J. Prevention of chronic diseases by tea: possible mechanisms and human relevance [J]. Annual Review of Nutrition, 2013, 33: 161-181.
[13] 夏涛, 高丽萍. 类黄酮及茶儿茶素生物合成途径及其调控研究进展[J]. 中国农业科学, 2009, 42(8): 2899-2908.
Xia T, Gao L P. Research progress on biosynthesis pathway and regulation of flavonoids and catechins [J]. Scientia Agricultura Sinica, 2009, 42(8): 2899-2908.
[14] Weisshaar B, Jenkins G I. Phenylpropanoid biosynthesis and its regulation [J]. Current Opinion in Plant Biology, 1998, 1(3): 251-257.
[15] Furukawa T, Eshima A, Koiya M, et al. Coordinate expression of genes involved in catechin biosynthesis incells [J]. Plant Cell Reports, 2002, 21(4): 385-389.
[16] 夏涛, 高丽萍, 刘亚军, 等. 茶树酯型儿茶素生物合成及水解途径研究进展[J]. 中国农业科学, 2013, 46(11): 2307-2320.
Xia T, Gao L P, Liu Y J, et al. Advances in biosynthesis and hydrolysis of catechins from tea tree[J]. Scientia Agricultura Sinica, 2013, 46(11): 2307-2320.
[17] 宛晓春. 茶树次生代谢[M]. 北京: 科学出版社, 2015.
Wan X C. Secondary metabolism of tea plant [M]. Beijing: Science Press, 2015.
[18] 陆建良, 林晨, 骆颖颖, 等. 茶树重要功能基因克隆研究进展[J]. 茶叶科学, 2007, 27(2): 95-103.
Lu J L, Lin C, Luo Y Y, et al. Advances in cloning of important functional genes from[J]. Journal of Tea Science, 2007, 27(2): 95-103.
[19] Stafford H A. Flavonoid metabolism pathway to proanthocyanindins (condensed tannins), flavan-3-ols, and unsubstituted flavans [M]. New York: CRC Press, 1990.
[20] Stafford H A, Lester H H. The conversion of ()-dihydromyricetin to its flavan-3, 4-diol (leucodelphinidin) and to ()-gallocatechin by reductase extracted from tissue cultures ofand[J]. Plant Physiology, 1985, 78(4): 791-794.
[21] Punyasiri P, Abeysinghe I, Kumar V, et al. Flavonoid biosynthesis in the tea plant: properties of enzymes of the prominent epicatechin and catechin pathways [J]. Archives of Biochemistry & Biophysics, 2004, 431(1): 22-30.
[22] Niemetz R, Gross G G. Enzymology of gallotannin and ellagitannin biosynthesis [J]. Phytochemistry, 2005, 66(17): 2001-2011.
[23] Gross G G. From lignins to tannins: forty years of enzyme studies on the biosynthesis of phenolic compounds [J]. Phytochemistry, 2008, 69(18): 3018-3031.
[24] Liu Y, Gao L, Liu L, et al. Purification and characterization of a novel galloyltransferase involved in catechin galloylation in the tea plant () [J]. Journal of Biological Chemistry, 2012, 287(53): 44406-44417.
[25] Zhong K, Zhao S Y, Jönsson L J, et al. Enzymatic conversion of epigallocatechin gallate to epigallocatechin with an inducible hydrolase from[J]. Biocatalysis, 2009, 26(4): 306-312.
[26] Wei C, Hua Y, Wang S, et al. Draft genome sequence ofvar.provides insights into the evolution of the tea genome and tea quality [J]. Proc Natl Acad Sci U S A, 2018, 115(18): E4151-E4158.
[27] Luo Y, Yu S, Li J, et al. Molecular characterization of WRKY transcription factors that act as negative regulators of O-Methylated catechin biosynthesis in tea plants (L.) [J]. J Agric Food Chem, 2018, 66(43): 11234-11243.
[28] Wang P, Zhang L, Jiang X, et al. Evolutionary and functional characterization of leucoanthocyanidin reductases from[J]. Planta, 2018, 247(1): 139-154.
[29] 牛义岭, 姜秀明. 植物转录因子MYB基因家族的研究进展[J]. 分子植物育种, 2016, 14(8): 2050-2059.
Niu Y L, Jiang X M. Research progress of plant transcription factor MYB gene family [J]. Molecular Plant Breeding, 2016, 14(8): 2050-2059.
[30] Martin C, Paz-Ares J. MYB transcription factors in plants [J]. Trends in Genetics, 1997, 13(2): 67-73.
[31] Verdonk J C, Haring M A, Tunen A J, et al. ODORANT1 regulates fragrance biosynthesis in[J]. Plant Cell, 2005, 17(5): 1612-1624.
[32] Bomal C, Bedon F, Caron S, et al. Involvement ofMYB1 and MYB8 in phenylpropanoid metabolism and secondary cell wall biogenesis: a comparative in planta analysis [J]. Journal of Experimental Botany, 2008, 59(14): 3925-3939.
[33] Schaart J G, Dubos C, Romero De La Fuente I, et al. Identification and characterization of MYB-bHLH-WD40 regulatory complexes controlling proanthocyanidin biosynthesis in strawberry [J]. The New Phytologist, 2013, 197(2): 454-467.
[34] An X H, Tian Y, Chen K Q, et al. MdMYB9 and MdMYB11 are involved in the regulation of the JA-induced biosynthesis of anthocyanin and proanthocyanidin in apples [J]. Plant Cell Physiol, 2015, 56(4): 650-662.
[35] Tian J, Zhang J, Han Z Y, et al. McMYB12 transcription factors co-regulate proanthocyanidin and anthocyanin biosynthesis in[J]. Scientific Reports, 2017, 7(1): 43715. doi: 10.1038/srep43715.
[36] James A M, Ma D, Mellway R, et al. Poplar MYB115 and MYB134 transcription factors regulate proanthocyanidin synthesis and structure [J]. Plant Physiology, 2017, 174(1): 154-171.
[37] Wang N, Qu C, Jiang S, et al. The proanthocyanidin-specific transcription factor MdMYBPA1 initiates anthocyanin synthesis under low temperature conditions in red-fleshed apple [J]. The Plant J, 2018, 96(1): 39-55.
[38] Xu W, Dubos C, Lepiniec L. Transcriptional control of flavonoid biosynthesis by MYB-bHLH-WDR complexes [J]. Trends in Plant Science, 2015, 20(3): 176-185.
[39] Terrier N, Torregrosa L, Ageorges A, et al. Ectopic expression of VvMybPA2 promotes proanthocyanidin biosynthesis in grapevine and suggests additional targets in the pathway [J]. Plant Physiol, 2009, 149(2): 1028-1041.
[40] Gesell A, Yoshida K, Tran L T, et al. Characterization of an apple TT2-type R2R3 MYB transcription factor functionally similar to the poplar proanthocyanidin regulator PtMYB134 [J]. Planta, 2014, 240(3): 497-511.
[41] Mellway R D, Tran L T, Prouse M B et al. The wound-, pathogen-, and ultraviolet B-responsivegene encodes an R2R3 MYB transcription factor that regulates proanthocyanidin synthesis in poplar [J]. Plant Physiology, 2009, 150(2): 924-941.
[42] Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in[J]. Curr Opin Plant Biol, 2001, 4(5): 447-456.
[43] Liu R, Wang Y, Tang S, et al. Genome-wide identification of the tea plant bHLH transcription factor family and discovery of candidate regulators of trichome formation [J]. Sci Rep, 2021, 11(1): 10764. doi: 10.21203/rs.3.rs-148784/v1.
[44] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta DeltaC(T)) method [J]. Methods, 2001, 25(4): 402-408.
[45] Chen C, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data [J]. Mol Plant, 2020, 13(8): 1194-1202.
[46] Dubos C, Stracke R, Grotewold E, et al. MYB transcription factors in[J]. Trends Plant Sci, 2010, 15(10): 573-581.
[47] Stracke R, Ishihara H, Huep G, et al. Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of theseedling [J]. Plant J, 2007, 50(4): 660-677.
[48] Gonzalez A, Zhao M, Leavitt J M, et al. Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex inseedlings[J]. Plant J, 2008, 53(5): 814-827.
[49] Lepiniec L, Debeaujon I, Routaboul J M, et al. Genetics and biochemistry of seed flavonoids [J]. Annu Rev Plant Biol, 2006, 57: 405-430.
[50] Zhong R, Lee C, Zhou J, et al. A battery of transcription factors involved in the regulation of secondary cell wall biosynthesis in[J]. Plant Cell, 2008, 20(10): 2763-2782.
[51] Zhou J, Lee C, Zhong R, et al. MYB58 and MYB63 are transcriptional activators of the lignin biosynthetic pathway during secondary cell wall formation in[J]. Plant Cell, 2009, 21(1): 248-266.
[52] Sun B, Zhu Z, Cao P, et al. Purple foliage coloration in tea (L.) arises from activation of the R2R3-MYB transcription factor CsAN1 [J]. Sci Rep, 2016, 6: 32534. doi: 10.1038/srep32534.
[53] Wang X C, Wu J, Guan M L, et al.MYB4 plays dual roles in flavonoid biosynthesis [J]. Plant J, 2020, 101(3): 637-652.
[54] Ma D, Constabel C P. MYB repressors as regulators of phenylpropanoid metabolism in plants [J]. Trends Plant Sci, 2019, 24(3): 275-289.
[55] Agarwal M, Hao Y, Kapoor A, et al. A R2R3 type MYB transcription factor is involved in the cold regulation of CBF genes and in acquired freezing tolerance [J]. J Biol Chem, 2006, 281(49): 37636-37645.
[56] Kang Y H, Kirik V, Hulskamp M, et al. Thegene provides a positive feedback loop for cell fate specification in theroot epidermis [J]. Plant Cell, 2009, 21(4): 1080-1094.
[57] Jiang X, Huang K, Zheng G, et al. CsMYB5a and CsMYB5e fromdifferentially regulate anthocyanin and proanthocyanidin biosynthesis [J]. Plant Sci, 2018, 270: 209-220.
[58] Wang P, Ma G, Zhang L, et al. A sucrose-induced MYB (SIMYB) transcription factor promoting proanthocyanidin accumulation in the tea plant () [J]. J Agric Food Chem, 2019, 67(5): 1418-1428.
Expression Analysis and Functional Identification ofTranscription Factor in Tea Plants
WANG Yuyuan1, LIU Renjian1, LIU Shaoqun1, SHU Canwei2, SUN Binmei1, ZHENG Peng1*
1. College of Horticulture, South China Agricultural University, Guangzhou 510642, China; 2. College of Agriculture, South China Agricultural University/Guangdong Province Key Laboratory of Microbial Signals and Disease Control/Integrative Microbiology Research Center, Guangzhou 510642, China
Catechin is one of the most characteristic secondary metabolites in tea plants and the main component affecting tea quality and flavor. It possesses rich pharmacological effects, such as anti-oxidation, anti-virus, lipid lowering and weight loss, etc. In this study, the function of a catechin biosynthetic regulator CsTT2 was preliminarily identified using phylogenetic analysis, gene expression pattern analysis and molecular biology experiments. The results show that CsTT2 was a R2R3-MYB transcription factor that shares a branch withthe MYB transcription factor inthat regulates secondary metabolites. The expression levels ofand catechin biosynthesis genes were relatively high in the apical bud tissue of tea plants with higher total catechin content. The results of subcellular localization, yeast assay and dual luciferase reporting system further reveal thatwas located into the nucleus and the protein it encodes possessed transcriptional activation capacity. CsTT2 could bind to the promoter of a key catechin biosynthetic geneto activate its expression.
tea plant, R2R3-MYB transcription factor,, catechin biosynthesis
S571.1
A
1000-369X(2022)04-463-14
2021-12-27
2022-01-19
广州市科技计划项目(202102020290)、广东省自然科学基金项目(2018A030313089、2021A1515012091)
王玉源,女,硕士研究生,主要从事茶树遗传育种与分子生物学,wangyy@stu.scau.edu.cn。*通信作者:zhengp@scau.edu.cn

