橡胶树PR107和CATAS8-79中胶乳蛋白的差异分析及磷酸化蛋白的鉴定
王丹 徐兵强 孙勇 彭存智 常丽丽 仝征






摘 要:橡胶树是合成天然橡胶的重要植物。PR107和CATAS8-79是具有不同产排胶性状的2个无性系。在以往的研究中,胶乳中蛋白表达差异被认为是影响天然橡胶合成的关键因子之一。然而,这2个无性系之间与胶乳产量相关的蛋白质图谱尚未明确。本研究采用蛋白质组学方法对这些蛋白质进行鉴定,有助于探讨巴西橡膠树胶乳合成机理。经双向凝胶电泳和质谱鉴定分析,共获得65个差异表达蛋白信息。通过磷酸化蛋白质组分析,在PR107胶乳中鉴定出31个磷酸化蛋白质,含有74个磷酸化氨基酸残基,在CATAS8-79胶乳中鉴定出80个磷酸化蛋白质,含有166个磷酸化氨基酸残基。橡胶延伸因子/小橡胶粒子蛋白(REF/SRPP)家族成员被鉴定为差异表达蛋白和磷酸化修饰蛋白,这些蛋白在调节天然橡胶合成中起着重要作用。pro-hevein和hevamine也表现出不同的磷酸化修饰水平,它们主要在天然橡胶排胶过程中起作用。一种丝氨酸-苏氨酸蛋白磷酸酶激酶的磷酸化和去磷酸化修饰可能在天然橡胶合成中起调节作用。这些结果为天然橡胶生物合成调控机制的研究提供了新的理论依据。
关键词:巴西橡胶树;天然橡胶生物合成;磷酸蛋白质组;双向凝胶电泳(2-DE)中图分类号:S794.1 文献标识码:A
is an important plant for producing natural rubber. RP107 and CATAS8-79 are two clones of with different properties of rubber production and expulsion. The study of protein function in the latex may help understand the regulatory mechanism related to the properties of rubber production and expulsion. This study aimed to compare and analyze the difference in latex protein between RP107 and CATAS8-79 at the level of protein accumulation and post-translational modification. Through the two-dimensional gel electrophoresis (2-DE) analysis, 65 proteins derived from 88 spots were found to be accumulated differently in the latex between the 2 clones. Among the proteins, 44 proteins had high accumulation in the latex of PR107 and 21 had high accumulation in the latex of CATAS8-79. The high-accumulation proteins (HAPs) in the latex of CATAS8-79 were involved in intracellular organelles, external encapsulating structure, and membrane-bound organelles in terms of cellular component, and most of them had drug-binding activity and hydrolase activity. Different from CATAS8-79, the HAPs in the latex of PR107 participated in a catalytic complex, nonmembrane-bound organelles, and apoplasts. Most of them had protein-binding and transferase activities. Furthermore, some proteins related to natural rubber synthesis and latex agglutination were found in DAPs. The rubber elongation factors (REFs) and small rubber particle proteins (SRPPs) were identified. The two classes of proteins played an important role in natural rubber biosynthesis in rubber trees. Some proteins mediating rubber particle aggregation (RPA) and participating in response to tapping were found in DAPs too. To determinate the phosphorylated proteins and amino acids in the latex of the two clones, the phosphopeptides were enriched using a Fe-NTA Phosphopeptide Enrichment Kit and shotgun analysis was performed through the high-throughput Tandem Mass Spectrometer (MS/MS). 74 phosphorylated amino acid residues derived from 31 phosphorylated proteins, as well as 166 phosphorylated amino acid residues derived from 80 phosphorylated proteins, were identified in PR107 and CATAS8-79, respectively. Among the phosphorylated proteins, 25 proteins of PR107 and 74 proteins of CATAS8-79 were specific in terms of the phosphorylation amino acids. Between the two clones, the members of REF/SRPP protein family, which regulate the synthesis of nature rubber (NR) in the latex, have high differentiation capacity at both protein accumulation and phosphorylation modification levels. The pro-hevein and hevamine proteins, which influence the process of rubber expulsion, also showed diversity at the phosphorylation modification level. The phosphorylation and dephosphorylation of an serine/threonine protein phosphatase kinase might play a regulatory role in NR synthesis. The results could provide a new theoretical basis for the study of the regulatory mechanism of NR biosynthesis.
; natural rubber biosynthesis; phosphoproteome; two-dimensional gel electrophoresis (2-DE)
: 10.3969/j.issn.1000-2561.2022.05.004
巴西橡膠树()是目前最具商业价值的天然橡胶来源。天然橡胶是在橡胶树树皮下韧皮部乳管系统的乳管细胞中合成的,适当的割破树皮会获得胶乳。自首次证明胶乳中含有蛋白质以来,采用蛋白质组学技术研究发现,胶乳蛋白参与诸如天然橡胶的生物合成、对病原体的防御、伤口愈合和抗应激能力等代谢途径的调控。基于双向凝胶电泳技术(2-DE)结合质谱的鉴定技术首先应用于胶乳及其亚细胞组织中蛋白质的检测,确定胶乳中含有乳胶过敏原类蛋白质、几丁质酶类蛋白以及包括橡胶延伸因子(REF)/小橡胶粒子蛋白(SRPP)在内的天然橡胶合成关键蛋白。随着质谱技术的发展,高通量非凝胶液质连用质谱检测技术应用于胶乳蛋白质研究,大量胶乳蛋白质及其翻译后修饰位点被成功鉴定,稳定同位素标记相对和绝对定量(iTRAQ)技术的应用部分揭示了橡胶树胶乳中天然橡胶生物合成调控机制。目前,虽然巴西橡胶树胶乳中蛋白质功能的研究取得一定突破,但这些蛋白参与的代谢途径的确切调控机制仍不清楚,胶乳中与天然橡胶产量密切相关的调控机理尚未明确。
巴西橡胶树的产量是由不同因素决定的,其中一个关键因素是割胶后排胶的持续时间。几丁质酶,β-1,3-葡聚糖酶,hevein蛋白以及hevein蛋白前体与细胞骨架结合一起形成了阻碍胶乳流出的蛋白质网络,缩短了排胶时间。类糊精中的几丁质酶和葡聚糖酶作为凝固因子在橡胶颗粒聚集中起着关键作用。对橡胶树无性系CATAS7-33-97和CATAS8-79进行比较分析发现,水通道蛋白基因和乙烯生物合成关键基因表达增加,胶乳凝集因子基因表达减少等促进了割胶后排胶的过程。随着胶乳中茉莉酸途径和甲羟戊酸途径(MVA)的激活,质膜内在蛋白、蔗糖转运体、蔗糖合酶和3-羟基-3-甲基戊二酰辅酶A(HMG-CoA)合酶促进了天然橡胶的合成。然而,在橡胶树高产和低产品系之间,天然橡胶生物合成途径相关基因的表达水平没有发现显著差异。
橡胶树无性系PR107由印度尼西亚培育。对PR107的胶乳合成特性分析表明,PR107具有较强的蔗糖代谢和基础代谢能力,能优先利用蔗糖合成天然橡胶,具有较强的抵御非生物胁迫能力。CATAS8-79是中国热带农业科学院自主培育的一个无性系,具有高产、早熟等优良性状。CATAS8-79的流速和排胶持续时间高于PR107。基于CATAS8-79和PR107之间的胶乳转录组测序及差异表达分析,发现内源性乙烯水平、乳管细胞壁木质素含量和葡聚糖酶的表达与排胶持续时间有关。这2个无性系的差异基因组学、蛋白质组学和转录组学分析可以揭示天然橡胶合成的一些分子机制。
蛋白质翻译后修饰(PTM)是天然橡胶合成途径的重要调控方式之一。在这个过程中,一些激酶对蛋白质的磷酸化起着重要的作用。羟甲基戊二酰辅酶A还原酶(HMGR)是MVA途径中的一种关键酶,调节巴西橡胶树的天然橡胶合成途径。SUGDEN等发现SNF1相关蛋白激酶(SnRK1)对HbHMGR1活性的影响主要是调节其Ser577氨基酸残基的磷酸化水平。在菠菜叶中,SnRK1通过调节HMGR蛋白的非活化的Ser577氨基酸位点的磷酸化来调控类异戊二烯和蔗糖的合成。一些研究还表明,天然橡胶合成相关蛋白的PTMs是橡胶树的重要调控机制。在CATAS7-33-97中,许多与橡胶生物合成相关的蛋白质被磷酸化。从16种橡胶延伸因子(REF)和7种小橡胶粒子蛋白(SRPP)异构体中鉴定出138种独特的磷酸化肽,包含129种磷酸化氨基酸。TONG等也发现PR107和CATAS8-79中有许多REF和SRPP被磷酸化,其中一些是PR107或CATAS8-79所特有的。
尽管橡胶合成相关蛋白的磷酸化修饰是一种常见现象,但蛋白质磷酸化与天然橡胶合成之间的关系尚未被揭示。本研究的目的是通过蛋白质组学分析,发现PR107和CATAS8-79胶乳蛋白之间的差异积累和磷酸化修饰蛋白信息,为天然橡胶生物合成调控机制的研究提供理论依据。
材料与方法
材料
以橡胶树无性系PR107和CATAS8-79为研究对象,PR107是由云南省热带作物科学研究所于1957年由印度尼西亚引进的优良无性系品种,因其在栽培中后期的橡胶产量、耐刺激割胶、抗病性、立木材积量等方面表现优良,于1962年选为云南大规模推广种植品种。CATAS8-79是从1973年‘热研88-13’(母本)和‘热研217’(父本)的人工授粉苗中选出建立的具备早熟高产优良性质的无性系,2001年通过全国农作物品种审定委员会审定。研究用橡胶树种植在中国热带农业科学院儋州市试验农场。从每个无性系中选择30棵树(8年树龄,未开割)进行分析。
方法
1.2.1 膠乳采集及相关指标测定 割胶后,丢弃前20滴胶乳,在冰上用离心管收集随后排出的胶乳。收集的胶乳被带回实验室作进一步分析。收集割胶后1 min内流出的胶乳,检测胶乳体积,计算初始胶乳流速。同时收集割胶后流出的所有胶乳,当2滴胶乳滴落间隔超过1 h后停止收集,测定胶乳产量。每个生理指标测定30个生物学重复。
1.2.2 蛋白质提取及双向电泳 胶乳中的蛋白质提取过程如下:将3 mL胶乳添加到5 mL冷提取缓冲液(100 mmol/L乙二胺四乙酸、50 mmol/L维生素C、50 mmol/L硼砂、1%Triton X-100、1%交联聚乙烯吡咯烷酮PVPP、2% β-巯基乙醇、30%蔗糖和100 mmol/L Tris-HCl,pH 8.0)。旋涡震荡10 min后,向管中加入8 mL Tris饱和苯酚(pH>7.8),震荡10 min,随后4℃,15 000 ,离心15 min。收集上层酚相,加入5倍体积的预冷过饱和硫酸铵甲醇溶液,在–20℃沉淀12 h。沉淀用冰甲醇和丙酮洗涤,自然干燥,沉淀用蛋白质裂解缓冲液(2 mol/L硫脲、7 mol/L尿素、2% 3-[(3-胆酰胺丙基)二甲氨基]-丙磺酸盐CHAPS、1%IPG缓冲液、13 mmol/L二硫苏糖醇DTT)重新溶解。以BSA作为标准品,用Bradford法测定蛋白质样品的浓度。
从2个无性系的胶乳中提取各1300 μg蛋白质样品加入线性梯度IPG条(24 cm,pH 4~7)中,在25℃下持续水化18 h。等电聚焦后,用含有1% DTT的溶液ES(50 mmol/L Tris pH 8.8,30%甘油,6 mol/L尿素,2%SDS和0.002%溴酚蓝)平衡15 min,用含有4% 碘乙酰胺的ES平衡15 min。经SDS-PAGE(12.5%)进行蛋白质的电泳分离。凝胶用考马斯亮蓝染料染色12 h,并用图像扫描仪III获取凝胶图谱。使用ImageMaster 2D Platinum软件进行图像分析。每组样品进行3次生物学重复。
1.2.3 差异积累蛋白质的质谱鉴定 在3个生物学重复中,蛋白质相对表达丰度变化量大于2倍的蛋白点称为差异积累蛋白点(DAP)。人工切除凝胶上的DAP斑点并进行胰蛋白酶消化,大致过程如下:在室温下用50%乙腈(/)/50 mmol/L NHHCO溶液脱色至胶粒透明,在37℃条件下用含有5 pmol胰蛋白酶的100 mmol/L NHHCO酶解16 h。瞬时离心后,取0.8 µL酶解液加在基质辅助激光解吸电离(MALDI)板(384 Opti-TOF)样品位上,待其干燥后,再向每个样品上加入0.8 µL 5 mg/mL基质(氰基-4-羟基肉桂酸(CHCA)溶解于50%乙腈和0.1%三氟乙酸)。待样品完全干燥后,用5800 MALDI-TOF/TOF质谱仪进行鉴定。质谱数据通过Protein Pilot软件(5.0版)进行分析,并根据NCBI数据库进行检索(ftp://ftp.ncbi.nlm.nih.gov/genomes/Hevea_bra siliensis/protein/),该数据库包括58 062个蛋白序列和27 683 334个多肽信息。DAPs的成功鉴定标准为肽段评分大于32,值小于0.05,至少匹配2条可信多肽,匹配氨基酸序列覆盖率大于4%。
1.2.4 抗体制备及蛋白表达量检测(Western blotting) 构建携带DAPs目的基因的重组质粒pET-30a,并将其转化到大肠杆菌(Rosetta DE3)。用0.5 mmol/L IPTG对重组大肠杆菌进行蛋白诱导。目的蛋白经His标记纯化。分别用Freund完全佐剂和Freund不完全佐剂乳化后,分次皮下注射新西兰大白兔,经多次免疫后获得含有多克隆抗体的兔血清。获得的抗体血清用于PR107和CATAS8-79胶乳样品中蛋白表达量的检测:10 μg胶乳蛋白样品经SDS-PAGE电泳分离,将凝胶中的蛋白电转移到PVDF膜上,用5%脱脂牛奶封闭PVDF膜。用DAPs多克隆兔抗体和HRP结合抗兔IgG孵育PVDF膜。PVDF膜在黑暗的环境中经试剂盒显色和并用LAS-4000微型成像仪检测DAPs蛋白条带的图像。所有的Western blotting进行3次生物学重复。
1.2.5 PR107和CATAS8-79胶乳磷酸化蛋白的富集与鉴定 分别取1 mg PR107和CATAS8-79的胶乳蛋白用于磷酸化蛋白鉴定,酶解后的多肽使用Pierce Fe NTA磷酸肽富集试剂盒富集磷酸肽:将酶解后的肽段用200 μL结合缓冲液重新悬浮并加载到Fe-NTA自旋柱。在室温下孵育20 min后,将柱转移到微型离心管中,并在室温下以1000 离心1 min。使用C18柱和UltiMate 3000系统收集并分离磷酸肽。将收集的组分通过HPLC(Dionex Ultra 3000 nano)-ESI-MS/MS(Triple TOF 5600,ABsciex)系统进行分离鉴定。质谱原始数据使用ProteinPilot软件V5.0(ABsciex)分析,获得磷酸化蛋白质和氨基酸位点的信息。质谱鉴定成功的标准:未使用的置信度得分≥1.3,至少有2个匹配的多肽具有高于95%的置信度和低于1%的错误发现率(FDR),所有的样品进行3个生物学重复。
1.2.6 生物信息学分析 使用Blast2-GO软件对来自2-DE的DAPs和不同磷酸化蛋白进行GO基因通路分析,使用WEGO软件进行GO基因富集分析(http://wego.genomics.org.cn/)。
结果与分析
和胶乳指标测定
测定了无性系PR107和CATAS8-79的胶乳流速后发现,2个无性系间差异极显著。在CATAS8- 79中,割胶后的第1分钟内收集到约为(4.1±0.5)mL胶乳,而在PR107中只收集到(2.5±0.3)mL胶乳(图1A)。测量了来自同一无性系的不同植株的胶乳产量,其中CATAS8-79收集到(139±25.6)mL胶乳,而PR107仅收集到(53±13.9)mL胶乳(图1B)。
和胶乳中差异积累蛋白质的鉴定及分析
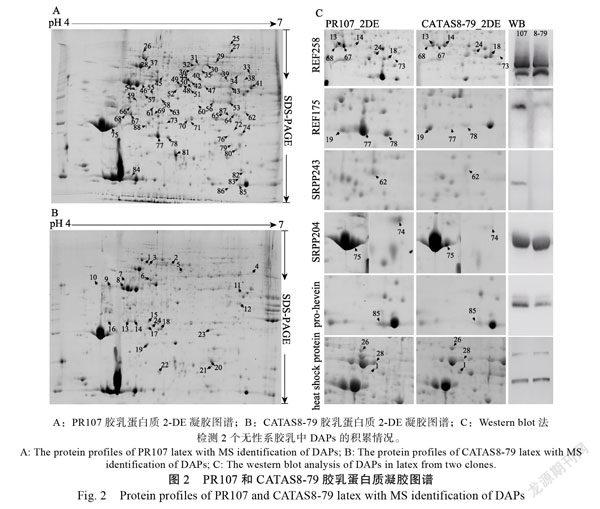
经胶乳蛋白质提取及双向凝胶电泳分离后,在无性系PR107的凝胶上检测到丰度Vol>0.01%的蛋白点1301±87个,丰度在0.005%~0.01%之间的蛋白点660±47个。在无性系CATAS8-79的凝胶上,检测到丰度Vol>0.01%的蛋白点1132±63个,0.005%~0.01%之间的蛋白点634±23个。其中88个蛋白点的丰度发生了2倍及以上的变化(<0.05)。PR107凝胶中检测到64个蛋白斑点的丰度高于CATAS8-79凝胶中相同蛋白斑点2倍以上(图2A)。CATAS8-79凝胶中检测到24个蛋白斑点的丰度高于PR107凝胶中的2倍以上(图2B)。这些斑点中含有的蛋白是PR107和CATAS8-79胶乳中差异积累蛋白质。使用MALDI-TOF/TOF-MS对88个蛋白斑点进行了鉴定,成功鉴定到65种差异蛋白质。其中44个蛋白在PR107胶乳中有高积累,21个蛋白在CATAS8-79胶乳中有高积累。
在这些差异蛋白中发现了一些与天然橡胶合成和胶乳凝集有关的蛋白质,并通过Western blot进行验证(图2C)。含有258个氨基酸的27.3-kDa REF(REF258, ref|XP_021653592.1)的7种异构体在2个无性系的胶乳中显示出差异积累(点13、14、18、24、67、68和73,表1)。这些异构体具有不同的等电点(pI)和分子量(MW)。其中3个(点67、68和73)在PR107胶乳中积累较高,而在CATAS8-79中积累较低。含有175个氨基酸的19.6-kDa REF(REF175, ref|XP_ 021653600.1)的2个异构体(点77和78)在PR107胶乳中积累较高,一个异构体(点19)在PR107胶乳中积累较低。与REF不同,含有204个氨基酸的22.3-kDa SRPP(SRPP204, ref|XP 021653597.1)的2种异构体(点74和75)和含有243个氨基酸的27.1-kDa SRPP(SRPP243, ref|XP 021662186.1)的一种异构体(点62)在2个无性系之间具有不同的表达水平,并且在PR107的胶乳中具有较高的积累。乙酰辅酶A乙酰转移酶在PR107的胶乳中有较高的积累(点41和43)(表1,图2A和图2B),该蛋白是橡胶生物合成中二磷酸异戊烯酯的来源,与橡胶产量和品质密切相关。
一些介导橡胶颗粒聚集(RPA)和参与割胶反应的蛋白质也被成功鉴定。在PR107中,橡胶蛋白前体pro-hevein(spot 85)在胶乳中有较高的积累(表1,图2C)。热休克70 kDa蛋白的2种异构体(点26和28)在PR107膠乳中存在积累差异,而在CATAS8-79胶乳中仅检测到一种异构体(点1)存在差异(表1,图2C)。2个无性系之间的western印迹也证实了胶乳中的pro-hevein和热休克70 kDa蛋白积累的差异(图2C)。
此外,CATAS8-79胶乳中的高积累蛋白(HAPs)在细胞成分上涉及胞内细胞器、外包囊结构和膜结合细胞器,其中大部分具有药物结合活性和水解酶活性。与CATAS8-79不同,PR107胶乳中的HAPs参与催化复合物、非膜结合细胞器和质外体,大多数具有蛋白质结合和转移酶活性。在CATAS8-79中,大多数HAPs参与初级代谢过程、有机物代谢过程、氮化合物代谢过程和分解代谢过程。此外,参与毒素分解代谢过程的蛋白质在CATAS8-79的胶乳中具有高积累,而参与响应非生物和生物刺激的蛋白质在PR107的胶乳中具有高积累(图3A)。
和胶乳中磷酸化蛋白质的鉴定
在PR107胶乳中鉴定出31个磷酸化蛋白,含有74个磷酸化氨基酸,置信度高于95%。这些磷酸化氨基酸包括56个丝氨酸残基、13个苏氨酸残基和5个天冬氨酸残基。在CATAS8-79胶乳中共鉴定出80种磷酸化蛋白,含有166个磷酸化氨基酸,这些磷酸化氨基酸包括141个丝氨酸残基、22个苏氨酸残基和3个天冬氨酸残基。在这些磷酸化蛋白中,有9个磷酸化蛋白包含17个磷酸化氨基酸(14个丝氨酸残基和3个苏氨酸残基)只是在PR107中鉴定到,有58个磷酸化蛋白包含103个磷酸化氨基酸(92个丝氨酸残基和11个苏氨酸残基)只在CATAS8-79中鉴定到。在PR107中,更多的磷酸化蛋白参与构成多种细胞成分,如膜、共质体、细胞连接和细胞外区域。在CATAS8-79中,更多的磷酸化蛋白参与多细胞组织过程、细胞成分组织或生物发生、色素沉着和免疫系统过程。与PR107相比,在CATAS8-79的胶乳中发现更多具有催化活性的蛋白质被磷酸化修饰(图3B)。
在这些磷酸化蛋白中,有7种蛋白参与了天然橡胶的合成、胶乳再生和胶乳排出时间的调控,但是它们在不同橡胶树无性系中的磷酸化修饰位点不同。在PR107中,REF258的第49个氨基酸残基被磷酸化(Ser49th),而在CATAS8-79中,是第3个氨基酸残基被磷酸化(Ser3rd)(表2,图4)。REF138的asp80、asp100和asp107仅在PR107中发生磷酸化。SRPP117的Ser45th、SRPP204的Ser26th、SRPP243的Ser109th和HMGR的Ser20th仅在CATAS8-79中发生磷酸化修饰(表2和图4)。pro-hevein在PR107和CATAS8-79胶乳中的磷酸化程度不同(表2)。
一些参与应激反应和细胞内信号转导的蛋白质在PR107和CATAS8-79之间也具有不同的磷酸化位点。在PR107胶乳中,Hsp70-Hsp90组织蛋白3(ref|xp_021641666.1)和应激相关蛋白(ref|xp_ 021653583.1和ref|xp_021653585.1)中发现了更多的磷酸化氨基酸(表2)。此外,在信号转导过程中起重要作用的pleckstrin同源结构域蛋白质(ref|xp_021682526.1和ref|xp_021659098.1)在CATAS8-79中有更多的氨基酸残基被磷酸化修饰。
讨论
橡胶蛋白前体和几丁质酶的积累和磷酸化差异可能是影响胶乳凝集的重要因素之一
在巴西橡胶树胶乳的C-血清、橡胶粒子和黄色体中,小橡胶粒子蛋白(SRPP)、葡聚糖酶(glucanase)、hevamine、hevein等蛋白通常與胶乳凝集有关。在本研究中,CATAS8-79的流速和总产胶量明显高于PR107,CATAS8-79中这些相关蛋白的表达或修饰情况可能会更倾向于增加胶乳的流动性,相互作用共同保持其较高的初始流速及最终高的产量。作为一种凝集素蛋白,橡胶树蛋白前体(pro-hevein)通过与橡胶粒子膜相互作用而显示出各种凝集特性。pro-hevein的N端结构域带有几丁质结合基序,C端结构域可以与几丁质结合活性协同提供凝集活性。在本研究中,PR107中pro-hevein的丰度是CATAS8- 79的3.08倍。它也被鉴定为磷酸化蛋白,在CATAS8-79中,Pro-hevein在Barwin样结构域的Thr149处被磷酸化,该位置是一个C末端诱导结构域。然而,在PR107中,pro-hevein的磷酸化位点位于Ser196th,更接近C末端(表2和图4)。本研究认为Pro-hevein的表达量和磷酸化修饰的差异可能是影响PR107和CATAS8-79胶乳流出速度和产量的因子之一。
hevamine是一种具有溶菌酶和几丁质酶活性的植物防御蛋白。在橡胶树中,hevamine通过堵塞乳管防止胶乳排出。诸多研究表明几丁质酶是决定割胶后胶乳流动持续时间的关键成员之一,在CATAS8-79中,割胶后胶乳几丁质酶基因(HbChit)下调表达。在本研究中,2个橡胶树无性系中的hevamine-A样蛋白(ref|xp_021671430.1)氨基酸残基的Thr157th均发生磷酸化修饰,但在PR107的胶乳中检测到更多含有Thr157th磷酸化位点的多肽,Thr157th位点的磷酸化修饰可能是hevamine-A行使其功能的重要修饰之一。
在胶乳凝集过程中,胶乳凝集素蛋白存在于黄色体膜上,与糖基化蛋白SRPP相互作用,充当连接橡胶粒子的多价桥梁。另一种可能是黄色体破裂后,黄色体膜内的凝集素蛋白结合位点暴露并与SRPP结合。橡胶粒子在黄色体膜碎片上的聚集导致橡胶凝结。SRPP在胶乳凝集过程中发挥重要作用。与PR107相比,CATAS8-79胶乳中鉴定到SRPP上具备更多的磷酸化氨基酸位点,SRPP的不同磷酸化水平可能影响橡胶树胶乳凝集过程。
磷酸化蛋白参与了和胶乳中橡胶合成的调控
天然橡胶的生物合成是橡胶树乳管的主要代谢过程,这一过程涉及20多种酶反应。在本研究中,SRPP204和SRPP243在蛋白表达水平和磷酸化修饰层面均与橡胶产量存在一定的相关性。REF258蛋白N端的ATP酶β-亚基结构域第49位的氨基酸残基仅在CATAS8-79胶乳中被磷酸化修饰。REF138中的3种氨基酸仅在PR107胶乳中磷酸化,REF/SRPP家族成员极有可能通过磷酸化修饰调控天然橡胶的合成。HMGR1是橡胶树胶乳中主要的胶乳代谢相关蛋白之一。天然橡胶产量与橡胶树中HbHMGR1表达水平显著正相关。在橡胶草中( R.),HMGR1在高橡胶产量橡胶草中表达量比较高,且与天然橡胶生产能力的相一致。在本研究中,PR107和CATAS8-79之间胶乳中HMGR1的蛋白积累没有差异,但是2个橡胶树无性系之间HMGR1在磷酸化水平上存在差异,可能与其产胶性状相关。
蛋白磷酸化激酶参与了和胶乳的生物过程调控
蛋白质磷酸化是调节许多细胞过程的重要机制,如原核生物和真核生物的细胞壁生物合成、生物膜形成和应激反应。各种氨基酸残基的磷酸化可控制蛋白质–蛋白质相互作用和蛋白质聚集体的形成。丝氨酸/苏氨酸蛋白激酶可通过在单个N-末端丝氨酸(苏氨酸)残基处特异性磷酸化来调节磷酸烯醇式丙酮酸羧化酶活性。在拟南芥中,STN7(丝氨酸/苏氨酸蛋白激酶7的同源物)中丝氨酸和苏氨酸残基磷酸化的动态调节对植物生长和环境适应至关重要。丝氨酸/苏氨酸蛋白激酶在调节膜相关功能、大分子生物合成过程和脂质代谢中起着重要作用。激酶很可能通过自身的磷酸化和去磷酸化影响这些生物过程。在本研究中,PR107和CATAS8-79胶乳中一些丝氨酸/苏氨酸蛋白激酶家族蛋白的氨基酸残基发生了磷酸化修饰并存在差异,这一发现暗示这种激酶家族在PR107和CATAS8-79的乳管细胞壁形成及应激反应过程中可能存在不同的调控机制。
结论
橡胶树无性系PR107与CATAS8-79在胶乳产量和割胶后胶乳排出持续时间上存在一定的差异。为了揭示天然橡胶合成和排胶的分子机理,本文对PR107和CATAS8-79的胶乳差异表达蛋白质和磷酸化蛋白质进行了比较分析。共检测到88个差异蛋白点含有65个差异积累蛋白。其中44个蛋白在PR107胶乳中有较高的积累量,21个蛋白在CATAS8-79胶乳中有较高的积累量,在CATAS8-79胶乳中鉴定出80个独特的磷酸化蛋白,包含166个磷酸化氨基酸。通过生物信息学分析,推测REF/SRPP超家族成员在2个无性系中通过蛋白表达和PTM调控天然橡胶合成的机制不同。基于某些形式的磷酸化修饰,橡胶蛋白前體和几丁质酶可能在胶乳凝集过程中起重要作用。一种丝氨酸–苏氨酸蛋白磷酸酶激酶家族成员的磷酸化和去磷酸化也可能参与天然橡胶的合成和胶乳的排出。
参考文献
[1] BEILEN J B V, POIRIER Y. Establishment of new crops for the production of natural rubber[J]. TRENDS in Biotechnology, 2007, 25(11): 522-529.
[2] D’AUZAC J, JACOB J L, PRÉVÔT J C, CLÉMENT A, GOHET E. The regulation of cis-polyisoprene production (natural rubber) from [J]. Research Singpost, 1997, 1(1): 273-332.
[3] SPENCE D. On the presence of oxydases in india-rubber, with a theory in regard to their function in the latex[J]. Biochemical Journal, 1908, 3(4):165-181.
[4] HABIB M A H, ISMAIL M N. latex proteomics: a review of analytical methods and the way forward[J]. Journal of Plant Research, 2021, 134(1): 43-53.
[5] POSCH A, CHEN Z, WHEELER C, DUNN M J, BAUR X Characterization and identifcation of latex allergens by two-dimensional electrophoresis and protein microsequencing[J]. Journal of Allergy and Clinical Immunology, 1997, 99(3): 385-395.
[6] SUBROTO T, VRIES H D, SCHURINGA J J, SOEDJANAATMADJA U M S, HOFSTEENGE J, JEKEL P A, BEINTEMA J J Enzymic and structural studies on processed proteins from the vacuolar (lutoid-body) fraction of latex of [J]. Plant Physiology and Biochemistry, 2001, 39(12): 1047-1055.
[7] DAI L J, XIANG Q L, LI Y, NIE Z Y, KANG G J, DUAN C F, ZENG R Z Rubber particle protein analysis of by two dimensional 16-BAC/SDS-PAGE and mass spectrometry[J]. Scientia Agricultura Sinica, 2012, 45(11): 2328-2338.
[8] RAHMAN A Y A, USHARRAJ A O, MISRA B B, THOTTATHIL G P, JAYASEKARAN K, FENG Y, HOU S, SU Y O, NG F L, LING S L Draft genome sequence of the rubber tree [J]. BMC Genomics, 2013, 14(1): 75.
[9] HABIB M A H, YUEN G C, OTHMAN F, ZAINUDIN N N, ISMAIL M N Proteomics analysis of latex from (clone RRIM 600)[J]. Biochemistry and Cell Biology, 2017, 95(2): 232-242.
[10] HABIB M A H, GAN C Y, LATIF A A, ISMAIL M N Unrestrictive identifcation of post-translational modifcations in latex[J]. Biochemistry and Cell Biology, 2018, 96(6): 818-824.
[11] DAI L J, KANG G J, NIE Z Y, LI Y, ZENG R Z Comparative proteomic analysis of latex from treated with Ethrel and methyl jasmonate using iTRAQ- coupled two-dimensional LC–MS/MS[J]. Journal of Proteomics, 2016, 132(1): 167-175.
[12] WANG D, XIE Q, SUN Y, TONG Z, CHANG L L, YU L, ZHANG X Y, YUAN B X, HE P, JIN X, DONG Y Y, LI H B, MONTORO P, WANG X C Proteomic landscape has revealed small rubber particles are crucial rubber biosynthetic machines for ethylene-stimulation in natural rubber production[J]. International Journal of Molecular Sciences, 2019, 20(20): 5082.
[13] SHI M J, LI Y, DENG S N, WANG D D, CHEN Y Y, YANG S G, WU J L, TIAN W M. The formation and accumulation of protein-networks by physical interactions in the rapid occlusion of laticifer cells in rubber tree undergoing successive mechanical wounding[J]. BMC Plant Biology, 2019, 19(1): 8.
[14] WANG X C, SHI M J, WANG D, CHEN Y Y, CAI F G, ZHANG S X, WANG L M, TONG Z, TIAN W M. Comparative proteomics of primary and secondary lutoids reveals that chitinase and glucanase play a crucial combined role in rubber particle aggregation in [J]. Journal of Proteome Research, 2013, 12(11): 5146-5159.
[15] CHAO J Q, YANG S G, CHENY Y, TIAN W M. Transcript profiling of during latex flow[J]. Frontiers in Plant Science, 2017, 8(1): 1904.
[16] TANG C R, XIAO X H, LI H P, FAN Y J, YANG J H, QI J Y, LI H B. Comparative analysis of latex transcriptome reveals putative molecular mechanisms underlying super productivity of [J]. PLoS One, 2013, 8(9): e75307.
[17] LI G H, WU Y, XIAO G X. Pattern of traits transmission heredity for cultivars in Yunnan province[J]. Tropical Agricultural Science & Technology, 2010, 33(1): 5-17.
[18] LI W G, GAO X S, ZHANG W S, FANG J L, HUANG H S. Breeding and selection of desirable rubber clone Reyan 8-79[J]. Chinese Journal of Tropical Crops, 2009, 30(10): 1389-1393.
[19] CHAO J Q, CHEN Y Y, WU S H, TIAN W M. Comparative transcriptome analysis of latex from rubber tree clone CATAS8-79 and PR107 reveals new cues for the regulation of latex regeneration and duration of latex flow[J]. Genomics Data, 2015, 5(C): 120-121.
[20] JAYASHREE R, NAZEEM P A, REKHA K, SREELATHA S, THULASEEDHARAN A, KRISHNAKUMAR R, KALA R G, VINEETHA M, LEDA P, JINU U, VENKATACHALAM P. Over-expression of 3-hydroxy-3- methylglutaryl-coenzyme A reductase 1 (hmgr1) gene under super-promoter for enhanced latex biosynthesis in rubber tree ( Muell. Arg.)[J]. Plant Physiology and Biochemistry, 2018, 127(1): 414-424.
[21] SUGDEN C, DONAGHY P G, HALFORD N G, HARDIE D G. Two SNF1-related protein kinases from spinach leaf phosphorylate and inactivate 3-hydroxy-3-methylglutaryl- coenzyme A reductase, nitrate reductase, and sucrose phosphate synthase [J]. Plant Physiology, 1999, 120(1): 257-274.
[22] TONG Z, WANG D, SUN Y, YANG Q, MENG X R, WANG L M, FENG W Q, LI L, WURTELE E S, WANG X C. Comparative proteomics of rubber latex revealed multiple protein species of REF/SRPP family respond diversely to ethylene stimulation among different rubber tree clones[J]. International Journal of Molecular Sciences, 2017, 18(5): 958.
[23] WITITSUWANNAKUL R, PASITKUL P, KANOKWIROON K, WITITSUWANNAKUL D. A role for a Hevea latex lectin-like protein in mediating rubber particle aggregation and latex coagulation[J]. Phytochemistry, 2008, 69(2): 339-347.
[24] BERTHELOT K, PERUCH F, LECOMTE S. Highlights on (pro) hevein proteins[J]. Biochimie, 2016, 127(1): 258-270.
[25] BERTHELOT K, LECOMTE S, COULARY-SALIN B, BENTALEB A, PERUCH F. prohevein possesses a conserved C-terminal domain with amyloid-like properties [J]. Biochimica et Biophysica Acta-Proteins and Proteomics, 2016, 1864(4): 388-399.
[26] ROZEBOOM H J, BUDIANI A, BEINTEMA J J, DIJKSTRA B W. Crystallization of hevamine, an enzyme with lysozyme/chitinase activity from latex[J]. Journal of Molecular Biology, 1990, 212(3): 441-443.
[27] JEKEL P A, HARTMANN B H, BEINTEMA J J. The primary structure of hevamine, an enzyme with lysozyme/ chitinase activity from latex[J]. FEBS Journal, 1991, 200(1): 123-130.
[28] GIDROL X, CHRESTIN H, TAN H L, KUSH A. Hevein, a lectin-like protein from (rubber tree) is involved in the coagulation of latex[J]. Journal of Biological Chemistry, 1994, 269(12): 9278-9283.
[29] WITITSUWANNAKUL R, PASITKUL P, JEWTRAGOON P, WITITSUWANNAKUL D. Hevea latex lectin binding protein in C-serum as an anti-latex coagulating factor and its role in a proposed new model for latex coagulation[J]. Phytochemistry, 2008, 69(3): 656-662.
[30] WU C T, LI Y, NIE Z Y, DAI L J, KANG G J, ZENG R Z. Molecular cloning and expression analysis of the mevalonate diphosphate decarboxylase gene from the latex of [J]. Tree Genetics & Genomes, 2017, 13(1): 22.
[31] LAN C T,NIE Z Y,ZENG R Z. The relationship between latex metabolism gene expression with rubber yield and related traits in [J]. BMC Genomics, 2018, 19(1): 897.
[32] PANARA F, LOPEZ L, DADDIEGO L, FANTINI E, FACELLA P, PERROTTA G. Comparative transcriptomics between high and low rubber producing R. plants[J]. BMC Genomics, 2018, 19(1): 875.
[33] JANCZAREK M, VINARDELL J M, LIPA P, KARAŚ M. Hanks-type Serine/Threonine protein kinases and phosphatases in bacteria: roles in signaling and adaptation to various environments[J]. International Journal of Molecular Sciences, 2018, 19(10): E2872.
[34] NICOT A S, LO VERSO F, RATTI F, PILOT-STORCK F, STREICHENBERGER N, SANDRI M, SCHAEFFER L, GOILLOT E. Phosphorylation of NBR1 by GSK3 modulates protein aggregation[J]. Autophagy, 2014, 10(6): 1036-1053.
[35] TAYBI T, PATIL S, CHOLLET R, CUSHMAN J C. A minimal Serine/Threonine protein kinase circadianly regulates phosphoenolpyruvate carboxylase activity in crassulacean acid metabolism-induced leaves of the common ice plant[J]. Plant Physiology, 2000, 123(4): 1471-1481.
[36] TROTTA A, SUORSA M, RANTALA M, LUNDIN B, ARO E M. Serine and threonine residues of plant STN7 kinase are differentially phosphorylated upon changing light conditions and specifically influence the activity and stability of the kinase[J]. The Plant Journal, 2016, 87(5): 484-494.
[37] WU F L, LIU Y, JIANG H W, LUAN Y Z, ZHANG H N, HE X, XU Z W, HOU J L, JI L Y, XIE Z, CZAJKOWSKY D M, YAN W, DENG J Y, BI L J, ZHANG X E, TAO S C. The Ser/Thr protein kinase protein-protein interaction map of [J] Molecular & Cellular Proteomics, 2017, 16(8): 1491-1506.

