MALDI-TOF MS自建数据库对非结核分枝杆菌鉴定的应用价值*
徐茂锁,刘亚隽,张慧,周聪,齐伟强,申春梅
(上海市第五人民医院a.检验科,b.院感科,上海 200240)
近年来,我国非结核分枝杆菌(non-tuberculous mycobacteria,NTM)发病率呈上升趋势,全球范围内NTM疾病发病率和患病率也迅速上升[1-2]。基质辅助激光解吸电离飞行时间质谱(matrix assisted laser ionization time of flight mass spectrometry,MALDI-TOF MS)技术现广泛应用于微生物领域,其对NTM菌种鉴定国内外均有报道,但在Bruker MALDI-TOF MS使用中,需使用分枝杆菌数据库鉴定才能取得满意效果[3-5]。本实验室Bruker MALDI-TOF MS搭载的数据库为MBT DB5989数据库,包含41种分枝杆菌信息,但目前已发现NTM 190余种,可见该数据库对NTM鉴定能力尚有不足。MALDI-TOF MS自建数据库可补充商品数据库外的预测图谱信息,对数据库进行有效完善,从而提高鉴定准确率[6]。本研究利用分子生物学测序确认NTM菌种信息并构建自建数据库,以分子生物学测序结果为金标准,分析MALDI-TOF MS自建数据库辅助MBT DB5989数据库对NTM鉴定的可行性,评价MALDI-T0F MS自建数据库对NTM鉴定的应用价值。
1 资料与方法
1.1一般资料 收集2018年1月—2021年5月上海市第五人民医院微生物实验室分枝杆菌液体培养阳性、抗酸染色确认且液体培养物MPT64抗原检测阴性的分枝杆菌,剔除同一患者重复菌株后,共73株,其中,男性41株,女性32株,患者年龄中位数66岁;标本类型:1株为引流液,72株为痰液,主要来源科室依次为结核门诊、结核病房、观察室及ICU。
1.2仪器与试剂 Microflex质谱仪(德国Bruker公司),MGIT 320全自动分枝杆菌培养仪(美国BD公司),Mastercycler nexus PCR仪(德国Eppendorf公司),DYY-6C型电泳仪(北京六一生物科技公司),ABI 3739XL全自动DNA测序仪(美国应用生物系统公司);结核分枝杆菌MPT64抗原检测试剂盒(胶体金法,韩国标准诊断公司),中性罗氏培养基(上海申启生物科技公司),抗酸染色液(珠海贝索公司),质谱试剂IVD细菌试验标准品(IVD Bacterial Test Standard,IVD BTS)和IVD HCCA基质溶液(德国布鲁克·道尔顿公司),无水乙醇(中国赛默飞世尔公司),甲酸、乙腈(美国Sigma-Aldrich公司),DNA-EZ Reagents V DNA提取液和PCR扩增试剂2×SanTaq PCR Mix预混液(上海生工生物公司),质谱分析软件Flexcontrol 3.4和Biotyper OC 3.1(德国Bruker公司)。
1.3方法
1.3.1NTM菌株的筛选 分枝杆菌液体培养阳性,液体培养物进行抗酸染色确认和MPT64抗原胶体金法检测,筛选MPT64抗原阴性分枝杆菌。
1.3.2自建数据库用菌株及待检NTM菌株的确认 对MPT64抗原阴性分枝杆菌使用DNA-EZ Reagents V DNA提取液处理,80 ℃金属浴5 min后取上清液作模板DNA。应用16S RNA编码基因(16SDNA)、热休克蛋白65编码基因(hsp65)和RNA聚合酶的β亚基(rpoB)进行PCR扩增,引物序列参照文献[7-8]设计,见表1,由上海生工生物公司合成。PCR反应体系:2×SanTaq PCR Mix预混液25 μL,DNA模板1 μL,上、下游引物(10 μmol/L)各2 μL,双蒸水20 μL。PCR反应参数:98 ℃ 2 min;98 ℃ 10 s,57 ℃ 5 s,72 ℃ 40 s,35个循环;72 ℃ 5 min,4 ℃保存。PCR产物经10 g/L琼脂糖凝胶电泳确认,送上海生工生物公司使用ABI 3739XL全自动DNA测序仪进行一代测序,同源序列比对使用美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库“Microbes”目录下“blastn”模块进行分析,以16SDNA序列比对结果为准,如无法区分,依次参考hsp65和rpoB序列比对结果确定MPT64抗原阴性分枝杆菌菌种。菌种信息确定后,以首次分离的NTM菌种作为自建数据库用菌株,其余均为待检菌株。

表1 hsp65、rpoB和16S DNA的引物序列
1.3.3质谱标本的前处理 质谱前处理方法依据文献[9-10]和厂家建议对甲酸提取法进行改良。具体操作:取适量新鲜菌落转移至含150 μL蒸馏水的1.5 mL离心管中,涡旋震荡形成均匀的混悬液,95 ℃ 30 min热灭活处理,待冷却后加入450 μL无水乙醇,涡旋震荡2 min,14 000 r/min离心菌体细胞2 min,重复1次,弃上清液,37 ℃干燥5 min待乙醇完全挥发,加入20 μL乙腈和适量0.5 mm直径的玻璃珠,涡旋震荡2 min,加入20 μL 70%甲酸溶液,涡旋震荡2 min,14 000 r/min离心微生物提取物2 min,将1 μL提取物移取到靶板上晾干,覆盖1 μL IVD HCCA基质溶液并晾干。
1.3.4MALDI-TOF MS自建数据库数据采集和建立 使用FlexControl 3.4软件采集图谱,软件设置:采用线性正离子模式,激光频率60 Hz,加速电压为20.00 kV,延迟提取电压18.10 kV,聚焦电压6.00 kV,延迟时间为150 ns,激光功率37.0%。图谱采集前使用IVD BTS手动校准仪器, 图谱采集时手动采集数据,确保指纹图谱质量,信号强度>10 000。建库的NTM,每株需制备8个靶位,每个靶位采集3张蛋白质图谱,计24张图谱,使用Biotyper软件Show Normalized Spectra in GelView模块评估图谱之间重复性,除去低质量的图谱,使用Creat MSP模块创建该菌种的预测图谱,其他NTM自建数据库用菌株同等要求建立各自预测图谱,构建自建数据库。
1.3.5自建数据库的重复性和准确性验证 重复性验证:选取菌种信息确认的4株脓肿分枝杆菌、3株堪萨斯分枝杆菌和3株偶发分枝杆菌(来源:9株临床分离株,1株室间质评菌株)进行固体培养,出现肉眼可见菌落时进行首次检测,每天3次,连续3 d,比较质谱结果符合率;准确性验证:选取菌种信息确认的脓肿分枝杆菌、胞内分枝杆菌和堪萨斯分枝杆菌各10株(来源:临床分离株)进行固体培养,当出现肉眼可见菌落时进行检测,共30个检测结果,比较质谱结果与分子生物学测序结果之间的符合率。
1.3.6MALDI-TOF MS鉴定 使用Biotyper OC 3.1软件调入待检NTM图谱,以“自建数据库+MBT DB5989数据库”鉴定待检NTM作实验组,以“MBT DB5989数据库”鉴定待检NTM作对照组,记录MALDI-TOF MS第一位鉴定结果及得分,鉴定结果得分≥1.7为可信,得分<1.7为不可信。以分子测序结果为金标准,分析实验组与对照组对NTM的鉴定结果符合率。
1.4统计学分析 采用DNASTAR SeqMan软件分析序列峰图,DNASTAR SeqBuilder软件编辑序列,MEGA-X软件绘制并构建进化树聚类分析。采用SPSS 20.0软件处理统计数据,计数资料以百分比表示,对照组与实验组鉴定效能比较使用配对χ2检验,以P<0.05为差异有统计学意义。
2 结果
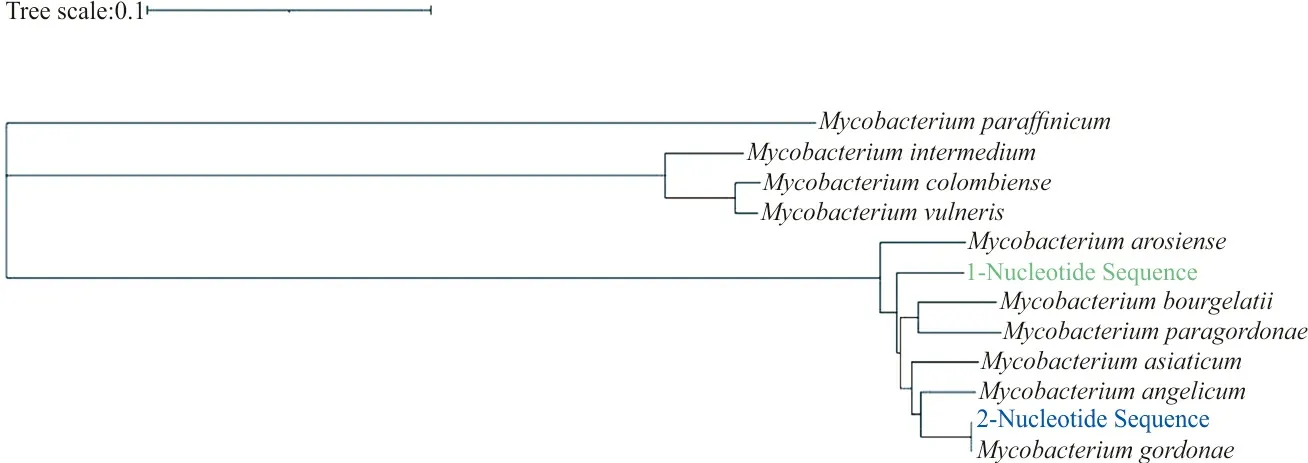
2.1分子生物学鉴定结果 应用hsp65、rpoB和16SDNA3对同源序列比对鉴定,73株MPT64抗原检测阴性分枝杆菌均能鉴定到种水平,其中72株为NTM,1株为结核分枝杆菌。72株NTM包括:胞内分枝杆菌21株、脓肿分枝杆菌17株、堪萨斯分枝杆菌13株、鸟分枝杆菌4株、偶发分枝杆菌4株、缓黄分枝杆菌3株、哥伦比亚分枝杆菌3株、戈登分枝杆菌3株、类戈登分枝杆菌2株和海分枝杆菌2株。10株NTM自建数据库用菌株同源序列比对结果见表2。自建库用类戈登分枝杆菌16SDNA、hsp65和rpoB基因Blast鉴定结果序列匹配率依次为99.08%、98.74%和99.39%,利用MAGE-X软件基于16SDNA、hsp65和rpoB基因进行序列比对计算遗传距离(p-distance)并构建进化树聚类分析,见图1、图2、图3。

表2 10株自建数据库用菌株的菌种信息

注:1-Nucleotide Sequence,戈登分枝杆菌自建库菌株;2-Nucleotide Sequence,类戈登分枝杆菌自建库菌株。

注:1-Nucleotide Sequence,戈登分枝杆菌自建库菌株;2-Nucleotide Sequence,类戈登分枝杆菌自建库菌株。
注:1-Nucleotide Sequence,戈登分枝杆菌自建库菌株;2-Nucleotide Sequence,类戈登分枝杆菌自建库菌株。
2.2NTM自建数据库的验证 自建数据库重复性与准确性验证符合率均为100%。
2.3MALDI-TOF MS鉴定结果 实验组和对照组分别对62株待检NTM进行鉴定,以分子生物学测序结果为金标准,对照组NTM鉴定率为51.61%(32/62),实验组NTM鉴定率为95.16%(59/62),差异有统计学意义(χ2=30.102,P<0.001);剔除MBT DB5989数据库中缺失的分枝杆菌后,对照组NTM鉴定率为86.48%(32/37),实验组NTM鉴定率为97.30%(36/37),差异无统计学意义(χ2=2.902,P=0.088),见表3。实验组鉴定结果中1株类戈登分枝杆菌被错误鉴定为戈登分枝杆菌,得分1.713,戈登与类戈登分枝杆菌自建库菌株MALDI-TOF MS蛋白质指纹图谱见图4。

图4 MALDI-TOF MS戈登与类戈登分枝杆菌自建库菌株蛋白质指纹图谱

表3 MALDI-TOF MS对62株待检NTM的鉴定效能
3 讨论
近年来,NTM病呈快速增多趋势,NTM肺病感染症状可与肺结核病相似,鉴别诊断困难,且NTM对很多一线抗结核药物天然耐药,并有研究发现某些特定菌种对药物的耐药机制与特定耐药基因有关[11-12]。因此,快速准确的鉴定NTM,对NTM感染治疗和结核病防治至关重要。
MALDI-TOF MS通过微生物中提取的肽和蛋白质成分获取指纹图谱,使用评分算法匹配参考图谱实现微生物的鉴定,已迅速取代传统生化和表型分析鉴定方法[13]。分枝杆菌细胞壁组成成分复杂且稳定,胞内核糖体蛋白质难以释放,指纹图谱难以获取,易导致分枝杆菌鉴定失败。95 ℃ 30 min热处理有助细菌灭活和增加细胞壁通透性,0.5 mm直径的玻璃珠漩涡震荡或研磨有助于细菌物理破壁释放蛋白质[9-10]。研究表明,使用改良甲酸提取法处理分枝杆菌,可提升Bruker质谱商品数据库对分枝杆菌的鉴定率和准确性,但在Vitek质谱中却并不适用[14]。
利用MALDI-TOF MS自建数据库功能可对数据库进行有效完善,从而提升鉴定准确率,已有报道表明自建数据库可提升对酵母菌和丝状真菌的鉴定能力[15-16]。本研究建立的自建数据库包含10种NTM预测图谱信息,实验组对62株待检NTM的鉴定率较对照组由51.61%提升至95.16%;剔除MBT DB5989数据库欠缺的分枝杆菌后,实验组NTM鉴定率较对照组由86.48%提升至97.30%,表明自建数据库辅助MBT DB5989数据库可显著提升对NTM的鉴定效能,主要体现在MBT DB5989数据库缺失的分枝杆菌上。综上,自建数据库可丰富MBT DB5989数据库信息,使其对数据库外的分枝杆菌具备鉴定能力,提升MALDI-TOF MS对NTM的鉴定效能。MALDI-TOF MS自建数据库具有共享功能,可为不同终端提供互助支持,使其临床应用价值更为突出。
类戈登分枝杆菌由Kim等[17]基于hsp65基因序列测定鉴定病原菌发现并命名为类戈登分枝杆菌49061型,并证明16SDNA、hsp65和rpoB基因序列的独特性。类戈登分枝杆菌49061型与戈登分枝杆菌ATCC1447016SDNA基因序列相似度为99.04%,构建的16SDNA进化树聚类分析表明两者亲缘关系密切,可见类戈登分枝杆菌16SDNA基因序列虽具有独特性,但与戈登分枝杆菌16SDNA基因序列高度相似,无法作为鉴别两者的特征性序列;类戈登分枝杆菌与戈登分枝杆菌hsp65基因序列相似度为95.10%,BLAST比对鉴定序列匹配率为98.74%,构建的hsp65进化树聚类分析显示两者虽具有相近亲缘关系,但可以对两者进行鉴别,可见hsp65基因可作为鉴定类戈登分枝杆菌的特征性序列;类戈登分枝杆菌与戈登分枝杆菌rpoB基因序列相似度为97.39%,BLAST比对鉴定序列匹配率为99.39%,构建的rpoB进化树聚类分析显示多种NTM亲缘关系密切,无法作为鉴别两者的特征性序列。综上,16SDNA和rpoB基因序列测定无法鉴别菌种,仅hsp65基因序列测定可对类戈登分枝杆菌进行准确鉴定。
MBT DB5989数据库缺失类戈登分枝杆菌预测图谱,戈登分枝杆菌与类戈登分枝杆菌蛋白质指纹图谱显示,位置3 766、5 653、6 352、7 243和10 619 m/z附近存在相似高强度特征性波峰,两者蛋白质指纹图谱相似,是导致此次鉴定错误的原因,这与杨本善等[18]研究使用Bruker Mycobacteria Library 3.0数据库鉴定类戈登分枝杆菌的结果相一致。Bruker Mycobacteria Library 5.0数据库在3.0版基础上丰富了类戈登分枝杆菌预测图谱,委托布鲁克(北京)科技有限公司上海分公司采用Bruker Mycobacteria Library 5.0数据库对待检类戈登分枝杆菌进行鉴定,结果正确,得分2.115。可见MALDI-TOF MS数据库的选择直接影响NTM鉴定结果。
本研究存在一定的局限性。本研究中MALDI-TOF MS自建数据库用菌株使用的是临床分离株,虽经分子生物学测序,但非标准模式菌株,其生化特征和蛋白质表达的稳定性无法保证;收集的NTM的种类和数量较少,无法覆盖所有NTM临床分离株,对本文未涉及和数量较少的NTM菌株,则需要大范围、多中心的合作研究以丰富MALDI-TOF MS自建数据库对NTM的鉴定能力评价。
综上,MALDI-TOF MS数据库选择直接影响NTM鉴定结果,MBT DB5989数据库对NTM鉴定能力尚有不足,自建数据库可辅助MBT DB5989数据库显著提升对NTM的鉴定效能,这项工作也可为MALDI-TOF MS鉴定数据库外其他微生物提供借鉴。

