中国汉族人群66个InDel基因座的遗传多态性
王雪倩,张庆珍,程鹏,董婷婷,李卫国,周喆,王升启
中国汉族人群66个InDel基因座的遗传多态性
王雪倩1,2,张庆珍2,程鹏2,董婷婷2,李卫国1,周喆2,王升启2
1. 河南师范大学生命科学学院,新乡 453007 2. 军事科学院军事医学研究院辐射医学研究所,北京 100850
插入/缺失多态性(insertion/deletion polymorphism, InDel)遗传标记是一类由DNA片段的插入或者缺失形成的变异形式,在降解检材身份识别领域展现出优势。本文从dbSNP数据库中自主筛选66个InDel基因座,基于高通量测序系统建立了一套多重复合扩增体系(66-plex InDels)。通过评估66-plex InDels在251名中国汉族人群的群体遗传学参数,分析其法医学应用价值。研究结果表明:经过Bonferroni法校正,66个InDel基因座在中国汉族人群中均符合Hardy-Weinberg平衡定律(>0.000 758),各基因座间处于连锁平衡状态。各基因座平均观测杂合度(Ho)为0.482,平均期望杂合度(He)为0.483,平均个体识别力(DP)为0.612,平均多态性信息含量(PIC)为0.365,累计个体识别率(TDP)为0.999 999 999 999 999 999 999 999 999 428 18。66个InDel基因座的二联体累计非父排除概率(CPEduo)为0.999 739,三联体累计非父排除概率(CPEtrio)为0.999 999 999 417。结果表明:66个InDel基因座在中国汉族人群中具有良好的遗传多态性,可独立应用于法医个体识别和亲权鉴定研究。
插入/缺失多态性;中国汉族人群;个体识别;亲权鉴定
短串联重复序列(short tandem repeat, STR)因灵敏度高、鉴别能力强的特点,已广泛应用于个体识别和亲权鉴定领域[1]。但STR突变率相对较高,约为10–3,在亲权鉴定中可能存在误判[2];且片段较长(100~500 bp),在降解检材中检测成功率较低。插入/缺失多态性(InDel)作为新型遗传标记,在人类基因组中分布广泛;突变率明显低于STR,约为10–8;扩增子较短(<200 bp),更适用于高度降解样本的检测;其二态结构更易于分型和检测[3~5]。
目前已有一些研究针对不同国家、不同群体设计了InDel遗传标记多重扩增体系,通过评估基因座的群体遗传学相关参数,为个体识别和亲权鉴定提供了基础数据。国内外应用比较广泛的InDel商品化分型试剂盒包括Investigator®DIPplex试剂盒(德国Qiagen公司)和AGCU InDel 50试剂盒(无锡中德美联生物技术有限公司)。Investigator®DIPplex试剂盒以德国人群基因频率作为主要筛选依据,对中国人群的鉴定效力较低[6]。30个InDel在北京汉族群体中累积个体识别率(total discrimination power, TDP)为0.999 999 999 985[7],在贵州仡佬族和苗族人群中的TDP分别为0.999 999 999 34和0.999 999 996 86[8],但在西班牙人群中TDP高达0.999 999 999 999[9]。AGCU InDel 50试剂盒是我国研制的商品化分型试剂盒,包含47个常染色体InDel基因座,2个Y染色体InDel基因座和1个性别鉴定基因座(Amelogenin)[10]。47个常染色体InDel基因座在汉族人群中的累积个体识别率(TDP)和累计非父排除概率(cumulative probability of exclusion, CPE)分别为0.999 999 999 999 999和0.9997[11]。虽然47个常染色体InDel基因座鉴定效力大于常规STR体系的TDP[12](0.999 999 999 98),能够满足个体识别需求,但CPEtrio<0.9999[13],亲权鉴定效力较低,有待进一步提高。
联合检测更多的InDel基因座可以提高体系的鉴定效力,独立完成个体识别和亲权鉴定研究。本文从dbSNP数据库中自主筛选66个常染色体InDel基因座和一个Amelogenin性别基因座,基于高通量测序系统自主构建了一个66 InDel + Amel的多重复合扩增体系(66-plex InDels)。通过251个中国汉族无关个体样本的DNA分型检测结果,分析66个InDel在中国汉族人群中的群体遗传学参数进行分析,为个体识别和亲权鉴定提供基础数据。
1 材料与方法
1.1 InDel筛选流程
本研究基于以人类GRCh37.hg19作为参考基因组的dbSNP数据库(https://www.ncbi.nlm.nih.gov/snp/)数据集,采用VCFtools[14]和plink软件[15]筛选出在东亚人群中有较高鉴定效力的InDel基因座。具体筛选标准如下:(1)位于常染色体内含子区域,全局次要等位基因频率(global minor allele frequency, GMAF)在0.4~0.5之间的二元多态性插入/缺失序列,得到17,374个基因座;(2)满足哈德温伯格平衡(Hardy-Weinberg equilibrium, HWE)定律,HWE>0.05,杂合度>0.4,得到1263个基因座;(3)符合连锁平衡检验(LE),2<0.01,得到987个基因座;(4)不同群体间(东亚、南亚、欧洲、美洲、非洲)群体分化指数()<0.06,得到549个基因座;(5)InDel基因座片段长度>1 bp,得到172个基因座;(6)同一染色体上,InDel基因座之间物理距离≥10 MB,得到93个基因座。将上述筛选标准得到的93个基因座采用Primer Premier 5.0和MFEprimer软件[16]进行多重引物设计,考虑到引物特异性、引物长度、退火温度和引物结构设计等因素,以确保每对引物能够均衡地扩增,最终确定66个InDel基因座用于复合扩增。66个InDel基因座未见与医疗敏感信息相关的文献报道。
1.2 样本收集和DNA提取
根据知情同意原则,采集100名中国汉族无关个体外周血样和151名中国汉族无关个体外周血液制成的FTA血卡,包括河南汉族40例、北京汉族28例、内蒙汉族21例、山东汉族21例、云南汉族20例、广东汉族20例、重庆汉族19例、山西汉族18例、江西汉族18例、湖南汉族16例、浙江汉族16例、安徽汉族14例。外周血样均采用QIAamp®DNA Investigator试剂盒(德国Qiagen公司)提取DNA。采用Qubit 3.0荧光定量仪(美国Thermo Fisher公司)进行DNA定量,–20℃保存备用。151名无关个体外周血液制成FTA血卡后,通风干燥处保存备用。本研究已通过军事医学科学院军事医学研究院伦理委员会批准(No. AF/SC-08/02.101)。
1.3 PCR扩增和文库制备
采用MultiSeq®Custom Panel (IGMU258V1)(嘉兴艾吉泰康生物科技有限公司)进行PCR多重复合扩增(具体引物信息见附表1),总扩增体系为30 μL,其中Enhancer buffer NB (1N) 3.5 μL,Enhance buffer M 2.5 μL,Primer 5 μL,IGT-EM808 polymerase mixture 10 μL,模板DNA 5 μL (1 ng/μL)或直径1 mm的FTA血卡样本DNA,去离子水补至30 μL。实验过程中,采用DNA女性标准品9947A和DNA男性标准品9948作为阳性对照,去离子水作为阴性对照。使用ProFlex™PCR扩增仪(美国Applied Biosystems公司)进行扩增,PCR扩增条件:95℃ 3 min 30 s;98℃20 s,60℃ 4 min,18个循环;最后72℃ 10 min,4℃保存。
PCR产物通过AMPure XP磁珠(美国Beckman Coulter公司)纯化,加入PCR产物13.5 μL,Index-i5 1 μL,Index-i7 1 μL,Enhance buffer M 2.5 μL,去离子水2 μL。通过ProFlex™PCR扩增仪(美国Applied Biosystems公司)进行扩增,PCR扩增条件:95℃ 3 min 30 s;98℃ 20 s,58℃ 1 min,72℃ 30 s,9个循环;最后72℃ 5 min,4℃保存。
1.4 高通量测序和InDel分型
样本文库使用Agilent 2100生物分析仪(美国Agilent公司)和7500实时荧光定量PCR (美国Thermo Fisher公司)进行质控和定量,随后制备10 pM DNA混合文库,采用MiSeq v2试剂盒(2×150循环) (美国Illumina公司)在MiSeq高通量测序仪(美国Illumina公司)进行高通量测序。采用CLC Genomics Workbench 20.0(德国Qiagen公司)对66个InDel基因座和Amel性别基因座进行分型分析。首先通过QC for sequencing Reads和Trim Reads功能对fastq序列进行质控,包括原始数据的质量评估及data trimming (去除原始测序reads中质量分数<30的低质量reads和接头序列)。根据扩增区域配置的bed文件,导入“Structural Variant Caller”模块;根据InDel位置及变异信息配置vcf文件,导入“Identify Known Mutations from mappings”模块。根据公安部发布的公共安全行业标准GA/T 1693- 2020《法庭科学DNA二代测序检测规范》,将“Identify Known Mutations from mappings”参数Minimum coverage设置为100、Detection frequency为软件默认值20%。由此得到每个基因座的分型结果。
1.5 统计分析
采用GenAIEx 6.5[17]软件计算66个InDel基因座的等位基因频率(allele frequency, AF)、观测杂合度(observed heterozygosity, Ho)、期望杂合度(expected heterozygosity, He),对各基因座进行Hardy- Weinberg平衡和基因座间的连锁不平衡检验,并计算典型父权指数(typical paternity index, TPI)。采用Modifie-PowerStates软件包[18]计算个体识别率(discrimination power, DP)、累计个体识别率(TDP)、多态性信息含量(polymorphism information content, PIC)。采用Cervus 3.0软件(http://www.fieldgenetics. com/pages/download.jsp)计算二联体累计非父排除概率(cumulative power of exclusion in duos, CPEduo)和三联体累计非父排除概率(cumulative power of exclusion in trios, CPEtrio)。
2 结果与分析
2.1 66-plex InDels基因座遗传信息
IGMU试剂盒对251份中国汉族无关个体的样本均可进行有效扩增,经CLC Genomics Workbench 20.0软件分析,所有样本质量分数Q30均大于95%,扩增子平均测序深度为1550X±1200,其多重PCR产物片段主要分布范围在150~180 bp,Agilent 2100生物分析仪质控无非特异性扩增。具体InDel基因座遗传信息及阳性标准品(9947A、9948)分型见表1。251个中国汉族人群的基因型信息见附表2。

表1 66个InDel基因座遗传信息及阳性标准品分型
1) rs#为InDel基因座在dbSNP数据库的ID号;2) InDel基因座在人类基因组数据库(GRCh37.p19)染色体上的物理位置;3)参考基因(Ref)在本体系中命名为1,突变基因(Alt)在本体系中命名为2。
2.2 Hardy-Weinberg平衡及连锁不平衡检验
对66个基因座进行Hardy-Weinberg平衡检验,除rs146109101、rs3061475、rs2307807三个基因座外,其他63个基因座基因型分布均符合Hardy- Weinberg平衡(>0.05),具体值见表2。经Bonferroni法矫正,将值设为0.000 758 (0.05/66),66个InDel基因座值均大于0.000 758,符合Hardy- Weinberg平衡。将66个基因座两两组合进行连锁不平衡检验,共产生2145 (2145 = [×(–1)]/2,为研究基因座的数目[4])个组合,具体基因座间连锁不平衡值见附表3。经Bonferroni矫正,=0.000 023 3 (0.05/2145),66个基因座均未发现配对连锁现象。因此,可以进行TDP、CPEduo、CPEtrio的计算。
2.3 66-plex InDels在中国汉族人群中群体遗传学参数调研
经GenAIEx 6.5软件统计分析,除rs79444593基因座的次要等位基因频率(minor allele frequency, MAF)低于0.1之外,其余65个InDel基因座的次要等位基因频率分布在0.28~0.5之间,平均值为0.436,表明其在中国汉族人群中等位基因频率分布比较均衡,66个InDel基因座等位基因频率分布见图1。经统计发现,Ho、He、DP、PIC、TPI最小值均在rs79444593基因座出现。除rs79444593外,其余65个基因座观测杂合度(Ho)为0.390~0.562,期望杂合度(He)为0.409~0.500,两者比较相差较小;个体识别率(DP)为0.562~0.653,DP越高,代表该遗传标记在识别不同个体方面的效能越强;多态性信息含量(PIC)为0.325~0.375,PIC越高,遗传标记的信息量越多;累计父权指数(TPI)为0.820~1.141。累计个体识别概率(TDP)为0.999 999 999 999 999 999 999 999 999 428 18。具体基因座参数见表2。除rs79444593基因座外,其他基因座在中国汉族人群中的多态性较高,66-plex InDels适用于中国汉族人群的个体识别研究。
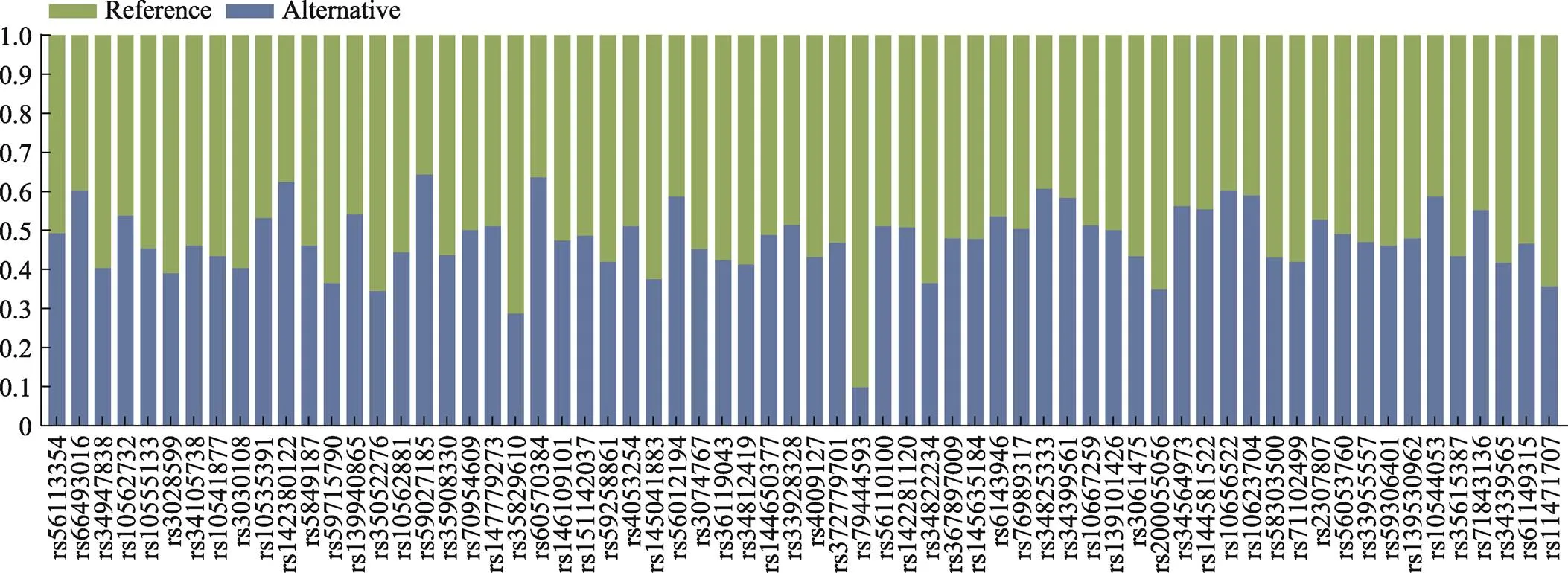
图1 66个InDel基因座等位基因频率分布
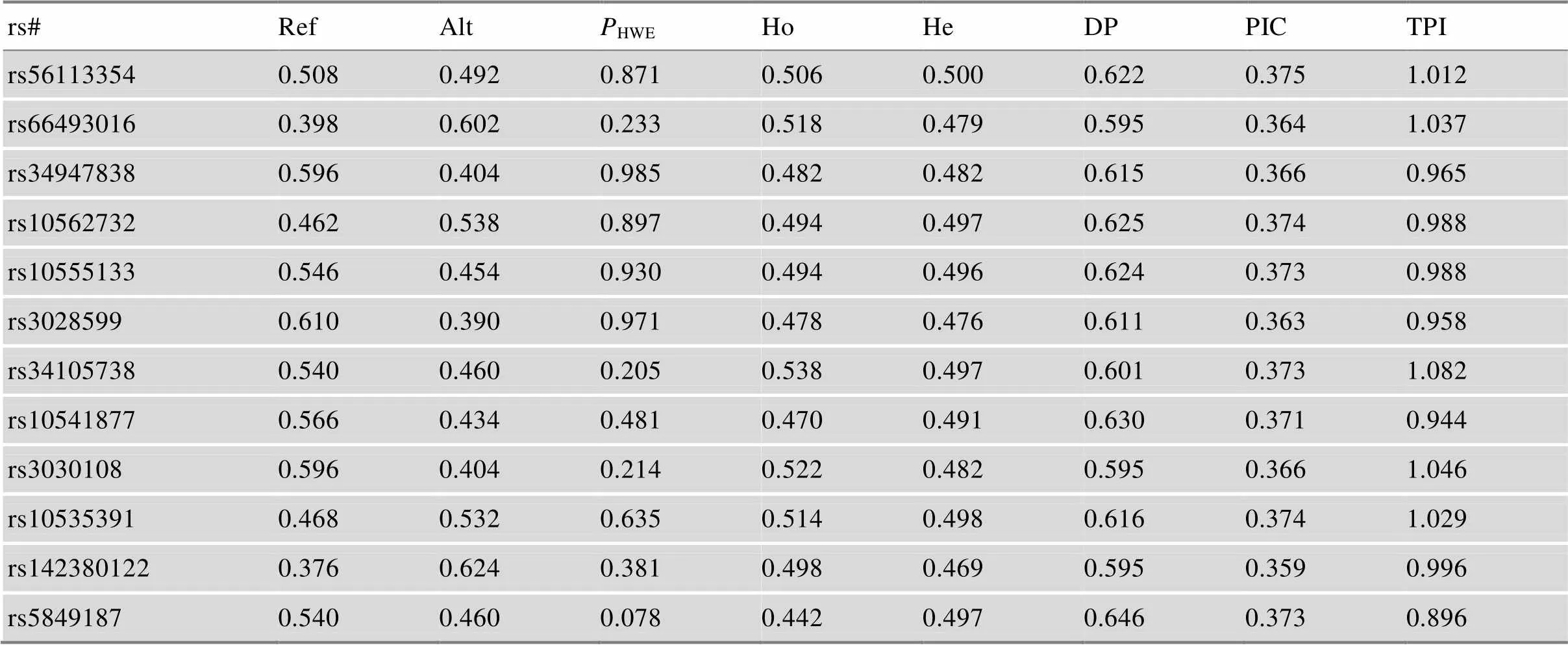
表2 66个InDel基因座法医遗传学参数(n=251)

续表
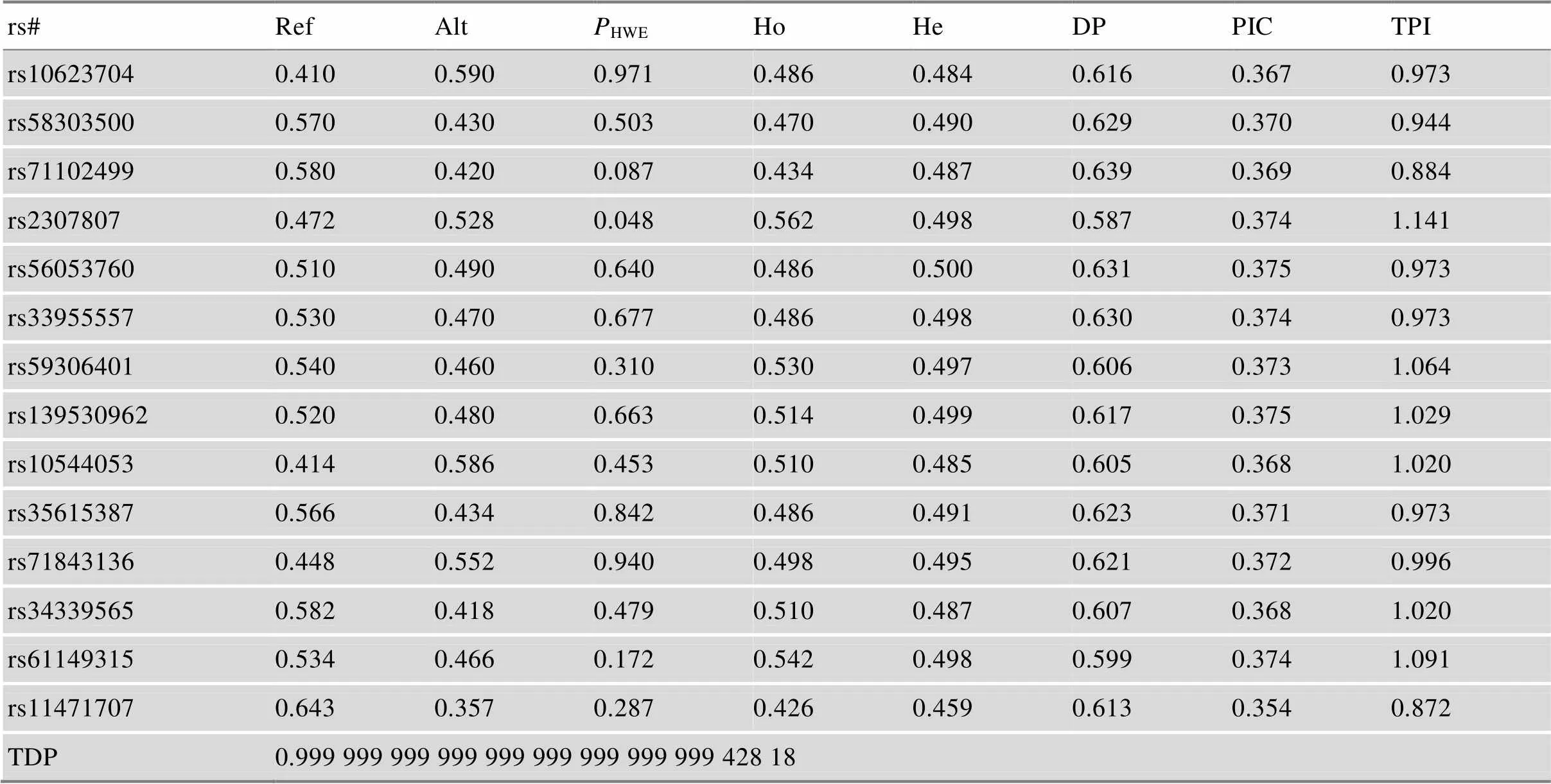
续表
2.4 66-plex InDels在个体识别研究中的应用
Investigator®DIPplex试剂盒和AGCU InDel 50试剂盒作为常用的商品化InDel 分型体系,在个体识别和亲权鉴定领域应用比较广泛。将66-plex InDels与Investigator®DIPplex、AGCU InDel 50以及常用STR商品化试剂盒(DNA Typer 21™)的鉴定效力进行比较。Investigator®DIPplex、AGCU InDel 50、DNA Typer 21™在中国汉族人群的累计个体识别效率(TDP)分别为0.999 999 973 69[19]、0.999 999 999 999 999 999 8[2]和0.999 999 999 999 999 999 999 998 85[20]。AGCU InDel 50体系的累计个体识别率高于常用STR体系TDP (0.999 999 999 98[12]),表明该体系可以单独应用于个体识别研究。本研究66-plex InDels的累计个体识别率(TDP)为0.999 999 999 999 999 999 999 999 999 428 18,远高于Investigator®DIPplex、AGCU InDel 50及DNA Typer 21™试剂盒,表明66-plex InDels识别不同个体的效力更强。
2.5 66-plex InDels在亲权鉴定案件中的应用
对于亲权鉴定的案件分析,采用Investigator®DIPplex 体系,中国江苏汉族的二联体累计非父排除概率(CPEduo)为0.998 933 978,三联体累计非父排除概率(CPEtrio)为0.997 806 392[6]。AGCU InDel 50试剂盒在广州汉族中三联体累计非父排除概率(CPEtrio)为0.999 732 16[2]。两个商品化试剂盒三联体累计非父排除概率(CPEtrio)均小于0.9999[21],对于二联体案件鉴定效力更低,不能单独适用于亲权鉴定案件。如表3所示,66-plex InDels二联体非父排除概率(PEduo)为0.016~0.125,三联体非父排除概率(PEtrio)为0.142~ 0.281,在进行亲权鉴定分析时,基因座非父排除概率越高,排除非亲生父亲的效能越高。经Cervus软件分析,251个无关个体样本在66-plex InDels中的CPEduo为0.999 739,CPEtrio为0.999 999 999 417。66-plex InDels的CPEtrio比20个STR基因座(CPEtrio= 0.999 999 996[20])鉴定效力更强,表明66-plex InDels可单独应用于三联体亲权鉴定案件的分析,但对于二联体案件系统效能未达到法医亲权鉴定的要求,需要其他商品化试剂盒联合使用。四个体系在中国汉族人群中鉴定效能对比见表4。
3 讨论
STR是目前法医物证学身份鉴定领域最常用的多态性遗传标记,其复合扩增体系的检测长度一般分布在100~500 bp之间,不适合检测高度降解和低拷贝的样本[22]。InDel作为二等位基因遗传标记,遗传标记扩增片段均小于200 bp,适合降解检材的分型[23]。本研究自主构建基于Illumina测序平台的多重复合扩增体系(66-plex InDels),包含66个常染色体InDel基因座和一个Amel性别基因座。
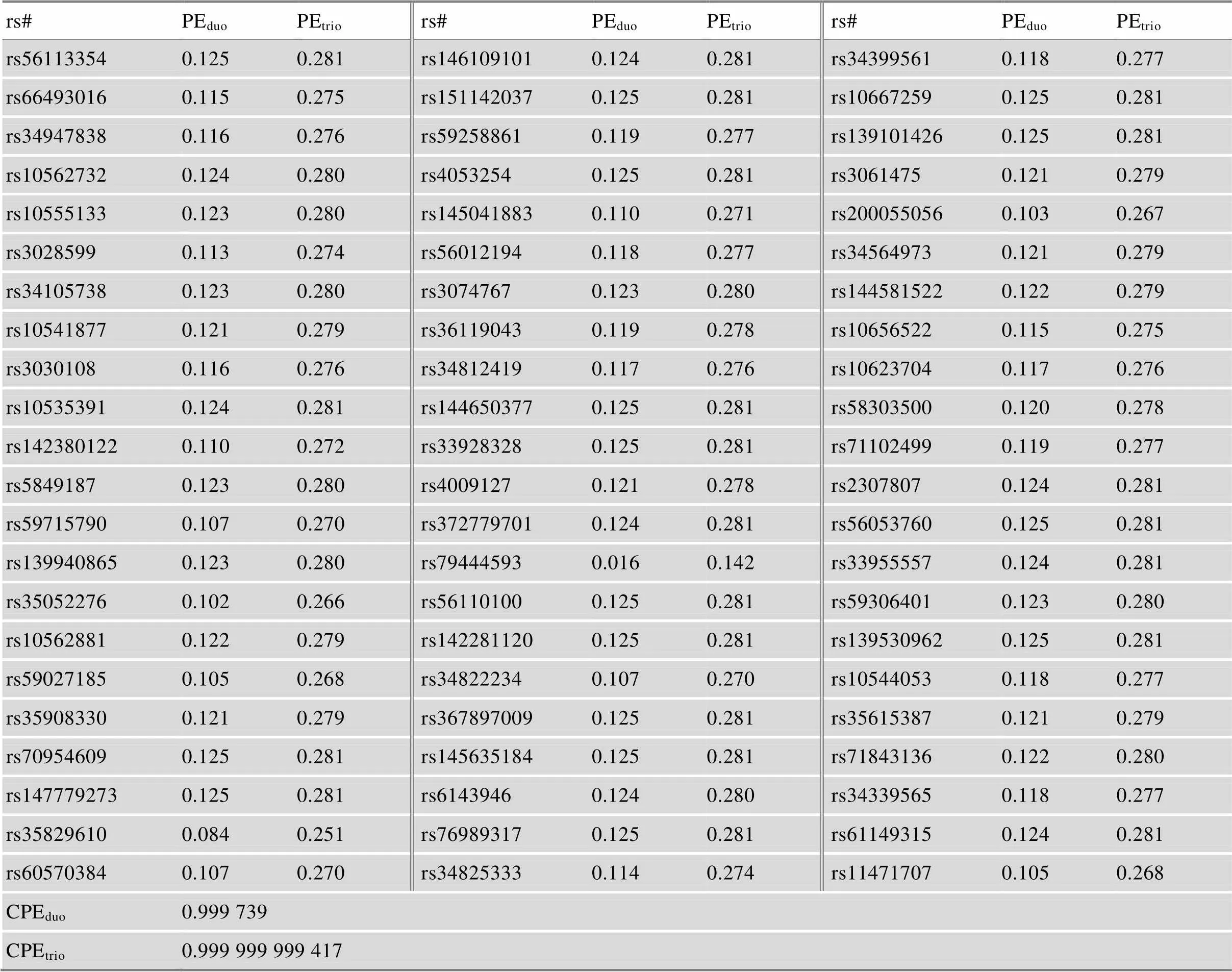
表3 66个InDel基因座二联体非父排除概率及三联体非父排除概率(n=251)

表4 不同体系鉴定效能比较
通过对中国汉族251个无关个体进行遗传多态性调查,评估66-plex InDels体系的遗传学参数。结果表明,66个InDel基因座分布在18条染色体上,在中国汉族人群中,其平均次要等位基因频率为0.431;平均观测杂合度为0.482;平均期望杂合度为0.483;平均匹配概率为0.388;平均个体识别率为0.612;平均多态性信息含量为0.365;平均累计父权指数为0.974。所有基因座经过Bonferroni校正后,均符合Hardy-Weinberg平衡定律(>0.000 758)且两两之间不存在连锁不平衡现象(>0.000 023 3)。其中,rs79444593基因座在国际千人基因组计划东亚人群中的MAF为0.4385,但该基因座在本研究中国汉族人群中MAF为0.0976,表明该位点在中国汉族人群中遗传多态性较低、鉴定效力较差。考虑到随机抽样也可能导致误差,在后续的实验中,可以通过增加样本量来尽量消除随机抽样误差。
前期研究表明约60个InDel基因座的个体识别鉴定效力可以等同于目前法医鉴定中常用的商品化STR试剂盒(如Sinofiler试剂盒包含15个STR基因座)[24]。DP是法医个体识别鉴定的重要指标,表示两个随机个体表型不同的概率。DP越大,代表个体识别效力越强。对66-plex InDels进行统计分析,其累计个体识别概率(TDP)为0.999 999 999 999 999 999 999 999 999 428 18,累计非父排除概率(CPE)为0.999 999 999 417。TDP高于DNA Typer 21™试剂盒中20个常染色体STR鉴定效力,同时CPE> 0.9999,因此,66-plex InDels可以单独应用于个体识别和亲权鉴定,也可以作为已有商品化试剂盒的补充基因座,进行复杂亲缘关系鉴定。
本研究不足之处在于个别基因座(rs79444593)在中国汉族人群中遗传多态性较差,后续多重复合扩增体系的优化中,可以将该位点进行替换,也可增加更多InDel基因座来提高66-plex InDels的系统效能。同时,后续研究可以分析中国人群其他种族的遗传多态性,评价66-plex InDels在中国不同群体中的法医学应用价值,进行群体间的遗传学参数的均衡性研究,探索66-plex InDels在中国人群中的普适性。
本研究获得了66个常染色体InDel基因座多重复合扩增体系,通过对66-plex InDels在中国汉族人群中的法医群体遗传学分析,为InDel基因座在中国人群中的个体识别和亲权鉴定研究提供了遗传背景数据。
附录:
附加材料见文章电子版www.chinagene.cn。
[1] Song F, Lang M, Li LY, Luo HB, Hou YP. Forensic features and genetic background exploration of a new 47-autosomal InDel panel in five representative Han populations residing in Northern China., 2020, 8(5): e1224.
[2] Pan XY, Liu CH, Du WA, Chen L, Han XL, Yang XY, Li Y, Liu C. Genetic polymorphism and forensic application of 47 autosomal InDel loci in 5 Chinese ethnic groups., 2020, 36(4): 531–537.
潘习勇, 刘长晖, 杜蔚安, 陈玲, 韩晓龙, 杨幸怡, 李越, 刘超. 47个常染色体InDel位点在中国5个民族中的遗传多态性及法医学应用. 法医学杂志, 2020, 36(4): 531– 537.
[3] Zhang XR, Shen CM, Jin XY, Guo YX, Xie T, Zhu BF. Developmental validations of a self-developed 39 AIM- InDel panel and its forensic efficiency evaluations in the Shaanxi Han population., 2021, 135(4): 1359–1367.
[4] Jin XY, Wei YY, He YF, Guo YX, Mei T, Meng HT, Zhang YD, Kong TT, Zhu BF. Genetic polymorphisms of 30 InDel loci in Ewenki ethnic group from Inner Mongolia.2017, 33(3): 271–276+280.
靳小业, 魏媛媛, 贺永锋, 郭瑜鑫, 梅婷, 孟昊天, 张玉党, 孔婷婷, 朱波峰. 内蒙古鄂温克族人群30个 InDel位点遗传多态性. 法医学杂志, 2017, 33(3): 271–276+ 280.
[5] Tozzo P, Delicati A, Frigo AC, Caenazzo L. Comparison of the allelic alterations between InDel and STR markers in tumoral tissues used for forensic purposes., 2021, 57(3): 226.
[6] Hong L, Wang XG, Liu SJ, Zhang YM, Ou XL, Chen Y, Chen WH, Sun HY. Genetic polymorphisms of 30 InDel loci in Guangdong Han population., 2013, 34(2): 299–304.
洪丽, 王小广, 刘素娟, 张胤鸣, 欧雪玲, 陈勇, 陈维红, 孙宏钰. 30个插入/缺失多态性位点在中国广东汉族人群中的遗传多态性. 中山大学学报(医学科学版), 2013, 34(2): 299–304.
[7] Bai RF, Jiang LZ, Zhang Z, Shi MS. Genetic polymorphism and forensic application of 30 InDel loci of Han population in Beijin., 2013, 35(12): 1368–1376.
百茹峰, 姜立喆, 张中, 石美森. 北京汉族群体30个常染色体InDel位点群体遗传学及法医学研究.遗传, 2013, 35(12): 1368–1376.
[8] Liu YJ, Hu M, Shi MS, Li XB. Analysis on the genetic polymorphisms of 30 InDel loci in Gelao and Miao ethnic population in Guizhou., 2021, 18(15): 2162–2168+2172.
刘亚举, 胡萌, 石美森, 李学博. 贵州仡佬族和苗族人群30个插入/缺失位点遗传多态性分析. 检验医学与临床, 2021, 18(15): 2162–2168+2172.
[9] Haidar M, Alsaleh H, Haddrill PR. Population genetics of 30 insertion/deletion polymorphisms in the Kuwaiti population., 2020, 134(3): 985–986.
[10] Wang MG, He GL, Gao S, Jia FQ, Zou X, Liu J, Wang SY, Ye ZW, Hou YP, Wang Z. Molecular genetic survey and forensic characterization of Chinese Mongolians via the 47 autosomal insertion/deletion marker., 2021, 113(4): 2199–2210.
[11] Chen L, Du W, Wu WB, Yu A, Pan XY, Feng PP, Feng CL, Li C, Xu LD, Liu CH, Liu C. Developmental validation of a novel six-dye typing system with 47 A-InDels and 2 Y-InDels., 2019, 40: 64–73.
[12] Wang YL, Song ZX, Gu J, Bai HR, Zhou YY, Zhang JY, Yang Y, Shi WY, Chen LQ. Forensic genetic study and application of 30 autosomal InDel loci in Chinese Mongolia population residing in Chifeng., 2019, 23(05): 359–366.
王亚丽, 宋振祥, 顾捷, 白慧茹, 周圆圆, 张佳怡, 杨越, 史唯一, 陈丽琴. 内蒙古赤峰地区蒙古族人群30个常染色体InDel的遗传多态性. 生命科学研究, 2019, 23(05): 359–366.
[13] Wang MY, Yu H. Analysis of genetic polymorphism of 19 short tandem repeat loci in Henan Han population., 2021, 30(22): 4067–4071.
王美莹, 于皓. 19个短串联重复序列基因座在河南汉族人群中的遗传多态性分析. 河南医学研究, 2021, 30(22): 4067–4071.
[14] Danecek P, Auton A, Abecasis G, Albers CA, Banks E, DePristo MA, Handsaker RE, Lunter G, Marth GT, Sherry ST, McVean G, Durbin R. The variant call format and VCFtools., 2011, 27(15): 2156–2158.
[15] Mo SK, Ren ZL, Yang YR, Liu YC, Zhang JJ, Wu HJ, Li Z, Bo XC, Wang SQ, Yan JW, Ni M. A 472-SNP panel for pairwise kinship testing of second-degree relatives., 2018, 34: 178–185.
[16] Qu WB, Shen ZY, Zhao DS, Yang Y, Zhang CG. MFEprimer:multiple factor evaluation of the specificity of PCR primers., 2009, 25(2): 276–278.
[17] Peakall R, Smouse PE. GenAlEx 6.5:genetic analysis in Excel. Population genetic software for teaching and research- an update., 2012, 28(19): 2537–2539.
[18] Zhao F, Wu XY, Cai GQ, Xu CC. Modified-Powerstates software in forensic biostatistics., 2003, 18(5): 297–298+312.
赵方, 伍新尧, 蔡贵庆, 许传超. Modified-Powerstates软件在法医生物统计中应用. 中国法医学杂志, 2003, 18(5): 297–298+312.
[19] Pan M, Ju XB, Liu YT, Cui H, Gu M, Zhou HY. Genetic polymorphism of 30 InDel loci in Han population from Jiangsu province., 2017, 33(6): 611–614+618.
潘猛, 居晓斌, 刘燕婷, 崔鹤, 顾民, 周惠英. 江苏汉族人群30个插入/缺失位点的遗传多态性. 法医学杂志, 2017, 33(6): 611–614+618.
[20] Yang X, Mu R, Cao YW, Wang X, Li CX, Cai JC, Liu HJ, Ma WH. Genetic polymorphism of 20 STR loci in Kazakh, Hui and Han population in Shihezi region of Xinjiang., 2021, 30(4): 339–347.
杨秀, 牟睿, 曹雨薇, 王郗, 李春香, 蔡君成, 刘焕杰, 马温华. 新疆石河子地区哈萨克族、回族、汉族人群20个STR基因座的遗传多态性研究. 激光生物学报, 2021, 30(4): 339–347.
[21] Wang W, Zhao L, Jiang L, Liu J, Huang MS, Li RR, Liu JJ, Ma Q, Wang YY, Li CX. Constructing a multiplex PCR system for personal identification by the InDel polymorphism of Chinese population, 2017, 42(01): 1–8.
王玮, 赵蕾, 江丽, 刘京, 黄美莎, 李冉冉, 刘佳佳, 马泉, 王英元, 李彩霞. 用于中国人群个体识别的InDe多重PCR系统的构建. 刑事技术, 2017, 42(01): 1–8.
[22] Hou YP. Forensic genetics., 2009: 72–73.
侯一平. 法医物证学. 北京: 人民卫生出版社, 2009: 72–73.
[23] Zhao SM, Zhang SH, Li CT. InDel_typer30:A multiplex PCR system for DNA identification among five Chinese populations., 2010, 26(5): 343–348+356.
赵书民, 张素华, 李成涛. InDel_typer30:用于中国5个主要民族DNA鉴定的多重PCR系统. 法医学杂志, 2010, 26(5): 343–348+356.
[24] Ji FQ, Teng J, Lai L, Han LL, Shen XL, Pu XD. A study on genetic polymorphisms of Han population in Fujian based on Sinofiler detection system., 2013, 34(21): 2835–2836+2839.
纪凤卿, 滕菁, 赖力, 韩丽丽, 沈晓丽, 浦晓东. 基于Sinofiler检测系统的福建汉族群体遗传多态性研究. 国际检验医学杂志, 2013, 34(21): 2835–2836+2839.
Genetic polymorphisms of 66 InDel loci in the Chinese Han population
Xueqian Wang1,2, Qingzhen Zhang2, Peng Cheng2, Tingting Dong2, Weiguo Li1, Zhe Zhou2, Shengqi Wang2
Insertion/deletion polymorphism (InDel) genetic markers refer to insertion or deletion of DNA fragments into genomic DNA, which have advantages in the identification of degraded samples. In this study, we independently screened 66 highly polymorphic InDel markers from the dbSNP database to establish a multiplex PCR system for forensic DNA identification using next-generation sequencing system (66-plex InDels). We assessed the population genetic data among 251 Chinese Han population using this system and evaluated their potential forensic application. The results showed that all 66 InDel loci conformed to the Hardy-Weinberg equilibrium (>0.000 758), and all the pairwise InDel loci were in linkage equilibrium after Bonferroni correction. The mean observed heterozygosity (Ho) was 0.482, the mean expected heterozygosity (He) was 0.483,the mean discrimination power (DP) was 0.612, the mean polymorphism information content (PIC) was 0.365, the total discrimination power (TDP) reached 0.999 999 999 999 999 999 999 999 999 428 18. The cumulative power of exclusion for 66 InDel loci was 0.999 739 in duo cases (CPEduo) and was 0.999 999 999 417 in trios cases (CPEtrio). The results show that the 66 InDel loci have high genetic polymorphisms in the Chinese Han population and can be used independently for forensic DNA identification and paternity testing.
insertion/delationpolymorphism (InDel); Chinese Han population; DNA identification; paternity testing
2022-01-06;
2022-03-15;
2022-03-24
王雪倩,在读硕士研究生,专业方向:分子生物学。E-mail: Wangxqer0122@163.com
李卫国,博士,教授,研究方向:分子生物学研究。E-mail: liwg0618@htu.edu.cn
周喆,博士,研究员,研究方向:分子生物学研究。E-mail: zhouzhe@bmi.ac.cn
王升启,博士,研究员,研究方向:基因分析与生物分子识别研究。E-mail: sqwang@bmi.ac.cn
10.16288/j.yczz.22-006
(责任编委: 朱波峰)

