基于生物信息学分析构建转录因子E2F1调控微RNA及其靶向基因网络
朱文韬,曲浩宁,何群
(中国医科大学生命科学学院生物信息教研室,沈阳 110122)
E2F是调控基因表达的重要转录因子家族,E2F1在1986年首次被ROVESDI发现,并由HELIN等从E2F家族中分离提出。E2F1是人体中重要的细胞周期正向调控因子,已有研究[1]发现E2F1在多种肿瘤如肺癌、结肠癌、乳腺癌中发挥促癌作用。
微RNA(microRNA,miRNA) 是一类长约 18~25个核苷酸的非编码RNA,与mRNA 转录本的 3’-UTR结合,可以对靶基因的表达形式进行调控[2]。它们对各种生物学过程具有调控作用,与细胞发育、细胞增殖、细胞分化及细胞凋亡等有关,其表达作用方式受转录因子调控影响,近年来转录因子与miRNA的调控网络已成为研究热点[3]。
然而,目前研究[4]多关注于E2F1及miRNA参与细胞通路,对E2F1与miRNA存在的内在调控机制尚无研究阐明。本研究基于生物信息学分析,构建TF-miRNA-mRNA调控网络,旨在为癌症治疗的药物研发提供潜在的靶点。
1 材料与方法
1.1 泛基因组筛选存在E2F1差异表达的肿瘤
UALCAN(http://ualcan.path.uab.edu/) 是基于TCGA构建的癌症组织数据库,用于分析癌症OMICS数据的全面、交互式的Web资源。其使用javascript和CSS在Perl-CGI构建了高质量图形。运用UALCAN进行E2F1泛基组表达分析,筛选pan-cancer view泛基因组图谱中存在E2F1表达差异的肿瘤。
1.2 E2F1调控miRNA靶基因筛选
1.2.1 差异表达miRNA筛选(differential expression miRs screening,DEG-miRNAs):Oncomir是一个在线数据库(http://www.oncomir.org/),提供30种癌症的miRNA表达谱。登陆Oncomir网站,分析E2F1存在差异表达肿瘤组的miRNA表达数据,下载各肿瘤组差异表达的miRNA数据。筛选条件为P≤ 0.05,score >50,将差异表达结果集合列为DEG-miRNAs。
1.2.2 基因转录调控数据库(gene tranion regulation database,GTRD)-miRNAs的预测:GTRD(http://gtrd.bio-uml.org/) 是一个通过ChIP-seq实验鉴定的人类和小鼠转录因子结合位点(transcription factor binding sites,TFBSs) 数据库。从ENCODE和SRA中获得原始ChIP-seq数据,并一致处理。利用GTRD,在transcription factor target genes中选择物种(organism):Human;转录因子(transcription factor):E2F1 QO1094;转录因子结合位点(TF binding site location):Promoter[-1 000,+100],得到E2F1调控靶基因并从中筛选保留靶基因中的miRNA,设为GTRD-miRNAs数据集。
利用venny2.1.0网站(https://bioinfogp.cnb.csic.es/tools/venny/index.html),分别对每个肿瘤组的DEGmiRNAs和GTRD预测的E2F1靶基 因GTRD-miRs数据集取交集,设为每个肿瘤组潜在的E2F1靶基因Pre-miRNAs,将所有肿瘤组的共有Pre-miRNAs设置为E2F1最终的miRNA靶基因GTRD-miRNAs。
1.3 E2F1-miRNA-mRNA调控网络构建
1.3.1 Target-mRNAs的预测与分析:使用TargetScan(http://www.targetscan.org/),TargetMiner(http://www.mybiosoftware.com/targetminer-microrna-target-prediction.html),TarBase(http://microrna.gr/tarbase/) 分别预测候选mRNA,并将预测结果进行交集筛选,最终取得Target-miRNAs的靶基因集Target-mRNAs。将Target-mRNAs与DEGs进行交集筛选,获得候选Target-miRNAs的靶基因mRNA组。
1.3.2E2F1高表达肿瘤的基因差异表达分析:登陆TCGA数据库,下载E2F1存在差异表达肿瘤组的mRNA-seq数据,利用R语言edgeR包进行基因差异表达分析,mRNA的筛选阈值为log2FC> 1.5且P<0.01,并将表达数据进行标准化处理,将表达下调的差异结果集合列为DEGs。
1.4 分析构建可视化TF-miRNA-mRNA网络
运用Cytoscape(3.7.1版本) 构建可视化TF-miRNAmRNA可视化网络。为分析其功能,进一步运用DAVID6.8数据库对取得的靶基因mRNAs进行基因本体(gene ontology,GO) 分析和京都基因与基因组百科全书(Kyoto encyclopedia of genes,KEGG) 通路分析,筛选条件均为P≤ 0.05。采用STRING(https://stringdb.org/) 数据库构建靶基因组的蛋白-蛋白相互作用(protein-protein interactions,PPI),并应用Cytoscape(3.7.1版本) 对STRING数据库构建出的PPI网络结果进行分析,将分析结果导入Metascpae构建可视化网络,进一步运用Cytoscape的Hubba插件对该网络进行枢纽基因功能富集。
2 结果
2.1 泛基因组中E2F1差异表达结果
存在E2F1表达差异的肿瘤组织共20个,其中E2F1表达上调的肿瘤组织18个,分别为BRCA、CESC、CHOL、COAD、ESCA、HNSC、KICH、KIRP、READ、PRAD、BLCA、KIRC、LIHC、LUAD、LUSC、THCA、STAD和UCEC,E2F1表达下调的肿瘤组织2个,分别为THYM和PCGC。见图1。

图1 E2F1转录因子在泛基因组中的表达状况Fig.1 The figure illustrates the expression status of E2F1 transcription factor in the pan-genome
2.2 E2F1调控miRNA基因预测及其筛选结果
2.2.1 转录因子结合位点预测:GTRD数据库预测转录因子的靶基因,取得E2F1靶基因集24 467个,其中miRNA 543个,列为GTRD-miRNAs。
2.2.2 DEG-miRNAs预测结果:为了进一步对E2F1调控miRNA靶基因进行筛选,登陆OncomiR(http://www.oncomir.org/),分析E2F1存在差异表达肿瘤组的miRNA表达情况,并下载各肿瘤组差异表达的miRNA。为进一步缩小范围,利用venny2.1.0对GTRD预测的GTRD-miRNAs和各肿瘤组对应下调的miRNA取交集,筛选各肿瘤组中E2F1的潜在靶miRNA,列集合为Pre-miRNAs并分别与GTRD-miRNAs取交集并进行可视化处理(图2),将所取得的所有肿瘤组潜在靶miRNA与GTRD-miRNAs的共交集作为E2F1的候选miRNA,列为DEGs-miRNAs。

图2 GTRD-miRNAs预测结果与泛基因组肿瘤差异基因交集结果Fig.2 The intersection results between the GTRD-miRNAs prediction results and the pan-genomic tumor difference genes
运用VENNY2.1.0将GTRD-miRNAs与DEG-miRNAs的预测结果与DEGs取交集,取得肿瘤组中表达上调的miRNA:hsa-miR-7,hsa-miR-9,hsa-miR-13,hsamiR-21,hsa-miR-22,hsa-miR-24,列为Target-miRNAs靶集,见表1。
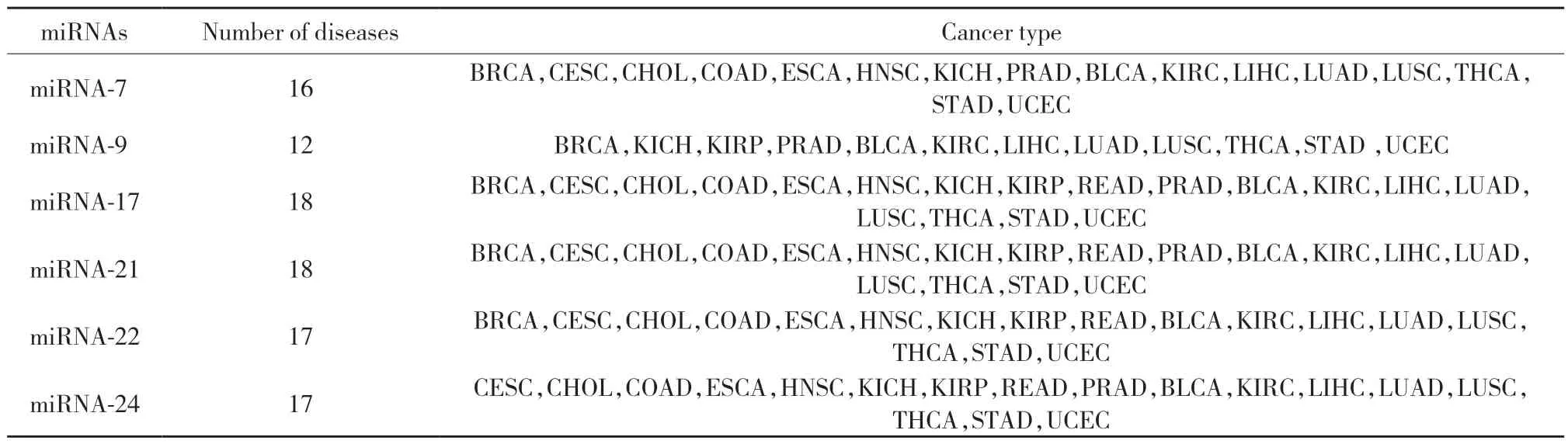
表1 Target-miRNAs数据表Tab.1 Target-miRNAs data table
2.3 Target-mRNAs的交集预测与定位结果
使用TargetScan,TargetMiner,TarBase交集预测得到miRNA-7,miRNA-9,miRNA-14,miRNA-21,miRNA-22,miRNA-24的靶mRNAs分别为224个,444个,83个,74个,130个和72个。进一步采用VENNY2.1.0将Target-miRNAs与DEGs取交集作为目标miRNA基因簇的靶mRNAs。miRNA-7的mRNA靶基因12个,miRNA-9的mRNA靶基因12个,miRNA-14的mRNA靶基因2个,miRNA-21的mRNA靶基因14个,miRNA-22的mRNA靶基因30个,miRNA-24的mRNA靶基因16个,见表2。

表2 miRNA-mRNA数据表Tab.2 miRNAs-miRNAs data table
2.4 基因调控网络的可视化构建分析
为宏观展现E2F1在泛基因组中的调控过程,进一步对E2F1转录因子调控的miRNA-7,miRNA-9,miRNA-14,miRNA-21,miRNA-22,miRNA-24及受其调控的关键靶基因进行可视化网络构建,以图谱展现转录因子泛基因组中E2F1通过下调miRNA参与基因表达调控的作用途径,见图3。
GO分析结果显示,生物过程(biological process,BP) 层面上述基因组主要参与白血病抑制因子信号通路,癌胚素-M介导的信号通路,NK T细胞分化的调控,肾小球形态发生等生物学功能;细胞组成(cellular component,CC) 层面,主要参与细胞的孔复合体,异染色质的构成;细胞组成(molecular function,MF) 层面,基因组有与蛋白质、受体结合的功能。下载导出GO富集分析结果,将上述所有功能节点导入Metascape进行可视化分析,见图4。

图4 基于转录因子调控网络的KEGG与GO分析Fig.4 KEGG and GO analysis for transportation factor regulatory network
KEGG通路分析结果显示,上述基因组主要参与micorRNAs in cancer(Hsa05206) 信号通路,该通路昼夜节律,朊病毒等信号代谢通路,共有9个枢纽基因参与此通路调节(P< 0.05)。
运用Cytoscape中CytoHubba插件筛选关键基因构建相互作用PPI网络图(图5),富集显示排在前5位的枢纽基因分别为CD34、TGFBR2、MEF2C、SOX4和VCL。

图5 PPI网络构建与Hubba分析Fig.5 PPI network construct and Hubba analysis
3 讨论
E2F1是E2F转录因子家族的重要一员,参与人体生命活动中重要的细胞周期和细胞凋亡环节,E2F1促进 S 期的细胞增多,同时通过p53依赖和非依赖途径诱导细胞凋亡[2]。近来研究[5-6]显示,E2F1在多种肿瘤中过表达,其作用为促癌基因,且表达水平与肿瘤的发生、发展及预后密切相关。
研究构建的TF-miRNA网络构建展现了泛肿瘤组中6个抑制表达的miRNAs与其所调控靶基因之间的关系。miRNA-7参与多种细胞途径,其正常表达多在肿瘤细胞中起抑癌作用。miRNA-7在肿瘤进展和MDR调控中的作用已被验证[7];肿瘤细胞中miRNA-9的下调被证实限制了抗癌治疗的有效性。miRNA-9在胃癌细胞中的表达被证实与胃癌细胞的恶性生物学行为密切相关[8-9];miRNA-17基因簇被证实提升肝癌细胞的自我更新、增殖、迁移能力的功能[10];miRNA-21的差异表达与肿瘤细胞的高增殖、低凋亡、高侵袭和转移潜能相关[11];miRNA-22在结消化系统癌症患者血清中的表达下调现象与癌症的临床病理特征密切相关[12];miRNA-24正常表达可靶向CARMA3抑制胃癌细胞的增殖、进而促进胃癌细胞凋亡[13]。上述miRNAs已被研究[14]证实与多种癌症发生发展存在密切联系,本研究构建的KEGG通路亦可佐证此结论。然而,目前已有研究多针对单个miRNA调控展开,miRNAs基因簇在泛基因组肿瘤间的整体调控机制尚乏研究,其上游基因调控机制仍待探索。
GO结果显示,相关基因组在生物学功能层面主要参与癌胚素-M介导的信号通路,该信号通路可调节基因激活、细胞存活、增殖和分化等生物功能。近年来,此通路研究热度逐年上升[15]。KEGG分析结果显示,上述miRNA通过降解靶mRNA或抑制蛋白翻译来调控基因的转录表达[16]。特异性miRNAs的上调(过表达) 可能导致抑癌基因表达[17],而其下调则会导致癌基因表达的增加[18],这两种情况都会导致细胞的增殖、分化和凋亡,进而导致肿瘤的生长和发展。研究[19]显示,不同癌症中miRNA的表达存在很大差异。
本研究直观展现了E2F1所调控的miRNAs在肿瘤中广泛参与的复杂生理过程。通过构建 TF-miRNAmRNA 调控网络,为未来泛基因组癌症治疗药物提供了治疗靶点的理论基础。同时为癌症的预后分析、鉴别与诊断提供了潜在的应用价值。

