新型冠状病毒侵染所需宿主基因TMPRSS2、CTSL、PIKFYVE、TPCN2重要功能变异分析
陈慧珍,杨健康

【摘要】 目的 研究新型冠狀病毒(SARS-CoV-2)侵染所需宿主基因TMPRSS2、CTSL、PIKFYVE、TPCN2的重要功能变异在全世界不同人群间的分布差异。
方法 使用GTEx数据库,在新型冠状病毒的主要侵染器官肺中分析TMPRSS2、CTSL、PIKFYVE、TPCN2基因的eQTL位点。使用1 k GP(千人基因组计划)数据库,系统分析上述4个基因的错义变异在世界不同人群间的分布差异,并使用PolyPhen-2和SIFT软件预测错义突变导致的氨基酸替代是否影响蛋白功能。
结果 在肺中发现了4个q值小于0.05的eQTL位点,分别为TMPRSS2基因的rs35074065、CTSL基因的rs2378757、PIKFYVE基因的rs12475932、TPCN2基因的rs930786。TMPRSS2、CTSL、PIKFYVE、TPCN2基因的4个eQTL位点可能与上述4个基因在东亚人群中的较低表达水平有关。在TMPRSS2、CTSL、PIKFYVE、TPCN2基因上分别有10、6、15、30个有害错义变异,大部分变异都是低频变异,其中大多数仅在一个人群中具有特异性。唯一的例外是TPCN2基因的变异位点rs78034812,它在全球的等位基因频率大于1%,在东亚人群中具有最高的等位基因频率且该位点在物种间具有保守性。
结论 TMPRSS2、CTSL、PIKFYVE、TPCN2基因存在重要功能变异,这些变异的等位基因频率在不同人群间存在差异,可能影响基因的表达和功能,从而影响人群对新型冠状病毒的易感性以及感染后症状的差异。
【关键词】 新型冠状病毒;TMPRSS2;CTSL;PIKFYVE;TPCN2;人群;变异
中图分类号:R181.3 文献标志码:A DOI:10.3969/j.issn.1003-1383.2022.02.002
Analysis of important functional variation of host genes required for SARS-CoV-2: TMPRSS2, CTSL, PIKFYVE and TPCN2
[HJ1][HJ]
CHEN Huizhen, YANG Jiankang
(Laboratory of Biochemistry and Molecular Biology of School of Basic Medical Sciences, Dali University, Dali 671000, Yunnan, China)
【Abstract】 Objective To study the distribution differences of important functional variants of the host genes TMPRSS2, CTSL, PIKFYVE and TPCN2 required for SARS-CoV-2 infection among different populations in the world.
Methods GTEx database was used, and the eQTL loci of the TMPRSS2, CTSL, PIKFYVE, and TPCN2 genes in the lungs of the main infecting organs of the SARS-CoV-2 were analyzed. 1k GP (1000 Genomes Project) database was used, and the distribution of missense variants of the above-mentioned 4 genes among different populations in the world were systematically analyzed, and PolyPhen-2 and SIFT software were used to predict whether amino acid substitutions caused by missense mutations to affect protein function.
Results 4 eQTL loci with q value less than 0.05 were found in the lungs, including rs35074065 of TMPRSS2 gene, rs2378757 of CTSL gene, rs12475932 of PIKFYVE gene, and rs930786 of TPCN2 gene. The 4 eQTL loci of TMPRSS2, CTSL, PIKFYVE, and TPCN2 genes may be related to the lower expression levels of the above-mentioned 4 genes in East Asian populations. There were 10, 6, 15, and 30 deleterious missense variants in TMPRSS2, CTSL, PIKFYVE, and TPCN2 genes, respectively. Most of the variants were low-frequency variants, and most of them were specific in only one population. The only exception was rs78034812, a variant in the TPCN2 gene, which had an allele frequency greater than 1% worldwide. It had the highest allele frequency in East Asian populations and this locus was conserved among species.
Conclusion There are important functional variations in TMPRSS2, CTSL, PIKFYVE, and TPCN2 genes. The allele frequency of these variants vary among different populations, which may affect gene expression and function, thereby affecting the population's susceptibility to the SARS-CoV-2 and the difference in symptoms after infection.
【Key words】 SARS-CoV-2; TMPRSS2; CTSL; PIKFYVE; TPCN2; population; mutation
新型冠状病毒肺炎是指由一种β新型冠状病毒,新型致病性SARS冠状病毒2(SARS-CoV-2)引起的传染性呼吸道疾病[1],以发热、咳嗽、头痛、乏力、呼吸困难等症状为主要临床表现的病毒性肺炎[2]。全球SARS-CoV-2的流行构成全球卫生紧急情况[3]。SARS-CoV-2侵染细胞依赖于病毒刺突(S)蛋白与细胞受体ACE2的结合[4],以及跨膜丝氨酸蛋白酶2(TMPRSS2基因)和组织蛋白酶L(CTSL基因)等蛋白酶对病毒S蛋白的活化[5]。SARS-CoV-2侵染293/hACE2细胞的过程主要是通过内吞作用介导的,而阻断PIKfyve(PIKFYVE基因)和TPCN2(TPCN2基因)能强烈抑制SARS-CoV-2进入细胞,这说明PIKfyve和TPC2对病毒进入细胞至关重要[6]。
TMPRSS2、CTSL、PIKFYVE、TPCN2基因对于SARS-CoV-2侵染宿主细胞都发挥了重要的作用,因此研究上述基因的重要功能变异在全世界不同人群间的分布差异也具有重要的意义。许多基因变异位点,包括编码区错义突变和表达定量性状位点(eQTL),都可能影响这些基因的表达和功能。进一步在世界不同人群中对这4个基因进行遗传分析,有助于解释疾病的症状及易感性差异。现将对4个基因的遗传变异,包括eQTL位点和功能编码区错义变异在人群中的分布差异进行分析,旨在为新型冠状病毒在世界不同人群中传播的流行病学研究提供参考。
1 材料与方法
1.1 TMPRSS2、CTSL、PIKFYVE、TPCN2基因eQTL的人群分布
新型冠状病毒主要靶器官是肺,因此分析肺组织的eQTL位点[7]。使用GTEx数据库[8],选择q值小于0.05的位点作为与肺组织表达相关的eQTL位点。1 k GP(千人基因组计划)的数据库[9]包括2504个个体的全基因组突变位点数据。从1 k GP的数据库中下载TMPRSS2、CTSL、PIKFYVE、TPCN2基因的eQTL位点的等位基因频率数据。分别统计南亚、欧洲、东亚、美洲和非洲5类人群的等位基因频率分布。
1.2 TMPRSS2、CTSL、PIKFYVE、TPCN2基因错义突变的人群分布
从1 k GP数据库中下载TMPRSS2、CTSL、PIKFYVE、TPCN2基因编码区的错义突变数据,并使用PolyPhen-2和SIFT软件预测错义突变导致的氨基酸替代是否影响蛋白功能。SIFT软件中,预测结果小于0.05则为有害变异。PolyPhen-2软件中,预测结果大于0.4470则为有害变异。分别计算错义突变和有害错义突变的等位基因频率的世界主要人群分布,并用Ensembl数据库[10]检查部分重要突变的物种保守性。
2 结 果
2.1 TMPRSS2、CTSL、PIKFYVE、TPCN2基因eQTL位点的人群分布
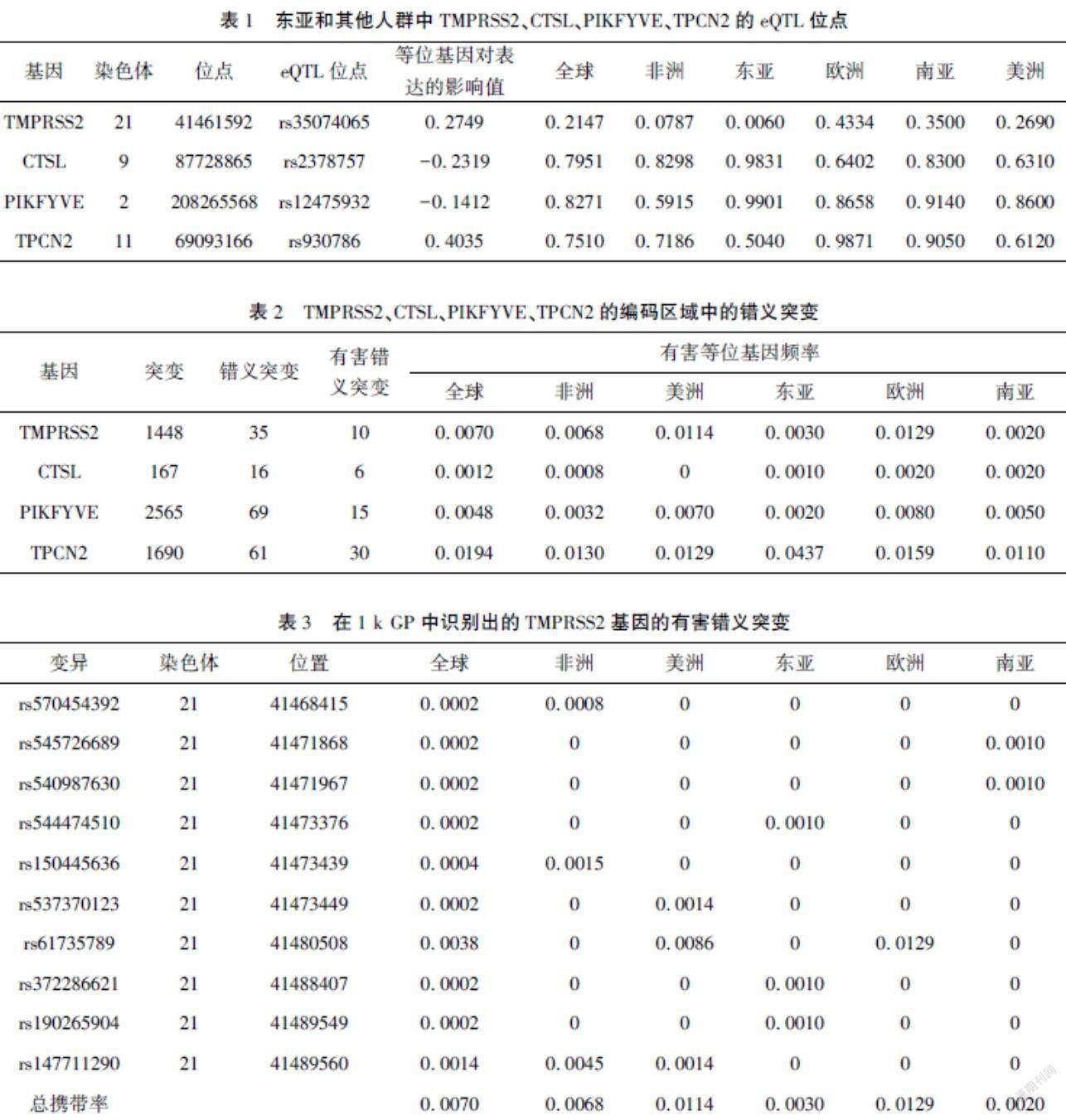
本研究使用GTEx数据库,在肺组织中发现了4个q值小于0.05的eQTL位点,分别为TMPRSS2的rs35074065、CTSL的rs2378757、PIKFYVE的rs12475932、TPCN2的rs930786。使用1 k GP数据比较了不同人群中4个eQTL位点的等位基因频率分布差异,结果表明,位于TMPRSS2基因上的eQTL变异rs35074065(等位基因对表达的影响值为0.2749)在东亚人群中的等位基因频率最低(0.0060)。与其他人群相比,在东亚人群中TMPRSS2基因上的rs35074065和TPCN2基因上的rs930786兩个eQTL变异的等位基因频率明显低于其他人群。而其他两个eQTL变异(CTSL的rs2378757和PIKFYVE的rs12475932)具有更高的等位基因频率。例如,位于CTSL基因中的eQTL变异rs2378757(等位基因对表达的影响值为-0.2319)的等位基因频率在东亚人群中最高(0.9831)。见表1。
2.2 TMPRSS2、CTSL、PIKFYVE、TPCN2基因错义突变的人群分布
本研究统计了来自1 k GP数据库的TMPRSS2、CTSL、PIKFYVE、TPCN2基因编码区所有错义突变的数量和频率分布(表2),重点对这4个基因的有害错义突变的人群分布进行了分析。
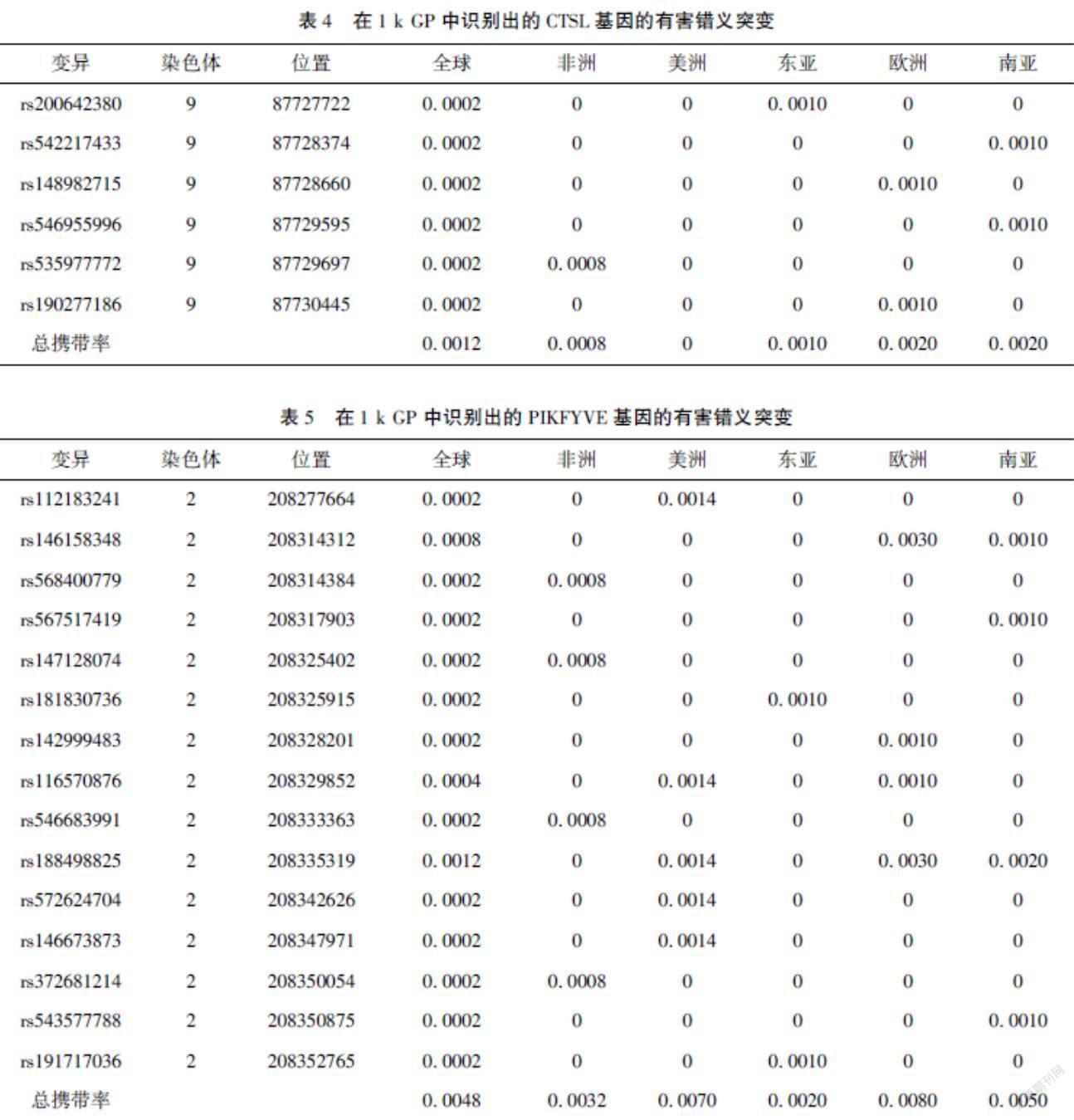
表3结果显示TMPRSS2基因中10个有害错义突变的等位基因频率。所有这些变异在5个人群中都是低频变异,且其中大多数仅在一个人群中具有特异性。例如,rs544474510、rs372286621、rs190265904这三个变异位点对于东亚人群是特异的,其他人群中不存在相关突变。表4~表6的结果分别显示,CTSL、PIKFYVE、TPCN2中有6个、15个、30个有害错义突变。除TPCN2基因上的rs78034812等位基因频率>0.01外,这些大多数变异在所有人群中都是低频变异。
我们试图评估这4个基因的所有有害错义突变的总携带率。这4个基因的总携带率也很低(表2),但在人群之间差异很大。TPCN2具有比其他三个基因更多的有害错义突变。东亚人群中的总携带率是全球人口的两倍。
3 讨 论
新型冠状病毒具有很强的传播性和致病性,容易引起呼吸道感染,导致病毒性肺炎[11]。截至北京时间2021年4月13日,全球累计确诊病例达1亿3千万人,累计死亡病例达290万余人。这些数字仍以惊人的速度增长,这使全世界的公共卫生和经济受到巨大影响[12]。目前为止,尚无针对新型冠状病毒肺炎的特效治疗手段。我们通过研究TMPRSS、CTSL、PIKFYVE、TPCN2基因上的功能变异位点,鉴定这些基因中自然发生的重要功能变异,这些变异可能影响不同群体间基因功能或表达的差异。
SARS-CoV-2的生命周期可分为三个阶段:进入宿主细胞及细胞内的运输,病毒基因组的复制,以及新病毒粒子的包装成熟和排出[13]。目前已有研究发现,SARS-CoV-2的蛋白(NSP1、NSP5、ORF8等)可与人类宿主蛋白相互作用。并发现不同新型冠状病毒蛋白可参与多种复合物的形成和生物学过程,包括DNA复制、表观遗传学和基因表达的调控、囊泡运输、脂类修饰等。如NSP5可能抑制人类HDAC2蛋白转运到细胞核中,并可能影响HDAC2介导的炎症和干扰素反应。ORF8可参与调控内质网对蛋白质加工过程[14]。另有研究表明,宿主细胞RNA结合蛋白(TIA1、SND1、IGF2BP1、DDX42等)可与SARS-CoV-2 RNA基因组相互作用。此外,下调4个基因TIA1、SND1、IGF2BP1、DDX42的表达显著降低了受感染细胞中SARS-CoV-2基因组RNA的总量[15]。
目前已有研究表明,新型冠状病毒除了对肺有影响外,还会对胃肠道产生影响,常表现为腹泻、恶心和呕吐[16]。同时,SARS-CoV-2也可以侵染神经细胞,使机体出现头痛、头晕等症状[17]。极少数患者甚至还会出现幻觉、躁动等精神方面的表现[18]。不仅如此,SARS-CoV-2还会导致急性肾损伤、急性心肌损伤等严重的并发症[19~20]。
本研究系统地分析了TMPRSS2、CTSL、PIKFYVE、TPCN2基因中重要的功能变异,包括eQTL变异。结果显示与其他人群相比,在东亚人群中TMPRSS2基因中的rs35074065和TPCN2基因中的rs930786这两个eQTL变异的等位基因频率明显低于其他人群。相反,其他两个eQTL变异(CTSL基因中的rs2378757和PIKFYVE基因中的rs12475932)在东亚人群中比其他人群具有更高的等位基因频率。这些发现表明,TMPRSS2、CTSL、PIKFYVE、TPCN2基因的eQTL位点可能与它们在东亚人群中的较低表达水平相关。
除TPCN2基因中的rs78034812外,其他大多数变异在所有人群中都是低频变异。rs78034812在东亚人群具有最高的等位基因频率,频率高达3.87%,奇怪的是非洲人群中不存在该变异。而此变异会导致TPCN2蛋白第681位氨基酸残基从丝氨酸变为亮氨酸,从亲水性氨基酸变为疏水性氨基酸。使用Ensembl数据库检查这个变异的保守性,发现这个变异在灵长类动物和哺乳动物中都是保守的。因此,此变异与TPCN2蛋白功能的关系值得进一步研究。双孔通道(TPCs)是位于宿主细胞内溶酶体区室膜上的钙阳离子通道,SARS-CoV-2依赖于这些细胞器进行感染和复制。TPCs的两种亚型TPC1和TPC2主要分布在溶酶体内,而TPC2主要位于晚期溶酶体区室[21]。TPCs直接参与钠外流,调节细胞内渗透压,使得囊泡表面积与体积比发生显著变化。因此,TPCs抑制剂可抑制溶酶体的融合潜力进而限制病毒侵染[22]。有研究表明,抑制剂粉防己碱、柚皮素阻断TPC2活性时,SARS-CoV-2侵染细胞的能力会被抑制[6,23]。
另外,我们发现虽然这4个基因的有害错义突变总携带率很低,但在人群之间差异很大。TPCN2具有比其他三个基因更多的有害错义突变。东亚人群中的总携带率是全球人口的两倍。不同人群之间功能变异的等位基因频率差异表明,遗传基础的多样性可能影响人群之间相应基因的功能。
参 考 文 献
[1] CHUGH H,AWASTHI A,AGARWAL Y,et al.A comprehensive review on potential therapeutics interventions for COVID-19[J].Eur J Pharmacol,2021,890:173741.
[2] 何青林,劉芳丽,姬新颖,等.新型冠状病毒肺炎相关并发症研究进展[J].河南大学学报(医学版),2020,39(6):391-396.
[3] PHAN T.Novel coronavirus:from discovery to clinical diagnostics[J].Infect Genet Evol,2020,79:104211.
[4] HOFFMANN M,KLEINE-WEBER H,SCHROEDER S,et al.SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor[J].Cell,2020,181(2):271-280.e8.
[5] VIVEK-ANANTH R P,RANA A,RAJAN N,et al.In silico identification of potential natural product inhibitors of human proteases key to SARS-CoV-2 infection[J].Molecules,2020,25(17):E3822.
[6] OU X Y,LIU Y,LEI X B,et al.Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV[J].Nat Commun,2020,11(1):1620.
[7] 王淮,杨健康.新型冠状病毒复制依赖基因MTHFD1的表达及功能[J].重庆师范大学学报(自然科学版),2020,37(4):140-146,封3.
[8] GTEX CONSORTIUM,HUMAN GENOMICS.The Genotype-Tissue Expression (GTEx) pilot analysis:multitissue gene regulation in humans[J].Science,2015,348(6235):648-660.
[9] 1000 GENOMES PROJECT CONSORTIUM,AUTON A,BROOKS L D,et al.A global reference for human genetic variation[J].Nature,2015,526(7571):68-74.
[10] BUSHEHRI A,BAREZ M R,MANSOURI S K,et al.Genome-wide identification of human-and primate-specific core promoter short tandem repeats[J].Gene,2016,587(1):83-90.
[11] 嚴新忠,洪大富,程智,等.新型冠状病毒(SARS-CoV-2)采样方法的现状与展望[J].天津科技大学学报,2020,35(6):1-6.
[12] AHMED R,HASAN R,SIDDIKI AMAMZ,et al.Host range projection of SARS-CoV-2:South Asia perspective[J].Infect Genet Evol,2021,87:104670.
[13] WONG J P,DAMANIA B.SARS-CoV-2 dependence on host pathways[J].Science,2021,371(6532):884-885.
[14] GORDON D E,JANG G M,BOUHADDOU M,et al.A SARS-CoV-2 protein interaction map reveals targets for drug repurposing[J].Nature,2020,583(7816):459-468.
[15] SUN L,LI P,JU X,et al.In vivo structural characterization of the SARS-CoV-2 RNA genome identifies host proteins vulnerable to repurposed drugs[J].Cell,2021,184(7):1865-1883.e20.
[16] LI LY,WU W,CHEN S,et al.Digestive system involvement of novel coronavirus infection:prevention and control infection from a gastroenterology perspective[J].J Dig Dis,2020,21(4):199-204.
[17] LV P,PENG F,ZHANG Y Q,et al.COVID-19-associated meningoencephalitis:a care report and literature review[J].Exp Ther Med,2021,21(4):362.
[18] ELKHALED W,BEN ABID F,AKHTAR N,et al.A 23-year-old man with SARS-CoV-2 infection who presented with auditory hallucinations and imaging findings of cytotoxic lesions of the corpus callosum (CLOCC)[J].Am J Case Rep,2020,21:e928798.
[19] LI N,ZHU L,SUN L,et al.The effects of novel coronavirus (SARS-CoV-2) infection on cardiovascular diseases and cardiopulmonary injuries[J].Stem Cell Res,2021,51:102168.
[20] MIGLIACCIO M G,DI MAURO M,RICCIOLINO R,et al.Renal involvement in COVID-19:a review of the literature[J].Infect Drug Resist,2021,14:895-903.
[21] HEISTER P M,POSTON R N.Pharmacological hypothesis:TPC2 antagonist tetrandrine as a potential therapeutic agent for COVID-19[J].Pharmacol Res Perspect,2020,8(5):e00653.
[22] FILIPPINI A,D'AMORE A,PALOMBI F,et al.Could the inhibition of endo-lysosomal two-pore channels (TPCs) by the natural flavonoid naringenin represent an option to fight SARS-CoV-2 infection?[J].Front Microbiol,2020,11:970.
[23] CLEMENTI N,SCAGNOLARI C,D'AMORE A,et al.Naringenin is a powerful inhibitor of SARS-CoV-2 infection in vitro[J].Pharmacol Res,2021,163:105255.
(收稿日期:2021-09-28 修回日期:2021-12-06)
(编辑:梁明佩)

