基于线粒体COI基因和D-loop区联合序列的杂交鲫与野生鲫群体遗传特征研究
杨聪慧,黄雪雪,戴承华,许效玮,王 石,刘庆峰,张 纯,周 毅,刘少军
(省部共建淡水鱼类发育生物学国家重点实验室/湖南师范大学生命科学学院,湖南 长沙 410081)
0 引言
【研究意义】鲫(Carassius auratus)是我国淡水水域中最常见的鱼类之一,其肉质鲜美,营养价值高,在我国淡水养殖业中占据重要地位。野生鲫生长速度极其缓慢,长至一斤需要5~10年,故鲫鱼养殖主要依赖人工培育的优良品种,包括由优良地方品系选育的彭泽鲫和方正银鲫,以及利用杂交等手段培育的湘云鲫和异育银鲫等(颜金鹏等,2007;丁立云等,2017;刘庆峰,2018)。随着鲫鱼养殖规模的逐年扩大,其品种也逐渐推陈出新。合方鲫是以日本白鲫(C.cuvieri)为母本、红鲫(C.cuvierired var.)为父本,通过远缘杂交技术培育获得的新型优质品种,具有生长速度快、抗逆性强、肉质细嫩、营养价值高等特点,已在全国多地推广养殖(王静等,2015;刘庆峰,2018;Liu et al.,2018,2019a,2019b)。新型同源二倍体类鲫(New homodiploid crucian carp-like species,NCRC)品系来源于鲤(Cyprinus carpioL.,♀)×团头鲂(Megalobrama amblycephala,♂)的杂交组合,具有无口须、侧线鳞、鳍条数目、体色等外形特征,与鲫非常相似(Wang et al.,2017,2021;焦妮,2019)。NCRC品系头部较小、体背较高,在育种实践中具有良好应用潜力,可作为鱼类物种进化研究的基础材料。因此,对合方鲫、NCRC等新型优良鲫及其养殖周边地区的野生鲫群体进行遗传特征研究,有助于为鱼类遗传育种开拓新的种质资源,同时为鱼类种质资源保护提供参考依据。【前人研究进展】当前的研究认为鲫属包括3个种,分别是分布在欧洲和我国额尔齐斯河流域的黑鲫(C.carassius)、分布在日本的白鲫(C.auratus cuvieri),以及广泛分布于欧亚大陆和东方各岛屿的鲫复合种(C.auratuscomplex)。鲫复合种因地理分布广、表型变异大,又被分成不同亚种,包括我国的普通鲫鱼(C.auratus auratus)和银鲫(C.auratus gibelio),日本的关东鲫(C.auratus langsdorfii),以及其他地方种群,如彭泽鲫、普安鲫、高背鲫等(董传举等,2020)。鲫属物种分类极其复杂,不同种群形态相似,因此通常需要借助分子标记来研究物种鉴定、系统发育及群体遗传分化等问题。线粒体基因组具有遵循母系遗传、进化速率快、重组率较低等特点(刘东东等,2022),其编码区的COI、COII和Cytb基因及非编码区的D-loop序列等均为最常用的DNA分子标记。Komiyama等(2009)、Wang等(2013)基于多个线粒体基因片段的分子系统发育研究结果表明,金鱼与自然水域中的野生鲫为同一物种,且最早可能驯化于长江下游的安徽、浙江等地,与历史文献记载一致。至今,基于线粒体基因组的鲫属物种鉴定、系统发育及群体遗传分化研究已有较多报道。Murakami等(2001)利用线粒体D-loop区对多个鲫群体进行系统发育进化分析,建议将白鲫从亚种地位提升到独立的物种;Takada等(2010)基于多个线粒体基因片段的系统发育分析,将鲫复合种划分为日本支系和欧亚大陆支系;Gao等(2012)基于线粒体D-loop区和Cytb基因对鲫属物种的谱系地理结构进行分析,验证了日本种群与我国大陆种群的遗传分化,并建议将欧亚大陆的鲫复合种划分为6个地理种群;程磊等(2012)利用线粒体COI基因验证了DNA条形码物种鉴定方法在鲫属物种划分中的可行性。鲫属群体中四倍体与六倍体同时存在,甚至发现少量八倍体,其多倍体的起源问题一直是鲫属物种的研究热点之一(董传举等,2020)。已有多个研究利用线粒体基因和核基因片段分析四倍体鲫和六倍体鲫的群体遗传分化情况,结果证实六倍体鲫群体来自同域四倍体鲫群体的多次独立多倍化起源(Luo et al.,2014;Liu et al.,2017a,2017b)。【本研究切入点】在鱼类遗传育种领域,随着野生资源的减少,人工培育和改良的优质鱼类品系逐渐成为鱼类种质资源的重要组成。合方鲫和NCRC作为新型优良杂交鲫鱼品系,其研究主要集中在经济性状或遗传物质变异方面(王静等,2015;Wang et al.,2017,2021;Liu et al.,2019a,2019b),而针对群体遗传的相关研究较少,因此亟待开展杂交鲫饲养群体的群体遗传特征及遗传分化研究,为其品系的延续和品种改良提供基础数据。【拟解决的关键问题】以合方鲫、NCRC、实验红鲫及湖南省的地方野生鲫群体为研究对象,对其线粒体COI基因片段和D-loop区进行扩增测序,解析各群体的遗传多样性、系统发育关系、群体结构和群体历史动态等,旨在揭示杂交鲫群体与野生鲫群体的种质资源现状,为开展鲫育种工作提供科学依据。
1 材料与方法
1.1 试验材料
供试鲫鱼样本包括2个杂交品系、1个实验室养殖品系和3个野生群体。杂交品系为湖南师范大学省部共建淡水鱼类发育生物学国家重点实验室培育的合方鲫(HFJ,30尾)和NCRC(27尾);实验室养殖品系为南华大学实验动物学部吴瑞生教授课题组培育的实验红鲫C1HD系(RCC,16尾);野生鲫样本于2018年9月—2019年1月分别采自湖南省长沙市望城区(WC,14尾)、湖南省长沙市长沙县(CSX,30尾)和湖南省常德市(CD,25尾)。
1.2 基因组DNA提取及PCR扩增
每个样本取尾鳍组织置于-20 ℃冰箱保存,使用基因组提取试剂盒Tissue DNA Kit(OMEGA)提取基因组DNA。扩增线粒体COI基因片段的引物为COI-F(5'-ACCCACCGCCTAAACACTCG-3')和COI-R(5'-ACTTCTGGGTGACCAAAGAATCA-3')(程磊等,2012),扩增线粒体D-loop区的引物为CR-F(5'-TTAAAGCATCGGTCTTGTAA-3')和CR-R(5'-G CCCTGAAATAGGAACCAGA-3')(Liu et al.,2017b)。PCR反应体系30.0 μL:DNA模板3.0 μL,上、下游引物各1.5 μL,2×RapidTaqMaster Mix 15.0 μL,TE Buffer 9.0 μL。扩增程序:94 ℃预变性5 min;94 ℃30 s,55 ℃(扩增COI基因片段)或50 ℃(扩增D-loop区)退火30 s,72 ℃50 s,进行30个循环;72 ℃延伸10 min。使用1.0%琼脂糖凝胶电泳检测PCR扩增产物,阳性扩增产物送至生工生物工程(上海)股份有限公司进行测序。
1.3 数据分析
采用SeqMan 7.1对测序得到的DNA序列峰图进行人工校对和拼接,并以MUSCLE进行多重序列比对分析。分别基于COI基因、D-loop区及二者联合序列,使用DnaSP 6.12进行群体遗传多样性分析,包括单倍型多样性(Haplotype diversity,h)和核苷酸多样性(Nucleotide diversity,π);基于COI基因和D-loop区联合序列进行Tajima’sD和Fu’sFs中性检验,结合错配分布分析(Mismatch distribution analysis)评估群体历史动态;基于COI基因和D-loop区联合序列,通过MEGA 7.0.26计算群体内和群体间的遗传距离,运用PopART中的TCS Network绘制单倍型网络(Haplotype network),再以Structure 2.3.4推测群体遗传结构;利用Arlequin 3.5.2.2计算群体间的遗传分化系数(Population differentiation,FST),并进行分子变异方差分析(Analysis of molecular variance,AMOVA)。
同时基于COI基因和D-loop区联合序列的单倍型数据进行系统发育进化分析,在GenBank下载鲤(KF856965.1)为外群,以及黑鲫(JQ911695.1)、日本白鲫(AP011237.1)和关东鲫(NC002079.1)共同分析;使用MEGA 7.0.26 基于Kimura 双参数模型(Kimura 2-parameter,K2P)构建邻接法(Neighborjoining,NJ)系统发育进化树,其置信度采用Bootstrap为1000次进行重复检验;经jModelTest 2.1.10评估后,以HKY+I+G为进化模型,分别通过最大似然法(Maximum likelihood,ML)和贝叶斯推断法(Bayesian inference,BI)构建系统发育进化树。其中,ML分析使用IQtree 1.6.12,其分支置信度评估Bootstrap 设 为1000 次 重 复;BI 分 析 采 用MrBayes 3.2.7,马尔科夫链蒙特卡洛(Markov chain Monte Crlo,MCMC)模拟运行1×106代,1000代取样1次,通过后验概率评估分支置信度。
2 结果与分析
2.1 序列特征及遗传多样性分析结果
对合方鲫品系(HFJ,30个样本)、NCRC品系(27个样本)、实验红鲫品系(RCC,16个样本)、望城野生鲫(WC,14个样本)、长沙野生鲫(CSX,30个样本)和常德野生鲫(CD,25个样本)共6个群体的线粒体COI基因片段和D-loop区进行PCR扩增,结果获得长度为687和427 bp的目的序列各142条。在线粒体COI基因序列中,检测到保守位点643个(占93.6%),变异位点44个(占6.4%);碱基组成为A占26.2%、T占28.5%、G占16.7%和C占28.6%,A+T为54.7%;共鉴定出14个单倍型,各群体的h介于0.214~0.780,π介 于0.00035~0.00218(表1)。在 线 粒 体D-loop区中,检测到保守位点385个(占90.2%),变异位点42个(占9.8%);碱基组成为A占36.5%、T占30.3%、G占13.7%和C占19.5%,A+T为66.8%,具有明显的A/T偏好;共鉴定出21个单倍型,各群体的h介于0.214~0.863,π介于0.00165~0.01039(表1)。此外,与COI基因序列相比,D-loop区虽然较短,但其遗传变异更大,符合线粒体基因组非编码区的特征。
将COI基因与D-loop区进行联合,其总序列长度为1114 bp,共鉴定出25个单倍型,对应的h为0.907,π为0.02373(表1)。其中,3个野生鲫群体均具有较高的h和π,最高的是常德野生鲫群体(h=0.877,π=0.00532);2个杂交鲫品系及实验红鲫品系的h和π相对较低,h最低的群体为NCRC品系(h=0.214),π最低的群体为合方鲫品系(π=0.00086)。可见,线粒体COI基因和D-loop区联合序列可提供更多的遗传信息,因此本研究的系统发育、群体遗传结构和历史动态等分析均使用联合序列进行计算。

表1 基于线粒体COI基因、D-loop区及联合序列的6个鲫群体遗传多样性分析结果Table 1 Genetic diversity analysis based on mitochondrial COI gene,D-loop region and their joint sequences
2.2 系统发育关系及单倍型网络结构
基于线粒体COI基因和D-loop区联合序列,从6个鲫群体中共鉴定出25个单倍型(Hap1~Hap25)。其中,单倍型Hap4在3个野生鲫群体中均有分布,单倍型Hap18为NCRC品系和常德野生鲫群体共享,单倍型Hap20为NCRC品系和实验红鲫品系共享,剩余22个单倍型为各群体的特有单倍型。通过TCS Network绘制的单倍型网络结构(图1)可知,3个野生鲫群体及实验红鲫品系的单倍型交错在一起,且相距较近,表明这些群体间无明显的谱系分化。合方鲫品系和NCRC品系的单倍型均与其他群体相距较远,且形成相对独立的聚群。
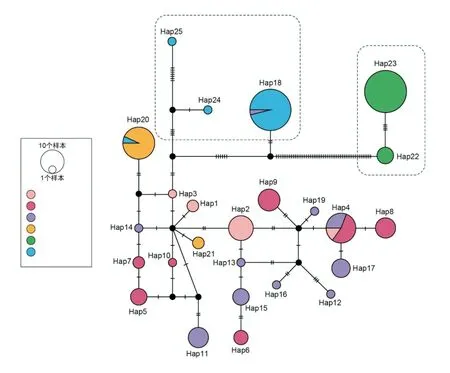
图1 基于线粒体COI基因和D-loop区联合序列的单倍型网络结构图Fig.1 Haplotype network structure chart based on joint sequences of mitochondrial COI gene and D-loop region
为进一步解释单倍型网络结构,分别通过NJ、ML和BI等3种方法对6个鲫群体进行系统发育进化分析,3种方法获得的拓扑结构基本一致,因此仅展示BI系统发育进化树(图2),并在节点位置标注每种方法的置信度。由构建的系统发育进化树(图2)可看出,3个野生鲫群体和实验红鲫品系的大部分单倍型紧密聚集为一支,但各群体尚未形成单系群;NCRC品系聚在该分支外部,也未形成单系群;合方鲫品系与日本白鲫聚在一起,以较高的置信度(BI=1,ML=96/99,NJ=100)单独形成一个分支,与黑鲫、鲤共同组成系统发育进化树的基部。

图2 基于线粒体COI基因和D-loop区联合序列构建的BI系统发育进化树Fig.2 BI phylogenetic tree based on joint sequences of mitochondrial COI gene and D-loop region
2.3 群体遗传结构特征
基于线粒体COI基因和D-loop区联合序列,对6个鲫群体进行遗传距离和群体间遗传分化分析,结果如表2所示。6个鲫群体的群体内平均遗传距离为0.0027,遗传距离最小的是合方鲫品系(0.0009),最大的是常德野生鲫群体(0.0054);群体间的平均遗传距离为0.0253,其中,望城野生鲫群体与长沙野生鲫群体间的遗传距离最小(0.0038),合方鲫品系与其他5个鲫群体间的遗传距离均较大,为0.0539~0.0628。6个鲫群体两两间均可检测到遗传分化,其FST介于0.05417~0.98331;长沙野生鲫群体与常德野生鲫群体间的FST最小,为0.05417,达显著水平(P<0.05,下同);合方鲫品系与其他5个鲫群体间的FST较大(0.95147~0.98331),且均达极显著水平(P<0.01,下同)。

表2 基于线粒体COI基因和D-loop区联合序列的6个鲫群体遗传分化和遗传距离分析结果Table 2 Genetic differentiation and genetic distances analysis of the 6 curcian carp populations based on joint sequences of mitochondrial COI gene and D-loop region
基于线粒体COI基因和D-loop区联合序列,对6个鲫群体进行Structure聚类分析,推测群体遗传结构。设群体分组数目K为1~6,各种分组重复运算10次,基于ΔK的评判标准,较合适的分组方式如图3望城野生鲫WC长沙野生鲫CSX常德野生鲫CD实验红鲫RCC合方鲫HFJ新型同源二倍体类鲫NCRC所示。当6个鲫群体分为2组时(K=2),合方鲫品系独自为一组,其他5个鲫群体聚为一组;分为3组时(K=3),合方鲫品系和NCRC品系各自为一组,其余4个鲫群体聚为一组;分为4组时(K=4),合方鲫品系、NCRC品系和实验红鲫品系各自为一组,而3个野生鲫群体聚为一组。

图3 基于线粒体COI 基因和D-loop区联合序列的6个鲫群体遗传结构Fig.3 Genetic structure based on joint sequences of mitochondrial COI gene and D-loop region of the 6 crucian carp populations
基于Structure聚类分析结果的3种分组方式,对6个鲫群体进行AMOVA分析,进一步探究其群体结构,以揭示群体的遗传变异来源。结果(表3)显示,在K=2、K=3、K=4不同分组方式下,均显示遗传变异主要来自于组间(约90.00%),但组间遗传分化系数(FCT)不具显著性(P>0.05);组内群体间和群体内的遗传变异相对较小(低于10.00%),但组内的群体间遗传分化系数(FSC)和FST均达极显著水平。此外,组间变异占比随分组增多而升高,当K=4时拥有最大组间变异(90.14%),说明2个杂交品系及实验红鲫品系与3个野生鲫群体间已出现一定的遗传分化。
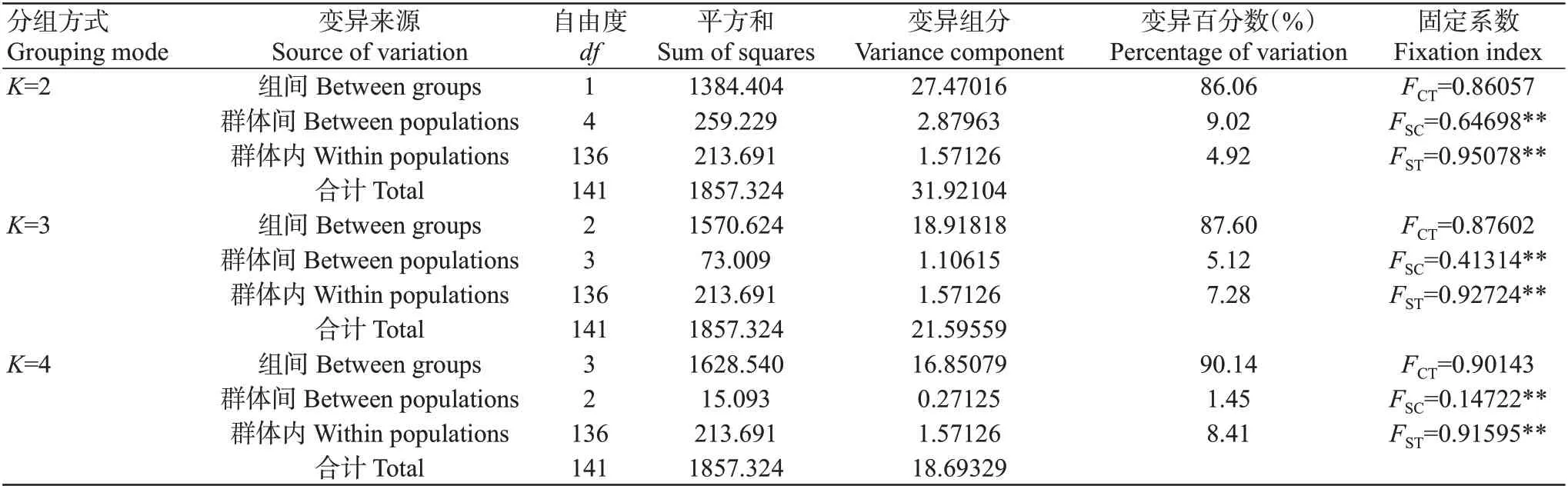
表3 基于线粒体COI基因和D-loop区联合序列的6个鲫群体AMOVA分析结果Table 3 AMOVA analysis of joint sequences of mitochondrial COI gene and D-loop region of the 6 crucian carp populations
2.4 群体历史动态检验结果
基于线粒体COI基因和D-loop区合序列,利用Tajima’sD和Fu’sFs模型对6个鲫群体的群体历史动态进行检验,结果(表4)显示,NCRC品系的Tajima’sD检验为显著性负值,表明其可能经历过群体扩张事件,或受到选择压力及瓶颈效应的作用;其他群体的Tajima’sD和Fu’sFs检验结果均为非显著性偏离零值,符合中性进化的假设。但核苷酸错配分布分析结果(图4)显示,6个鲫群体的核苷酸错配分布曲线均呈现多峰,表明各群体在近期历史上并未经历过明显的群体扩张。

图4 基于线粒体COI基因和D-loop区联合序列的6个鲫群体核苷酸错配分布检验结果Fig.4 Nucleotide mismatch distribution analysis based on mitochondrial COI gene and D-loop region of the 6 crucian carp populations

表4 基于线粒体COI基因和D-loop区联合序列的6个鲫群体中性检验分析结果Table 4 Neutrality test analysis based on mitochondrial COI gene and D-loop region of the 6 crucian carp populations
3 讨论
遗传多样性分析是研究和保护生物多样性的重要环节。本研究对6个鲫群体的线粒体COI基因和D-loop区进行扩增测序,并进行群体遗传多样性分析,结果显示,鲫线粒体COI基因和D-loop区序列均具有明显的A/T偏好,符合硬骨鱼线粒体基因组的序列特征。根据Grant和Bowen(1998)提出的群体多样性判断标准,无论是线粒体COI基因、D-loop区序列还是二者联合序列的分析结果均显示3个野生鲫群体为高单倍型多样性(h>0.500),而合方鲫品系、NCRC品系和实验红鲫品系为低单倍型多样性(h<0.500);6个鲫群体的线粒体COI基因均为低核苷酸多样性(π<0.005),而D-loop区序列表现为长沙野生鲫群体和常德野生鲫群体为高核苷酸多样性(π>0.005),二者联合序列显示常德野生鲫群体为高核苷酸多样性。D-loop处于线粒体非编码区,受到的选择压力较小,在大部分鲫样本中线粒体D-loop区的遗传变异程度明显高于线粒体COI基因,因此二者联合分析更能反应群体遗传多样性的变化情况。邓朝阳(2015)基于线粒体Cytb基因和D-loop区序列比较了彭泽鲫、方正银鲫、异育银鲫3个养殖品系和野生鲫群体间的遗传多样性及群体结构,结果发现养殖群体的遗传多样性明显低于野生鲫群体。类似研究结果在翘嘴鳜(成为为等,2020)、草鱼(欧阳美等,2021)、岩原鲤(岳华梅等,2021)、黄姑鱼(赵祥等,2021)和小黄鱼(郭丹丹等,2022)等鱼类的养殖群体中也得到证实,说明经济鱼类在人工繁殖过程中普遍存在多态性降低的现象,因此需要警惕养殖鱼类种质衰退等问题。
通过系统发育进化树和单倍型网络结构的构建,对6个鲫群体间的遗传进化关系进行推测,结果发现3个野生鲫群体并未按照采样地进行聚类,说明各地域野生鲫群体间的差异不显著,与刘科均(2020)收集分析湖南省内包括“湘资沅澧”流域及洞庭湖水域共41个地点鲫样本的结论一致,即各群体的形态差异和遗传分化未达亚种水平,尚未形成较稳定的地方群体。此外,虽然实验红鲫品系与野生鲫群体间不存在共享单倍型,但紧密聚集在一起,未能形成独立一支,可能与本研究选用的分子标记较少有关。王余德等(2021)利用线粒体4个基因片段(COI、COII、Cytb基因和D-loop区)的联合序列实现了对实验红鲫品系的有效鉴别。NCRC品系虽然与实验红鲫品系和野生鲫群体间存在共享单倍型,但相距较远。Wang等(2020)对NCRC(F1~F3)全线粒体基因组进行测序分析,结果发现其线粒体DNA与母本(鲤)差异较大,而与鲫更相似。Gu等(2022)利用线粒体基因和核基因标记研究NCRC的起源,结果显示其与鲫复合体间存在非常亲密的种内关系。综合本研究结果,推测NCRC确属于鲫复合体的一种,但与野生鲫也有一定的遗传差异。本研究还发现,在系统发育进化树上合方鲫品系的单倍型独自聚在一起,并与日本白鲫聚为一支,表明其线粒体来源于母本(日本白鲫),与刘庆峰(2018)的研究结果一致。同时,本研究再次验证了白鲫物种的独立性,均符合此前研究中鲫属的物种划分标准(Murakami et al.,2001;刘良国等,2010;Takada et al.,2010;程磊等,2012;Gao et al.,2012)。
在DNA条形码技术中,通常将1%~2%的种间遗传距离作为物种鉴定的标准(Hebert et al.,2003a,2003b)。本研究进一步利用遗传距离和群体间的FST来检测各鲫群体的遗传分化水平,结果显示,6个鲫群体的群体内遗传距离均小于1%,说明各群体内部的遗传变异较小。两两群体间的遗传距离分析也发现,3个野生鲫群体和实验红鲫品系间的遗传距离均小于1%,遗传差异不明显;NCRC品系与这4个鲫群体间的遗传距离处于1%~2%,出现一定的遗传差异;合方鲫品系与其他5个群体间的遗传距离均大于2%,高达5%~7%,遗传差异较大。根据Wright(1965)提出基于FST判断群体间遗传分化程度的标准,本研究中3个野生鲫群体间的遗传分化较小(0.05<FST<0.15),而实验红鲫品系、合方鲫品系及NCRC品系与除各自群体外的其他5个鲫群体均已产生高度遗传分化(FST>0.25)。可见,3个野生鲫群体间遗传差异较小,遗传分化不明显;实验红鲫品系与野生鲫群体间的遗传差异虽然较小,但已产生明显的遗传分化;NCRC品系与其他鲫品系的遗传差异较大,遗传分化明显;合方鲫品系与其他鲫群体的遗传差异和遗传分化均最明显。本研究的AMOVA分析结果展示了3种较理想的分组情况(K=2、K=3和K=4),且发现随着分组数目的增加,组间遗传变异百分比也逐渐增加,在分组结果中依次将合方鲫品系、NCRC品系、实验红鲫品系与野生鲫群体分离开,进一步佐证了系统发育进化和遗传分化分析的结果。合方鲫品系的遗传分化是因其母本白鲫与鲫复合种不属于同一物种所导致(刘庆峰,2018);实验红鲫品系的遗传分化可能与养殖过程中的瓶颈效应、遗传漂变等因素有关;而NCRC品系的遗传分化除了受瓶颈效应、遗传漂变等因素影响外,可能还与其杂交起源及复杂的遗传背景相关联(Wang et al.,2020);野生群体间并未产生明显的遗传分化可能与各地水域开放交互导致的鲫群体繁育自由、基因流频繁等因素有关(刘科均,2020)。
从群体历史动态检验结果来看,在Tajima’sD检验中仅有NCRC品系为显著性负值,表明其可能发生过规模扩张或定向选择;但在Fu'sFs检验中6个鲫群体的结果均不显著,核苷酸错配分布曲线也呈现多峰,考虑到NCRC品系的Tajima’sD检验结果并未达极显著水平,故推测6个鲫群体在近期历史上保持相对稳定,未曾经历过明显的群体扩张。
4 结论
合方鲫品系、NCRC品系及实验红鲫品系3个养殖群体与3个野生鲫群体间已产生明显的遗传分化,且存在一定程度的遗传多样性降低等问题。因此,在后续的鲫育种工作中应适当扩大繁育亲本数量,避免近亲繁殖和瓶颈效应,注重保护养殖群体种质资源的遗传多样性。

