盐芥叶片应答盐胁迫的蛋白质组学分析
常丽丽 彭存智 王丹 仝征 黄超 徐兵强






摘要: 盐芥是研究耐盐机理的模式植物。为从蛋白质水平揭示盐芥响应盐胁迫的分子机制,本研究采用同位素标记相对和绝对定量(iTRAQ) 技術对不同NaCl浓度处理7 d的盐芥叶片进行差异蛋白质组学分析。结果表明,盐芥叶片中共鉴定到4 607个蛋白质,其中281个蛋白质的表达丰度显著增加,95个蛋白质的表达丰度显著降低。盐胁迫差异表达蛋白质的KEGG代谢通路和蛋白质互作网络分析结果表明,促进植株光合作用可帮助盐芥适应低盐环境;抑制叶绿素和支链氨基酸合成、调控应激反应基因的表达是盐芥应对中盐环境的重要因素;而有效清除活性氧、提高渗透物质积累量和增加能量供应可能是盐芥耐受高盐环境的关键。本研究结果为揭示盐芥应答盐胁迫的分子响应机制提供了理论基础。
关键词: 盐芥;盐胁迫;差异蛋白质;iTRAQ
中图分类号: 文献标识码: A 文章编号: 1000-4440(2022)01-0049-16
Abstract: Eutrema salsugineum is a model plant for salt tolerance study. To reveal the molecular mechanism of E. salsugineum in response to salt stress at protein level, isobaric tags for relative and absolute quantification (iTRAQ) technology was used to do differential proteomics analysis on E. salsugineum leaves treated under different NaCl concentrations for seven days. The results showed that, a total of 4 607 proteins were identified from leaves of E. salsugineum, among which the expressional abundance of 281 proteins were significantly up-regulated, while the expressional abundance of 95 proteins were significantly down-regulated. Results of KEGG metabolic pathway and protein-protein interaction network analysis for differentially expressed proteins under salt stress showed that, promotion of photosynthesis was helpful for E. salsugineum to adapt to environment with low salinity. Inhibition of chlorophyll and branched chain amino acids synthesis, as well as expression regulation of stress-responsive genes were important factors for E. salsugineum to survive in an environment with medium salinity. Moreover, the key points for E. salsugineum to tolerate high salinity condition might be effective elimination of reactive oxygen species (ROS), elevating accumulation of osmolytes and increasing energy supply. The results can provide theoretical basis for revealing molecular mechanism of E. salsugineum in response to salt stress.
Key words: Eutrema salsugineum;salt stress;differentially expressed protein;isobaric tags for relative and absolute quantification (iTRAQ)
盐胁迫是自然界中主要的非生物胁迫之一,土壤中的高浓度盐分会使植物体内离子失衡,产生渗透压力,从而严重影响植物的生长和发育[1]。盐芥是一年生草本植物,属真盐生植物,多生长于盐渍化土壤中。盐芥是拟南芥的近亲[2-3],同样具有生长周期短、基因组构成小(约为拟南芥的2倍)、种子数目多、可利用农杆菌侵染花序法进行遗传转化试验、遗传转化效率高等特点。因此,近几年盐芥被提出作为耐盐分子机理研究的理想模式植物[2,4-5]。虽然盐芥和拟南芥有很多相似的特性,但两者之间的耐盐性却存在很大差异。通过转录组和蛋白质组学技术分析拟南芥和盐芥在响应盐胁迫的差异时,发现拟南芥中存在的盐胁迫响应基因,不管是否处于盐胁迫环境,在盐芥中均能高丰度表达[6-7],说明盐芥在面对逆境时是“有备无患”;而且盐芥中存在特有的新陈代谢途径,使得其在盐胁迫条件下能减少由植物激素所诱导的生理修饰[8],从而能在一定程度上承受更大的胁迫压力。同时研究发现,盐胁迫条件下,盐芥根系的磷酸化蛋白质组发生改变,这些变化的磷酸化蛋白质参与了信号转导、活性氧清除、能量途径、蛋白质合成以及蛋白质折叠等过程[9],这些研究为将来从磷酸化水平上研究盐芥的耐盐机制提供了重要的理论依据。
目前,人们已从盐芥中鉴定到一些与盐胁迫耐受相关的基因和调控因子,如编码K+/Na+转运蛋白基因(ThHKT1)[10-11]、编码細胞质膜和液泡膜Na+/H+逆向转运蛋白基因(ThNHX1)和(ThSOS1)[12-14]、焦磷酸酶基因(TsVP) [15-17]、高亲和性K+转运体(ThHAK5)[18]、脯氨酸合成基因(ThP5CS)[19]、Cu/Zn超氧化物歧化酶基因(ThCSD)[20]等,且部分基因的生物学功能已得到一定的阐述。我们前期对盐芥叶片和叶绿体响应盐胁迫的比较蛋白质组学的研究结果表明,盐芥可通过Na+液泡区隔化,积累渗透调节物质(如淀粉、可溶性糖、脯氨酸等),以及维持光合效率和生长发育等方式来适应盐胁迫环境[21-22]。本研究在已有的研究基础上,应用先进的同位素相对标记与绝对定量技术(iTRAQ)对不同盐浓度处理条件下的盐芥叶片进行比较蛋白质组学研究,并重点分析不同盐浓度处理对盐芥叶片蛋白质表达丰度的影响,期望揭示盐芥适应不同盐浓度条件的蛋白质化学机制,为揭示盐芥耐盐分子机制提供参考。
1 材料与方法
1.1 试验材料与试验设计
将盐芥种子直接点播在基质土(营养土∶蛭石=1∶1,体积比)中,放置在培养箱中进行培养。培养条件:温度为白天22 ℃,晚上20 ℃;相对湿度为60%±5%;16 h光照,8 h黑暗。种子发芽后,用1/2浓度Hoagland营养液每3 d浇灌1次。2个月后,挑选长势一致的盐芥植株用含有0 mmol/L、200 mmol/L(低盐)、400 mmol/L(中盐)和600 mmol/L(高盐)NaCl的1/2浓度Hoagland营养液进行浇灌,每天更换新的营养液,每个盐浓度处理36株植株。收集连续浇灌7 d(根据前期的预试验确定的盐处理时间)的盐芥叶片,每12株为1个生物学重复,液氮速冻,-80 ℃保存备用。
1.2 盐芥叶片蛋白质的提取、酶解及iTRAQ分析
参照Wang等[23]的BPP法提取盐芥叶片总蛋白质,用预冷的甲醇和丙酮清洗蛋白质沉淀,然后溶解在蛋白质裂解液(7 mol/L尿素,2 mol/L硫脲,4% CHAPS(一种非变性的两性离子型去垢剂),30 mmol/L Tris-HCl,pH 8.5)中。以牛血清白蛋白(BSA)为标准溶液,通过Bradford测定法测定蛋白质浓度[24]。取100 μg用不同浓度的NaCl处理的盐芥叶片总蛋白质,用胰蛋白酶进行消化后,使用iTRAQ试剂分别标记0 mmol/L、200 mmol/L、400 mmol/L和 600 mmol/L NaCl处理的上述叶片总蛋白质酶解产物后,再进行等量混合。采用强阳离子变换(SCX)色谱柱(4.6 mm×250.0 mm, Aqua C18, 5 μm, 100 )对iTRAQ标记的肽段进行预分级,再通过液相串联质谱对标记肽进行鉴定,获取差异表达肽段信息。
1.3 质谱数据搜库
使用Proteinpilot软件(Version 4.5)对质谱结果进行搜库,搜索数据库为自建的盐芥蛋白质数据库,蛋白数据从美国国立生物技术信息中心(NCBI)网站(https://www.ncbi.nlm.nih.gov/genome/12266)上下载。搜库参数设置为:酶类为Trypsin,错误剪切位点数为1,固定修饰位点为Carbamidomethyl(C),可变修饰位点为Oxidation(M)。将0 mmol/L NaCl处理盐芥叶片蛋白质作为对照组,200 mmol/L NaCl、400 mmol/L NaCl、600 mmol/L NaCl作为处理组,按处理组与对照组离子的峰面积比值,选择置信度在95%以上的结果进行报告。质谱鉴定出的可信蛋白质筛选参数应满足:可信度在95%以上的肽置信水平,至少鉴定出2条肽段,且错误发现率(FDR)≤1%。
1.4 质谱数据生物信息学分析
通过KEGG途径分析对可信鉴定的蛋白质参与的代谢通路进行富集。同时,对盐胁迫差异表达蛋白质(变化倍数>1.50或<0.67,P<0.05)进行KEGG代谢途径和蛋白质间相互作用网络分析(https://string-db.org/cgi/input.pl)。
1.5 部分盐胁迫差异表达蛋白质对应基因的qRT-PCR分析
提取用不同盐浓度处理的盐芥叶片总RNA,取1 μg总RNA反转录成cDNA,并将反转录后的cDNA样品稀释5倍后用于qRT-PCR分析,每个样品至少重复3次qRT-PCR试验。取1 μl稀释后的cDNA加入到SYBR Green PCR Master mix体系中,使用Mx3005P荧光定量PCR仪进行qRT-PCR试验,盐芥的actin基因(NCBI基因登录号312283264)作为内参基因。用于qRT-PCR的引物序列详见表1。
2 结果与分析
2.1 盐芥叶片蛋白质种类分析及盐胁迫差异表达蛋白质的筛选
应用iTRAQ技术结合AB5600+高端生物质谱仪对盐芥叶片总蛋白质进行定量分析,3次iTRAQ重复鉴定出的可信蛋白质数分别为3 750个、3 897个和3 901个,共计4 607个蛋白质至少在1次重复中鉴定到的可信的盐芥叶片总蛋白质,其中3 811个蛋白质至少在2次重复试验中得到可信鉴定(图1A)。这3 811个可信蛋白质参与了18条代谢途径,其中碳水化合物代谢途径参与的蛋白质种类最多(988个蛋白质),其次是翻译类蛋白质(895个蛋白质)与折叠、分类和降解类蛋白质(830个蛋白质)、氨基酸代谢(579个蛋白质)、信号转导(547个蛋白质)、运输和分解代谢(522个蛋白质)、脂质代谢(496个蛋白质)、环境适应(452种蛋白质)、能量代谢(416种蛋白质)等代谢途径类蛋白质(图1B)。
以0 mmol/L NaCl处理的盐芥叶片为对照,盐胁迫处理后共检测到376个盐胁迫差异表达蛋白质(变化倍数>1.5或<0.67,P<0.05)(图1C),其中281个蛋白质的表达丰度显著增加,95个蛋白质的表达丰度显著降低;其中低盐处理(200 mmol/L NaCl)检测到57个差异表达的蛋白质,其中表达丰度增加的蛋白质43个,丰度降低的蛋白质14个;中盐环境(400 mmol/L NaCl)引起108个蛋白质的表达丰度发生显著改变,24个显著增加,84个显著降低;高盐条件下(600 mmol/L NaCl)共有263个蛋白质的表达丰度发生显著改变,252个表达丰度增加,11个表达丰度降低。通过比较不同盐浓度处理条件下的差异表达蛋白质数量发现,盐处理浓度越高,表达丰度发生显著改变的蛋白质数目越多,说明盐芥植株需要调控更多蛋白质的表达来应对高盐环境。
2.2 盐胁迫差异表达蛋白质的KEGG代谢通路分析
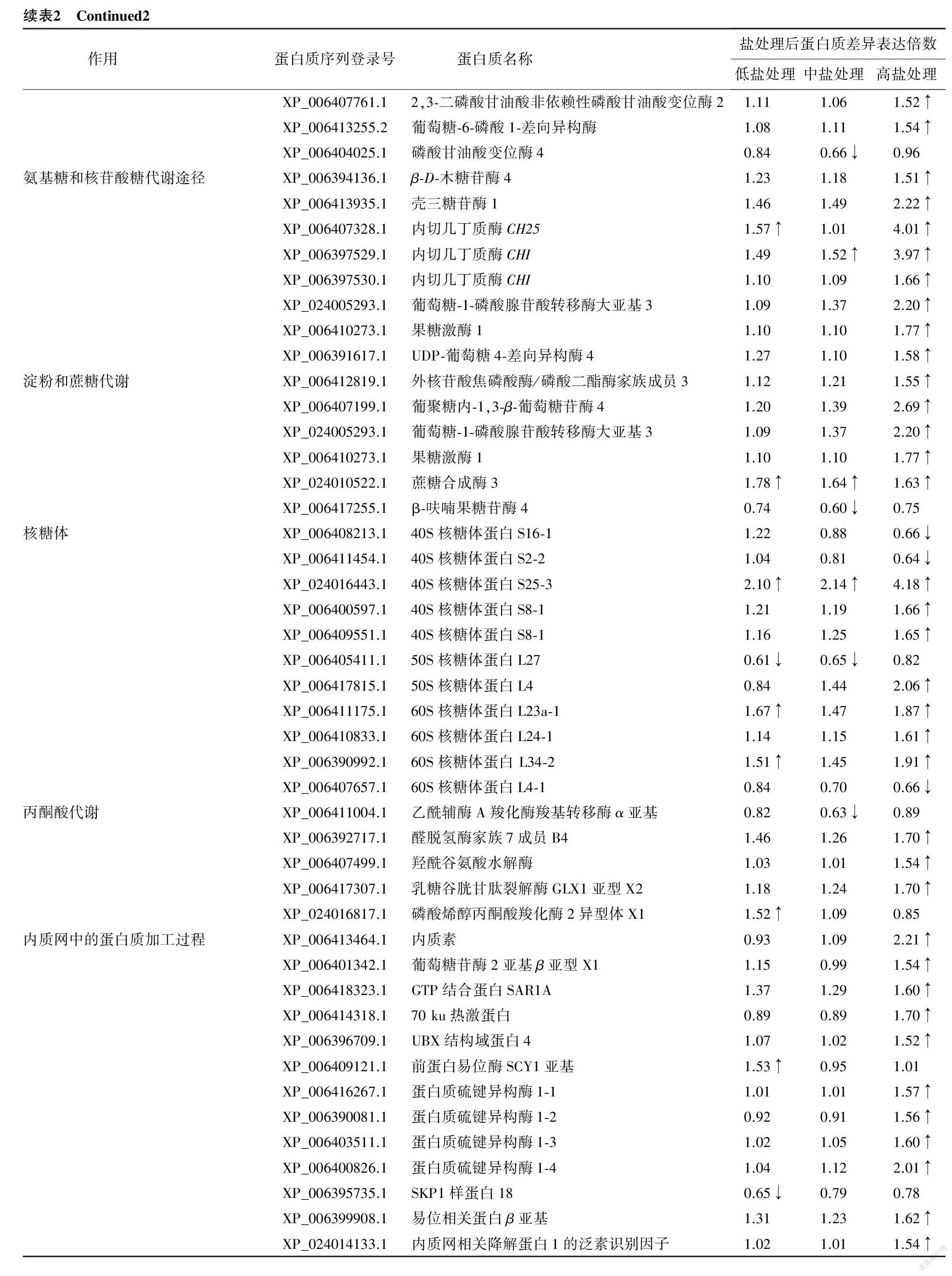
对376个盐胁迫差异表达蛋白质参与的代谢通路进行分析,发现它们主要富集到26条代谢途径中(仅统计蛋白质数量≥4个的代谢途径)(图2、表2),包括能量代谢、其他氨基酸代谢、氨基酸代谢、碳水化合物代谢途径等。其中能量代谢包括光合作用-天线蛋白、氧化磷酸化和硫代谢;谷胱甘肽代谢、硒化合物代谢和β-丙氨酸代谢属于其他氨基酸代谢途径;缬氨酸、亮氨酸和异亮氨酸生物合成、酪氨酸代谢、精氨酸和脯氨酸代谢、半胱氨酸和甲硫氨酸代谢、甘氨酸、丝氨酸和苏氨酸代谢属于氨基酸代谢;碳水化合物代谢途径包括半乳糖代谢、氨基糖和核苷酸糖代谢、糖酵解/糖异生、丙酮酸代谢、乙醛酸和二羧酸代谢、淀粉和蔗糖代谢。此外,卟啉和叶绿素代谢、核糖体、RNA转运、内质网中的蛋白质加工过程、苯丙素生物合成、嘌呤代谢、2-氧代羧酸代谢等代谢途径也受到盐胁迫的影响(图2)。
进一步比较不同盐浓度处理条件下差异表达蛋白质富集的代谢通路后发现,低盐浓度处理显著诱导了光合作用天线蛋白和参与氧化磷酸化途径的蛋白质的丰度积累,包括叶绿素a/b结合蛋白1(CP1,XP_006415537.1)、CP2(XP_024012787.1)、CP4(XP_006404376.1)、CP6(XP_006403520.1)、CP24 10B(XP_006416877.1)、光系统Ⅰ反应中心亚基Ⅲ(PsaF,XP_006415339.1)和ATP酶亚基8(ATPase8,XP_006397344.2)。中等盐度处理却显著降低了大部分参与卟啉和叶绿素代谢、缬氨酸生物合成、亮氨酸生物合成和异亮氨酸生物合成以及半胱氨酸和甲硫氨酸代谢途径蛋白质的表达,其中参与卟啉和叶绿素代谢途径的蛋白质为胆色素原脱氨酶(PBGD,XP_006399317.1)、尿卟啉原脱羧酶2(UROD2,XP_006411313.1)、谷氨酸-1-半醛2,1-氨基变位酶2(GSAM2,XP_006404223.1)、原叶绿素还原酶B(PORB,XP_006413074.1)、镁螯合酶亚基ChlI-1(CHLI1, XP_006414096.1),以及脱镁叶绿素加氧酶(PAO, XP_006419093.1);参与缬氨酸、亮氨酸和异亮氨酸生物合成途径的蛋白质包括3-异丙基苹果酸脱氢酶3(IMDH3,XP_006399931.1)、3-异丙基苹果酸脱水酶小亚基1(IPMD-S1,XP_006411585.1)、乙酰乳酸合酶1(ALS1,XP_006404237.1)、蘇氨酸脱水酶(THD1,XP_024015515.1);参与半胱氨酸和甲硫氨酸代谢途径的蛋白质有6个,包括1-氨基环丙烷-1-羧酸氧化酶2(ACO2,XP_006391897.1)、ACO4(XP_006418070.1)、S-腺苷甲硫氨酸合酶(SAMS,XP_006396376.1)、SAMS2(XP_006397900.1和 XP_006406736.1)、SAMS3(XP_006410860.1)和胱氨酸裂解酶(CORI3,XP_006413537.1)(图3)。高盐浓度处理条件下,大部分参与谷胱甘肽代谢、精氨酸和脯氨酸代谢、糖降解/糖异生,以及氨基糖和核苷糖代谢途径的蛋白质表达丰度显著增加,包括10种谷胱甘肽S-转移酶(GST-Z1、GST-U16、GST-U17、GST-U19、GST-U27、GST-F3、GST-F8、GST-F9、GST-DJAR2和GST-DHAR1)、磷脂氢过氧化物谷胱甘肽过氧化物酶6(PHGP6,XP_006396880.1)、谷胱甘肽过氧化物酶7(GPX7,XP_006412543.1),以及吡咯啉-5-羧酸还原酶(PYCR,XP_006399997.1)、δ-1-吡咯啉-5-羧酸合酶A(P5CS-A,XP_006411212.1)、精氨酸酶1(ARG1,XP_006397249.1)、醛脱氢酶家族7成员B4(ALDH7B4,XP_006392717.1)、磷酸甘油酸变位酶4(PGM4,XP_006404025.1)、醛糖1-差向异构酶(ALE1,XP_006404339.1)、2,3-二磷酸甘油酸非依赖性磷酸甘油酸变位酶2(PMG2,XP_006407761.1)、NADPH依赖性醛酮还原酶(AKR,XP_006410968.1)、葡萄糖-6-磷酸1-差向异构酶(GLPE,XP_006413255.2)、UDP-葡萄糖4-差向异构酶4(UGE4,XP_006391617.1)、β-D-木糖苷酶4(XYL4,XP_006394136.1)、内切几丁质酶(CHI,XP_006397529.1和XP_006397530.1)和CH25(XP_006407328.1)、果糖激酶1(FRK1,XP_006410273.1)、壳三糖苷酶1(CHIT1,XP_006413935.1)和葡萄糖-1-磷酸腺苷酸转移酶大亚基3(GLGL3,XP_024005293.1)(图3)。这些结果表明,盐芥可能通过调控不同的代谢途径来适应不同的盐胁迫环境。
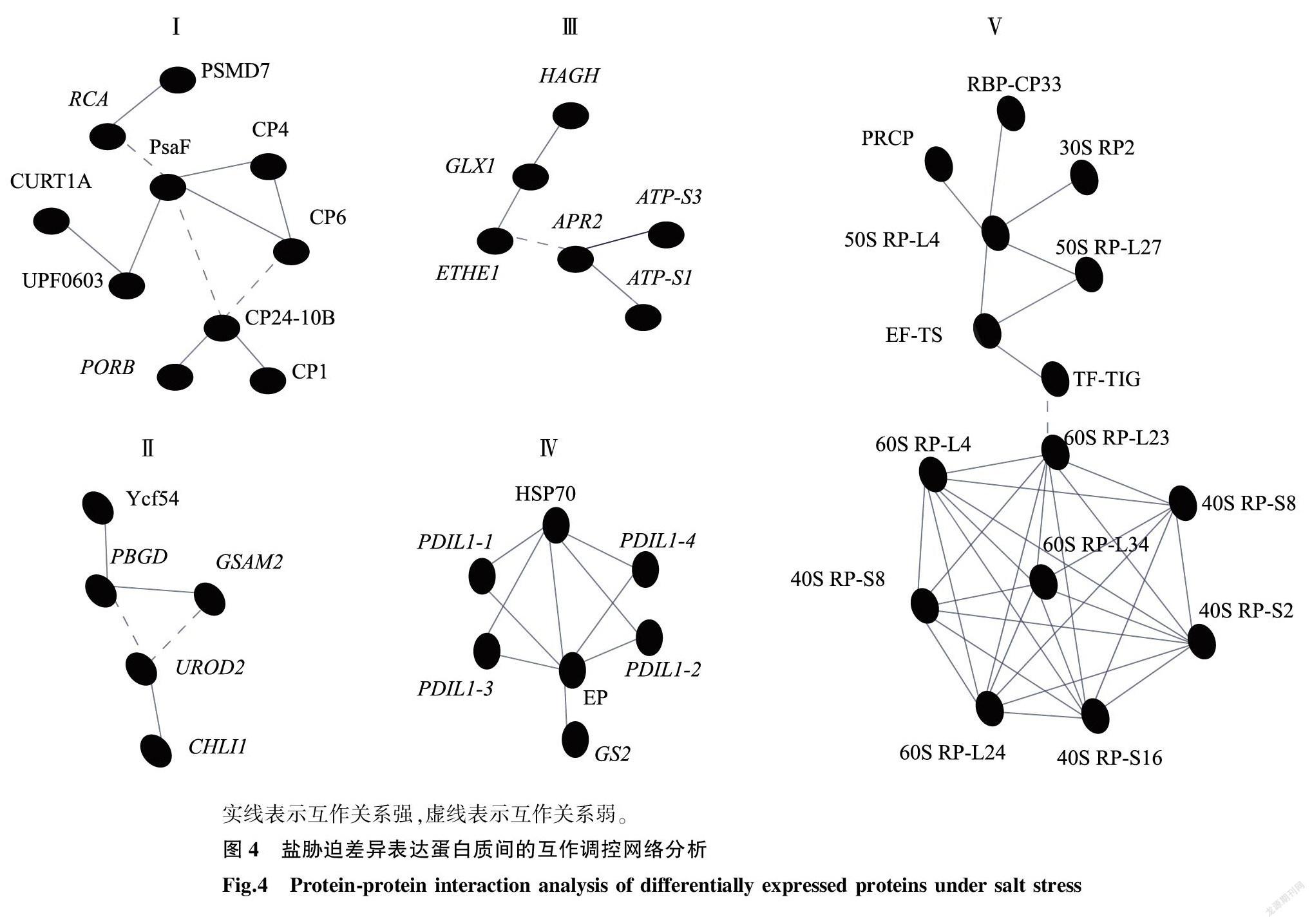
2.3 盐胁迫差异表达蛋白质间的互作调控网络分析
STRING数据库可用来分析蛋白质间的相互作用,本研究采用最高置信度(得分大于0.9)和马尔可夫聚类(MCL)算法来呈现盐胁迫差异表达蛋白质之间的互作调控网络,结果如图4所示。第Ⅰ类蛋白质主要参与光合作用、叶绿素合成和蛋白质降解途径,含有10个蛋白质:CP1、CP4、CP6、CP24-10B、PsaF、核酮糖二磷酸羧化酶/加氧酶(Rubisco)活化酶(RCA,XP_006411205.1)、弯曲类囊体蛋白1A(CURT1A,XP_006396295.1)、PORB、26S蛋白酶体非ATP酶调节亚基7(PSMD7,XP_006399036.1)和UPF0603蛋白At1g54780(UPF0603,XP_006392487.1)。第Ⅱ类包括5个蛋白质,主要参与卟啉和叶绿素代谢,分别是GSAM2、PBGD、UROD2、CHLI1和未知蛋白质LOC18016835(Ycf54,XP_006401109.1)。第Ⅲ类包括6个蛋白质,主要参与硫代谢和丙酮酸代谢,包括5′-腺苷酸还原酶2(APR2,XP_006391919.1)、过硫化双加氧酶ETHE1(ETHE1,XP_006392779.1)、羟酰谷氨酸水解酶(HAGH,XP_006407499.1)、ATP-硫酰酶1(ATP-S1,XP_006406118.1)和ATP-硫酰酶3(ATP-S3,XP_006414665.1)、乳酰谷胱甘肽裂解酶(GLX1,XP_006417306.1)。第Ⅳ类含有7个蛋白质,包括葡萄糖苷酶2(GS2,XP_006401342.1)、内质素(EP,XP_006413464.1)、蛋白质二硫键异构酶1-1(PDIL1-1,XP_006416267.1)、蛋白质二硫键异构酶1-2(PDIL1-2,XP_006390081.1)、蛋白质二硫键异构酶1-3(PDIL1-3,XP_006403511.1)和蛋白质二硫键异构酶1-4(PDIL1-4,XP_006400826.1),以及70 000热激蛋白(HSP70,XP_006414318.1),这些蛋白质主要参与内质网中的蛋白质加工过程。第V类含有15个蛋白质,包括RNA结合蛋白CP33(RBP-CP33,XP_006403809.1)、含五肽重复序列蛋白(PRCP,XP_006418504.1)、延伸因子(EF-TS,XP_006412882.1)、触发因子蛋白TIG(TF-TIG,XP_006401478.1),以及11个核糖体蛋白(RP),如30S RP2(XP_006403837.1)、40S RP-S2(XP_006411454.1)、40S RP-S8(XP_006400597.1, XP_006409551.1)、40S RP-S16(XP_006408213.1)、50S RP-L4(XP_006417815.1)、50S RP-L27(XP_006405411.1)、60S RP-L4(XP_006407657.1)、60S RP-L23(XP_006411174.1)、60S RP-L24(XP_006410833.1)和60S RP-L34(XP_006390992.1),这些蛋白质主要参与蛋白质翻译过程。蛋白质-蛋白质相互作用分析结果表明,盐芥可通过多条代谢途径间的相互协同作用来适应盐胁迫环境。
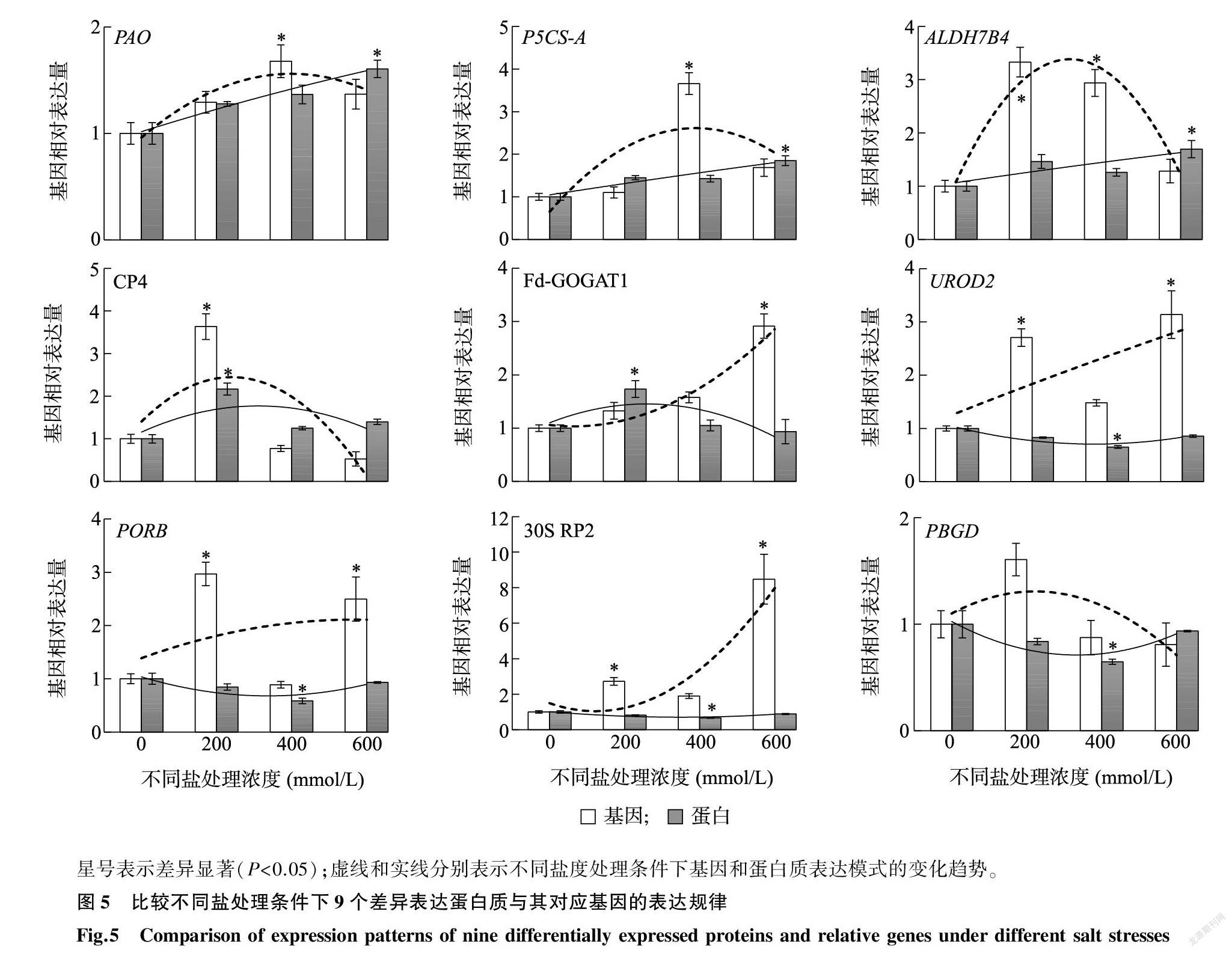
2.4 部分盐胁迫差异表达蛋白质的基因表达规律分析
为了评估蛋白质表达丰度与其相应基因表达水平之间的相关性,本研究利用qRT-PCR对9个典型的盐胁迫差异表达蛋白质进行基因表达分析。结果(图5)表明,8个盐胁迫差异表达蛋白质的编码基因在盐处理后显著上调表达,仅1个蛋白质(PBGD)对应的基因表达水平在盐处理后无明显变化。其中CP4基因表达水平仅在低盐条件下显著升高,PAO和P5CS-A基因表达水平在中盐环境下显著升高,而Fd-GOGAT1基因表达水平在高盐处理条件下显著升高。低盐和中盐处理显著诱导了ALDH7B4基因的表达,而低盐和高盐环境却显著上调UROD2、PORB和30S RP2基因的表达(图5)。
比较9个盐胁迫差异表达蛋白质与其相应基因的表达变化趋势后发现,PAO、P5CS-A和CP4的蛋白质表达模式与其编码基因的表达变化规律基本相似,而ALDH7B4、Fd-GOGAT1、UROD2、PORB、30S RP2和PBGD的蛋白质与编码基因的表达变化趋势却不一致(图5)。
盐胁迫处理后基因表达水平和蛋白质表达丰度不一致的现象也在其他植物中出现,如小麦[25]、甜菜[26]等,具体原因还需要进一步分析才能确定。
基于上述研究结果和已有的文献报道,我们提出一个盐芥适应不同盐浓度胁迫环境的可能模式(图6)。
3 讨论
当植物受到高盐胁迫时,植物体内的离子和渗透压会失去平衡,并由此产生次级胁迫(如氧化胁迫),从而严重影响植株的生长和发育,甚至导致植株死亡[1]。植物通过建立新的离子和渗透动态平衡、缓解或消除氧化损害、调控生长等措施来适应新的胁迫环境[5]。已有研究结果表明,在盐胁迫条件下,盐芥主要将Na+累积在地上部分,并将Na+泵入液泡内;同时,盐芥的渗透调节能力也较强,可通过降低生长速率来适应渗透胁迫[27]。本研究利用定量蛋白质组学iTRAQ技术,对0 mmol/L、200 mmol/L、400 mmol/L和600 mmol/L NaCl处理7 d条件下的盐芥叶片进行差异蛋白质组学分析,发现376个蛋白质的表达丰度在盐处理后发生显著改变。根据这些差异表达蛋白质的功能分类和代谢途径分析,推测盐芥适应不同盐浓度胁迫处理7 d条件下的可能分子机制。
3.1 增强的光合作用和能量供应可能是盐芥适应低盐环境的关键
对许多真盐生植物来说,适当的盐浓度可以促进它们的生长和发育。研究结果表明,300 mmol/L NaCl处理可显著促进真盐生植物盐角草的叶片肉质化和茎直径增加[28];盐生植物海马齿在200~300 mmol/L NaCl处理条件下表现出最佳的生长状态[29];200 mmol/L NaCl处理可显著增加盐地碱蓬的根系干质量和鲜质量以及根系的总长度[30]。100 mmol/L NaCl处理盐芥表现出最佳生长状态,而200 mmol/L和400 mmol/L NaCl处理抑制了叶片的生长发育,但没有表现出任何毒害症状[31],这与我们以前的研究结果[21]相一致。本研究发现光合作用天线蛋白的基因和蛋白质的表达丰度在200 mmol/L NaCl处理条件下均显著增加,结合未发生显著变化的叶绿体显微结构[22,31]以及升高的总叶绿素含量和叶绿素a与叶绿素b比值的研究结果[21,31],推測盐芥可能通过增加叶绿素含量和提高光合作用天线蛋白的表达量来吸收更多的光能。同时,200 mmol/LNaCl处理可诱导PsaF和ATPase8上调表达,将吸收的更多光能传递到光反应中心,促进盐芥的光合作用,从而合成更多的有机物和增加能量供应来帮助盐芥应对低盐环境,这些研究结果与部分植物[32-33]响应盐胁迫的比较蛋白质组学研究结果相一致。
3.2 抑制叶绿素与支链氨基酸的合成和调控胁迫响应基因的表达可能是盐芥在中盐环境下存活的重要因素
光合色素是光合作用系统的重要组成部分,主要包括高等植物中的叶绿素a、叶绿素b和类胡萝卜素,其中叶绿素是主要的捕光和反应色素。许多研究结果表明,盐胁迫通常会降低植物的叶绿素含量[34-36]。同样,我们之前的研究发现400 mmol/L和600 mmol/LNaCl处理均显著降低了盐芥叶片的叶绿素含量[21]。本研究共鉴定到6个参与卟啉和叶绿素代谢的盐胁迫差异表达蛋白质(PBGD、UROD2、PORB、GSAM2、CHLI1和PAO),其中PBGD可催化4个胆色素原分子转化为尿卟啉原Ⅲ,是叶绿素生物合成的关键步骤[37];UROD催化尿卟啉原Ⅲ脱羧为共卟啉原Ⅲ[38];PORB是叶绿素生物合成中的关键酶,可催化原叶绿素还原为叶绿素[39];GSAM催化谷氨酸-1-半醛异构化为5-氨基乙酰丙酸,而5-氨基乙酰丙酸是四吡咯(如叶绿素和血红素)的通用前体[40];CHLI1是Mg螯合酶的一个亚基,可将Mg2+插入原卟啉IX中,完成叶绿素生物合成的第一步[41];PAO是叶绿素分解代谢的关键调节酶[42]。在盐胁迫条件下,水稻幼苗中PBGD的活性[43]和番茄幼苗中PBGD的表达水平[44]均显著下降;耐盐和盐敏感棉花中PORB的表达丰度亦受到显著抑制[45]。同时,盐胁迫下水稻幼苗、番茄幼苗和盐敏感棉花中的叶绿素含量也明显降低。本研究结果与这些研究结果相似,即参与叶绿素生物合成的蛋白质(PBGD、UROD2、PORB、GSAM2和CHLI1)的表达丰度在400 mmol/LNaCl处理后显著降低,而参与叶绿素分解代谢的PAO表达丰度却在600 mmol/LNaCl处理条件下显著增加,由此推测盐芥可能通过降低叶绿素生物合成中关键蛋白质的表达量来降低叶片中的叶绿素含量,从而适应中盐胁迫环境;而高盐胁迫环境增强了叶绿素的分解代谢,从而引起叶绿素含量降低。
非生物胁迫可强烈诱导支链氨基酸(BCAA)的降解[46-47]。亮氨酸、缬氨酸和异亮氨酸属于BCAA,但亮氨酸、缬氨酸和异亮氨酸在响应胁迫过程中的功能可能并不完全相同[48]。本研究共鉴定到4个盐胁迫差异表达蛋白质(IMDH3、IPMD-S1、ALS1和THD1)参与到缬氨酸、亮氨酸和异亮氨酸生物合成途径。已有研究结果表明,靶向干扰禾谷镰刀菌中编码IMDH的FgLEU2A基因的表达,可显著降低其对渗透和氧化胁迫的耐受性[49];反义抑制番茄IPMD的表达可提高果实中BCAA的水平[50];使用乙酰乳酸合成酶(ALS)抑制剂并不干扰干旱胁迫诱导的BCAA积累过程[51]。在本研究中,400 mmol/LNaCl处理显著降低了IMDH3、IPMD-S1、ALS1和THD1的表达量,表明中度盐胁迫环境可能抑制了BCAA的合成。
此外,大多数参与半胱氨酸和甲硫氨酸代谢途径的蛋白质在400 mmol/L NaCl处理后表达丰度明显降低,其中ACO是乙烯生物合成中的关键酶,可将1-氨基环丙烷-1-羧酸转化为乙烯。在拟南芥中过量表达小麦TaACO1基因,可通过调控DREB1/CBF信号转导途径中的胁迫响应基因来降低转基因株系的耐盐性[52];但过量表达拟南芥ACO基因却提高了转基因株系的耐涝性[53],这些结果表明不同植物中的ACO可能在响应非生物胁迫时发挥不同的功能。SAMS可催化甲硫氨酸和ATP生成S-腺苷-L-甲硫氨酸(SAM),而SAM参与了乙烯、烟草胺和多胺的生物合成。研究结果表明,非生物胁迫可显著诱导SAMS的表达[54],且拟南芥中异源表达马铃薯和甜菜的SAMS,可通过上调与胁迫反应相关的基因表达量来提高转基因株系的胁迫耐受性[55-56]。结合本研究结果和现有报道,推测盐芥可能通过降低半胱氨酸和甲硫氨酸代谢中关键蛋白质的表达丰度来调控胁迫响应基因的表达,从而适应中度盐胁迫环境。
3.3 有效清除活性氧、累积渗透调节物质、增加能量供应可能是盐芥耐受高盐胁迫环境的关键
植物在受到盐碱、干旱、低温等逆境胁迫时,体内活性氧物质(ROS)含量会迅速上升,当ROS积累到一定水平后,会对植物造成损伤,而植物主要通过酶促抗氧化系统和非酶促抗氧化系统来清除多余的ROS,以平衡体内的ROS水平[57]。谷胱甘肽(GSH)是植物中重要的抗氧化剂,细胞中GSH的水平与生物和非生物胁迫响应密切相关[58-59]。GPX和GST是谷胱甘肽代谢途径中的关键酶。GPX在胁迫响应过程中发挥多种功能,如维持H2O2稳态、形成蛋白质复合体参与胁迫防御、作为ROS或氧化还原传感器促进不同信号通路间的相互协作[60]。GST含有大量多功能酶,在GSH介导的氧化还原调控和细胞解毒过程中发挥重要作用[61]。有研究结果表明,GPX和GST的表达量上调与许多植物的耐逆性提高密切相关,如拟南芥[62-63]、水稻[64]、小麦[65-66]、大麦[67]、大豆[68]等。目前已在盐芥中鉴定到GPX基因家族,并证实盐和渗透处理可诱导大多数GPX基因及其蛋白质的表达丰度增加[69]。本研究发现在高盐处理条件下,PHGP6、GPX7和10个GST蛋白质的表达丰度均显著增加,这与苹果树根响应低温胁迫和沙枣幼苗接种丛枝菌根真菌后响应盐胁迫的比较蛋白质组学结果相似[70-71]。由此推测盐芥可能通过促进GSH代谢途径中关键酶的表达来清除过量的ROS,从而适应高盐胁迫环境。
当植物受到非生物胁迫时可通过积累渗透调节物质来进行渗透调节[72]。渗透调节物质大致可分为2类:一类是从外部引入细胞的无机离子,另一类是细胞内合成的有机物质,包括可溶性糖(蔗糖、葡萄糖、果糖、半乳糖等)和含氮化合物(如脯氨酸、甜菜碱等),其中脯氨酸是许多植物中最有效的渗透调节物质之一[73-77]。本研究中,我们鉴定到3个参与精氨酸和脯氨酸代谢途径的关键蛋白质PYCR、P5CS-A和ALDH7B4。PYCR和P5CS-A是植物中脯氨酸合成的关键酶。许多研究结果表明,PYCR和P5CS的表达水平与胁迫诱导的脯氨酸合成密切相关[78-79]。ALDH不仅能将醛氧化成羧酸,通过减少脂质过氧化来响应胁迫环境[47],还能通过促进脯氨酸合成参与拟南芥对高盐胁迫的响应[80]。本研究发现,在高盐胁迫条件下PYCR、P5CS-A和ALDH7B4的蛋白质表达水平明显升高,结合不同NaCl浓度处理条件下盐芥叶片中脯氨酸含量急剧增加的研究结果[21],推测高盐胁迫提高脯氨酸代谢途径的蛋白质表达量可促进盐芥中脯氨酸的积累。此外,我们还发现盐芥叶片中淀粉和可溶性糖的含量分别在400 mmol/L和600 mmol/LNaCl處理时达到最大值[21],结合本研究结果,600 mmol/LNaCl处理下参与碳水化合物代谢的蛋白质(5个参与糖酵解/糖异生的蛋白质,8个参与氨基糖和核苷酸糖代谢的蛋白质)表达丰度均显著增加,由此推测高盐环境也促进了碳水化合物的积累。这些结果表明,积累的渗透调节物质(如脯氨酸、淀粉、可溶性糖等)可能是盐芥适应高盐胁迫环境的关键因素。
为了降低Na+的毒害效应,植物需要消耗大量三磷酸腺苷(ATP)来排除体内多余的Na+,或将Na+区隔化进液泡来建立新的离子和渗透平衡[5]。ATP合成酶可通过合成ATP为细胞提供能量[81]。本研究发现,高盐胁迫环境下ATP合成酶的G亚基和ε亚基的表达丰度均显著增加,这与盐芥已有的研究结果[21-22]和其他植物的研究结果相似[82-84]。通过ATP合成酶亚基表达丰度的增加来提高能量供应可能是盐芥应对高盐环境的重要策略。
本研究利用iTRAQ技术从盐芥叶片中鉴定到4 607个蛋白质,其中281个蛋白质的表达丰度显著增加,97个蛋白质的表达丰度明显降低,以响应不同NaCl浓度处理的胁迫环境,这些盐胁迫差异表达蛋白质主要参与能量代谢、碳水化合物代谢、氨基酸代谢、卟啉和叶绿素代谢等代谢途径。通过对不同盐浓度处理下的胁迫差异表达蛋白质进行KEGG代谢通路和蛋白质互作调控网络分析,基本明确了盐芥适应不同盐胁迫环境的可能分子机制,下一步将深入研究关键盐胁迫差异表达蛋白质的生物学功能,为揭示盐芥耐盐分子机制奠定理论基础。
参考文献:
[1] LIANG W J, MA X L, WAN P, et al. Plant salt-tolerance mechanism: a review[J]. Biochemical and Biophysical Research Communications, 2018, 495(1): 286-291.
[2] WARWICK S I, AL-SHEHBAZ I A, SAUDER C A. Phylogenetic position of Arabis arenicola and generic limits of Aphragmus and Eutrema (Brassicaceae) based on sequences of nuclear ribosomal DNA[J]. Canadian Journal of Botany-Revue Canadienne de Botanique, 2006, 84(2): 269-281.
[3] AMTMANN A, BOHNERT H J, BRESSAN R A. Abiotic stress and plant genome evolution. Search for new models[J]. Plant Physiology, 2005, 138(1): 127-130.
[4] BRESSAN R A, ZHANG C, ZHANG H, et al. Learning from the Arabidopsis experience. The next gene search paradigm[J]. Plant Physiology, 2001, 127(4): 1354-1360.
[5] ZHU J K. Plant salt tolerance[J]. Trends in Plant Science, 2001, 6(2): 66-71.
[6] TAJI T, SEKI M, SATOU M, et al. Comparative genomics in salt tolerance between Arabidopsis and Arabidopsis-related halophyte salt cress using Arabidopsis microarray[J]. Plant Physiology, 2004, 135(3): 1697-1709.
[7] PANG Q, CHEN S, DAI S, et al. Comparative proteomics of salt tolerance in Arabidopsis thaliana and Thellungiella halophila[J]. Journal of Proteome Research, 2010, 9(5): 2584-2599.
[8] ARBONA V, ARGAMASILLA R, GMEZ-CADENAS A. Common and divergent physiological, hormonal and metabolic responses of Arabidopsis thaliana and Thellungiella halophila to water and salt stress[J]. Journal of Plant Physiology, 2010, 167(16): 1342-1350.
[9] ZHOU Y J, GAO F, LI X F, et al. Alterations in phosphoproteome under salt stress in Thellungiella roots[J]. Chinese Science Bulletin, 2010, 55(32): 3673-3679.
[10]蔡小寧,杨 平,贲爱玲,等. 盐芥ThHKT1基因的克隆[J], 江苏农业科学, 2006(6): 21-24.
[11]唐 宁,杨 平. 盐芥ThHKT1基因的生物信息学分析[J]. 药物生物技术, 2008, 15(6): 449-452.
[12]WU C, GAO X, KONG X Q, et al. Molecular cloning and functional analysis of a Na+/H+ antiporter gene ThNHX1 from a halophytic plant Thellungiella halophila[J]. Plant Molecular Biology Reporter, 2009, 27(1): 1-12.
[13]OH D H, GONG Q, ULANOV A, et al. Sodium stress in the halophyte Thellungiella halophila and transcriptional changes in a thsos1-RNA interference line[J]. Journal of Integrative Plant Biology, 2007, 49(10): 1484-1496.
[14]OH D H, LEIDI E, ZHANG Q, et al. Loss of halophytism by interference with SOS1 expression[J]. Plant Physiology, 2009, 151(1): 210-222.
[15]GAO F, GAO Q, DUAN X G, et al. Cloning of an H+-PPase gene from Thellungiella halophila and its heterologous expression to improve tobacco salt tolerance[J]. Journal of Experimental Botany, 2006, 57(12): 3259-3270.
[16]SUN Q H, GAO F, ZHAO L, et al. Identification of a new 130 bp cis-acting element in the TsVP1 promoter involved in the salt stress response from Thellungiella halophila[J]. BMC Plant Biology, 2010, 10(1): 90.
[17]LV S L, ZHANG K W, GAO Q, et al. Overexpression of an H+-PPase gene from Thellungiella halophila in cotton enhances salt tolerance and improves growth and photosynthetic performance[J]. Plant and Cell Physiology, 2008, 49(8): 1150-1164.
[18]ALEMN F, NIEVES-CORDONES M, MARTNEZ V, et al. Differential regulation of the HAK5 genes encoding the high-affinity K+ transporters of Thellungiella halophila and Arabidopsis thaliana[J]. Environmental and Experimental Botany, 2009, 65(2/3): 263-269.
[19]高秀華. 盐芥耐盐相关基因的功能研究[D]. 济南: 山东师范大学, 2006.
[20]XU X, ZHOU Y, WEI S, et al. Molecular cloning and expression of a Cu/Zn-containing superoxide dismutase from Thellungiella halophila[J]. Molecules and Cells, 2009, 27(4): 423-428.
[21]WANG X C, CHANG L L, WANG B C, et al. Comparative proteomics of Thellungiella halophila leaves from plants subjected to salinity reveals the importance of chloroplastic starch and soluble sugars in halophyte salt tolerance[J]. Molecular & Cellular Proteomics, 2013, 12(8): 2174-2195.
[22]CHANG L L, GUO A P, JIN X, et al. The beta subunit of glyceraldehyde 3-phosphate dehydrogenase is an important factor for maintaining photosynthesis and plant development under salt stress-Based on an integrative analysis of the structural, physiological and proteomic changes in chloroplasts in Thellungiella halophila[J]. Plant Science, 2015, 236: 223-238.
[23]WANG X C, SHI M J, LU X L, et al. A method for protein extraction from different subcellular fractions of laticifer latex in Hevea brasiliensis compatible with 2-DE and MS[J]. Proteome Science, 2010, 8(1): 35.
[24]BRADFORD M M. A rapid and sensitive method for the quantization of microgram quantities of protein using the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1/2): 248-254.
[25]YAN M, LU Z L, LI B, et al. Comparative proteomics reveals new insights into the endosperm responses to drought, salinity and submergence in germinating wheat seeds[J]. Plant Molecular Biology, 2021, 105(3): 287-302.
[26]LI J, CUI J, CHENG D, et al. iTRAQ protein profile analysis of sugar beet under salt stress: different coping mechanisms in leaves and roots[J]. BMC Plant Biology, 2020, 20(1): 347.
[27]劉爱荣,赵可夫. 盐胁迫对盐芥生长及硝酸还原酶活性的影响[J]. 植物生理与分子生物学学报, 2005, 31(5): 469-476.
[28]KATSCHNIG D, BROEKMAN R, ROZEMA J. Salt tolerance in the halophyte Salicornia dolichostachya Moss: growth, morphology and physiology[J]. Environmental and Experimental Botany, 2013, 92: 32-42.
[29]YI X, SUN Y, YANG Q, et al. Quantitative proteomics of Sesuvium portulacastrum leaves revealed that ion transportation by V-ATPase and sugar accumulation in chloroplast played crucial roles in halophyte salt tolerance[J]. Journal of Proteomics, 2014, 99: 84-100.
[30]郭建荣,郑聪聪,李艳迪,等. NaCl处理对真盐生植物盐地碱蓬根系特征及活力的影响[J]. 植物生理学报, 2017, 53(1): 63-70.
[31]GOUSSI R, MANA A, DERBALI W, et al. Comparative analysis of salt stress, duration and intensity, on the chloroplast ultrastructure and photosynthetic apparatus in Thellungiella salsuginea[J]. Journal of Photochemistry and Photobiology B-Biology, 2018, 183: 275-287.
[32]PANG Q, CHEN S, DAI S, et al. Comparative proteomics of salt tolerance in Arabidopsis thaliana and Thellungiella halophila[J]. Journal of Proteome Research, 2010, 9(5): 2584-2599.
[33]LIU Z, ZOU L, CHEN C, et al. iTRAQ-based quantitative proteomic analysis of salt stress in Spica Prunellae[J]. Scientific Reports, 2019, 9(1): 9590.
[34]KUMARI M P, SEKAR K. Effect of plant growth regulators on chlorophyll and carotenoid content of salinity stressed okra seedlings[J]. Asian Journal of Horticulture, 2008, 3(1): 54-55.
[35]XING W, WANG J, LIU H, et al. Influence of natural saline-alkalistress on chlorophyll content and chloroplast ultrastructure of two contrasting rice(Oryza sativa L. japonica) cultivars[J]. Australian Journal of Crop Science, 2013, 7(2): 289-292.
[36]SAYYAD-AMIN P, JAHANSOOZ M R, BORZOUEI A, et al. Changes in photosynthetic pigments and chlorophyll-a fluorescence attributes of sweet-forage and grain sorghum cultivars under salt stress[J]. Journal of Biological Physics, 2016, 42(4): 601-620.
[37]SHOOLINGIN-JORDAN P M. Porphobilinogen deaminase and uroporphyrinogen Ⅲ synthase: structure, molecular biology, and mechanism[J]. Journal of Bioenergetics & Biomembranes, 1995, 27(2): 181-195.
[38]ELDER G H, ROBERTS A G. Uroporphyrinogen decarboxylase[J]. Journal of Bioenergetics & Biomembranes, 1995, 27(2): 207-214.
[39]SCHOEFS B, FRANCK F. Protochlorophyllide reduction: mechanisms and evolutions[J]. Photochemistry & Photobiology, 2003, 78(6): 543-557.
[40]ECKHARDT U, GRIMM B, HRTENSTEINER S. Recent advances in chlorophyll biosynthesis and breakdown in higher plants[J]. Plant Molecular Biology, 2004, 56(1): 1-14.
[41]IKEGAMI A, YOSHIMURA N, MOTOHASHI K, et al. The CHLI1 subunit of Arabidopsis thaliana magnesium chelatase is a target protein of the chloroplast thioredoxin[J]. Journal of Biological Chemistry, 2007, 282(27): 19282-19291.
[42]PRUZINSK A, TANNER G, ANDERS I, et al. Chlorophyll breakdown: pheophorbide a oxygenase is a Rieske-type iron-sulfur protein, encoded by the accelerated cell death 1 gene[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(25): 15259-15264.
[43]TURAN S, TRIPATHY B C. Salt-stress induced modulation of chlorophyll biosynthesis during de-etiolation of rice seedlings[J]. Physiologia Plantarum, 2015, 153(3): 477-491.
[44]HU L, XIANG L, LI S, et al. Beneficial role of spermidine in chlorophyll metabolism and D1 protein content in tomato seedlings under salinity-alkalinity stress[J]. Physiologia Plantarum, 2016, 156(4): 468-477.
[45]GONG W, XU F, SUN J, et al. iTRAQ-based comparative proteomic analysis of seedling leaves of two upland cotton genotypes differing in salt tolerance[J]. Frontiers in Plant Science, 2017, 8: 2113.
[46]HILDEBRANDT T M, NUNES NESI A, ARAJO W L, et al. Amino acid catabolism in plants[J]. Molecular Plant, 2015, 8(11): 1563-1579.
[47]HUANG S P, ZENG Y L. Research progress on plant aldehyde dehydrogenase under adversity stresses[J]. Biotechnology Bulletin, 2015, 31(12): 8-14.
[48]HILDEBRANDT T M. Synthesis versus degradation: directions of amino acid metabolism during Arabidopsis abiotic stress response[J]. Plant Molecular Biology, 2018, 98(1/2): 121-135.
[49]LIU X, HAN Q, WANG J, et al. Two FgLEU2 genes with different roles in leucine biosynthesis and infection-related morphogenesis in Fusarium graminearum[J]. PLoS One, 2016, 11(11): e0165927.
[50]KOCHEVENKO A, FERNIE A R. The genetic architecture of branched-chain amino acid accumulation in tomato fruits[J]. Journal of Experimental Botany, 2011, 62(11): 3895-3906.
[51]HUANG T, JANDER G. Abscisic acid-regulated protein degradation causes osmotic stress-induced accumulation of branched chain amino acids in Arabidopsis thaliana[J]. Planta, 2017, 246(4): 737-747.
[52]CHEN D, MA X, LI C, et al. A wheat aminocyclopropane-1-carboxylate oxidase gene, TaACO1, negatively regulates salinity stress in Arabidopsis thaliana[J]. Plant Cell Reports, 2014, 33(11): 1815-1827.
[53]RAMADOSS N, GUPTA D, VAIDYA B N, et al. Functional characterization of 1-aminocyclopropane-1-carboxylic acid oxidase gene in Arabidopsis thaliana and its potential in providing flood tolerance[J]. Biochemical & Biophysical Research Communications, 2018, 503(1): 365-370.
[54]GUO Z, TAN J, ZHUO C, et al. Abscisic acid, H2O2 and nitric oxide interactions mediated cold-induced S-adenosylmethionine synthetase in Medicago sativa sub sp. falcata that confers cold tolerance through up-regulating polyamine oxidation[J]. Plant Biotechnology Journal, 2014, 12(5): 601-612.
[55]KIM S H, KIM S H, PALANIYANDI S A, et al. Expression of potato S-adenosyl-L-methionine synthase (SbSAMS) gene altered developmental characteristics and stress responses in transgenic Arabidopsis plants[J]. Plant Physiology and Biochemistry, 2015,87: 84-91.
[56]MA C, WANG Y, GU D, et al. Overexpression of S-adenosyl-l-methionine synthetase 2 from sugar beet M14 increased Arabidopsis tolerance to salt and oxidative stress[J]. International Journal of Molecular Sciences, 2017, 18(4): 847.
[57]APEL K, HIRT H. Reactive oxygen species: metabolism, oxidative stress, and signal transduction[J]. Annual Review of Plant Biology, 2004, 55(1): 373-399.
[58]MAY M J, VERNOUX T, LEAVER C, et al. Glutathione homeostasis in plants: implications for environmental sensing and plant development[J]. Journal of Experimental Botany,1998,49(321):649-667.
[59]CHOUDHURY F K, DEVIREDDY A R, AZAD R K, et al. Rapid accumulation of glutathione during light stress in Arabidopsis[J]. Plant & Cell Physiology, 2018, 59(9): 1817-1826.
[60]BELA K, HORVTH E, GALL , et al. Plant glutathione peroxidases: emerging role of the antioxidant enzymes in plant development and stress responses[J]. Journal of Plant Physiology, 2015, 176(1): 192-201.
[61]LABROU N E, PAPAGEORGIOU A C, PAVLI O, et al. Plant GSTome: structure and functional role in xenome network and plant stress response[J]. Current Opinion in Biotechnology, 2015, 32: 186-194.
[62]MILLA M, MAURER A, HUETE A R, et al. Glutathione peroxidase genes in Arabidopsis are ubiquitous and regulated by abiotic stresses through diverse signaling pathways[J]. The Plant Journal, 2003, 36(5): 602-615.
[63]XU J, TIAN Y S, XING X J, et al. Over-expression of AtGSTU19 provides tolerance to salt, drought and methyl viologen stresses in Arabidopsis[J]. Physiologia Plantarum, 2016, 156(2): 164-175.
[64]KANG S G, JEONG H K, SUH H S. Characterization of a new member of the glutathione peroxidase gene family in Oryza sativa[J]. Molecules & Cells, 2004, 17(1): 23-28.
[65]GALL , CSISZR J, SECENJI M, et al. Glutathione transferase activity and expression patterns during grain filling in ag leaves of wheat genotypes differing in drought tolerance: response to water deficit[J]. Journal of Plant Physiology, 2009, 166(17): 1878-1891.
[66]CSISZR J, GALL A, HORVTH E, et al. Different peroxidase activities and expression of abiotic stress-related peroxidases in apical root segments of wheat genotypes with different drought stress tolerance under osmotic stress[J]. Plant Physiology & Biochemistry, 2012, 52: 119-129.
[67]REZAEI M K, SHOBBAR Z S, SHAHBAZI M, et al. Glutathione S-transferase (GST) family in barley: identification of members, enzyme activity, and gene expression pattern[J]. Journal of Plant Physiology, 2013, 170(14): 1277-1284.
[68]CHAN C, LAM H M. A putative lambda class glutathione S-transferase enhances plant survival under salinity stress[J]. Plant and Cell Physiology, 2014, 55(3): 570-579.
[69]GAO F, CHEN J, MA T, et al. The glutathione peroxidase gene family in Thellungiella salsuginea: genome-wide identification, classification, and gene and protein expression analysis under stress conditions[J]. International Journal of Molecular Sciences, 2014, 15(2): 3319-3335.
[70]LI L J, LU X C, MA H Y, et al. Comparative proteomic analysis reveals the roots response to low root-zone temperature in Malus baccata[J]. Journal of Plant Research, 2018, 131(5): 865-878.
[71]JIA T, WANG J, CHANG W, et al. Proteomics analysis of E. angustifolia seedlings inoculated with arbuscular mycorrhizal fungi under salt stress[J]. International Journal of Molecular Sciences, 2019, 20(3): 788.
[72]CHEONG M, YUN D J. Salt-stress signaling[J]. Journal of Plant Biology, 2007, 50(2): 148-155.
[73]KHAN T A, YUSUF M, AHMAD A, et al. Proteomic and physiological assessment of stress sensitive and tolerant variety of tomato treated with brassinosteroids and hydrogen peroxide under low-temperature stress[J]. Food Chemistry, 2019, 289: 500-511.
[74]BANDURSKA H, NIEDZIELA J, PIETROWSKA-BOREK M, et al. Regulation of proline biosynthesis and resistance to drought stress in two barley (Hordeum vulgare L.) genotypes of different origin[J]. Plant Physiology And Biochemistry, 2017, 118: 427-437.
[75]尹 秀,王 俊,张二豪,等. PEG-6000浸种处理对甘青青兰种子萌发及幼苗抗旱性的影响[J].江苏农业科学,2020,48(13):168-172.
[76]殷世航,周 赛,黄霄宇,等. 中蔗系列新品种对干旱胁迫的响应及抗旱性评价[J].南方农业学报,2020,51(6):1339-1345.
[77]任保兰,耿建建,吕 亚,等. 辣木幼苗对淹水胁迫的生理响应及耐涝性综合评价[J].南方农业学报,2021,52(3):789-796.
[78]PREZ-ARELLANO I, CARMONA-ALVAREZ F, MARTNEZ A I, et al. Pyrroline-5-carboxylate synthase and proline biosynthesis: from osmotolerance to rare metabolic disease[J]. Protein Science, 2010, 19(3): 372-382.
[79]SINGH P, TIWARI A, SINGH S P, et al. Proline biosynthesizing enzymes (glutamate 5-kinase and pyrroline-5-carboxylate reductase) from a model cyanobacterium for desiccation tolerance[J]. Physiology and Molecular Biology of Plants, 2013, 19(4): 521-528.
[80]DEUSCHLE K, FUNCK D, HELLMANN H, et al. A nuclear gene encoding mitochondrial Δ1-pyrroline-5-carboxylate dehydrogenase and its potential role in protection from proline toxicity[J]. The Plant Journal, 2001, 27(4): 345-356.
[81]TIKHONOV A N. pH-dependent regulation of electron transport and ATP synthesis in chloroplasts[J]. Photosynthesis Research, 2013, 116(2/3): 511-534.
[82]WANG L X, PAN D Z, LI J, et al. Proteomic analysis of changes in the Kandelia candel chloroplast proteins reveals pathways associated with salt tolerance[J]. Plant Science, 2015, 231: 159-172.
[83]JI W, CONG R, LI S, et al. Comparative proteomic analysis of soybean leaves and roots by iTRAQ provides insights into response mechanisms to short-term salt stress[J]. Frontiers in Plant Science, 2016, 7: 573.
[84]LIU Z, ZOU L, CHEN C, et al. iTRAQ-based quantitative proteomic analysis of salt stress in Spica Prunellae[J]. Scientific Reports, 2019,9(1): 9590.
(責任编辑:陈海霞)
1889500783337

