基于环境DNA 宏条形码的太湖流域底栖动物监测与生态健康评价
金 珂,张丽娟,张 伟,张 翔,陈 桥,杨江华,张 咏,张效伟,3
1.南京大学环境学院,江苏 南京 210023
2.江苏省环境监测中心,江苏 南京 210023
3.江苏省环境保护化学品安全与健康风险研究重点实验室,江苏 南京 210023
对生态环境中物种多样性的精准监测是科学开展生态环境保护与管理的前提。 底栖动物占据淡水生态系统的核心位置,其种类与数量的分布情况对淡水生态系统的健康状况具有一定的指示作用,因此,常被用于淡水生态系统监测与健康评估[1]。 传统底栖动物监测主要通过采集生物样本,依据形态学方法进行鉴定。 然而,样品挑拣和鉴定的难度大、周期长,需要消耗大量的人力和时间,且部分近缘种的形态特征非常相似,难以区分,对鉴定人员的专业要求较高[2-3]。 这些问题使得以形态学鉴定为主的底栖动物监测方法在我国生态环境监测工作中难以得到大规模应用。
环境DNA 宏条形码(eDNA Metabarcoding)技术是一种用于检测生物多样性的新方法[4],其基础来自DNA 条形码(DNA Barcoding)技术[5-8],在生物监测中具有巨大的应用潜力。 生物在环境中活动时,其DNA 会不断释放到周围的环境介质中。 从环境介质中提取的包含了物种多样性信息的DNA 物质被称为环境DNA[9-12]。 环境DNA 宏条形码技术使用通用引物对环境DNA 样品进行聚合酶链式反应(Polymerase Chain Reaction,PCR)扩增,结合高通量测序技术,可产生上百万条反映物种多样性的序列。 通过对序列进行分析,可以识别环境中的物种组成、群落结构等多样性信息。 与传统形态学监测方法相比,环境DNA宏条形码技术能够更快速、更精确、更经济[13-15]地识别水生态系统中生物群落的细微变化[14,16-17]。
环境DNA 宏条形码技术在底栖动物监测和生物评价上尚存在一些不确定的问题[18]。 目前常用的底栖动物环境DNA 提取方法包括水样提取、研磨提取、酒精浸提3 种方法。 其中,通过水样提取的环境DNA 在物种识别方面与形态学识别结果的一致性较低[19]。 尽管研磨破碎经挑拣的底栖动物并提取其DNA 可以获得更高的物种识别率,但该方法的操作相对烦琐。 酒精浸提法则是从用于保存标本的“清洁”乙醇固定剂中获得底栖动物群落的环境DNA[11,20-23]。 最近,有少数研究者开始使用保存环境样品的“脏”乙醇作为底栖动物环境DNA 的提取来源[24-25],表明其具有用于底栖动物生物监测的潜力。 但受时间、人力、经济等因素的限制,早期的方法研究与示范仅限于少数样品或监测位点,鲜有在流域尺度上的实际应用与验证研究。
太湖流域水环境治理和生态恢复工作受到广泛关注。 通过监测太湖流域底栖动物多样性,可以有效揭示太湖流域水环境受胁迫水平和水生态健康修复状况。 对于太湖流域底栖动物监测而言,既要对流域内各点位的底栖动物物种进行识别,也要在此基础上进行流域生态健康评价。 然而,我国尚未在流域尺度上开展过基于环境DNA的底栖动物多样性监测。 对此,本研究同步采用环境DNA 宏条形码技术和形态学鉴定方法,在太湖流域布点开展底栖动物群落监测,详细比较了两种方法在物种识别、生态评价方面的一致性,并系统地分析了将环境DNA 宏条形码技术应用于底栖动物监测的可行性与有效性。
1 实验方法
1.1 采样点布设与环境样品采集
2019 年8—11 月,在太湖流域共布设65 个采样点位,同时采用环境DNA 宏条形码技术与传统形态学方法开展底栖动物监测(图1)。 采集环境样品时,使用1/16 m2Peterson 采泥器采集水体底部沉积物样品,每个点位采集8 夹。 在没有沉积物的点位,使用D 网(宽0.3 m,孔径425 μm)采集足量样品。 在溪流点位,使用踢网(1 m×1 m,425 μm)采集足量样品。 将采集到的样品置于孔径为425 μm 的筛网内淘洗,挑去大块石头、植物枝干后,保留剩余的环境基质,于4 ℃冷藏带回实验室保存。 连续采样时,应清洗器材以避免交叉污染。 将样品均匀混合后,分成两等份,一份用于形态学鉴定,另一份用于环境DNA 监测中的酒精浸提处理。 研究路线及技术路线见图2。
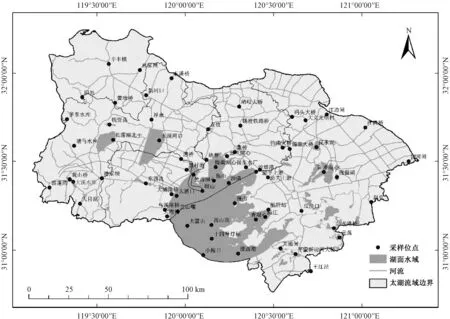
图1 太湖流域底栖动物采样点Fig.1 Sampling sites of macroinvertebrate in Taihu Lake Basin
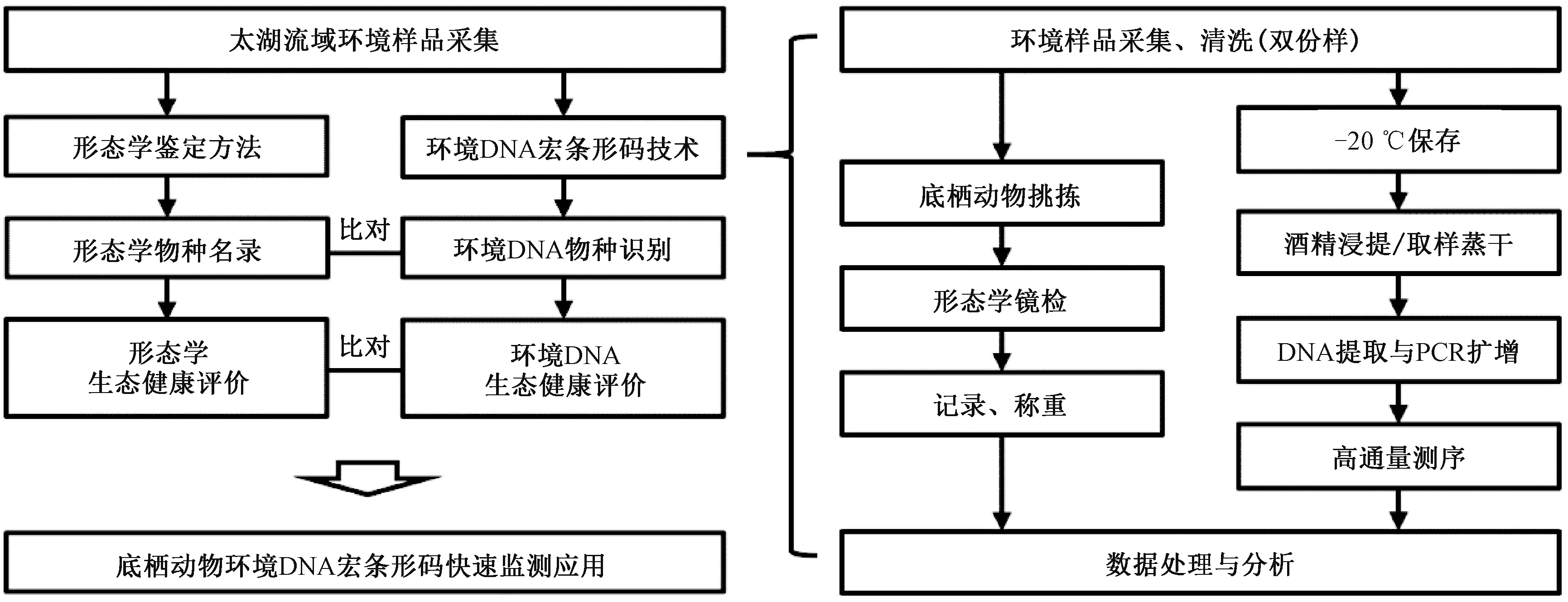
图2 底栖动物监测研究路线与技术路线Fig.2 Research flowchart and technical approaches of macroinvertebrate monitoring
1.2 形态学鉴定
返回实验室后,将用于形态学鉴定的样品4 ℃冷藏,并于24 h 内挑拣出其中的底栖动物个体。 在解剖镜和显微镜下对底栖动物个体进行鉴定,同时通过比对参考资料,将其鉴定至种或属,并计数、称重,计算生物量和生物密度[26-28]。
1.3 环境样品前处理
用于环境DNA 监测的样品可于-20 ℃储存。解冻后,参考MARTINS 等[25]的方法进行前处理。将包含底栖动物的环境基质样品置于2 L 玻璃缸中,根据环境基质样品的总重加入无水乙醇。 1 g样品换算为1 mL,加入3.5 倍环境基质样品体积的无水乙醇。 轻轻摇晃混匀后,盖上盖子,在室温下静置14 d,随后抽取2 mL 浸提液,保存在无菌离心管中。 在50 ℃的真空环境中蒸干离心管内的乙醇,得到干燥的组织样品。 为保证组织样品留在离心管底部,可在真空离心机中蒸干。
1.4 环境DNA 提取与PCR 扩增
1.4.1 DNA 提取
对于干燥后的组织样品,用DNeasy 试剂盒(德国Qiagen,69504)提取组织DNA。 首先向干燥的组织样品中加入裂解缓冲液和蛋白酶K,涡旋均匀后,56 ℃水浴裂解3~4 h。 后续操作主要包括DNA 吸附上柱、洗涤杂质、洗脱DNA,参照试剂盒说明书进行。 使用荧光计( 美国Invitrogen,Qubit 2.0)测定DNA 浓度。
1.4.2 PCR 扩增
PCR 扩增体系为50 μL,包含25 μL 的2×PrimeStar Max DNA 聚合酶(日本 TAKARA,R045A),5 μL DNA 模板(总量约20 ng),上下游引物各1.25 μL,无酶水17.5 μL。 上游引物为mlCOⅠintF,下游引物为dgHCO2198,其中,下游引物带有一段唯一的短核苷酸序列,用于标记不同样品。 PCR 程序:98 ℃预变性3 min;98 ℃变性15 s,46 ℃复性30 s,72 ℃延伸45 s,35 个扩增循环;72 ℃延伸5 min;4 ℃保存。 PCR 产物通过2%琼脂糖凝胶电泳的检测后,进一步纯化。
上游引物mlCOⅠintF 序列:5’-GGWACWGG WTGAACWGTWTAYCCYCC-3’。
下游引物dgHCO2198 序列:5’-TAAACYTC AGGRTGACCRAARAAYCA-3’。
DNA 提取和PCR 扩增中使用的器材和试剂均为无菌状态或做灭菌处理,操作过程中避免交叉污染。
1.5 文库构建与高通量测序
将各个点位样品的PCR 产物按照相等的DNA 量混合。 混合后的DNA 样品用DNA 纯化与分选磁珠(南京诺唯赞,VAHTS N411)再次纯化,利用DNA 文库构建试剂盒(南京诺唯赞,VAHTS ND702)构建测序文库。 利用Ion Torrent测序平台(美国Life Technologies,S5)进行测序。使用的所有器材和试剂均为无菌状态或做灭菌处理,操作过程中避免交叉污染。
1.6 数据分析
1.6.1 生物信息学分析
测序完成后,使用Python 中的SeqIO 将FASTQ 文件转化为FASTA 格式,使用QIIME(v1.8.0)剔除低质量序列(Q<20)、错配序列和长度<250 bp 的序列。 以上操作均在Bio-Linux 8系统下完成。 利用USEARCH7 进行可操作分类单元(Operational Taxonomic Units,OTU)聚类,相似度阈值为97%。 利用QIIME 将OTU 的代表性序列与本土物种数据库进行比对,相似性阈值设定为85%,采用相似性最高的序列对应的物种信息对OTU 进行注释。
1.6.2 环境DNA 与形态学监测数据的比较分析
为评估环境DNA 方法在底栖动物物种识别上的有效性和可靠性,本研究从分类单元检出数、优势物种识别结果、物种检出频次等方面,对两种方法的监测结果进行了比较,以探究两种方法的一致性。 其中,对于检出频次上的相关性,分别采用了检出频次和检出种次两个指标进行比较。 检出频次表示某分类单元在多少个点位被检出。 检出种次进一步考虑了所隶属的分类单元的数目,将隶属于某一类群的所有物种在所有点位出现的次数进行加和,作为一项指标进行比较。 例如,环棱螺属有铜锈环棱螺和梨形环棱螺2 个物种被检出,其中,铜锈环棱螺在26 个点位被检出,梨形环棱螺在25 个点位被检出,则可以说环棱螺属共计检出51种次。
1.6.3 生物指数计算
参考已有的太湖流域生物指数研究成果[29],利用物种鉴别结果计算底栖动物完整性指数(Benthic-Index of Biotic Integrity,B-IBI),并评 价每个点位的底栖动物生态健康状况。 单项指数包括:
B1:软体动物分类单元数。 B1(监测值)= 样品中的软体动物种类数。
B2:第一优势种优势度得分。 形态学方法中,B2(监测值)= 第一优势种生物密度/点位总生物密度;环境DNA 方法中,B2(监测值)= 第一优势OTU 序列数/点位总底栖动物序列数。
B3:大型底栖动物生物监测工作组(Biological Monitoring Working Party,BMWP)打分值。 B3(监测值)= BMWP=∑ti,ti为物种i所属科的科级敏感值。
基于环境DNA 方法和形态学方法监测结果,分别计算单项指数和B-IBI,计算方法见表1 及公式(1)。 结合计算结果,对点位进行生态质量等级划分,并进行比较。
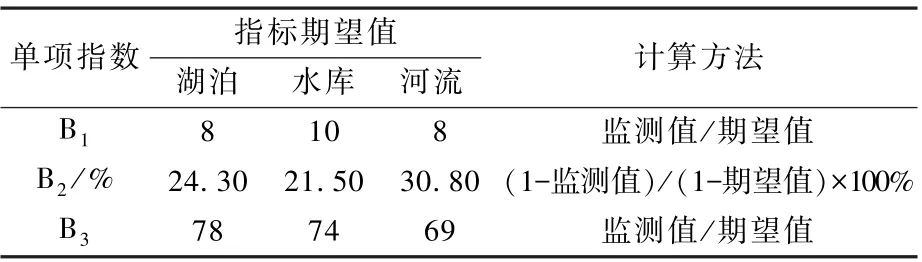
表1 淡水大型底栖无脊椎动物完整性单项指数计算方法Table 1 Index calculation method for the integrity of freshwater macroinvertebrates
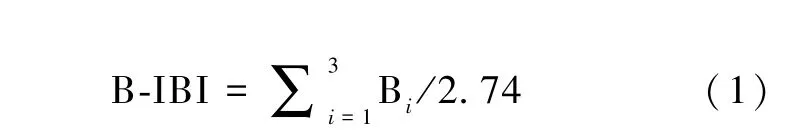
式中:Bi为第i个生物指数的值。
2 结果分析
2.1 底栖动物多样性监测
2.1.1 物种识别
使用环境DNA 方法在65 个点位检测出107种底栖动物,隶属3 门6 纲14 目35 科73 属。 底栖动物共计出现1 308 种次,平均每个点位发现底栖动物20.5 种。 通过环境DNA 方法在65 个点位的样品中获得了共计1 643 777 条高质量序列,其中828 415 条序列(50%)可注释到370 个OTU。 共有243 个OTU 被注释到了107 种底栖动物,代表了760 331 条序列,包括环节动物、软体动物、节肢动物等主要底栖动物类群(图3)。
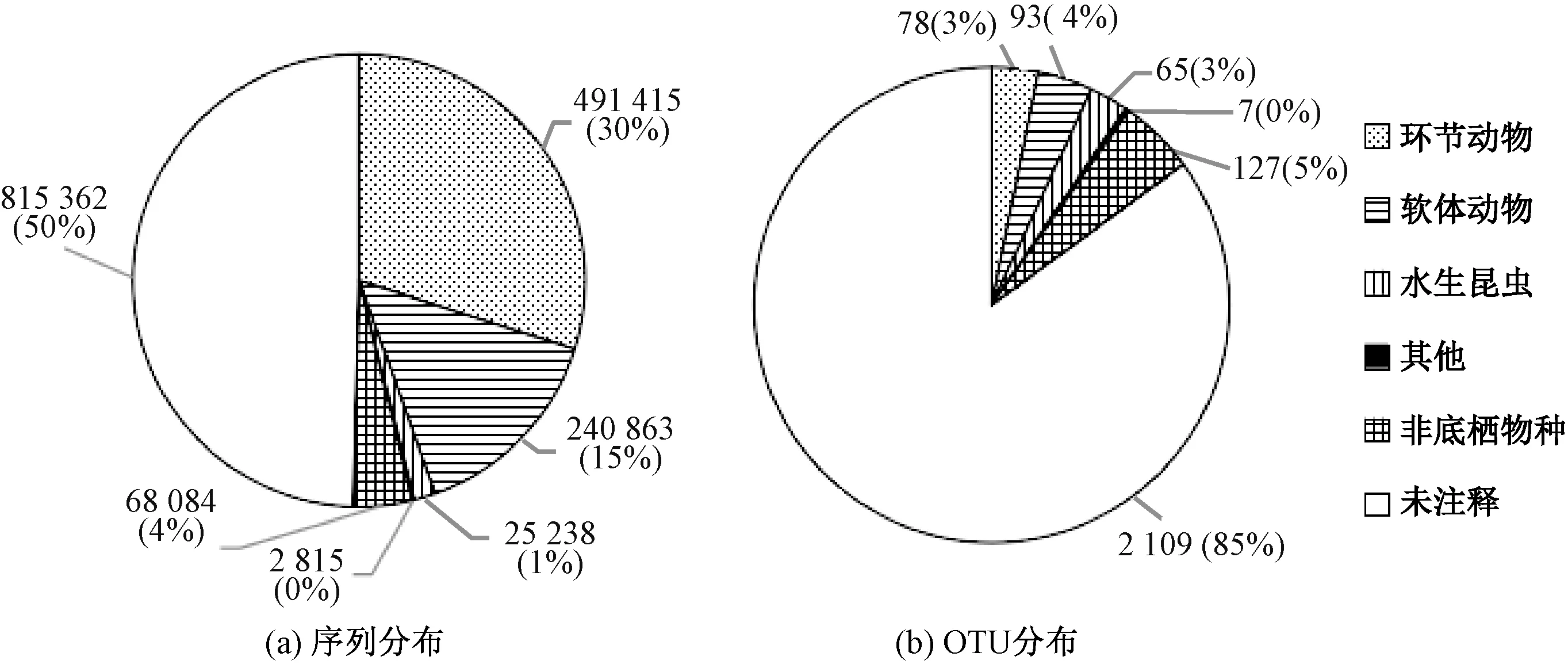
图3 环境DNA 监测注释结果Fig.3 Annotated results of eDNA monitoring
基于形态学方法在65 个点位共检出69 种底栖动物,隶属3 门8 纲21 目33 科55 属。 底栖动物共计出现515 种次,平均每个点位发现7.7 种。环境DNA 方法平均检出物种数是形态学方法的近3 倍,在科、属、种分类水平上均比形态学方法检出了更多的分类单元数,分别是形态学方法的106%、132%、155%(图4)。 采用环境DNA 方法检出物种数较多的点位及其物种数:阳澄湖心,39种;大港桥,35 种;钓渚大桥,33 种;观山桥,32种;东西氿,30 种。
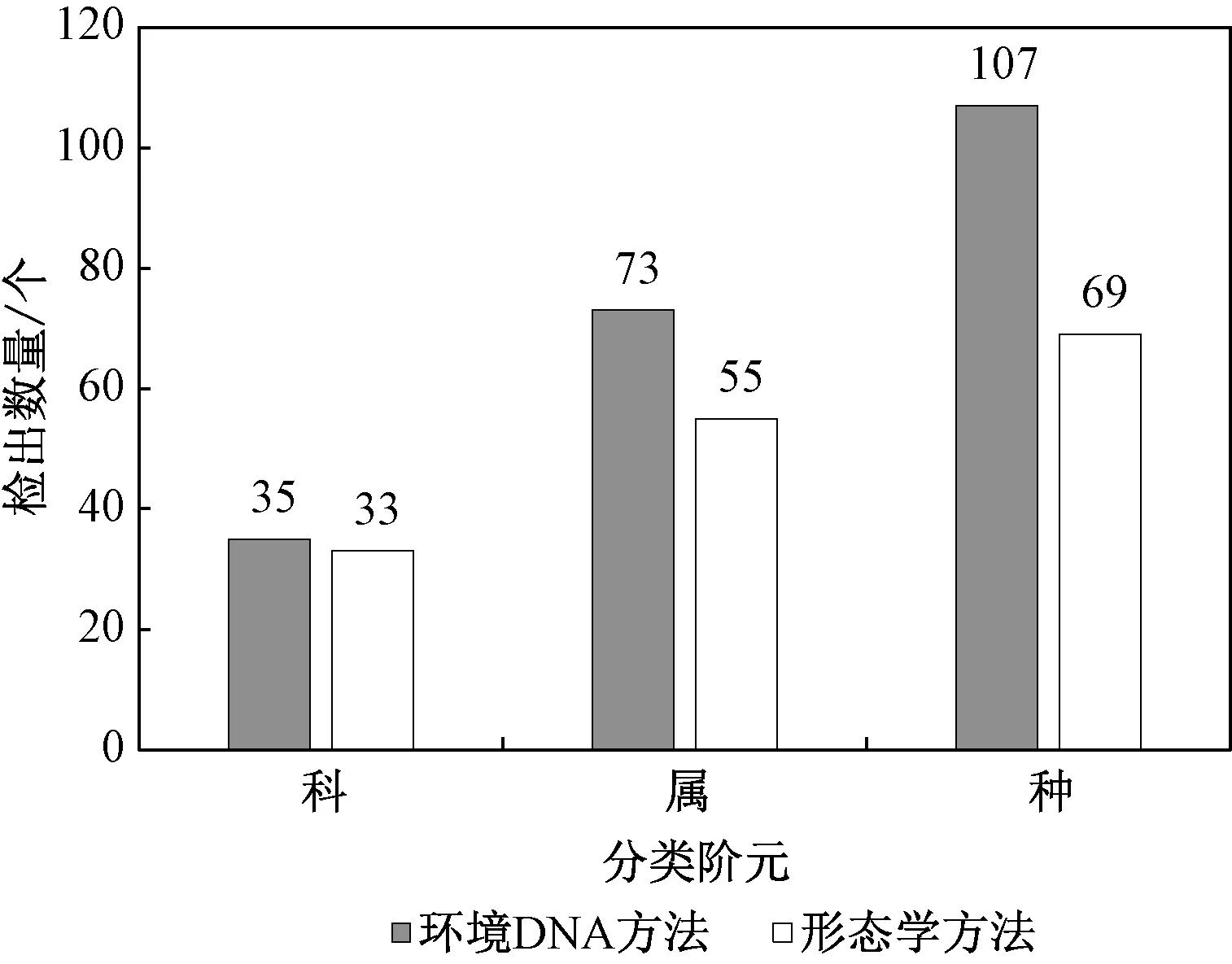
图4 两种方法在不同分类阶元的检出数目比较Fig.4 Comparison of the number of taxadetected by the two methods
2.1.2 分类单元交叉情况
在科级水平,环境DNA 方法共检出35 个科,形态学方法共检出33 个科,其中有13 个科为两种方法共同检出,占形态学方法检出总科数的近40%。 共有44 个形态学物种隶属于交叉的13 个科,占据了形态学方法检出总物种数的64.7%、所有个体数的94.9%和所有生物量的99.2%(表2,图5)。 同时,共同检出的科所代表的物种占据了环境DNA 方法检出的所有底栖动物类群中66.3%的OTU 和76.4%的序列数,说明环境DNA 方法产生的信息大部分都来源于形态学方法鉴定出的主要底栖动物类群。
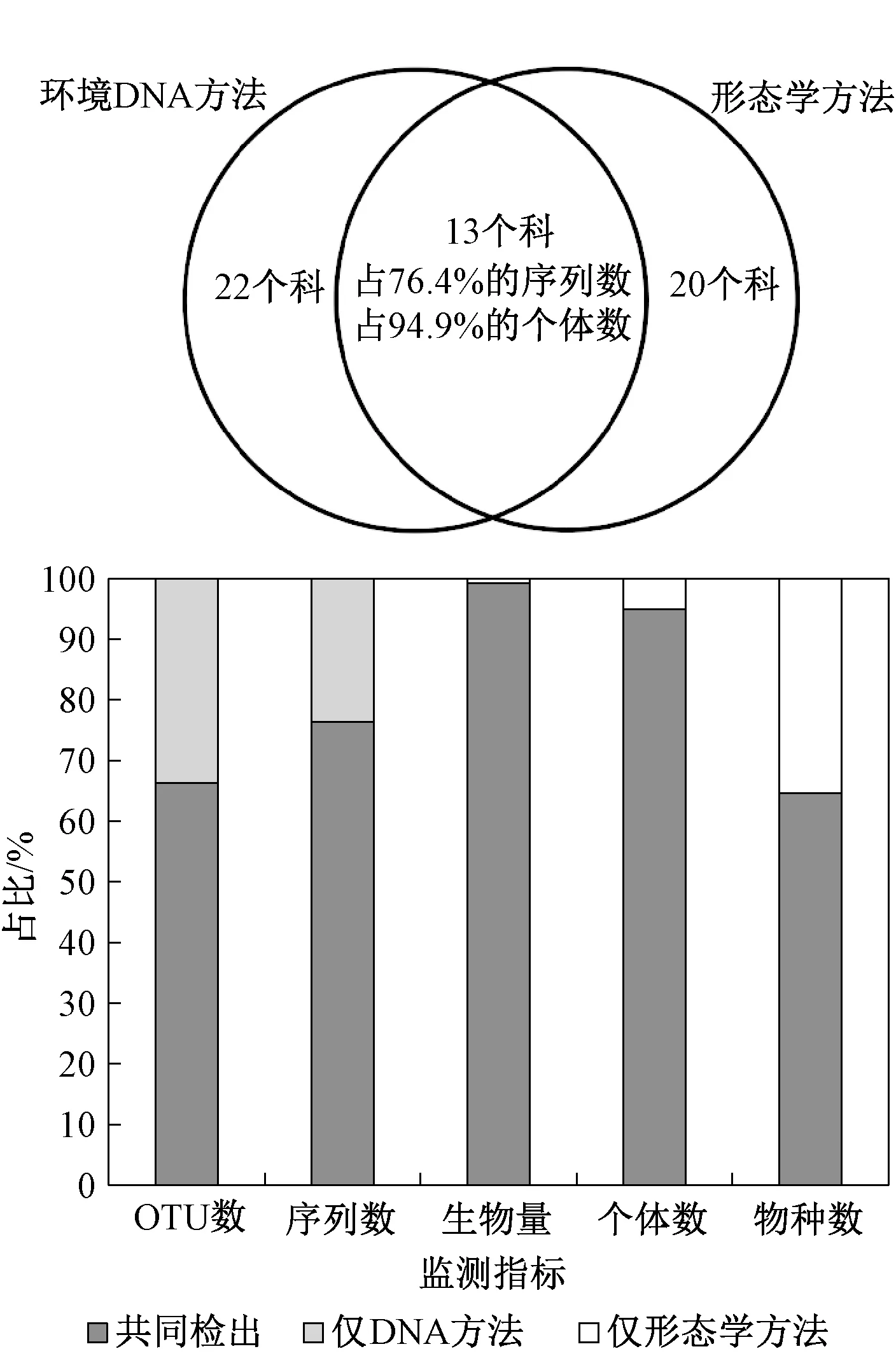
图5 科级别的交叉率Fig.5 Overlaps at family level
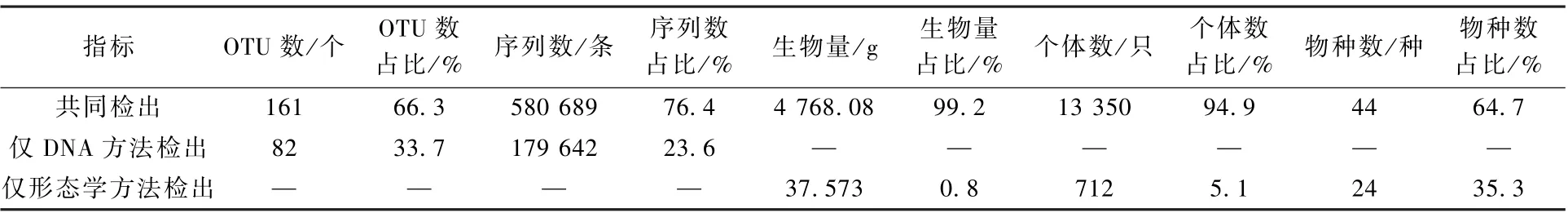
表2 各项指标在科级别的交叉情况Table 2 Overlaps of indexes in family level
在属水平上,环境DNA 方法共检出73 个属,形态学方法共检出55 个属,其中有25 个属为共同检出,占形态学检出总属数的45.5%。 共同检出的25 个属包括30 个形态学物种,占据了形态学检出总物种数的48.4%、所有个体数的94.2%及所有生物量的80.1%(图6,表3)。 同时,共同检出的属所代表的物种也占据了环境DNA 方法检出的大部分OTU(58.7%)和序列数(90.6%)。
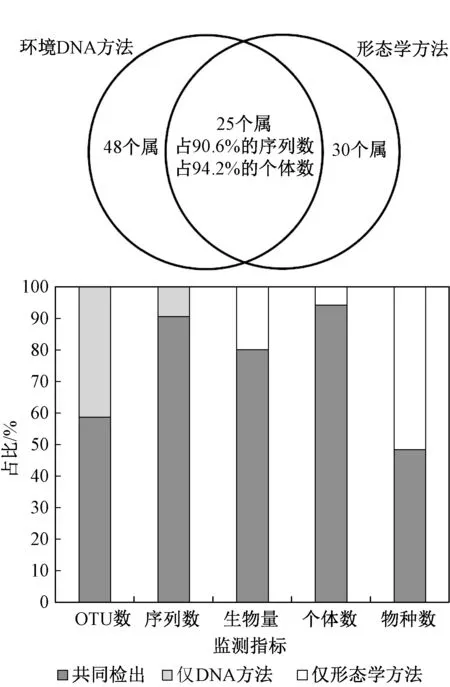
图6 属级别的交叉率Fig.6 Overlaps at genus level
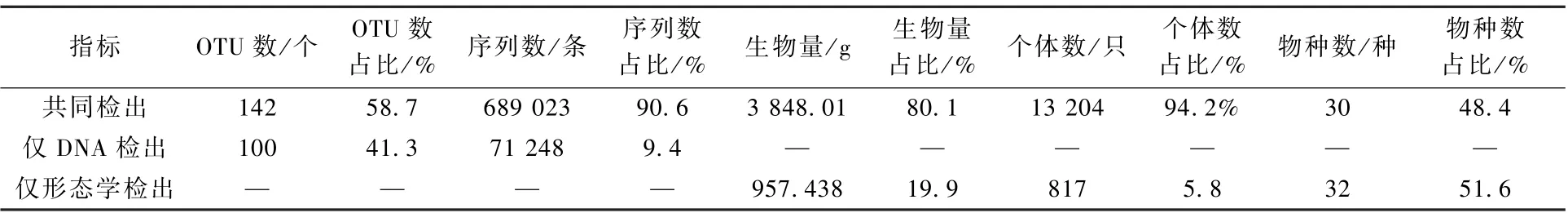
表3 各项指标在属级别的交叉情况Table 3 Overlaps of indexes in genus level
2.1.3 优势物种与检出频次比较
在环境DNA 方法和形态学方法监测结果中,检出频次和第一优势物种次数的前五名非常一致(表4)。 与形态学方法相比,环境DNA 方法物种检出频次、第一优势物种次数、科检出种次、属检出种次的前五名,分别有80%、60%、80%、60%是一致的。 总体而言,环境DNA 方法监测结果表明,颤蚓科水丝蚓属和尾鳃蚓属、田螺科环棱螺属、蚬科蚬属是太湖流域广泛分布的底栖动物类群。
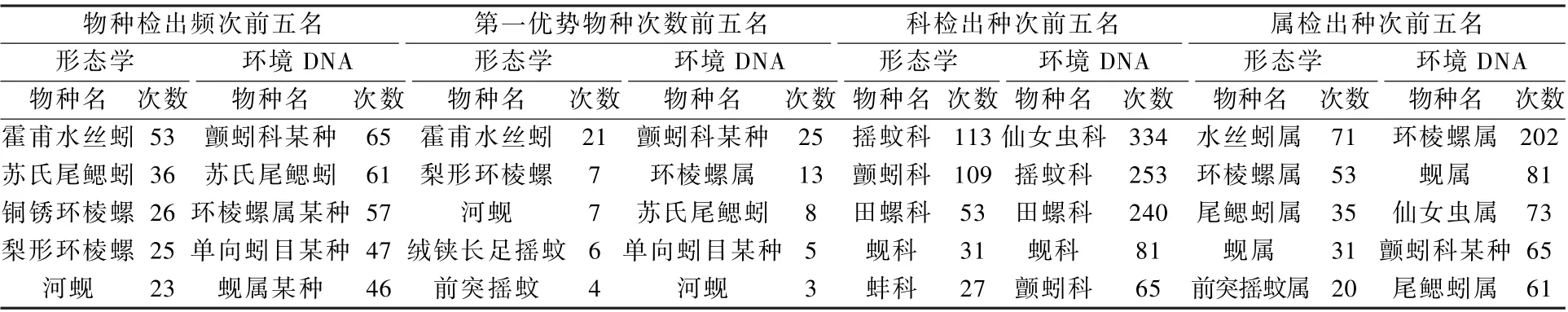
表4 优势物种对照Table 4 Comparison of dominant species 次
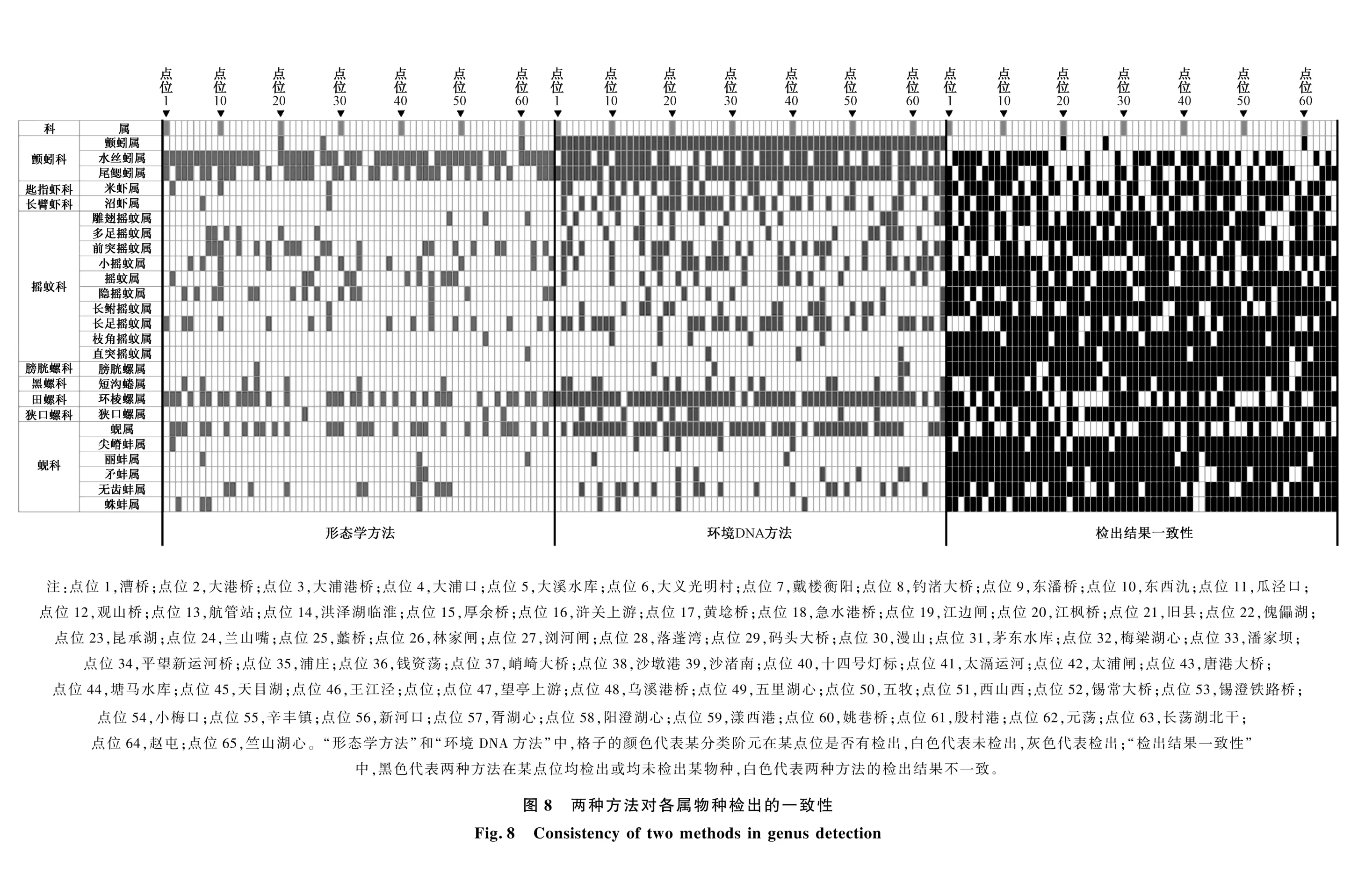
在底栖动物属水平的检出频次和检出种次上,环境DNA 方法与形态学方法呈显著线性相关(P<0.000 1,图7)。 底栖动物流域分布特征存在较高的一致性,1 625 个点位-物种单元格中,共有1 175 个在两种方法中获得了一致的检出结果,一致率达到了72.3%(图8)。 同时,热点图也直观地表明,水丝蚓属、尾鳃蚓属、环棱螺属、蚬属、前突摇蚊属、长足摇蚊属等类群是太湖流域较为普遍存在的类群,均被两种方法高频率地检出。
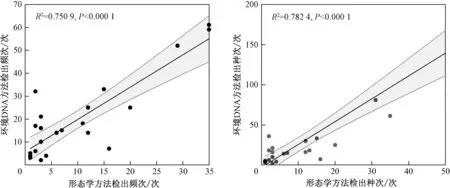
图7 两种方法对各属检出率的比较Fig.7 Comparison of the detection rate of each genus by two methods
2.2 环境DNA 方法预测生物量
摇蚊科和颤蚓科是太湖流域最为常见的底栖动物类群,也是重要的水生态指示类群[30]。 本研究发现,对于摇蚊科和颤蚓科底栖动物,在各点位测得的该类群序列数与形态学方法记录的个体数及生物量之间,具有显著的相关性(P<0.05,图9)。 其中,摇蚊科的相关性较强,无论是序列数与个体数(R2=0.737,P<0.001),还是序列数与生物量(R2=0.545,P<0.001),都存在非常显著的相关性。 而颤蚓科序列数与个体数、生物量之间的相关性较弱,但仍然显著(P<0.05)。 上述结果表明,基于环境DNA 获得的序列数可以用于定量预测摇蚊科和颤蚓科的个体数和生物量。
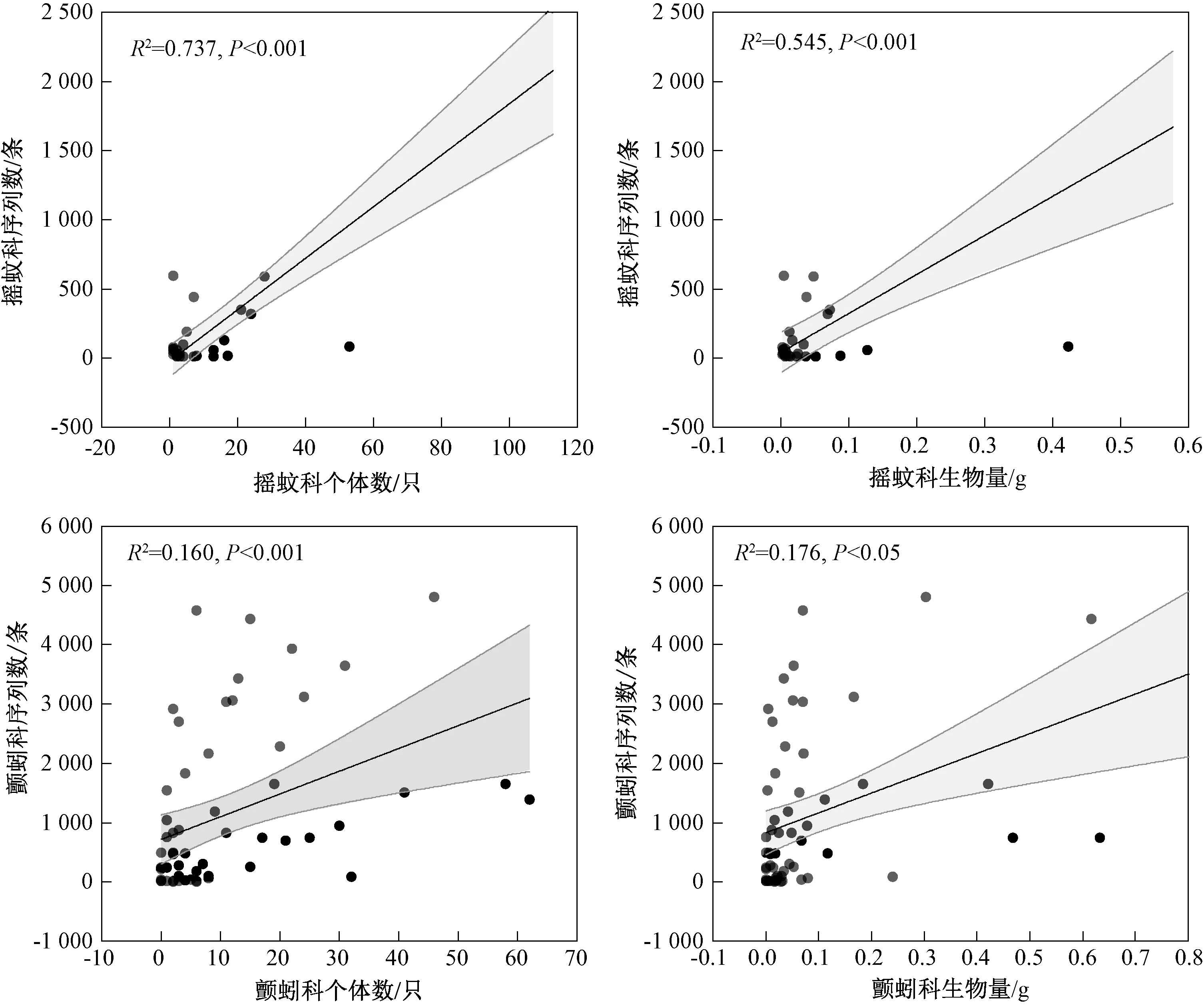
图9 序列数与个体数、生物量的相关关系Fig.9 Correlation between reads and individuals or biomass
2.3 生态评价
2.3.1 生物指数
基于环境DNA 方法与形态学方法监测结果计算得到的软体动物分类单元数(R2=0.381)、BMWP 打 分 值(R2= 0.287)、 B-IBI 值(R2=0.235)显著线性相关(P<0.000 1,图10)。 软体动物分类单元数和BMWP 打分值是底栖动物生态评价中的常用指标,也是太湖流域B-IBI 值的组成部分。 由图10 中的箱型图可知,采用环境DNA 方法得到的B-IBI 值能够较好地区分基于形态学方法监测结果划分的不同生态健康等级的点位,对于参考点(良)、受损点(差)能够很好地进行区分,箱体无重叠。
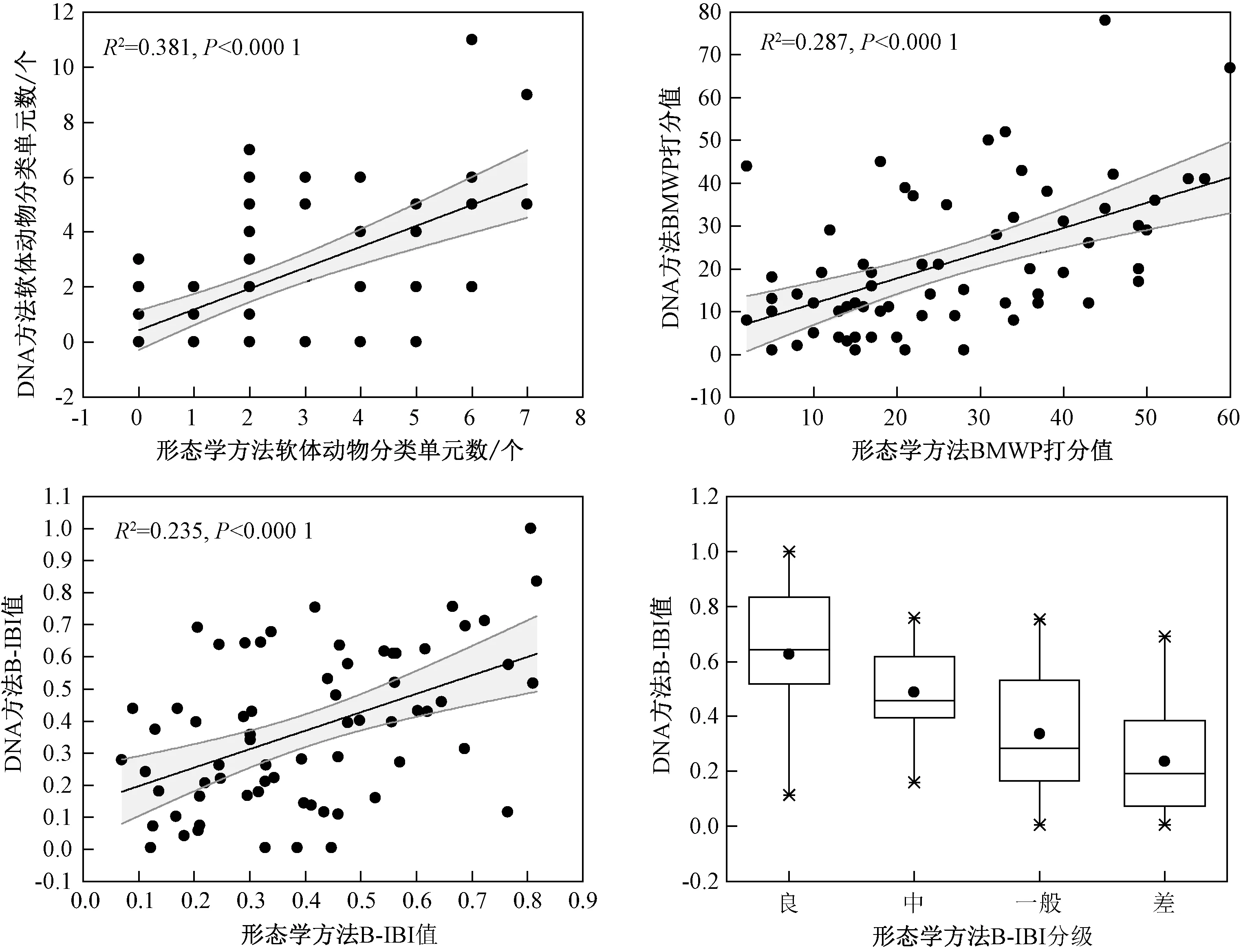
图10 两种方法计算生物指数的相关性Fig.10 Correlation of biological indices by two methods
2.3.2 流域尺度生态健康评价
在对点位进行生态健康等级划分方面,根据环境DNA 方法监测数据计算得出的B-IBI 结果与基于形态学方法得出的结果基本一致。 40%的点位取得了与形态学方法一致的B-IBI 等级,绝大多数点位(94%)的B-IBI 等级和形态学方法评价结果的偏差在1 级以内。 利用插值法绘制B-IBI 值的地理分布图,反映出整个太湖流域江苏片区的底栖动物完整性状况(图11)。 环境DNA方法监测结果显示,在流域尺度上,太湖东西两岸、太湖东北(钓渚大桥周边)、太湖流域西侧(大溪水库周边)的底栖动物完整性状况较好。 形态学方法监测结果呈现出类似的格局,两种方法的高值区分布高度一致。
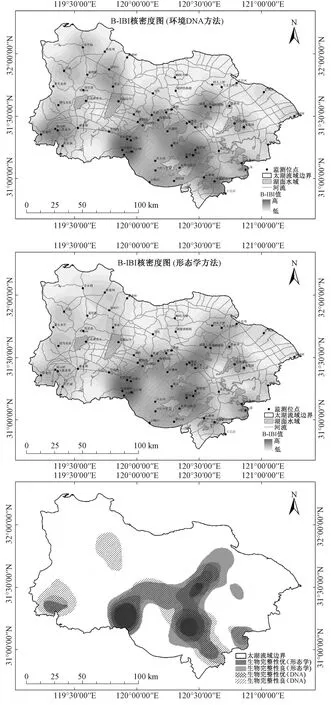
图11 太湖流域底栖动物完整性Fig.11 The integrity of macroinvertebrate in Taihu Lake Basin
3 讨论
3.1 物种鉴别的一致性
本研究采用的酒精浸提法是一种适用于底栖动物监测的环境DNA 样品前处理方法,相较于组织研磨或者标本浸提等前处理方式,酒精浸提无需对其中的底栖动物个体进行挑拣和初步分类,大大降低了前处理的时间和人力成本,也减小了交叉污染的风险。 相较于水样提取,利用底栖基质更能反映采样点原位的群落组成,样品中的底栖动物密度也更大。 本研究环境DNA 方法注释结果中,有54%的序列和90%的OTU 来自非靶向物种(非底栖动物),这是环境DNA 宏条形码监测中普遍存在的现象。 这些信号可能来源于浮游动植物或其他后生动物,也可能是由于物种条形码数据库中底栖动物数据的缺失[20,31]。
总体而言,环境DNA 方法在流域物种丰富度计算、常见种识别上,与形态学方法具有较高的一致性。 无论是在科还是属的分类水平上,环境DNA 方法与形态学方法的共同检出结果涵盖了形态学方法检出的大部分物种、绝大部分个体和生物量,代表了太湖流域底栖动物中分布较为广泛、检出频次较高、物种优势度较高的类群。 在本研究中,采用环境DNA 方法检出了形态学方法监测结果中40%的科和45.5%的属,共同检出的科和属分别涵盖了形态学方法检出物种数的64.7%和48.4%。 这一比例与DEINER 等[32](科水平73%交叉)、MACHER 等[31](EPTs 类群64%交叉)、MÄCHLER 等[33](属水平62%交叉)的研究结果相近。 而GLEASON 等[19]的研究表明,在利用水样环境DNA 进行底栖动物监测时,其结果与组织研磨样品环境DNA 分析结果的一致性较低。 此外,本研究中,在形态学监测中检出频率较高的分类单元,在环境DNA 方法中同样被高频检出,即环境DNA 方法在对流域常见种的识别上与形态学方法具有较高的一致性,两种方法对于判断某物种在流域范围内是否广泛存在具有相近的效果。 AYLAGAS 等[34]发现,利用形态学方法检出的优势物种同样也是利用环境DNA 方法识别出的优势物种。 HAJIBABAEI 等[35]的研究则表明,对于个体数不少于2 的物种,通过对标本进行酒精浸提,可以达到100%的检出率,这也印证了上述结论。 因此,使用酒精浸提法直接处理环境样品,并结合环境DNA 宏条形码技术进行底栖物种识别,在结果上与形态学方法具有较高的一致性。
环境DNA 方法比形态学方法识别出了更多的物种,既体现在总分类单元数目上,也体现在点位平均值上。 环境DNA 方法能够识别出在形态学挑拣中因肉眼识别困难而容易遗漏的物种,例如仙女虫科的某些种类。 尽管在数量上,两种方法共同检出的科较少,但共同检出的科都是具有很高代表性的类群,如摇蚊科、颤蚓科、田螺科、蚌科等,这些科的检出频次和种次都处于所有科的前列。 与科级水平的情况类似,共同检出的属也都是具有很高代表性、检出频次和种次较高的类群,如环棱螺属、蚬属、水丝蚓属、尾鳃蚓属、长足摇蚊属、小摇蚊属等。 而环境DNA 方法检出次数较少的物种主要为沙蚕、蛭等动物。 此类动物在接触高浓度乙醇后会迅速固缩,其组织释放较慢,可能导致浸提效率较低。 另外,一些毛翅目、蜻蜓目的物种在环境DNA 方法中的检出次数较少,可能是由于个体数少且出现率低,降低了环境DNA方法的检出率。 在MACHER 等[31]的研究中,环境DNA 宏条形码方法对EPT 类群(蜉蝣目/衤责翅目/毛翅目)的识别率仅有8%(4/51),说明现有方法对该类群的检出能力可能较弱。
在流域尺度的空间分布格局上,环境DNA 方法监测结果与形态学方法监测结果呈现多方面的一致性。 两种方法在检出频次和检出种次上均呈现显著正相关(R2>0.7,P<0.000 1)。 在形态学鉴定中频繁检出的分类单元,在环境DNA 方法中同样频繁检出;在形态学鉴定结果中较为罕见的分类单元,在环境DNA 方法中同样较少检出。 在太湖流域,采用环境DNA 方法和形态学方法获得了相似的物种分布图谱,两种方法的总体检出一致率达到了72.3%。 因此,环境DNA 方法在物种包括优势种的识别上是有效的,与形态学方法具有较高的一致性,能够用于太湖流域尺度的底栖动物多样性监测。
3.2 定量关系
环境DNA 方法在优势物种的定量分析上与形态学方法具有一致性。 其中,摇蚊科和颤蚓科物种在环境样品中的生物密度和生物量,与环境DNA 方法检出的序列数显著正相关(P<0.05),尤其是摇蚊科的相关性较强,R2分别为0.737 和0.545。 摇蚊科和颤蚓科的生物密度往往非常大,采用人工计数的方法较为烦琐。 此时,环境DNA数据提供的帮助就不容忽视。 对物种生物密度和生物量的估算是环境DNA 研究中的一项重要挑战。 目前,已有一些研究表明,环境DNA 可以定量表征浮游动物和鱼类等类群的生物多样性[36-37],但是对于底栖动物的定量研究还较少且不深入。 CAREW 等[38]和LINARD 等[20]研究发现,在标本浸提液样品中,部分底栖动物的生物量与序列数具有相关性。 AYLAGAS 等[34]还发现了个体数与序列数之间的显著相关性。 但在大块环境样品的浸提液中,将环境DNA 测序数据用于物种定量的可行性尚不明确,亦有研究认为难以发掘其定量关系[39]。 尽管底栖动物定量监测不是本研究的主要目标,但本研究的结果表明,在复杂的环境样品中,存在DNA 序列数与底栖动物生物密度、生物量之间的相关性。
3.3 生态评价的一致性
在流域尺度上开展的生态评价表明,由环境DNA 监测数据推导出的B-IBI 值具有显著的辨识度。 环境DNA 监测结果在软体动物分类单元数、BMWP 打分值和B-IBI 值上,与形态学监测结果具有一致性,可用于评估水生态健康状况。 在流域生态健康空间格局的描绘上,两种方法的高值区高度重合,具有一致性。 已有部分研究表明,将环境DNA 宏条形码技术用于生态评价,可以获得与形态学方法类似的结果。 例如,CLARKE 等[40]和MARTINS 等[41]分别在针对浮游动物和底栖动物群落的研究中发现,通过环境DNA 方法和形态学方法可以得到相似的β 多样性,尽管两种方法得到的α 多样性之间差距较大。 这与本研究结果类似,即两种方法在物种识别上的结果并不完全一致,但在生态评价上的结果较为相近。EMILSON 等[42]和SERRANA 等[43]的研究表明,基于DNA 方法和形态学方法检出的底栖动物群落,对理化因子的环境梯度有着相似的响应。 而ERDOZAIN 等[44]研究发现,相比形态学方法,环境DNA 方法检出的底栖动物群落对环境变量的响应较弱。 MARSHALL 等[45]发现,基于两种方法对底栖动物的监测结果,可以对栖息地类型进行类似的区分。 此外,有证据表明,使用多引物组合可以更加全面地解释群落结构,使生物评价的结果更加准确[41,46]。 综上所述,尽管在物种识别层面有时略有出入,但这并未成为将环境DNA 宏条形码技术应用于水体生态健康评价的障碍。 将环境DNA 监测结果用于太湖流域底栖动物完整性评价是可行的,并且在很大程度上与形态学方法的评价结果是一致的。
4 结论与展望
将环境DNA 宏条形码技术与形态学方法同步用于针对太湖全流域的底栖动物监测,监测结果表明,两者在物种识别和生态评价方面均具有较高的一致性。 环境DNA 方法可以识别出太湖流域底栖动物中分布较为广泛、检出频次较高、优势度较高的类群,且能比形态学方法识别出更多的物种。 两种方法在各物种的检出频次上具有较强的相关性(P<0.000 1)。 基于环境DNA 方法得到的B-IBI 值与形态学监测结果显著相关,且在底栖动物完整性的流域空间格局描绘上高度一致。 此外,两种方法还在生物量、序列数等方面呈现出一定的定量关系。
未来,需进一步完善本土物种DNA 条形码数据库,以提高测序数据的物种注释率和注释结果的可靠性。 此外,生物密度和生物量是生物监测和评估中的重要信息,因此,建立基于环境DNA的定量生物监测技术具有重要意义。 综上,环境DNA 宏条形码技术在大规模、多点位底栖动物多样性快速监测及相关评价工作中具备较高的可靠性,具有广泛的应用前景,未来有望显著提高我国水生态系统生物监测和生态健康评估的技术水平。

