罗源地区秀珍菇栽培菌株亲缘关系初步分析*
施乐乐
(福建省食用菌技术推广总站,福建 福州 350000)
秀珍菇(Pleurotus pulmonarius) 学名为肺形侧耳,又名袖珍菇、姬平菇,隶属于担子菌门(Basidiomycota) 伞菌目 (Agaricales) 侧耳科 (Pleurotaceae) 侧耳属(Pleurotus)[1],20世纪 90年代由中国台湾引进内地试种并取得成功[2]。罗源县是福建省秀珍菇主产区,2020年全县秀珍菇产量达14.2万吨,产值9.7亿元,栽培秀珍菇成为菇农创收致富的重要途径。目前罗源秀珍菇主栽品种主要有:秀57、秀63、秀93、秀98、秀912、台秀122等,上述品种由不同栽培企业在不同时期从中国台湾地区不同机构引进,经过多年继代扩繁,栽培企业又对引进品种进行了重命名,造成罗源秀珍菇栽培品种名称混乱,同种异名或异种同名现象极为普遍,不利于秀珍菇产业的健康发展。
近年来,分子标记技术已广泛应用于食用菌等大型真菌的分类、鉴定,其具有模板需求量少、操作简便、多态性高等诸多优点[3-5],分子标记技术的出现弥补了传统分类鉴定方法的不足,进一步提高了食用菌分类、鉴定的准确性。研究共收集罗源地区秀珍菇栽培菌株16株,并以福建农林大学菌物研究中心保藏的1株野生秀珍菇菌株为对照,通过平板拮抗试验及RAPD、ISSR、SRAP分子标记技术对其进行亲缘关系分析,以期在分子水平上为解决罗源秀珍菇栽培品种混乱问题提供数据支撑和理论依据。
1 材料与方法
1.1 供试菌株
供试菌株共17株,其中16株收集自罗源秀珍菇栽培企业及菇农,另有1株为福建农林大学菌物研究中心提供。供试菌株来源信息详见表1。

表1 供试秀珍菇菌株Tab.1 Tested strains of Pleurotus pulmonarius
1.2 培养基
马铃薯葡萄糖(PD) 液体培养基:去皮马铃薯200 g切成小块,加1 000 mL水,水煮30 min,双层纱布过滤,滤液中加入2%葡萄糖,并补足因蒸发失去的水分,pH自然,121℃灭菌20 min。
马铃薯葡萄糖(PDA)固体培养基:需另加入2%琼脂,其余成分同马铃薯葡萄糖(PD)液体培养基。
1.3 平板拮抗试验
将不同秀珍菇菌株两两接种至平板中,设置3个重复,菌丝萌发后,每天观察平板菌落交界处的拮抗情况及平板菌落形态差异情况,做好记录统计,无拮抗记为“-”,有拮抗记“+”,“+”的数量表示拮抗的强弱。另用“*”表示菌落形态差异情况,“*”的数量表示菌落形态差异的程度。
1.4 分子标记试验
用改良CTAB法[6]提取17个供试菌株DNA,提取后测定DNA浓度,经凝胶电泳检测合格后,进行RAPD、ISSR和SRAP三种标记PCR。引物及反应体系见表2和表3。
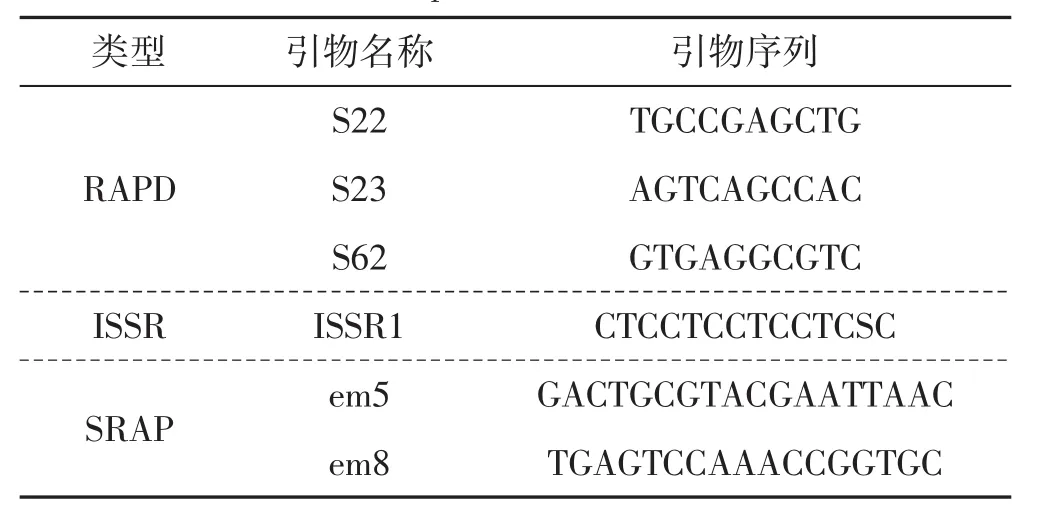
表2 PCR引物Tab.2 The primers of PCR reaction

表3 PCR反应体系Ta.3 The PCR reaction system
RAPD-PCR反应程序为:94℃预变性7 min;94℃变性 1 min,34℃退火 1 min,72℃延伸 1 min,35个循环;72℃延伸10 min;4℃保存。
ISSR-PCR反应程序为:94℃预变性5 min;94℃变性1 min,48℃退火 1 min,72℃延伸 1 min,35个循环;72℃延伸10 min;4℃保存。
SRAP-PCR反应程序为:94℃预变性5 min;94℃变性1 min,35℃退火 1 min,72℃延伸 1 min,5个循环;94℃变性1 min,50℃退火1 min,72℃延伸1 min,35个循环;72℃延伸10 min;4℃保存。
1.5 数据统计和分析
在RAPD、ISSR和SRAP扩增结果电泳图谱中,按凝胶同一位置上DNA条带的有无进行统计,有条带用“1”表示,无条带用“0”表示。采用NTSYSpc-2.10e分析软件计算样品间的遗传相似性系数,同时用UPGMA进行聚类分析,构建聚类图。
2 结果与分析
2.1 平板拮抗试验
不同秀珍菇菌株平板拮抗试验结果见表4。

表4 不同秀珍菇菌株平板拮抗与菌落差异情况Tab.4 Plate antagonism and colony morphology difference of Pleurotus pulmonarius
由表4可知,菌株P916、P624、57-0、GD三代之间不存在拮抗,菌株P620和金秀之间不存在拮抗,菌株P615和X57之间不存在拮抗,菌株P626和P650之间不存在拮抗,其余菌株之间均存在拮抗;观察不同供试菌株的菌落形态,发现供试秀珍菇菌株之间的菌落形态均存在一定差异。但平板观察结果相对主观,仅能作为菌株亲缘关系初步判断的依据。
2.2 分子标记试验
对提取的17株秀珍菇菌株DNA进行RAPDPCR、ISSR-PCR和SRAP-PCR扩增,扩增所得的DNA图谱见图1~图3。
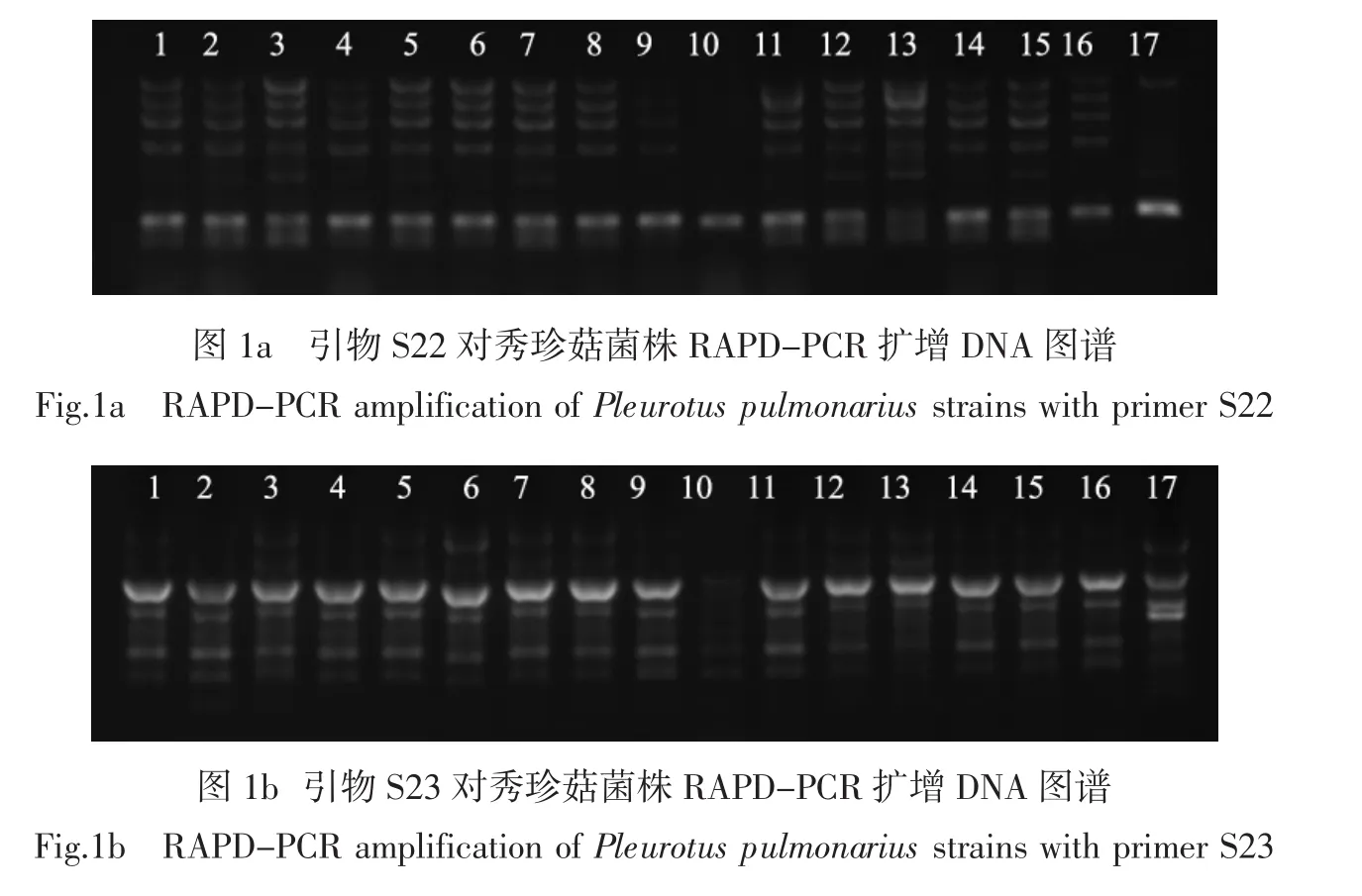

图1 秀珍菇菌株RAPD-PCR扩增DNA图谱Fig.1 RAPD-PCR amplification of Pleurotus pulmonarius strains

图2 引物ISSR1对秀珍菇菌株ISSR-PCR扩增DNA图谱Fig.2 ISSR-PCR amplification of Pleurotus pulmonarius strains with primer ISSR1
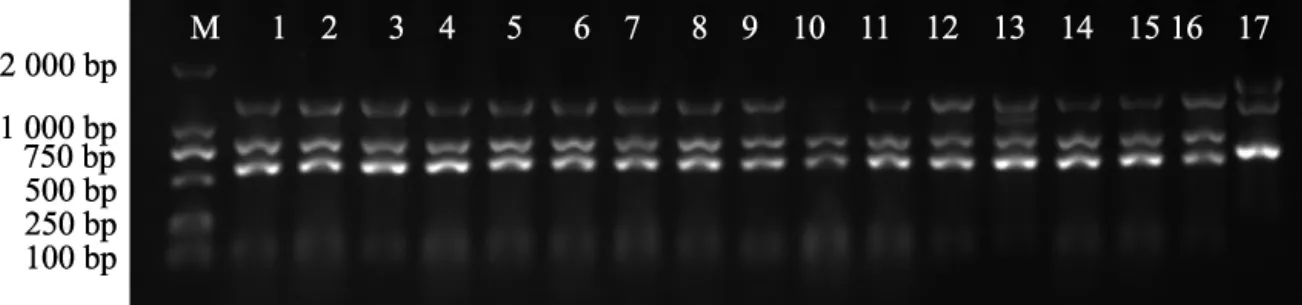
图3 引物em5-em8对秀珍菇菌株SRAP-PCR扩增DNA图谱Fig.3 SRAP-PCR amplification of Pleurotus pulmonarius strains with primer em5-em8
由图1~图3DNA图谱可知,第10泳道的P627和第17泳道的对照菌株秀2与其他供试菌株的电泳条带差异较大,这表明P627和秀2与其他供试菌株的遗传差异较大。
2.3 遗传距离综合聚类分析
计算各供试菌株的相似系数,并构建遗传距离综合分析聚类图,结果见图4。

图4 秀珍菇遗传距离综合分析聚类图Fig.4 Cluster diagram of Pleurotus pulmonarius strains
由图4可知,17株供试菌株遗传相似系数在0.45~1.00。当遗传距离D为0.68时,P627和秀2各被聚为1个单一群体,其余菌株被聚为一个复合群体,这表明P627和秀2与其余供试菌株的亲缘关系较远,遗传差异较大,除了P627和秀2外,其余供试菌株的亲缘关系均较近;当遗传距离D为1.00时(D为1,表示很可能为同一菌株),P605和P625被聚为一个群体,P916、P624、57-0、GD三代被聚为一个群体,这表明菌株P605和P625可能分属同一菌株,菌株P916和P624、57-0、GD三代可能分属同一菌株。
3 讨论与结论
传统的分类学鉴定方法是指在微观和宏观的层面上对食用菌的内部结构和外部形态进行分类鉴定的方法[7]。其通过直观观察的方式在一定程度上能对食用菌类群进行划分,但由于食用菌的形态特征常因生境的不同而存在较大差异,且近缘物种间表观特征相似,使得单纯依靠传统分类学鉴定方法鉴定食用菌存在局限性[8]。随着生物技术的快速发展,真菌分类学家结合核酸分子生物学鉴定技术建立了更为有效的分类鉴定方法,即分子标记。分子标记能直接反映生物个体或种群间基因组的差异,因其不受材料的限制,且具有数量多、多态性高、准确性可靠、不影响生物个体目标性状表达的特点,已被广泛应用于食用菌的分类鉴定中。
研究通过平板拮抗试验及分子标记技术对供试秀珍菇菌株进行亲缘关系分析,试验结果表明:P627菌株和野生对照菌株秀2与其余15株供试菌株的亲缘关系较远,而其余15株供试菌株之间的亲缘关系均较近。在亲缘关系较近的供试菌株中,P605和P625可能分属同一菌株,P916和P624、57-0、GD三代可能分属同一菌株。研究后续将会开展所有供试菌株的出菇试验,通过供试菌株农艺性状的比较以进一步确定各供试菌株的亲缘关系。
——福建省罗源第一中学简介之二
——福建省罗源第一中学简介之一

