三种DNA条形码对卤虫分类和进化的比较研究
左佳俊,徐 倩,邓元告,梁雪娇,韩学凯,隋丽英
(亚洲区域卤虫参考中心,天津科技大学海洋与环境学院,天津 300457)
卤虫是一种广温性、耐高盐的小型甲壳类浮游动物,分布于日晒盐场和内陆盐湖高盐水域中.目前普遍认为卤虫包含8大种类[1],其中5种为来自旧大陆的A. salina、A. urmiana[2]、A. sinica[3]、A. tibetiana[4]和Artemiasp.[5],2种为来自新大陆的A. franciscana[5-6]和A. persimilis[7],1个孤雌生殖种,被命名为A. parthenogenetica[8].同时因地域和生境的不同,形成不同的品系.营养丰富的卤虫是水产苗种极佳的鲜活饵料.不同品系卤虫卵及其无节幼体的品系特征不同,在大小、营养和孵化特性等方面差别较大,而这些差异也决定了不同品系的卤虫作为开口饵料的效果不同[9].单一地理卤虫品系中存在着不同种的卤虫混杂共存现象,这给卤虫分类和系统进化研究工作带来挑战.
DNA条形码是在种内遗传变异始终低于种间遗传变异的假设下,利用短而标准化的基因区域作为内部标记进行物种和品系鉴定[10].如今该技术已被应用于卤虫种的分类,特别是形态特征相似卤虫种的鉴别[11].目前,已有多种DNA条形码技术被成功应用于卤虫的分类和系统发育研究.如线粒体DNA细胞色素氧化酶亚基I(COI)基因,因其序列相对保守,但又有足够的变异,而成为鉴定卤虫种最常用的DNA条形码[12-14].内部转录间隔区(ITS)序列为中度保守序列,ITS基因的转录产物在rRNA形成之前即被剪掉,不参与成熟核糖体的形成,因此面对的选择压力相对较小,进化速率快,被认为是在品系水平上对生物体进行分类的有效DNA标记[15-16]. 研究[17-19]表明ITS1可以作为卤虫系统发育研究的有效DNA标记.对于线粒体核糖体RNA基因,16S或12S核糖体RNA基因也被认为是卤虫系统发育研究的有效标记[18-20].16S rRNA与12S rRNA基因序列常常被单独使用,但是一些物种间的进化关系仅显示于某些基因中,而通过“联合”途径直接利用所有序列位点提供进化信息,系统发育分析结果可能会更接近于正确的物种树.虽然目前关于DNA条形码已有较多相关研究,但是将16S rRNA与12S rRNA基因区域视为一个整体作为卤虫DNA条形码的研究则较少.同时也缺乏对这些不同DNA条形码的比较研究,特别是在卤虫的分类和鉴定方面.
本研究旨在通过比较线粒体基因条形码(COI基因和16S-12S rRNA基因)和核基因条形码(ITS1基因)的基因片段,建立适合于卤虫种水平鉴定的DNA条形码,并利用这3个标记对48个卤虫样品进行遗传距离和进化关系分析.
1 材料与方法
1.1 卤虫样本
本研究所用48个卤虫卵样本来自天津科技大学亚洲区域卤虫参考中心.4个参照卤虫卵样本ARC1709、ARC1187、ARC1188和ARC1321取自比利时根特大学卤虫参考中心.称量虫卵样品25mg,置于盛有10mL 3%稀释卤水的试管中进行水合;水合3h后,在充气状态下加入100µL脱壳液(8%的NaClO溶液与30%的NaOH溶液的体积比为20∶1)去壳,3min后用清水冲洗去壳卵,直至无NaClO气味.将冲洗后的去壳卵收集于1.5mL EP管中,75%酒精冲洗1次,灭菌ddH2O冲洗3次.处理好的卤虫卵于-20℃临时保存,并用于下一步DNA提取.
1.2 DNA提取、PCR扩增和测序
将处理保存的卤虫卵运用研磨法制备组织液,用TIANGEN®动物组织DNA提取试剂盒从组织液中提取全基因组DNA.利用Primer Premier 6.06(http://www.premierbiosoft.com/primerdesign/)软件从GenBank数据库中获得卤虫的基因序列,设计引物.本研究将16S rRNA基因与12S rRNA基因结合在一起作为一个DNA标记,单独设计扩增16S-12S rRNA基因片段的引物.扩增COI、16S-12S rRNA和ITS1基因片段的引物见表1.

表1 用于扩增线粒体COI、16S-12S rRNA基因区域以及核内ITS1基因区域的引物Tab. 1 Primers designed for amplifying COI,16S-12S rRNA and ITS1 gene
对COI基因片段、16S-12S rRNA基因区域以及核内ITS1基因进行PCR扩增,反应体系(25μL):1μL模板DNA,12.5μL PremixTaqDNA聚合酶(日本Takara公司),上下游引物各1 μL,9.5 μL无菌ddH2O.扩增反应:4℃预变性5min;94℃变性1min,退火1min(COI、16S-12S rRNA、ITS1基因的PCR退火温度分别为54、56、58℃),72℃延伸1.5min,30个循环;72℃延伸10min.由于Shuanghu 2014、ARC1188、Yuncheng 2012、Inner Mongolia 2011、Naritusumu 2013、ARC1709和ARC1321样品的ITS1基因序列剪接失败,因此利用单个卤虫卵代替混合卵用于ITS1序列扩增,PCR反应中加入6μL模板DNA.PCR产物送华大基因公司测序.利用在线软件 Cloud Splicing(http://customer.genomics.cn/customer-self/home#/show/pinjie-view)进行基因拼接.COI、16S-12S rRNA和ITS1基因片段测得的正反向序列成功拼接后,获得的序列上传到GenBank数据库(表2),部分测序拼接未成功的则没有上传.

表2 实验所用卤虫卵样本信息Tab. 2 Information on Artemia cysts samples used in this study
1.3 数据分析
通过MEGA 6软件(http://www.mega.com/)[21]实现多序列比对、序列信息分析和Neighbor-Joining进化树的构建.Neighbor-Joining进化树构建使用最大组成似然法(maximum composite likelihood method),自展检测(Bootstrap test)设置为1000次.利用Genedoc 2.7.000(http://www.softpedia.com/get/Science-CAD/GeneDoc.shtml)软件进行多序列比对.整体转化/颠换比率(R)计算公式为
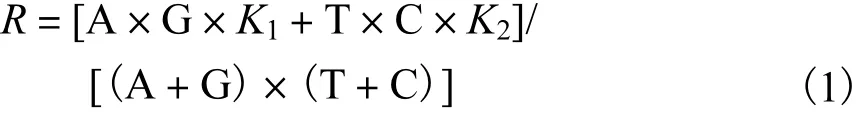
式中:A、T、C、G为碱基数,K1和K2分别代表嘌呤和嘧啶的转化/颠换比率.遗传距离使用最大复合似然法计算,并使用Kimura 2参数模型[22]进行分析.
2 结果与分析
2.1 序列扩增、测序和比对
分别用3种DNA分子标记对48份卤虫卵扩增测序.扩增的COI、16S-12S rRNA和ITS1基因片段长度分别为1597bp、1238bp和1089bp.对各个序列进行比对和修剪后,用于分析的最终片段长度分别为1197bp、1007~1020bp和676~942bp.本研究使用的所有序列均已保存在GenBank数据库中,通过BLAST序列比对结果见表2.BLAST结果表明,3种标记对卤虫种的鉴定结果一致.
分别对44份、47份和42份卤虫样本进行COI、12S-16S rRNA和ITS1基因的拼接.在使用混合卵时,同一样品的序列在电泳图上存在有规则的重叠峰,导致无法提供参考序列.为了排除卤虫卵样品被其他物种污染的可能性,进一步将单个卤虫卵作为实验材料.同一样本可获得理想序列,只有少数样本未能成功拼接,其中以ITS1测序样本最多,这可能与ITS1基因非编码区存在多拷贝有关.
分别对COI、16S-12S rRNA和ITS1基因片段进行多序列比对,未发现COI基因序列插入或缺失.与16S-12S rRNA基因序列中获得的少量插入不同,序列插入在ITS1基因中频繁出现.A. franciscana、A.persimilis和A. salina的样本插入或缺失较长,插入最长的有28个核苷酸(图1).

图1 部分ITS1核苷酸序列的多重比对结果Fig. 1 Multiple alignment of partial ITS1 nucleotide sequences
2.2 核苷酸组成和替代
各位点及密码子各位置上核苷酸组成见表3.COI、16S-12S rRNA和ITS1基因的A+T含量分别为62.5%(24.0%+38.5%)、66.1%(30.9%+35.2%)和53.1%(24.9%+28.2%).COI和16S-12S rRNA基因序列有轻微的A+T偏好性.16S-12S rRNA基因的碱基频率表现出A+T偏好性,相比于COI基因,15条16S-12S rRNA序列密码子各位上碱基比例的多态性更明显些.
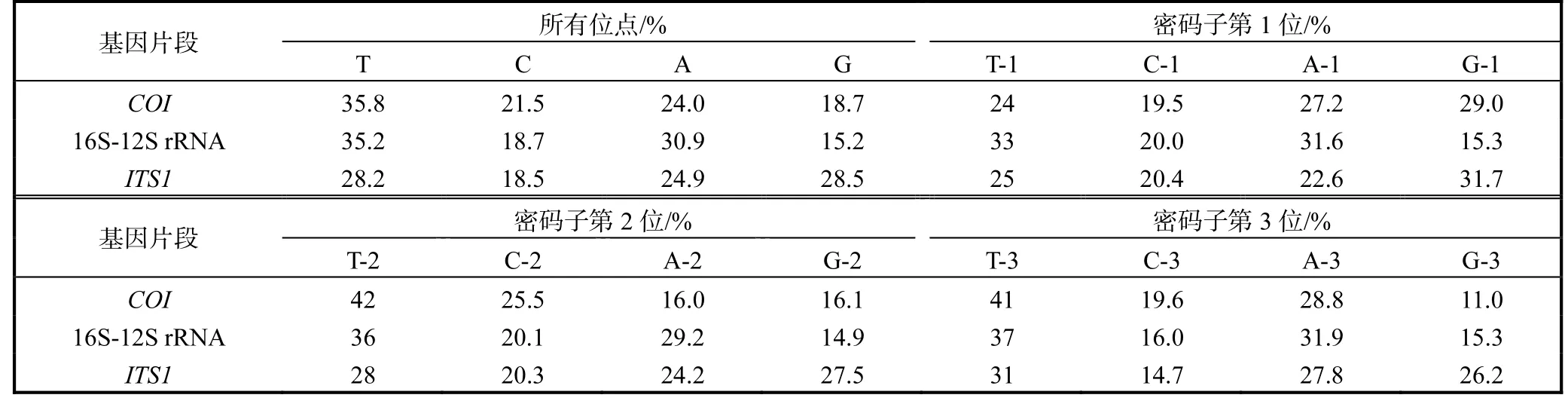
表3 各位点及密码子各位置上核苷酸组成Tab. 3 Percentage of nucleotide composition at all sites and three codon positions
序列的碱基替换矩阵见表4.与颠换替代率相比,转化替代率占主导地位.各基因片段最大转化/颠换值为COI基因片段的T对C为41.15%,16S-12S rRNA基因片段的A对G为32.95%,ITS1基因片段的T对C为19.17%.COI基因序列的转化/颠换的比率为K1=17.759(嘌呤)和K2=32.257(嘧啶).整体转化/颠换比率R值为13.41.16S-12S rRNA基因序列K1=8.326,K2=3.652,R=2.539.ITS1基因序列K1=2.131,K2=3.132,R=1.262.
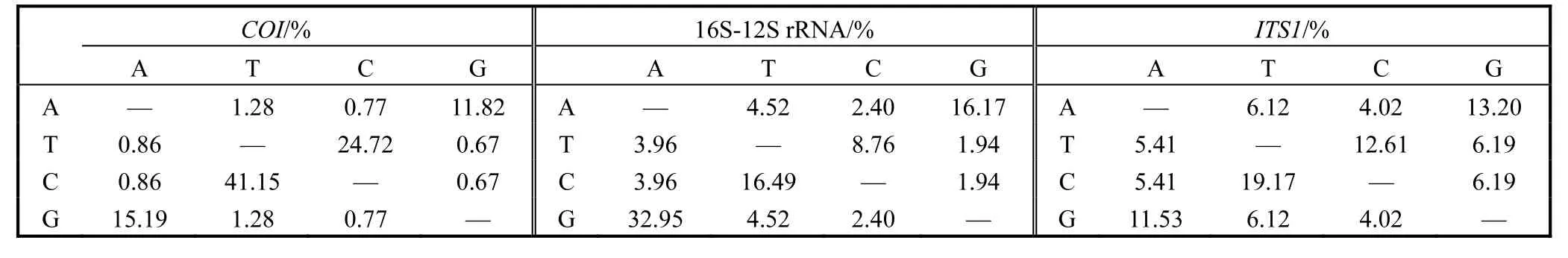
表4 序列的碱基替换矩阵Tab. 4 Nucleotide substitution matrix for markers
2.3 遗传距离
基于3个基因片段的种间(对角线下)和种内(对角线)遗传距离见表5.COI、16S-12S rRNA和ITS1序列的平均种内差异分别为0.6%、1.4%和0.6%.COI、16S-12S rRNA和ITS1序列的平均种间差异分别为16.2%、17.6%和9.1%.A. parthenogenetica、A.urmiana、A. tibetiana和A. sinica的种内和种间遗传距离均较低.
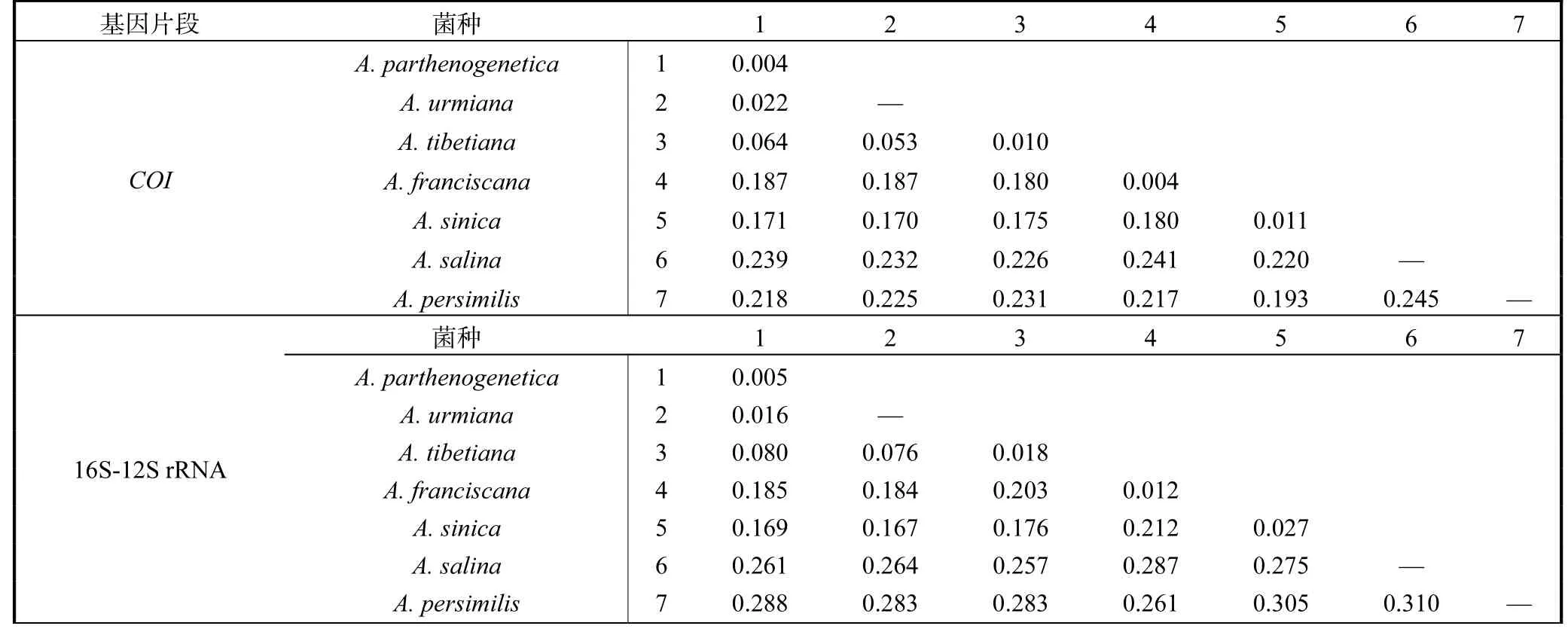
表5 基于3个基因片段的种间(对角线下)和种内(对角线)遗传距离Tab. 5 Interspecies(below diagonal)and intraspecies(diagonal)genetic distance based on three gene fragments of Artemia
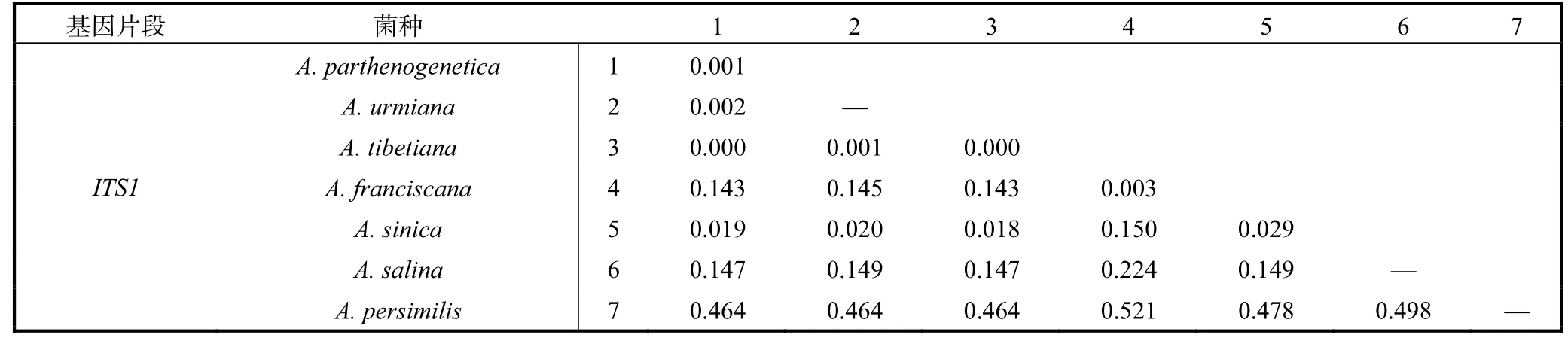
续表
2.4 系统进化树
采用Neighbor-Joining方法构建基于3个标记的系统进化树(图2—图4).

图2 基于COI基因序列构建的系统进化树Fig. 2 Phylogenetic tree based on COI gene sequences
根据COI基因系统进化树(图2),将样本分为7大类群.其中A. parthenogenetica与A. urmiana聚集后再与A. tibetiana聚集,形成明显的分枝.而A.franciscana和A. sinica聚集,再与上述3个种聚集.进化树的基部位置为A. persimilis,其次为A.salina.基于16S-12S rRNA基因的聚类结果(图3)与基于COI基因的结果比较相似.
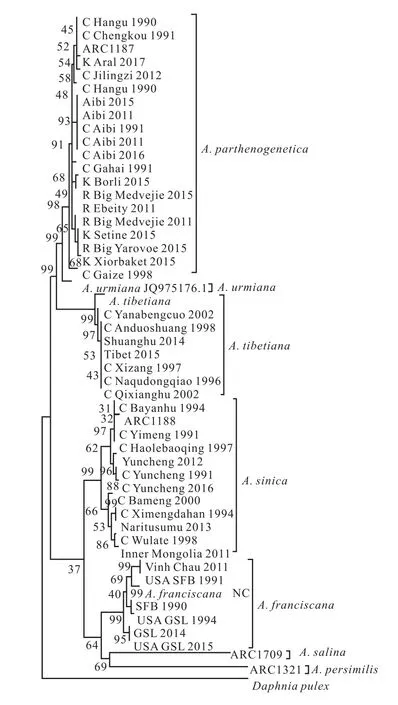
图3 基于16S-12S rRNA基因序列构建的系统进化树Fig. 3 Phylogenetic tree based on 16S-12S rRNAgene sequences
与COI不同的是,A. franciscana先与A. salina和A. persimilis聚集,然后与A. sinica聚集,进化树的根部也不再只是A. pesimilis.
基于ITS1基因构建的系统进化树(图4),A.franciscana、A. salina和A. persimilis的聚类情况与基于COI基因的结果相似.其余样本的分类结果则不一致,尤其是A. parthenogenetica、A. urmiana、A.tibetiana和A. sinica杂乱地聚集而无法分开.
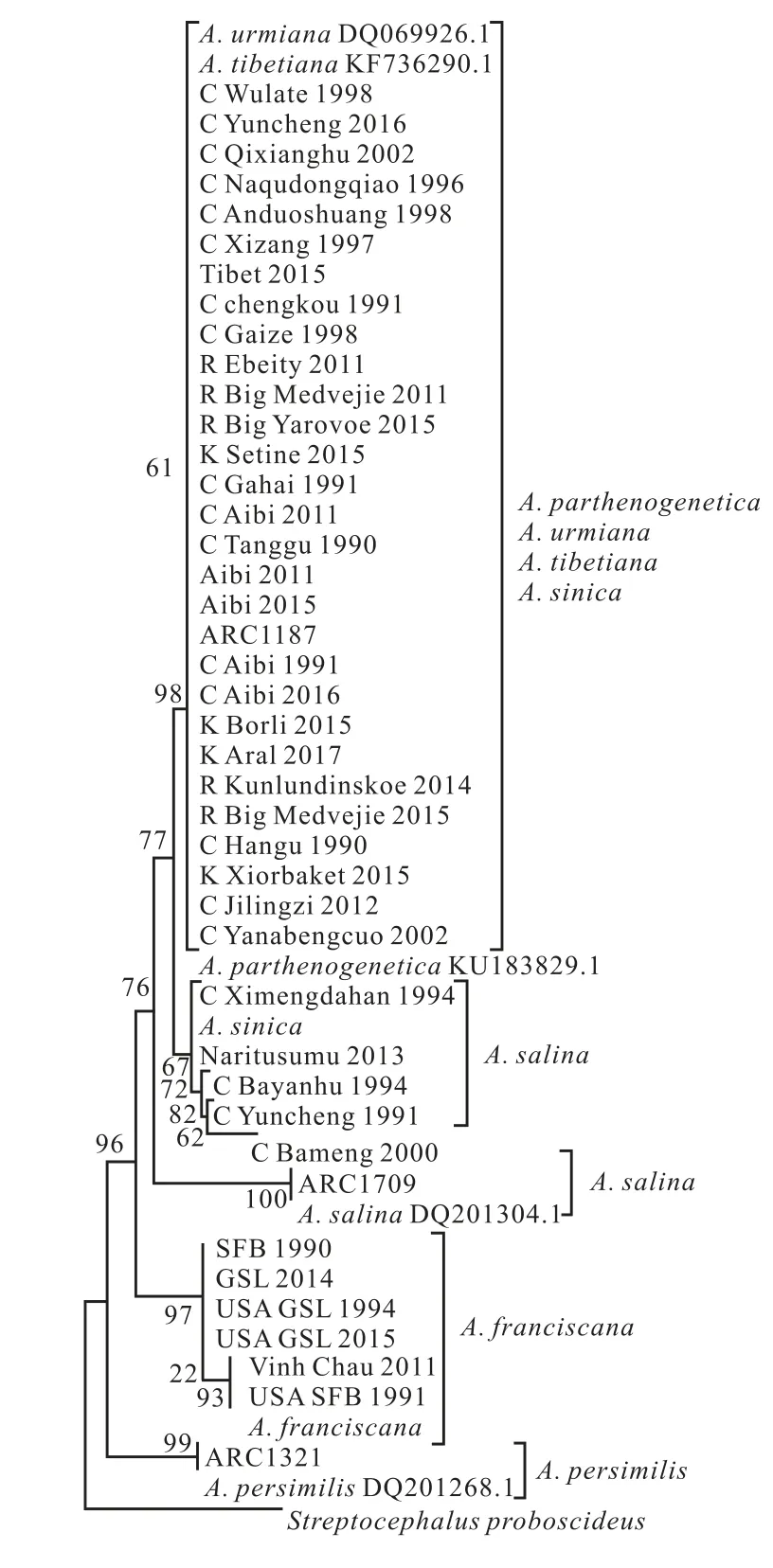
图 4 基于ITS1基因序列构建的系统进化树Fig. 4 Phylogenetic tree based on ITS1 gene sequences
3 讨 论
一般而言,鸟类、哺乳动物等高等生物的线粒体DNA中G+C含量较高,而无脊椎动物等低等生物的线粒体DNA中A+T含量较高[23-24].卤虫作为低等浮游甲壳动物,其COI和16S-12S rRNA基因序列有轻微A+T偏好性,而核基因ITS1则无明显A+T偏好性.替代率表示从一个碱基到另一个碱基的替代概率[25].众所周知,在线粒体和核基因中,转化替代比颠换替代更有优势[26-27],而在线粒体DNA(mtDNA)中这一优势更为显著[28].在本研究中,3个组基因片段的转换替换率均高于颠换替换率,这与上述原则一致.这可能是卤虫不同地方种群之间长期隔离没有基因交换导致的,而这将有利于维持卤虫种群繁衍和进化的稳定性.序列比对发现相较于COI和16S-12S rRNA基因序列,在ITS1基因序列插入中频繁出现,尤其A. franciscana、A. persimilis和A. salina的样本插入或缺失较长,说明A. francis-cana、A. persimilis和A. salina三者间ITS1基因的种间差异较大,这也是相关研究认为ITS1基因在卤虫鉴定方面具有较高潜力的原因之一.
遗传距离反映了不同物种间的遗传关系.研究表明COI基因标记在不同物种间的遗传差异应大于3%的阈值[11].本研究基于COI和16S-12S rRNA标记的种内距离数据均小于阈值3%,与理论相符.然而,由于某些物种间的亲缘关系非常密切,因此基于3个标记的种内变异并非都小于3%,如ITS1标记的种内距离数据超过3%.此外,本研究中一些亲缘关系密切的物种,3个标记的遗传距离矩阵中存在种内差异和种间差异的重叠.如在原始COI遗传距离数据中,样本ARC1188与Inner Mongolia 2011的种内差异为2.1%,大于序列A. urmianaJQ975176.1与A.parthenogeneticaKF707700.1的种间差异(数据未列出),这一方面可能是个别卤虫种之间的亲缘太近所致,另一方面可能是相关分子标记并不完全适用于近缘种间区分.
DNA条形码在物种分类和鉴定中起着重要的作用.为了比较和确定3种DNA条形码的有效性,构建卤虫系统进化树并进行聚类分析.COI和16S-12S rRNA系统进化树显示,卤虫样品被分为7个类群,与传统分类一致,而基于ITS1系统进化树的聚类则不同,将A. parthenogenetica、A. urmiana、A. tibetiana和两个A. sinica样本聚集.Baxevanis根据COI和16S-12S rRNA基因树将不同地区的两性卤虫样本聚类为6个类群(A. urmiana、A. tibetiana、A. sinica、A.franciscana、A. persimilis和A. salina)[19].也有研究[17]表明,两性卤虫根据COI序列可划分为6个不同的遗传类群,而ITS1树则显示不出完整的谱系,并由此推测孤雌生殖卤虫与A. urmiana和A. tibetiana的亲缘关系非常密切.这与本研究结果是一致的.
尽管ITS1基因区域被认为是卤虫种分类的潜在工具[12],本研究中A. parthenogenetica、A. urmiana和A. tibetiana的种内和种间遗传距离很低(0~0.7%),没有显示出ITS1基因的优势.结果表明,只有A.franciscana、A. sinica、A. salina和A. persimilis之间存在较大的遗传差异,而A. parthenogenetica、A. urmiana和A. tibetiana之间的种间差异不足以支持其在物种水平上分化.邹山梅[29]在以ITS1基因序列作为DNA条形码研究新腹足目贝类的系统发育关系时,也发现了ITS1序列没能成功地完成亚种水平的鉴定,其未能将N. festiva和N. festivaⅡ区分开.这可能的原因是尽管核基因ITS1在面对巨大的选择压力时具有很大的突变率,但它没有足够的时间产生新的变异,A. parthenogenetica、A. urmiana、A. tibetiana之间具有一些共同的单倍体也说明了这一点.此外,已有研究报道,A. urmiana和A. tibetiana亲缘关系非常密切,可能是一个复合种,A. tibetiana可能在这个复合种的起源和A. parthenogenetica的起源中发挥关键作用.这也部分解释了为什么在本研究中A.urmiana、A. tibetiana和A. parthenogenetica关系如此相近以至于ITS1基因很难将其分开.此外,考虑到样本量的影响,还需要大量的样本进一步验证ITS1基因用于卤虫种间和种内分类可行性.虽然应用于DNA条形码工作中的基因很多,但是目前卤虫系统分类研究所用基因条形码主要为COI、ITS1以及16S rRNA与12S rRNA.也有研究者利用核基因条形码Na/K ATPase开展卤虫系统分类学研究[30],发现Na/K ATPase基因可以区分两性生殖卤虫和孤雌生殖卤虫,但却难以显示完整的系统发育关系,这可能是由于线粒体基因比核基因具有更高的遗传变异.后期还需要开发更多高效基因条形码用于卤虫近缘种间或者品系鉴定研究.
本研究利用3种DNA条形码对卤虫进化史进行研究.在COI和ITS1基因构建的系统进化树中,除外群外,A. persimilis均位于基部位置,说明A. persimilis可能是一个原始分支.之前研究也通过构建COI基因的系统发育树,发现A. persimilis与A.sinica、A. urmiana、A. franciscana相比更接近物种的祖先种[12].也有研究发现,在ITS1基因构建的系统发育树中A. persimilis处于基部位置,而在COI基因树中A. salina比A. persimilis更为原始.但在本研究16S-12S rRNA构建的进化树中,祖先类群似乎是沿着两个方向进化的,一支进化为A. persimilis和A.salina,然后是A. franciscana和A. sinica,另一支进化为其他物种.本研究基于COI和ITS1进化树认为A. persimilis属于一个物种的祖先群,这与大多数研究相一致.Barigozzi[31]研究表明,至少在距今较近的一段时期里A. persimilis具有广泛的地理分布,这也间接证明了上述论点的可能性.
4 结 论
为了提高卤虫分类和鉴定的准确性和可靠性,本研究用多个DNA条形码技术对中国、哈萨克斯坦、俄罗斯等国的48个取自日晒盐场或盐湖的卤虫标本进行分类和进化研究.每个DNA标记扩增的基因片段均大于1kbp,以确保DNA条形码可获得更多的遗传信息.结果表明,COI和16S-12S rRNA序列对卤虫分类鉴定有效,而ITS1序列仅对A.franciscana、A. salina和A. persimilis等几种卤虫鉴别有效.COI和ITS1基因构建的系统发育树表明A.persimilis可能是一个原始分支,属于一个物种的祖先群.研究结果可为卤虫样本鉴定和卤虫资源保护与可持续开发利用提供重要工具和分析手段.

